基于形态与DNA条形码的中国鲾科鱼类分类研究
周 琪,章 群,唐楚林,徐 示,王业磷
(暨南大学生态系,广州 510632)
鲾科(Leiognathidae)隶属于鲈形目(Perciformes),广泛分布于西太平洋及印度洋海域,中国主要分布于南海及台湾海峡。鲾科鱼类为群居性小型鱼类,一般栖息于岸边泥沙地、河口或内湾,在沿岸软底质生态系统中占有重要地位,并具有一定经济价值和药用价值[1-2]。鲾科鱼类鳞片细小易脱落,体内有可与发光细菌相互作用的发光器官和组织。目前国际上对鲾科的分类标准不统一 ,如ITIS(Integrated Taxonomic Information System)和《南海鱼类检索》[3]将鲾科分为3个属:小牙鲾属(Gazza)、鲾属(Leiognathus)、仰口鲾属(Secutor);CHAKRABARTY等[4]依据外部形态、体长和体高的比例、发光器官和组织的特征等,将鲾科分为6个属:鲾属、金鲾属(Aurigequula)、项鲾属(Nechequula)、马鲾属(Equulites)、布氏鲾属(Eubleekeria)、光胸鲾属(Photopectoralis);SPARKS等[5]依据发光器官内在和外在是否存在性二态,认为鲾科由不存在性二态的鲾属和金鲾属以及存在性二态的Gazzinae亚科中8个属组成;Fishbase (http://www. fishbase. org/)记录全球鲾科至少有8属48种,其中中国8属19种;陈大刚等[6]记录全球鲾科有3属30种,中国有3属22种。鲾科鱼类无论是外部形态还是内部的发光器官和组织等特征具有极大的种间相似性[7],通过传统形态鉴定存在较大困难。目前国内外对于鲾科鱼类的研究报道多为生态资源调查[8]、发光器官和组织的性二态及其发光机制的研究[9]等。在分类方面,YU[10]对台湾海域的鲾科鱼类进行了分子上和形态上的比较分析。ABRAHAM等[11]根据详细的形态数据研究了印度西海岸16种鲾科鱼类的种间关系。但现有的国内外资料不足以阐明中国沿海鲾科鱼类的分类现状。
2003年HEBERT等[12-13]提出“DNA条形码”的概念,即通过一段易于扩增测序的标准基因序列进行物种鉴定;对动物界中11门13320个物种的线粒体细胞色素氧化酶Ⅰ亚基(COI)序列分析发现,除腔肠动物门(Cnidaria)外,多数物种种内遗传距离≤ 2%,种间遗传距离远大于(一般≥10×)种内遗传距离,并可对98%以上的物种进行分类。2005年WARD等[14]对澳大利亚等地海域207种鱼类的研究,验证了COI基因序列作为海洋鱼类DNA条形码标准基因序列的可行性。同年国际社会开始实施鱼类生命条形码计划,采用经权威分类鉴定标本的鱼类线粒体COI基因序列,以电子数据库形式,构建含有DNA条形码、图像和地理坐标的全球鱼类参考标准数据库。虽然DNA条形码技术有助于物种鉴定,而且对物种间群落关系多样性(如食物网结构复杂性)的研究有重要作用;但由于所使用的线粒体标记为母系遗传,在区分杂交种、近期分化物种时仍存在诸多问题[15],因而准确的物种分类需要DNA条形码和形态分类研究的相互参照及验证[16-17]。YING等[18]利用GenBank(http://www.ncbi.nlm.nih.gov)和BOLD (http://www. barcodinglife.org)下载的232条鲾科鱼类6属24种COI序列进行了DNA条形码分析,但并无中国黄斑鲾(L.bindus)、短吻鲾(L.brevirostris)、颈斑鲾(L.nuchalis)、短棘鲾(L.equulus)、坚鲾(L.stercorarius)序列分析,也没有进行形态分析等;JU[19]利用COI序列和形态特征在中国北部湾发现新纪录种Deveximentummegalolepis。
本研究对本实验室在中国沿海各地采集的鲾科鱼类进行分类整理和详细的形态测量,测定了每一形态种一定数量的标准COI序列;结合GenBank和BOLD中下载的序列,以期突破传统形态学根据表型性状鉴定的局限性,明确中国鲾科鱼类的分类地位,为保护鲾科鱼类生物多样性和开展海洋生态调查提供基础资料。
1 材料与方法
1.1 实验材料来源、形态测量与数据处理
参阅FishBase、《南海鱼类检索》[3]等资料,将2005—2018年采集于中国4省14个地点(表1)的142尾鲾科鱼类鉴定为3属10种,其中小牙鲾(G.minuta)14尾,G.sp.A10 尾,黄斑鲾 14尾,短吻鲾33尾,颈斑鲾26尾,短棘鲾20尾,细纹鲾(L.berbis)4尾,带纹鲾(L.striatus) 1尾,坚鲾1尾,仰口鲾(S.ruconius)19尾。参照ROHLF等[20]的几何形态测量法,测量体长、体高、头长、头高、吻长、眼径、尾柄长、尾柄高、背鳍长、胸鳍长、腹鳍长、臀鳍长、全长等13个常规性状;通过8个解剖学坐标点:A:吻端、B:头部末端、C:背鳍起点、D:尾鳍背部起点、E:峡部末端、F:腹鳍起点、G:臀鳍起点、H:尾鳍腹部起点构建框架结构,测量AB、BC、CD、DH、HG、GF、FE、EA、EB、EC、FB、FC、CG、GD、HC等15个框架结构测量性状间距离,形态测量精确到0.1mm。测量示意图见图1。为消除异速生长带来的变动,将除体长外的27个形态测量值按ELLIOTT等[21]的方法标准化处理后,通过SPSS22.0软件对27个度量特征进行主成分分析、判别分析和聚类分析。
1.2 DNA条形码实验与数据处理
参照乐小亮等[22]方法提取鱼类样本DNA。使用本实验室自行设计的引物Cx1F2:5′-ACCTGTGGCAATCACACGCTGAT-3′,Cx1R2:5′-CATGCCCATGTAGCCGAATGG-3′,进行PCR扩增,将扩增产物送往华大基因测序。从GenBank和BOLD下载中国及其邻近海域鲾科鱼类同源序列,通过MEGA6.0[23]软件进行人工校对,分析序列特征,计算基于Kimura 2-parameter(K2P)模型的遗传距离;构建邻接(Neighbor-Joining)树,经1000次重复抽样(Bootstraps)检测分支置信度。采用自动条形码间隙检索方法(automatic barcode gap discovery,ABGD)[24]将全部序列自动分组。本研究用于条形码分析的序列如表1所示。
2 结果与分析
2.1 鲾科鱼类形态测量多元分析
对147 尾 28个形态参数进行标准化处理后,发现测量值与标准体长间无显著相关性,表明成功消除了因异速生长造成的体长效应,使分析结果更准确。在主成分分析中,KMO(0.845)值接近1;Bartlett值为5 484.638,27个形态特征值种间差异极显著(P<0.001);说明主成分分析结果较为理想。第1、2主成分的累积贡献率为65.35%。其中对主成分1有主要贡献的是FB、FE、体高等,对主成分2有主要贡献的是臀鳍长、背鳍长和尾柄长等。主成分散点图(图2)显示,第一主成分中各种鱼类重叠部分较多,因此无法只通过主成分1区分不同鱼类;第二主成分中短棘鲾和仰口鲾与其它种类重叠部分较少。逐步判别分析结果(图2)显示,短吻鲾有3尾判入颈带鲾,2尾判入细纹鲾,判别成功率为78.8%。颈带鲾有4尾判入短吻鲾,判别率为84.6%。短棘鲾有1尾判入为带纹鲾,判别率为95.0%。其余7种鱼类判别成功率为100%。在交互验证分析中,G.sp.A、细纹鲾、仰口鲾判别结果与逐步判别分析结果相同,均为100%。坚鲾的判别率为0.0%,有且只有1尾判入小牙鲾;短棘鲾判别率为95%,有1尾被判入带纹鲾;带纹鲾判别率则为5%,有且只有1尾判入短棘鲾;小牙鲾、黄斑鲾、短吻鲾的结果低于逐步判别分析结果。逐步判别分析和交互验证分析的总体判别率分别为91.5%和85.2%。形态聚类图上(图3),短吻鲾与细纹鲾首先聚类,其次细纹鲾、小牙鲾、黄斑鲾、仰口鲾、短棘鲾、坚鲾依次加入聚类;其中颈带鲾和短吻鲾、G.sp.A和小牙鲾、带纹鲾和短棘鲾聚类。

表1 本研究中用于COI分析的鲾科鱼类样本的信息Tab1 Specimens information of Leiognathidae in the present study

图1 鲾科鱼类框架结构示意图(引自中国台湾鱼类资料库)Fig.1 Schematic drawing of morphomertric measurements of Leiognathidae(adopted from fishdb.sinica.edu.tw)注:1:体长;2:体高;3:头长;4:头高;5:吻长;6:眼径;7:尾柄长;8:尾柄高;9:背鳍长;10:胸鳍长;11:腹鳍长;12:臀鳍长;13:全长。A:吻端;B:头部末端;C:背鳍起点;D:尾鳍背部起点;E:峡部末端;F:腹鳍起点;G:臀鳍起点;H:尾鳍腹部起点。Note:1: body length; 2: body height; 3: head length; 4: head height; 5: snout length; 6: eye diameter; 7: caudal peduncle length; 8: caudal peduncle height; 9: dorsal fin length; 10: pectoral fin length; 11: ventral fin length; 12:anal fin length; 13: full length. A: rostral side; B: head end; C: dorsal fin starting point; D: caudal fin back starting point; E: isthmus end; F: pectoral fin starting point; G: anal fin starting point; H: caudal fin ventral starting point.

图2 10种鲾科鱼类主成分得分散点图(左)和判别函数得分(右)Fig.2 Scatter plot of component factor score (left) and discriminant scores (right) of 10 Leiognathid species

图3 基于形态测量的鲾科鱼类聚类图Fig 3 Dendrogram of morphological characters of 10 Leiognathid species
2.2 鲾科鱼类线粒体COI序列特征与DNA条形码分析
在105条平均长度为643bp(552~652bp)的序列中,没有碱基插入和缺失,多态位点225个,简约信息位点218个;A、T、C、G碱基组成分别是24.7%、29.0%、28.7%、17.5%。A+T含量(53.7%)高于C+G含量(46.2%),呈现出明显的碱基组成偏向性,与WARD等[14]的硬骨鱼研究一致;颠换比(R)为2.93,大于2.0,表明突变未达到饱和,适合系统发育分析。在邻接树上(图4),12种鱼聚类成11个bootstrap支持率为100%分支(Clade),分支间平均遗传距离[18.6%(7.9%~23.6%)]是分支内[0.3%(0.0%~1.0%)]的62倍。黄斑鲾聚类在2个分支:在Clade1中与短吻鲾混杂,平均遗传距离为0.2%;在Clade5则无混杂而独立成支。本研究测序的黄斑鲾L.bindusQ.BoA1、L.bindusQ.SanY1,4、L.bindusQ.HaiW1,3均聚类在Clade 5。曳丝鲾在Clade3中与细纹鲾混杂,Clade 3进一步分为subclade 1和subclade 2,subclade 1与细纹鲾混杂,subclade 2中曳丝鲾独立成支,平均遗传距离subclade间(5.8%)是subclade分支内(0.1%)的58倍。subclade1中细纹鲾与曳丝鲾种间遗传距离(0.2%)与种内(0.2%)相当,推测二者为同种。G.sp.A与小牙鲾种间遗传距离是种内遗传距离79倍,与宽身小牙鲾是35倍,支持了其为独立物种。其余种类单独聚类成支。根据ABGD递归分析(图5),当种内先验遗传距离范围为0.46%~3.59%时,本研究鲾科划分为12个假设种与上述形态学与分子结果分析相对应。另发现广东阳江坚鲾、海南博鳌带纹鲾、G.sp.A等为3个新纪录种。
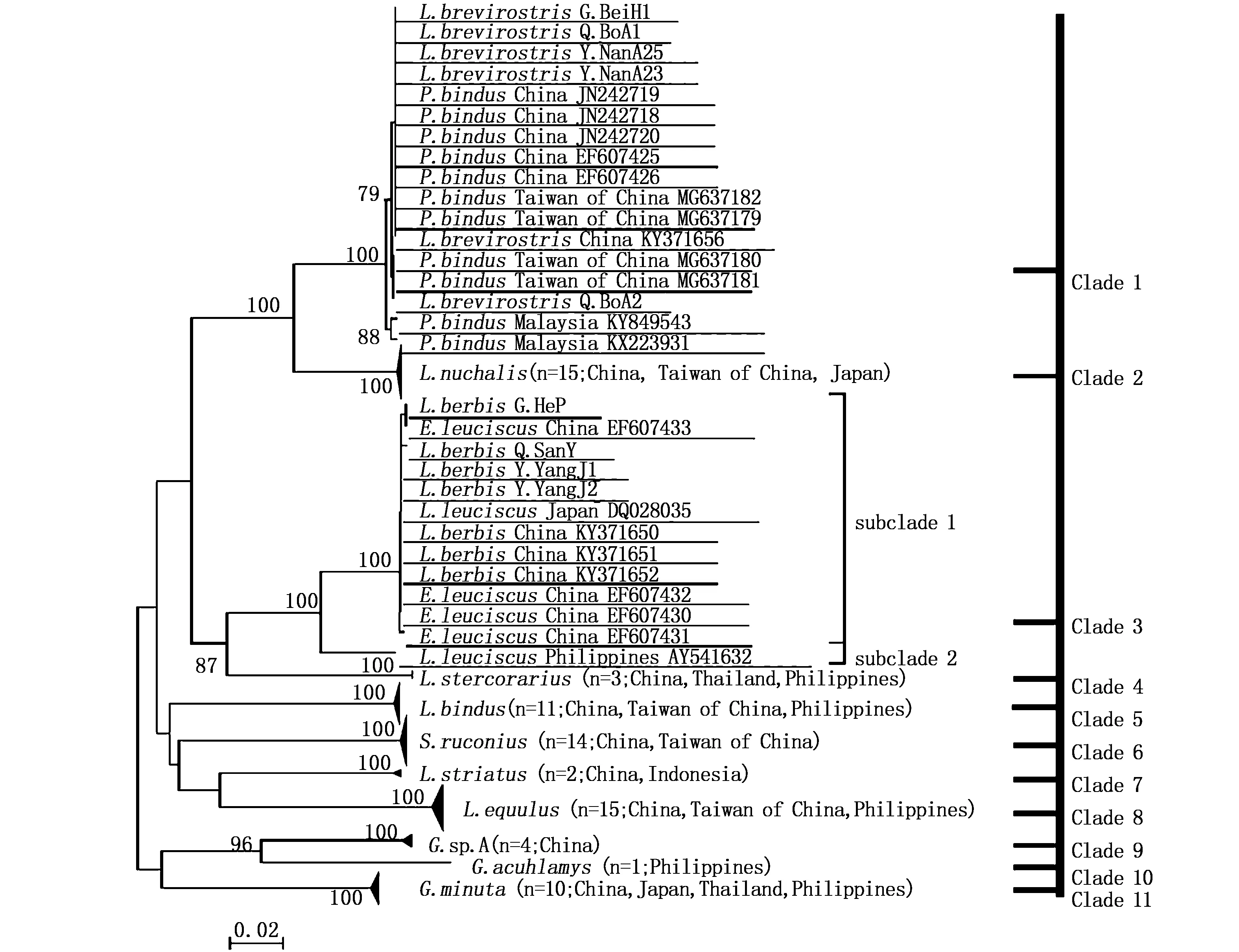
图4 COI序列K2P模型的鲾科NJ树Fig4 K2P-based NJ tree of COI sequences of Leiognathidae注:图中划线部分为混杂分支Note:The underlined part of the graph are branch intertwined with different species

图5 鲾科鱼类的ABGD图Fig.5 Automatic barcode gap discovery analyses of Leiognathidae注:横坐标表示先验种内分化值,纵坐标表示划分类群的数量Note: Abscissa means prior intraspecific divergence, ordinate means the number of groups produced
3 讨论
3.1 短吻鲾、黄斑鲾和颈带鲾的分类地位
DNA条形码序列用于物种鉴定的一般标准是种内遗传距离通常低于2%,且种间遗传距离是种内遗传距离的10倍以上[12-14]。在图3 Clade1中从GenBank下载的黄斑鲾与短吻鲾混杂在平均遗传距离(0.2%)小于≤2%的Clade1中;但本研究测定黄斑鲾L.bindusQ.BoA1、L.bindusQ.SanY1,4、L.bindusQ.HaiW1,3均聚类在Clade5上,2个分支间遗传距离为18.4%是分支内(0.15%)的123倍。因TALWAR等[25]否定了短吻鲾和黄斑鲾为同物异名的可能性,故推测混杂在Clade1中的黄斑鲾为短吻鲾,ABGD分析也支持这一推测。短吻鲾和颈带鲾第一和第二主成分都难以区分;形态聚类图上(图3)首先独自聚类,在判别分析图中重心没有重叠,表明二者表观形态较为相似,但也仍具有一些差异,需要仔细测量以避免形态鉴定出错。短吻鲾(Clade1)和颈带鲾(Clade2)分支间遗传距离为8.1%是分支内(0.15%)的54倍,支持将二者作为独立物种的观点。
3.2 细纹鲾与曳丝鲾的分类地位
细纹鲾与曳丝鲾间遗传距离仅为0.2%,不满足DNA条形码的分种标准[12-13]。一方面,二者体型相似,体侧均有蠕虫状细纹,区别仅在于细纹鲾背鳍第二棘条短于体高,曳丝鲾背鳍第二棘条呈丝状延长,长于体高;而鱼类捕捞及保存过程中操作不当,易使丝状的棘条残缺,影响物种鉴定。另一方面,种间生殖隔离机制薄弱时易出现杂交情况[26],杂交后代可能会偏向其中一种亲本的形态,而线粒体为母系遗传,造成邻接树上出现不同物种混杂在同一分支上的情况;推测二者为同种或为杂交所致。由于曳丝鲾序列仅为GenBank数据库下载,因此需要获得曳丝鲾标本进行形态和DNA条形码分析,补充测定核基因序列,并开展生物学特征研究,方可准确判断物种分类地位。
综上所述,本研究鉴定了中国常见10种鲾科鱼类,表明DNA条形码能够用于快速鉴定鲾科鱼类。由于线粒体为母系遗传,不能区分杂交物种,因而对于混杂成支的种类,需要进行详细形态特征的统计分析,并使用双亲遗传的核基因标记综合分析。由于本研究仍未能涉及所有中国鲾科鱼类物种,今后应扩大采样范围,增加样品种类及数量,并妥善保存样本的鳞式及鳍式等性状特征,以进一步明确中国鲾科鱼类的分类地位,为中国海洋渔业调查、种质资源的管理和开发提供科学依据。

