食源性致病菌生长延滞期建模的研究进展
岳思远,苏 亮,任鹏程,刘阳泰,王 翔,刘 箐,董庆利,*
(1.上海理工大学医疗器械与食品学院,上海 200093;2.国家食品安全风险评估中心,北京 100022)
食源性致病菌是一类以食品为传播媒介,可引起食物中毒的致病性微生物,是目前食源性疾病的主要诱因。由食源性致病菌引起的食源性疾病是国际性公共卫生问题。世界卫生组织2017年的报告表明,全球食源性疾病患者约6亿,由食源性疾病引起的死亡人数约42万,其中5岁以下儿童死亡人数约12.5万[1]。近年来我国建立了食源性疾病监测信息数据库,系统分析监测网地区14 年间8 000余起食物中毒案例,发现微生物性病原仍然是我国食源性疾病的主要病因(占30%~40%)[2]。同时,我国国家卫生健康委员会(原国家卫生和计划生育委员会)办公厅发布2017年第4季度的食物中毒公告显示,食物中毒事件报告数量和中毒人数最多的是微生物性食物中毒事件,分别占总报告数量和总中毒人数的34.6%和55.5%[3]。因此,需严格控制食品中食源性致病菌的数量及生长,以保证食品食用的安全性。
近年来,随着人们对食品安全的重视,预测微生物学也得到了良好的发展。通常情况下,微生物生长一般会经历4 个阶段:延滞期、对数期、稳定期及衰亡期。目前微生物延滞期仍未基于微生物生长机理明确定义,生物学上被普遍接受的定义是:微生物群体经历环境突变,自我调整后开始繁殖的时间[4]。几何意义上微生物延滞期是指微生物对数生长期达到最大比生长速率时,生长曲线切线的反向延长线和初始菌量水平延长线的交点在时间轴上的投影点与零时刻的时间间隔[5]。在传统预测微生物生长的模型中,延滞期常通过生长曲线确定。而应用数学模型预测微生物生长时,延滞期较最大比生长速率受更多因素影响,因此延滞期较最大比生长速率更难以准确获取。
预测微生物学作为食品风险评估的重要工具,已被广泛应用于食品安全风险控制中,然而因延滞期尚未明确定义,且影响因素较多,相较于最大比生长速率预测模型更难以准确估计微生物生长延滞期,因此延滞期研究仍具有较大的发展空间,需进一步探讨。以下分述微生物生长延滞期的测定方法和建模方法的研究进展。
1 延滞期的测定方法
现有技术并不能直接测定微生物生长延滞期,需通过已建立的微生物生长模型拟合生长曲线,间接获取延滞期。在预测微生物学中,微生物生长模型的建立需确定微生物生长曲线,微生物生长曲线的获取和记录可采用多种方法。基于微生物群体水平和单细胞水平下不同的延滞期概念,延滞期的测定分别从群体水平和单细胞水平进行论述。
1.1 群体生长延滞期的测定
目前测定微生物生长曲线的方法主要分为传统方法和分子生物学方法。表1中总结了应用传统方法和分子生物学方法测定微生物生长曲线进而通过模型拟合获取生长延滞期的相关研究。传统测定微生物生长曲线的方法是活菌计数法,因其不受菌悬液颜色及浊度的影响而成为测定食品中食源性致病菌的主流手段。但是该方法操作繁琐、耗时耗力,且应用该方法获取的生长参数准确性与数据点的数量及观测点的位置均相关[23],因此活菌计数法还需进一步改进。相较于活菌计数法,比浊法因其操作简便、省时省力的优点被广泛应用于微生物生长曲线的测定和建模研究中[12,24]。然而,比浊法的检测限仅能达到106~107CFU/mL,且只可用于液体培养基的测定,同时因测定的菌悬液浓度并不是活菌浓度而导致获得的生长参数有些许误差。建议今后的研究可基于技术层面对该方法进行优化,使其能更好地描述低浓度食源性致病菌的生长,为进一步研究微生物生长提供依据。

表1 应用各种方法测定食源性致病菌生长曲线的相关研究Table1 A summary of methods for determining the growth curve of foodborne pathogenic bacteria
随着分子生物学技术的不断发展,变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)技术和PCR等分子生物学技术也被应用于微生物生长曲线的测定,相较于传统方法,这些分子生物学技术具有省时省力、高效等优点,且可同时测定两种细菌的生长,极大地改善了传统方法的不足[25]。Liao Chao等[18]应用DGGE结合PCR技术测定了单增李斯特菌和副溶血性弧菌的生长情况,进而通过Baranyi模型拟合其生长曲线获取延滞期。传统PCR技术需结合DGGE技术才可定量测定微生物的生长情况,而实时荧光定量PCR(real-time quantitative PCR,qPCR)技术[26]的出现弥补了其缺陷,实现了从定性到定量的突破。Guan Zhengping等[27]将金黄色葡萄球菌的菌液接种于猪肉中进行培养,每隔一段时间提取生长后细菌的总基因组DNA,并对其进行qPCR分析,进而根据已建立的微生物浓度与qPCR的循环阈(cycle threshold,Ct)值间的标准曲线,定量描述猪肉中金黄色葡萄球菌的生长情况,最后应用模型拟合其生长曲线,间接获取生长延滞期。Ye Keping[20]和孙文烁[21]等也同样应用qPCR技术分别定量描述了猪肉中单增李斯特菌和即食南美白对虾中副溶血性弧菌的生长情况,并对其进行模型拟合,获取了延滞期。但因qPCR技术无法区分活菌和死菌,导致实验结果有一定的误差。基于此,Zhang Zhaohuan等[22]将叠氮溴化丙锭(propidium monoazide,PMA)与多重qPCR技术相结合,极好地定量描述了生鲜南美白对虾样品中副溶血性弧菌和单增李斯特菌的生长行为。虽然PMA的应用可区分活死菌,但其会对细菌产生一定的损伤。分子生物学技术的发展为预测微生物学注入了新的活力,加速了预测微生物学的发展。然而,任何方法均需辩证地看待,即每一种方法均各有优点和局限性,因此分子生物学技术并不能完全取代传统方法,仍需根据具体的实验目的选择合适的实验方法。
1.2 单细胞生长延滞期的测定
单细胞生长延滞期的研究对象为单细胞,其分布研究需获取大量的实验数据。因此,传统的活菌计数法并不适用于开展单细胞水平研究,需采用其他方法测定单细胞延滞期。
单细胞生长延滞期可通过直接观测单细胞分裂过程进行测定。最早可追溯到Kelly等[28]的相关研究,将少量微生物接种于琼脂薄膜上,并置于显微镜下观测微生物单细胞个体的分裂过程,结果表明,随着细胞传代次数的增加,分裂时间间隔随之下降。基于固体培养观测法,董庆利等[29]设计流动槽装置,显微观测铜绿假单胞菌单细胞的生长分裂情况,流动槽通过液体流动带走新分裂的子细胞,进而持续观测一个目标细胞多次分裂的情况。固体培养显微观测法和液体培养显微观测法的实质均是通过单细胞分裂时间计算单细胞延滞期。虽然显微观测法可直接观测单细胞生长分裂的过程,但是该法对仪器设备要求较高且耗时耗力。同时,采用显微观测法开展单细胞延滞期分布的研究难以获取大量实验数据。
比浊法通过间接推断计算单细胞生长延滞期。与群体水平的观测不同,比浊法应用Bioscreen FP-1100C型全自动微生物生长曲线分析仪实时连续记录单细胞生长情况,进而通过检测时间(the time to detection,Tdet)计算单细胞延滞期。比浊法中的检测时间是指初始OD值增加至对应于菌悬液菌落总数为107CFU/mL时OD值的时间。McKellar[30]通过不同稀释度的菌液与检测时间线性回归获得回归曲线的斜率(slope),进而计算最大比生长速率(μmax),计算公式如式(1)所示。

进而再采用已得的μmax和模型估计Tdet,预测得到的Tdet和实测Tdet的差值即为单细胞延滞期。此后,Baranyi等[31]在前人研究的基础上,改进了单细胞延滞期的计算方式。假设Bioscreen微型孔板100 孔中每个孔的细胞数为1,且单细胞生长数量服从泊松分布[32],同时,细菌在对数期以恒定生长速率(μmax)生长,则单细胞延滞期即可通过公式(2)计算,生长曲线如图1所示[5]。

式中:x0为初始细胞数/(CFU/mL);xdet为达到检测时间的细胞数/(CFU/mL);Tdet为初始OD值达到菌悬液菌落总数为107CFU/mL所对应OD值的时间/h;μ为对数期细菌生长的恒定生长速率/h-1;λ为单细胞生长延滞期/h。

图1 微生物生长曲线[5]Fig.1 Microbial growth curve[5]
经Bioscreen FP-1100C型全自动微生物生长曲线分析仪测定单细胞延滞期的比浊法可连续监测大量单细胞的生长情况且省时省力,已广泛用于单细胞延滞期分布的研究。
综上所述,虽然微生物群体延滞期和单细胞延滞期均可采用上述方法间接获得,但是微生物群体延滞期会因拟合模型不同而不同,同时微生物单细胞生长观测方法依然存在精度低、控制难、耗时长等问题。因此,需在明确延滞期定义的基础上,进一步改进已有的微生物观测手段以提高生长模型预测延滞期的准确性。
2 延滞期的建模方法
在预测微生物学中,微生物生长模型一般根据变量类型分为三大类:一级模型是描述恒定环境条件下微生物数量与时间的关系;二级模型是描述微生物生长参数(延滞期(λ)和最大比生长速率(μmax))与环境变量(如温度、pH值和水分活度等)间的关系;三级模型是基于一级模型和二级模型,应用计算机软件获取微生物生长参数[33]。
基于微生物生长延滞期的测定方法,微生物群体延滞期需通过生长模型拟合生长曲线间接获取。表2归纳了可描述微生物生长曲线的一级模型。

表2 预测微生物学中常见的一级模型Table2 Primary models in predictive microbiology
由表2可知,与其他模型相比,三段式线性模型因分段描述微生物生长较难实现拟合。而Gompertz模型和Logistic模型最初并不是基于微生物学概念建立的模型。同时,Gompertz模型描述的S型曲线中,拐点处的确定曲率会导致高估延滞期[39-40]。与其他模型相比,Huang模型与Baranyi模型有一定的生理学意义,均为机械模型。
在预测微生物学中,被人普遍接受的一级模型为Baranyi模型[37],该模型引入了细胞生理状态(Q)这一生理学概念,公式如式(3)、(4)所示。

将Baranyi模型式(3)中的Q(t)/(1+Q(t))为调整函数α(t)。α(t)描述微生物延滞期向对数生长期的转换。假设微生物生长由某一物质P(t)控制,且该物质的累积遵循Michaelis-Menten动力学,则α(t)的计算如式(5)所示。

式中:Kp为米氏常数,当微生物生长过程中环境条件改变时,调整函数α(t)取决于P(t)/Kp速率。P(t)/Kp速率用于表征细胞的生理状态Q(t)。Baranyi模型中延滞期可用细胞生理状态表示,公式如式(6)所示。

式中:Q(0)为细胞的初始生理状态;h0为α(t)与Q(t)在数学意义上的转换参数。
除延滞期一级建模外,还存在延滞期的二级建模。目前最常用的延滞期二级模型是Ratkowsky等[41]于1982年提出的平方根模型,用于描述微生物延滞期随温度的变化情况,具体表达式如式(7)所示。

式中:λ为微生物生长延滞期/h;b为平方根模型的系数;T为微生物培养温度/℃;Tmin为微生物理论最低生长温度/℃。
延滞期的二级模型仅适用于描述微生物延滞期随温度的变化,而微生物所处生长环境较为复杂,因此,延滞期二级模型存在一定的局限性,建议在建模过程中增加其他影响因素。
延滞期的获取一般需将实验获取的微生物生长数据输入到三级模型即计算机软件中,进而再选择软件中的一级模型进行拟合,获得相应的生长参数,其中包括延滞期。表3总结了目前国内外可获取延滞期的相关软件,其中以英国食品研究所开发的ComBase和美国农业部开发的病原菌模型程序(pathogen modeling program,PMP)最为著名[45]。由表3可知,每个软件中所包含的一级模型并不是很全面,因此,建议在日后的软件开发方面,尽可能将所有一级模型编入程序中,供使用者自由选择合适的生长模型进行延滞期的获取。
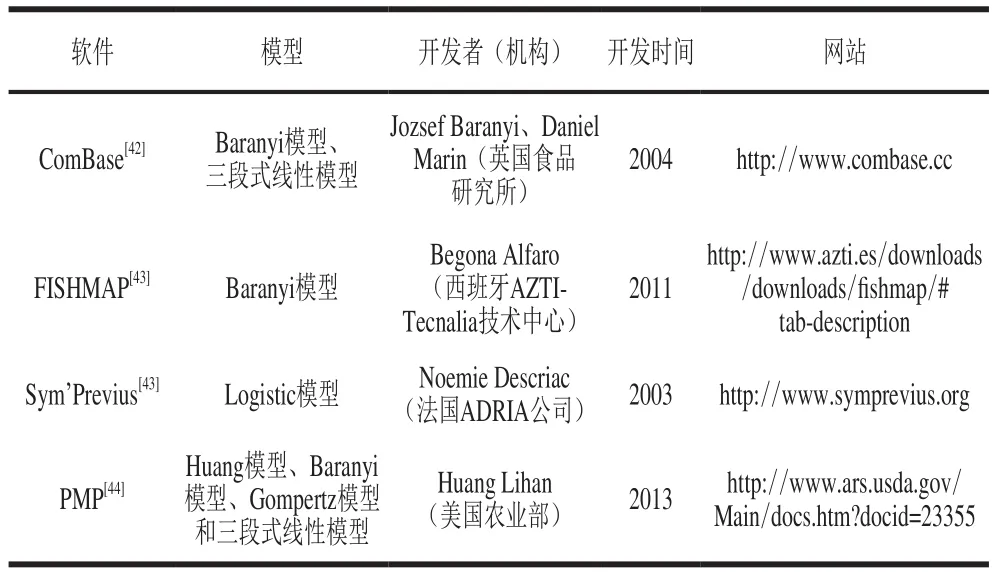
表3 预测微生物学中的相关软件Table3 Overview of predictive microbiology software tools
综上所述,虽然已有诸多一级模型可描述微生物生长,但是现有一级模型均是基于群体建立的,未考虑单细胞生长变异性。同时,因已有一级模型中的数学概念并不能通过实验验证,还需进一步改进已有模型或建立新模型以提高生长模型预测延滞期的准确性。除此之外,基于现有一级模型,还需在已有的计算机软件中添加部分一级模型,或开发新的覆盖所有一级模型的计算机软件,从而在应用计算机软件进行模型拟合时实现一级模型的最优选择。
3 结 语
虽然目前的已有研究可观测食源性致病菌的生长,也可应用模型拟合生长曲线获得生长延滞期,但仍需改进测定食源性致病菌生长的方法和拟合食源性致病菌生长的模型,进一步完善食源性致病菌生长延滞期建模工作。建议食源性致病菌生长延滞期建模及预测的研究方向如下:首先,应从机理出发,进一步明确食源性致病菌生长延滞期定义,为延滞期建模提供理论依据;其次,基于目前已有的延滞期观测手段,改进传统计数法耗时费力等问题,或优化分子生物学技术的精度及特异性,为延滞期的准确预测提供帮助;最后,延滞期建模中的模型仍存在一定问题,建议改进现有一级模型的部分参数或基于延滞期机理建立新的延滞期模型,同时优化已有三级模型或开发新的三级模型,使其覆盖所有延滞期模型,为延滞期建模的发展提供强有力的支持。

