基于定向进化的烷基间苯二酚合酶改造
包莹玲,赵威棣,吕旭浩,周靖阳,祝衢鑫,李 忠,陈小龙
(1.浙江工业大学 生物工程学院发酵工程研究所,浙江 杭州310014;2.浙江省桐庐汇丰生物科技有限公司,浙江 杭州311500)
烷基间苯二酚(Alkylresorcinols)是一种具有两亲性的酚醛脂类次级代谢产物,主要存在于植物、真菌及细菌中。在微生物中,烷基间苯二酚与其环境胁迫响应及拮抗作用有关[1]。在植物中,烷基间苯二酚有提高生物膜稳定性和通透性、保护植物免受氧化应激损伤以及产生化感物质等生理功能[2]。在哺乳动物中,烷基间苯二酚尤为重要,具有抗氧化、抑制脂氧合酶活性、抗癌和保护心脑血管等多种药理学功能[3]。随着研究的深入,烷基间苯二酚的生理功能不断得到阐明,已发展成一种新型的功能性因子,在食品、医疗和农药等行业具有广泛的应用前景。
烷基间苯二酚合酶(Alkylresorcinol synthases,ARS)属于研究蛋白结构与功能关系的重要模式分子——Ⅲ型聚酮合酶家族,其可催化脂肪酰CoA和丙二酰CoA进行脱羧、缩合、环化生成烷基间苯二酚。由于具有独特的底物识别和催化机制,ARS及其所属的PKS家族为人们进行酶的结构与功能研究提供了空前契机,成为新型药物研究和开发的理想材料[4]。定向进化及高通量筛选是获得酶优异突变体常用的方法之一[5,6]。本研究在成功实现ARS在温控自裂解大肠杆菌异源表达的基础上,结合易错PCR和定点随机突变构建ARS突变文库,并通过高通量筛选获得酶活力提高的突变ARS。在此基础上,对突变ARS进行底物选择性和酶动力学研究。研究结果不仅有助于深入探究ARS的催化机制,而且对进一步改造酶分子、扩大酶的发酵生产应用具有重要意义。
1 材料与方法
1.1 材 料
1.1.1 菌株、质粒
宿主菌Escherichia coli BL21(DE3)和可温控诱导裂解大肠杆菌的双控质粒pBM1230-ars均由实验室保存或构建。质粒pBM1230-ars主要包括:1)由温控启动子λcI857/pRpL串联噬菌体裂解基因SRRz构成的温控裂解元件;2)由lac启动子串联多克隆位点构成的IPTG(Isopropyl-β-D-thiogalactopyranoside)调控目的基因表达元件两部分。质 粒 pBM1230-ars 中 的 ars 基 因 (GenBank:Sb08g003170)来源于高粱,通过在线软件 Gen-Script[7]进行针对大肠杆菌密码子的偏好性分析及低使用率密码子同义转化后[8],交由青兰生物科技有限公司进行合成。
1.1.2 酶和试剂
质粒抽提试剂盒、DNA凝胶回收试剂盒和蛋白纯化试剂盒等均购于生工生物工程公司;随机突变试剂盒购于美国安捷伦公司;高保真酶、一步克隆试剂盒和定点突变试剂盒购于诺唯赞生物科技公司(南京);丙二酰-CoA、IPTG、氨苄青霉素(Ampicillin,Amp)和固蓝RR盐购于生工生物工程公司;其他试剂均为国产分析纯。脂肪酰-SNAC的合成基于Gilber方法[9]。引物合成和基因测序由北京擎科新业生物有限公司完成,引物设计如表1所示。

表1 易错PCR及定点随机突变的PCR引物列表1)Table 1 PCR prime for the error-prone PCR and site-random mutation
1.1.3 培养基
LB(Amp)培养基:蛋白胨10g/L,酵母粉5g/L,NaCl 10g/L,Amp 0.05g/L,pH 7.5,Amp与IPTG由0.22μm微孔滤膜过滤除菌,其他培养基成分均在121℃灭菌20min。固体培养基加入1.5%~2%的琼脂粉。
1.2 突变文库的构建
1.2.1 易错PCR突变文库的构建
以质粒 pBM1230-ars为 模板,以 Ep-ars-F 和Ep-ars-R为引物,通过随机突变试剂盒对目的基因进行易错 PCR 扩增。以 Ep-vec-F 和Ep-vec-R 为引物,通过高保真酶进行PCR扩增,将质粒pBM1230-ars线性化。将PCR产物分别经琼脂糖凝胶电泳验证后,使用DNA凝胶回收试剂盒进行回收、纯化,并通过快速克隆试剂盒基于同源臂进行同源重组环化质粒。将环化的重组质粒转化至E.coli BL21(DE3)感受态细胞中,涂布固体 LB(Amp)平板,挑选阳性克隆构建易错PCR突变文库。
1.2.2 定点随机突变文库的构建
以易错PCR获得的质粒pBM1230-ars(Ep-2)为模板,采用定点突变试剂盒进行定点随机突变,分别 以 Sd-ars-F,Sd-ars-R,Sd-vec-F 和 Sd-vec-R 为引物,进行定点随机突变扩增。成功扩增后的PCR产物用DpnI酶消化,以去除甲基化模板质粒,消化后产物可直接通过同源臂进行同源重组环化质粒。将环化的重组质粒转化至E.coli BL21(DE3)感受态细胞中,涂布固体LB(Amp)平板,挑选阳性克隆组成定点随机突变体库。
1.3 突变体文库的双控诱导表达
挑取阳性克隆接种于每孔装有200μL LB(Amp)培养基的96孔板中,30℃,200r/min培养过夜后,将20μL菌液接种于另一含有180μL LB(Amp)培养基的96孔板中,30℃,200r/min培养4h,加入终浓度为0.8mmol/L的IPTG于30℃诱导ARS表达4h后,升温至38℃热诱导裂解蛋白SRRz表达2h,最后将培养温度降至30℃继续裂解培养3h[10]。双控诱导表达结束后,将培养液于5 000 g离心5min后,收集上清液并加入等体积乙酸乙酯进行萃取,真空干燥后得到黄色油状产物用于突变文库筛选。
1.4 突变文库的高通量筛选
向含有提取产物的96孔板中加入固蓝RR盐工作液,以V(乙酸)∶V(甲醇)=1∶99的混合液为溶剂,配成质量分数为0.05%的固蓝RR盐储液,按V(固蓝RR盐储液)∶V(甲醇)=1∶4配制工作液,以质量分数为5%的K2CO3溶液为碱化剂,调节pH为10,室温下反应15min,烷基间苯二酚与固蓝RR盐特异性结合呈紫红色化合物,于酶标仪(Spectra Max M2,美国 Molecular Devices公司)上测定OD480并保存数据。以重组菌E.coli BL21(DE3)/pBM 1230-ars作为对照,将结果高于阳性对照的克隆挑出。
1.5 蛋白质分离纯化及突变酶的酶学性质研究
将筛选到的ARSSd-3接于50mL LB(Amp)培养基中进行双控诱导表达。表达结束后将培养液于5 000 g离心5min,收集上清液,采用蛋白纯化试剂盒中的Ni-柱纯化ARS,并经透析和蛋白超滤管浓缩后用于酶学性质的测定。
ARS酶活力单位(U)定义:每分钟催化产生1μmol 5-烷基间苯二酚所需的酶量。以0.3mmol/L棕榈酰-SNAC((C16∶0)-SNAC)为起始底物,以0.7mmol/L丙二酰-CoA为延伸底物,加入20μL粗 酶 液,以 50mmol/L,pH 7.0 的 K2HPO4-KH2PO4缓冲液补足200μL,于30℃反应60min。通过添加50μL甲醇终止反应并萃取产物[11]。13 000r/min离心1min,分离上层有机相,真空干燥后收集产物,并采用高效液相色谱法测其质量分数[12]。HPLC 检测条件:安捷伦 Zorbax SB-C18反相色谱柱(250mm×4.6mm,5μm)。流动相:线性梯度洗脱,50%~100%甲醇-水(含体积分数为0.1%的三氟乙酸),10min;等强度洗脱,100%甲醇,5min。流速1mL/min,检测波长286nm。在底物特异性试验中,除(C16∶0)-SNAC外,另选取其他脂肪酰-SNAC(月桂酰(C12∶0)-SNAC、肉蔻酰(C14∶0)-SNAC、肉蔻油酰(C14∶1)-SNAC、棕榈油酰(C16∶1)-SNAC、硬脂酰(C18∶0)-SNAC)作为底物,考察ARSSd-3底物特异性。
1.6 突变酶的结构预测和突变位点分析
以来源于Freesia hybrid的聚酮合酶晶体结构(PDB∶4WUM)为模板[13],采用 SWISS-MODEL在线分析服务器(http://swissmodel.expasy.org),对ARS的蛋白结构进行同源建模,并通过VMD分析ARS的结构,推测氨基酸突变点在酶蛋白空间结构中的位置以及对酶蛋白功能可能造成的影响。
1.7 数据分析
数据显著性分析采用Excel和SPSS 18软件进行,其中多重比较基于Duncan法进行。以P<0.05,P<0.01及P<0.001分别表示在统计学上存在差异、差异显著和差异极显著。酶动力学分析采用 GraphPad prism 7.0进行。
2 结果与分析
2.1 易错PCR的突变结果
经过两轮易错PCR及单次库容量达到5 000以上的高通量筛选,筛选得到的4株突变ARS表现出比野生ARS更高的酶活性(表2)。通过显著性分析发现,除ARSEp-4外,其余的酶活力均显著提高(P<0.05)。其中,包含266位点和256位点等2个突变位点的ARSEp-2酶活性最高,达到野生ARS的1.23倍。此外,ARSEp-1也包括2个突变位点,而ARSEp-3则包括1个突变位点。

表2 两轮易错PCR筛选及基因测序结果Table 2 Screening results and gene sequencing of mutant strains for error-prone PCR
2.2 定点随机饱和突变结果
2.2.1 基于模型的突变位点选择
ARS与所选的模板序列相似度为46%,序列同源性为54%,序列的覆盖率为0.94,符合建模要求。根据SWISS-MODEL自带系统对构造的同源模型进行打分,GMQE为0.76,QMEAN 为-2.03,说明用该模型进行结果预测是可靠的。
根据ARS模型(图1)对3个突变体共涉及的5个突变位点进行分析发现:199位点和228位点的氨基酸位于活性中心附近;256位点的氨基酸位于蛋白表面,推测该位点氨基酸的对ARS的结构有一定的影响;207位点和266位点的氨基酸均位于ARS活性中心腔内,这对聚酮链的延伸长度以及起始底物和产物的特异性具有决定作用。由于256,199和228位点远离活性腔,故选取207和266两个位点的氨基酸进行下一步的定点饱和突变改造。

图1 ARS的蛋白质建模分析图Fig.1 The 3Dmodel analysis of ARS
2.2.2 定点随机突变文库的筛选
对突变型Ep-2的位点207和266的氨基酸进行定点随机突变,筛选库容量均达到5 000以上,共筛选到3个酶活性提高的突变 ARSSd-1,ARSSd-2和ARSSd-3。通过显著性分析发现,各突变ARS酶活力均显著提高(P<0.001)。如表3所示,酶活性最高的ARSSd-3,其酶活力达到ARS的1.55倍。

表3 定点随机突变文库筛选结果Table 3 Screening results and gene sequencing of mutant for of site random mutation
2.3 野生型和突变体酶学性质表征
2.3.1 ARSSd-3底物特异性表征
以ARS为参照,考察突变ARS对不同起始底物的底物特异性,结果如图2所示:ARS与ARSSd-3一样,对不同长度及饱和度的脂肪酰-SNAC均能进行催化,且酶活均有所提升。与Cook等[14]以脂肪酰-CoA为起始底物进行底物选择性研究的结果相似,ARS偏向于选择碳链长度中等的(C14∶0)-SNAC为底物,且随着链长的增加,比酶活力迅速下降。ARSSd-3的底物范围略有增加时,也能很好地催化(C16∶0)-SNAC,这可能与活性腔中266位氨基酸的特异性进化与在不破坏酶的催化力的前提下快速实现产物多样性有关[15]。此外,当碳链长度相等时,ARS和ARSSd-3均更偏向于选择不饱和度为1的脂肪酰-SNAC。
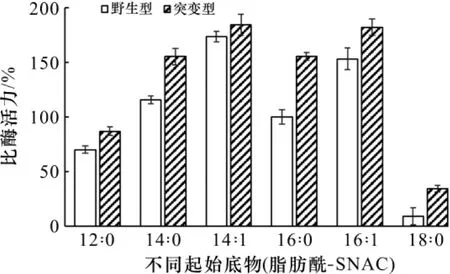
图2 不同起始底物对ARSSd-3酶活力的影响Fig.2 Enzymatic activities of ARSSd-3on different starter molecule-SNAC
2.3.2 ARSSd-3酶动力学分析
以(C16∶0)-SNAC 为起始底物,以丙二酰CoA为延伸底物,以ARS为参照,对ARSSd-3的酶动力学性质进行分析。如图3所示,ARSSd-3的反应速度显著高于ARS。当底物(C16∶0)-SNAC的浓度大于50μmol/L时,ARS和 ARSSd-3的酶促反应速率逐渐增大到最大值。

图3 不同底物浓度下的ARS和ARSSd-3的反应速度Fig.3 The reaction velocity of ARS and ARSSd-3with different(C16∶0)-SNAC concentration
通过 Michaelis-Menten方程分别对ARS和ARSSd-3的酶促反应进行拟合,模型的决定系数R2均大于90%,可用于对动力学参数的计算。ARS的米氏常数Km为(8.9±1.3)μmol/L,最大反应速率Vmax为(176.9±6.0)pmol/(min·mmol);ARSSd-3的Km为(6.8±1.3)μmol/L,Vmax为(234.1±9.8)pmol/(min·nmol)。ARSSd-3的Km值相较于 ARS有明显的降低,说明酶对底物的亲和力增强,更接近于 ARS和天然底物(C16∶0)-CoA的Km值(2.3±1.3)μmol/L[14]。此外,ARSSd-3的Vmax值均高于ARS,表明其催化活力有所提高。
3 结 论
通过结合易错PCR技术、定点随机突变技术和96孔板通量筛选技术,对ARS进行定向化分子改造,得到1株包含3个突变位点(A207S,M266A和T256S)的酶活显著提高的突变体ARSSd-3,表征后发现不仅其酶活提高至野生型的1.55倍,底物选择性向长链脂肪酰SNAC也略有偏移。此外,酶动力学性质分析发现:ARSSd-3的 Km为(6.8±1.3)μmol/L,对底物的亲和力增强;Vmax为(234.1±9.8)pmol/(min·nmol),催化活力有所提高。本研究有助于深入理解ARS的催化机制,为后续利用理性设计手段改造ARS,实现ARS的发酵生产提供了参考。

