植物N-糖链检测技术研究进展
何金霞, 贾晓晨, 王文霞, 胡建恩, 尹 恒*
1.大连海洋大学食品科学与工程学院, 辽宁 大连 116023;2.中国科学院大连化学物理研究所, 大连市糖类农用制剂工程研究中心; 辽宁省碳水化合物研究重点实验室, 辽宁 大连 116023
N-糖基化是普遍存在的蛋白质翻译后修饰方式之一,在各种复杂的生命活动中发挥着重要作用[1,2]。在哺乳动物中,N-糖基化对于调节胚胎发育、癌症发生发展和免疫防御等具有重要意义。如α-1,6-连接的核心岩藻糖基化可以通过扰乱转化生长因子(transforming growth factor,TGF)β1和表皮生长因子(epidermal growth factor,EGF)的信号传导而导致严重的肺功能障碍[3];在由TGF β1诱导的上皮-间质转化(epithelial-mesenchymal transition,EMT)过程中,N-糖链合成异常可影响胚胎的发育[4];唾液酸化的N-糖基化对于脑发育和乳腺癌的发生发展具有重要影响[4]。植物N-糖基化的研究相对较少,但近年来也得到了研究人员的重视。在植物中,N-糖基化对于细胞壁的形成[5]、生殖发育[6]和抵御病原菌入侵[7]等诸多生理过程具有重要意义。如拟南芥突变体lew3(leafwilting3)中编码α-1,2-甘露糖基转移酶ALG11的LEW3基因缺失会导致纤维素合成受损进而影响细胞壁的形成[8];拟南芥N-糖基化突变体alg3(asparagine-linkedglycosylation3)、stt3(staurosporinandtemperaturesensitive3aand 3b)、dad1(defenderagainstcelldeath1)和cgl1(complexglycan1)等均比野生型拟南芥更易感病,表明N-糖基化在抵御病原菌的防御反应中发挥着重要作用[9~12]。
植物和哺乳动物中蛋白质的N-糖基化模式不同,其差异主要出现在高尔基体阶段:植物中五糖核心结构修饰是核心α-1,3岩藻糖(α-1,3-Fuc)和β-1,2木糖(β-1,2-Xyl),这是植物所特有的,而哺乳动物中五糖核心结构修饰是α-1,6岩藻糖(α-1,6-Fuc);此外,植物中与分枝末端N-乙酰氨基葡萄糖(N-acetylglucosamine,GlcNAc)连接的是含有Lewis-a表位的β-1,3-半乳糖(β-1,3-Gal)和α-1,4岩藻糖(α-1,4-Fuc),而哺乳动物中与分枝末端GlcNAc连接的是含有Lewis-x表位的β-1,4-半乳糖(β-1,4-Gal)和α-1,3-Fuc,其末端还可被唾液酸进一步修饰(图1,彩图见图版二)[13~15]。正是植物和哺乳动物中N-糖基化修饰存在的差异,使得哺乳动物中N-糖基化的研究方法不完全适用于植物,这也是植物中N-糖基化研究相对滞后的原因之一。与哺乳动物相比,植物中关键蛋白N-糖链及不同生理条件下N-糖组的结构在很大程度上仍然是未知的,这就意味着糖链结构的解析对于揭示植物中N-糖基化功能至关重要。
而植物中糖链的表征工作也存在一定挑战:首先,植物来源的糖链较哺乳动物分枝少且丰度更低;其次,由于糖链在富集时易受色素等的干扰,且糖链往往缺乏生色团,从而使其在光谱或质谱检测时受阻。近年来,随着糖链标记试剂的开发以及质谱技术的发展,基于质谱(mass spectrometry,MS)的检测技术逐渐成为表征糖链的首要分析工具,各种糖链定量检测方法也相继建立。本文就近年来蛋白质N-糖链的释放、定量策略、可视化检测及其在植物中的应用进展进行归纳总结,以期为更好地开展植物中糖组学研究提供参考。
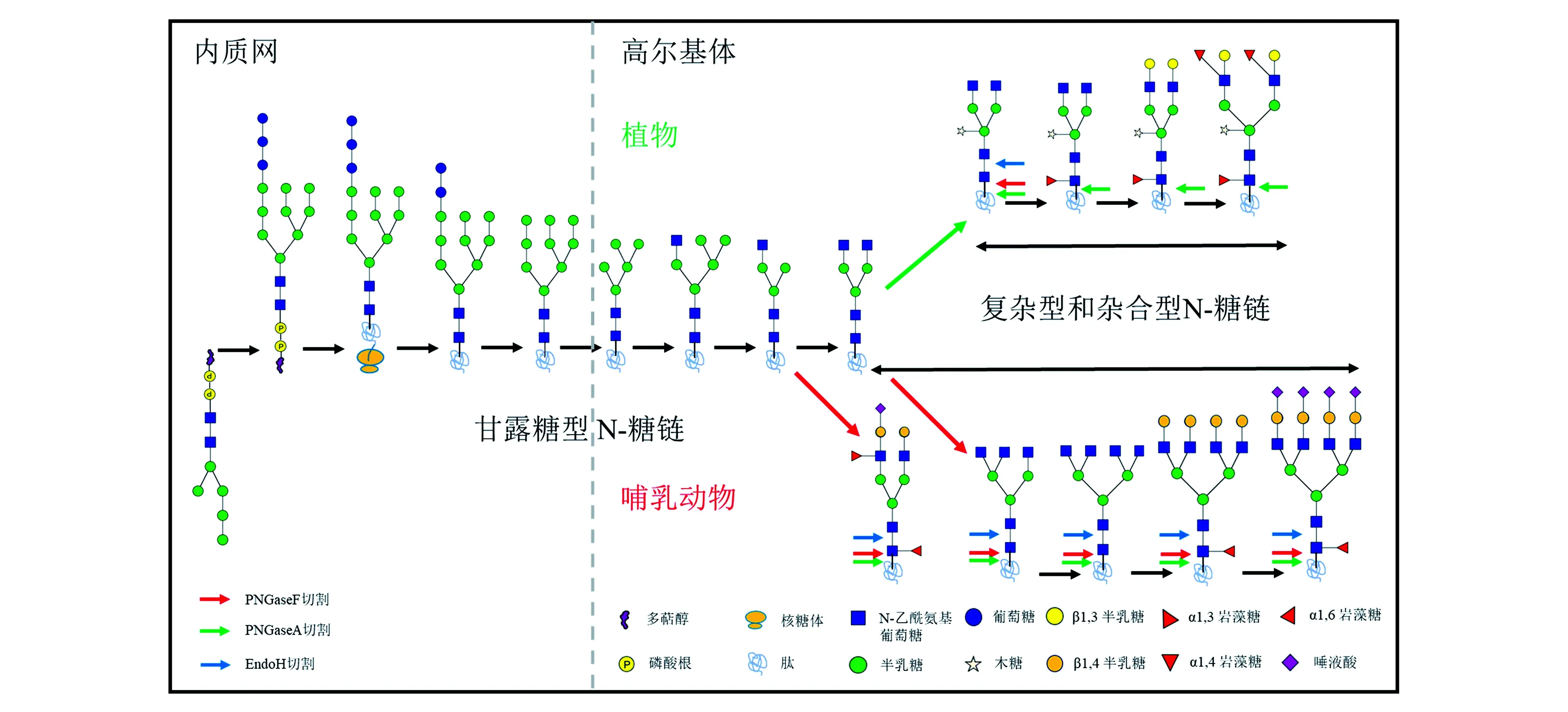
图1 植物和动物的蛋白质N-糖基化过程及差异[13]Fig.1 Process and difference of protein N-glycosylation in plants and animals[13].(彩图见图版二)
1 N-糖链的释放方法
N-糖链的释放方法主要有2种:酶切释放和化学释放。酶切释放主要依赖于肽N-糖酰胺酶(peptide-N-glycosidase,PNGase)A/F/H和糖苷内切酶(endo-β-N-acetylglucosaminidase,Endo)F/H。PNGase可特异性识别并水解连接N-糖链的天冬酰胺(asparagine,Asn)残基侧链的氨基与羰基间的酰胺键。常见的PNGase见表1[16],其中,PNGase F是一种具有普适性的肽N-糖酰胺酶,已被广泛应用于释放哺乳动物N-糖链,但其对植物中核心α-1,3-岩藻糖基化的N-糖链的释放效率较低。因此,植物N-糖链释放时通常将PNGase F和PNGase A联合使用,或单独用特异性较高的PNGase A或PNGase H+,以提高N-糖链的释放效率。Endo是一类糖苷水解酶,可特异性水解N-糖链五糖核心2个GlcNAc残基间的糖苷键,其有多个亚型,且每种亚型对水解的糖型有一定的专一性(表1),可利用这种专一性根据不同的实验目的选择合适的亚型。
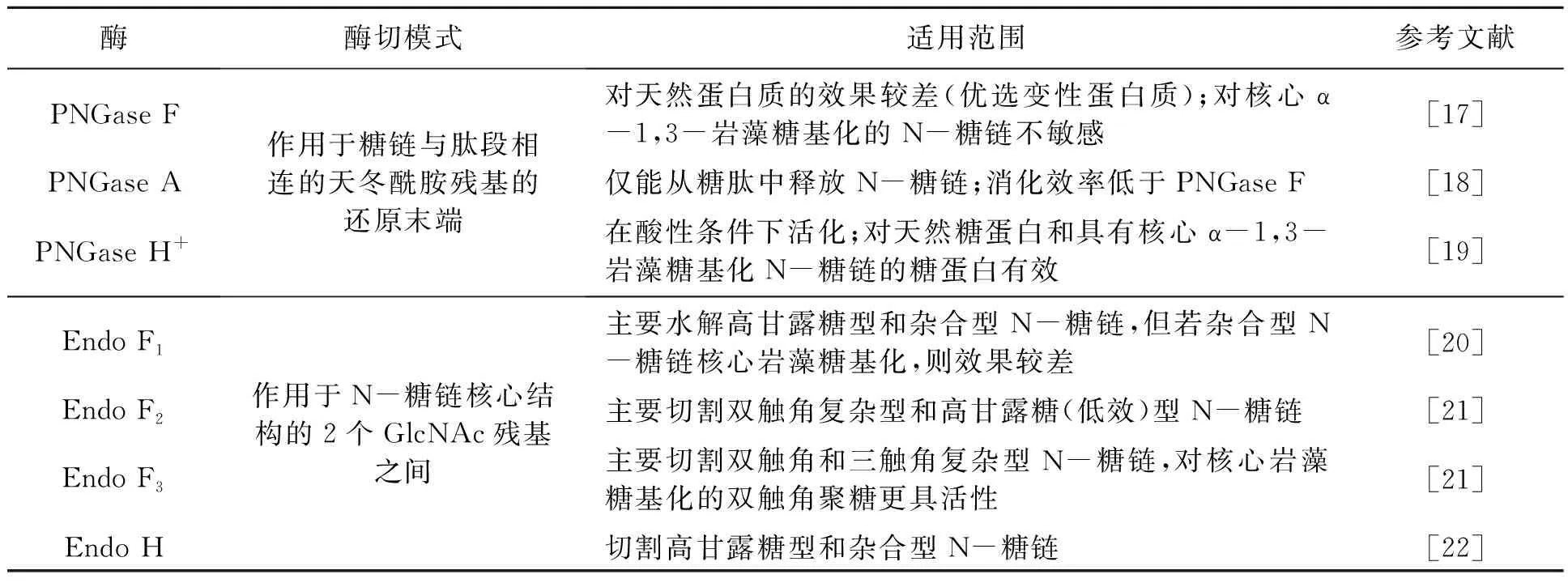
表1 PNGase和Endo切割N-糖链的特异性比较[16]Table 1 The comparison of cleavage specificity of PNGase and Endo for N-glycan[16].
综合考虑酶制剂商业化程度及其效率,利用PNGase A进行植物N-糖链的释放是目前应用最为广泛的方法。Wilson等[23]利用PNGase A从豚草种子的蛋白质中释放N-糖链,用2-氨基苯甲酰胺(2-amino benzamide,2-AB)标记后进行大小分级和反相高效液相(reversed phase-high performance liquid chromatography,RP-HPLC),最后用基质辅助激光解吸/电离飞行时间质谱(MALDI-TOF MS)进行检测,最终从豚草种子中鉴定出8种N-糖链,主要是截短型和复杂型N-糖链。采用同样的方法,从水稻种子、根和苗中共检测到15种N-糖链,其中,茎和根中主要是复杂型N-糖链,种子中主要是截短型N-糖链[24,25];而从拟南芥种子的蛋白质中共鉴定到18种N-糖链,主要是高甘露糖型N-糖链[26]。
然而,传统的酶切释放无法保持蛋白质的完整结构,因此难以从反应混合物中充分分离糖链[27,28],且在荧光标记或HPLC分析前,需要进行多个纯化步骤,这会导致糖链产生较大的损失,尤其是低丰度的糖链。南京农业大学Voglmeir和刘丽团队在传统方法的基础上进行了改进,即利用PNGase H+酶逐步处理4次以完全释放N-糖链,再结合2-AB衍生和亲水相互作用色谱-超高效液相色谱(hydrophilic interaction chromatography with ultra-performance liquid chromatography,HILIC-UPLC)纯化1次即可获得N-糖链,最终从银杏种子中鉴定到14种N-糖链,其中包括之前在银杏种子中未检测到的高甘露糖型N-糖链[29]。这种基于PNGase H+的释放策略允许在更温和的条件下进行多次水解,直到检测不到糖链的释放为止,且在整个过程中纯化与碳固相萃取只需1步,减少了糖链的损失,从而可以更为充分的分离糖链。
化学法释放N-糖链因具有广谱性且成本较低,近年来也得到了一定的发展。N-糖链的化学释放主要依赖于肼、次氯酸钠和氨水等化学试剂。肼解是利用无水肼以非还原性化学释放N-糖链[30],但无水肼具有高毒性且伴随脱乙酰化副反应的发生。次氯酸钠是通过氧化来释放N-糖链[31],此方法虽然能大规模释放N-糖链,但会使N-糖链还原末端的GlcNAc丢失。2014年,西北大学王仲孚团队报道了一种新的非还原性化学释放N-糖链的策略,用NaOH释放的N-糖链通过使用1-苯基-3-甲基-5-吡唑啉酮(1-phenyl-3-methyl-5-pyrazolone,PMP)标记以避免发生剥离反应,用此方法从拟南芥叶中检测到11种N-糖链,从烟草叶中检测到10种N-糖链[32]。2018年,该团队又建立了一种利用氨水释放N-糖链的新策略,该方法也成功应用于复杂的植物样品,从银杏种子的蛋白质中检测到8种N-糖链,获得了与PNGase A酶解释放相同的效果,但该方法存在核心α-1,3-岩藻糖基化N-糖链剥离的副反应[33]。随后,他们对该方法进行改进,将氨水释放的N-糖链经氰基硼氢化钠还原后用甲基化试剂标记,避免了核心α-1,3-岩藻糖基化N-糖链的剥离反应,最终成功从荞麦花粉中检测到15种N-糖链[34]。由此可见,化学法的不断发展为植物中N-糖链的低成本释放提供了可能。
2 N-糖链的定量策略
N-糖链组成、结构的复杂性及其在液相色谱-质谱联用(liquid chromatogram-mass spectro-metry,LC-MS)技术中的电离效率差等问题是定量糖组学研究所面临的巨大困难与挑战。为了更好的表征和定量糖组,N-糖链在LC-MS分析前通常需经过衍生化以改善其电离效率。传统的糖链定量策略主要依赖于比较衍生化糖链的色谱吸收峰面积,而基于MS的同位素标记定量是在糖链中引入稳定同位素(如2H、13C、15N、18O),使得在一次质谱扫描中标记轻、重同位素的糖链具有相同的色谱行为和离子化效率,此时成对出现的质谱峰信号的相对强度可以精确地反映样品中糖链的丰度比例[35]。目前,糖链同位素标记技术主要有化学标记、代谢标记和酶促18O标记(表2)。
化学标记是应用最为广泛的N-糖链标记方法,即将稳定同位素通过化学反应掺入糖链中。如苯胺(aniline)、PMP和精氨酸(arginine,Arg)等,通常用作质量标签标记于反应糖链还原末端的醛基上(图2,彩图见图版二),进而利用标记基团的轻、重同位素对其进行定量;碘甲烷(13CH3I或CD3I)则用作质量标签标记于糖链的羟基上,与普通的碘甲烷(CH3I)标记的糖链形成一定的质量差,从而实现质谱的相对定量[38,49,50]。如Atwood等[38]利用13CH3I和12CH2DI结合混合线性离子阱傅里叶变换离子回旋共振(Fourier transform ion cyclotron resonance,FTICR)质谱对人血清中23种N-糖链和鼠胚胎干细胞中5种N-糖链进行相对定量。该方法虽然应用广泛,但由于同位素质量差异极小(差异仅为0.002 922 Da),需使用高分辨率质谱仪,因此,不适合用常规质谱仪分析甲基化位点较少的N-糖链。此外,西北大学王仲孚团队利用非氘代和氘代苯胺结合电喷雾电离质谱(electrospray ionization mass spectrometry,ESI-MS)建立了人乳和牛乳中游离寡糖的相对定量研究方法[51];又基于PMP与RP-HPLC-UV-ESI-MS/MS,建立了一种用氨水快速释放O-糖链和N-糖链的方法[52]。虽然这些化学标记在植物中尚未得到充分推广,但其在复杂的动物样本中的应用为在植物中更好地开展糖组学定量研究开辟了道路。

表2 基于质谱的N-糖链同位素定量策略[16]Table 2 Mass spectrometry-based strategies for N-glycan quantitation[16].
代谢标记主要是利用生物体内的通路作用,将重同位素元素引入单糖分子,经过几代细胞培养,使目标单糖被完全标记。Lance Wells团队将酰胺-15N谷氨酰胺加入培养基作为细胞培养物的唯一氮源,使其通过己糖胺途径掺入到糖链中,从而建立了IDAWG(isotopic detection of aminosugars with glutamine)代谢标记定量策略[46]。标记后的糖链与普通酰胺-14N谷氨酰胺标记的糖链形成一定的质量差,从而实现质谱相对定量。此方法成功运用于小鼠和人胚胎干细胞N-糖链和O-糖链的相对定量[46,53]。其对培养的植物细胞N-糖链的相对定量具有较强的指导意义。
与化学标记和代谢标记相比,近年来发展的酶促18O标记的最大优势在于其标记反应在酶解过程中进行,操作简单。在H218O中利用Endo释放N-糖链时,18O被掺入糖链的还原末端(图3)[47]。但其仅在16O-和18O-标记的N-糖链之间产生2 Da的质量差异,在质谱分析中易造成同位素峰重叠。复旦大学杨芃原团队在酶促糖链18O标记反应完成后,向反应体系中加入氘代硼氢化钠,在糖链还原末端标记上18O的同时标记上1个氘原子[48],使分子量差异扩大到3 Da,大大降低了同位素峰的重叠程度,使糖链酶促定量分析结果更加可靠,并利用此方法对大鼠血清中25种N-糖链进行了相对定量分析。此方法对复杂的动物样品的适用性间接地为植物中N-糖链的酶促18O标记定量策略提供了参考。

图2 通过还原胺化反应使用Arg的GRIAL策略[36]Fig.2 GRIAL strategy using Arg by the reductive amination reaction[36].(彩图见图版二)
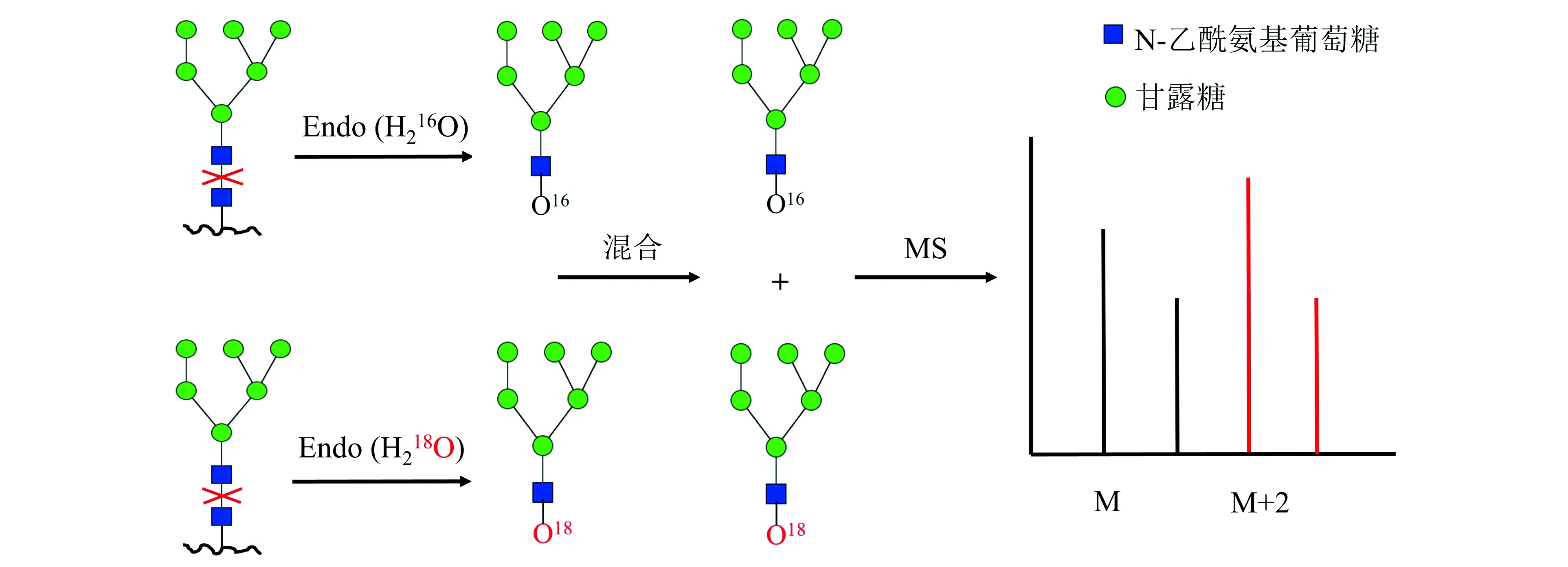
图3 在H218O中通过内切糖苷酶(Endo)从糖蛋白中释放和酶促18O标记的N-糖链策略[47]Fig.3 The strategy for release and 18O labeling of N-glycan from glycoproteins by endoglycosidase in H218O[47].
3 N-糖链的可视化检测
自1987年McCormick等[54]提出“科学计算可视化”(visualization in scientific computing)概念以来,“可视化技术”应运而生。此技术对于分析多维空间结构的具体信息进而阐明其生理机制具有非常重要的意义,并作为一门交叉学科应用于生物、医学等领域。目前,N-糖链的可视化检测方法主要有以下2大类。
第一大类是近年来在动物中建立的基于代谢标记的糖链可视化检测技术[55~58]。如将炔烃以代谢的方式掺入生物分子(包括核酸、蛋白质、脂质和聚糖)中,而炔烃在细胞的拉曼-沉默区域内具有独特的拉曼散射,将这种光谱特征与受激拉曼散射(stimulated raman scattering,SRS)显微镜相结合用于观察炔烃标记的活细胞生物分子成像[59];此外,可利用包封叠氮糖配体的靶向脂质体,选择性标记细胞培养物中特定细胞类型的糖链,通过靶向标记体内肿瘤相关糖链以达到可视化成像的目的[60]。
2016年,北京大学陈兴团队首次在拟南芥中报道了使用乙酰化N-叠氮乙酰葡糖胺(Ac4GlcNAz)作为化学报告分子的代谢标记N-糖链可视化检测技术[61]。此方法利用N-叠氮乙酰葡糖胺(GlcNAz)通过GlcNAc的补救途径掺入到N-糖链中,通过荧光探针进行点击化学标记,从而显示N-糖链的含量及分布情况(图4)。此项研究不仅证实了GlcNAc补救途径的存在,而且发现不同发育时期根组织中N-糖基化的分布模式不同,表明N-糖基化以阶段依赖性方式调节根发育。此项研究是糖链代谢标记在阐明植物N-糖基化功能中的实际应用,打破了植物中传统的糖链检测方法,实现了对植物体内N-糖链进行可视化监控,为阐明植物中N-糖基化功能提供了新思路和新方法。
另一种N-糖链可视化检测的方法是基于美国范德堡大学Caprioli教授于1997年提出的基于基质辅助激光解吸/电离(MALDI)质谱成像技术而得以实现的[62],经过20年的发展,2016年,Caprioli教授又提出了全新的质谱成像策略,即基于超快速基质辅助激光解吸/电离飞行时间质谱(MALDI-TOF)和超高分辨基质辅助激光解吸/电离和激光解吸/电离傅里叶变换离子回旋共振质谱(MALDI FT-ICR)的新一代质谱成像技术[63]。此技术借助超快速的质谱成像系统,将PNGase F通过溶剂喷雾器作为分子层应用于冷冻或福尔马林固定的组织上,并且使用基质辅助激光解吸/电离成像质谱(MALDI-IMS)检测指定组织区域中所有释放的N-糖链,从而全面观察复杂的生物组织样本。此技术在调控哺乳动物癌症和关节炎等疾病的N-糖基化可视化研究中得到了广泛的应用,如利用此技术检测到位于关节炎患者骨髓中的二唾液酸化双触角复杂型糖链NeuAc2Gal2GlcNAc2Man3GlcNAc2[64]和几乎全部位于平滑肌肉瘤区域的复杂型N-糖链GalGlcNAc2Man3GlcNAc2和GalGlcNAc2Man3GlcNAc2Fuc[65],上述研究结果揭示了特定N-糖链在疾病中的重要作用。此技术与传统糖链检测技术相比,其优势在于无需标记样本,也不需要经过繁琐的提取、纯化、富集等步骤,一次数据采集即可获取成千上万不同的分子空间分布信息,且能较好地呈现原位信息。虽然目前此技术尚未在植物N-糖基化研究中得到应用,但是未来在植物N-糖链空间可视化检测方面具有很大的空间,同时对于阐明N-糖基化机制的研究也有重要的指导意义。
4 展望
近年来,植物N-糖组的研究受到了广泛的关注,已在拟南芥、水稻、玉米等多种植物上开展了相关研究,但由于植物较哺乳动物样本的糖链丰度更低以及植物中糖链检测方法的不完善,使得植物中N-糖组的研究仍停留在对糖链检测方法的探索阶段。而最近发展起来的N-糖链代谢标记和质谱成像可视化检测等新技术在哺乳动物中的成功运用将极大地推动植物中N-糖组的研究,同时,植物中N-糖组的研究以及N-糖链检测技术的不断改进也将极大地推动整个植物糖生物学的发展。此外,由于糖链片段化谱的注释和二级结构的确定仍然是手动的,所以更全面的糖链数据库的构建与N-糖链相匹配的数据处理软件的开发将成为今后的重点发展方向。
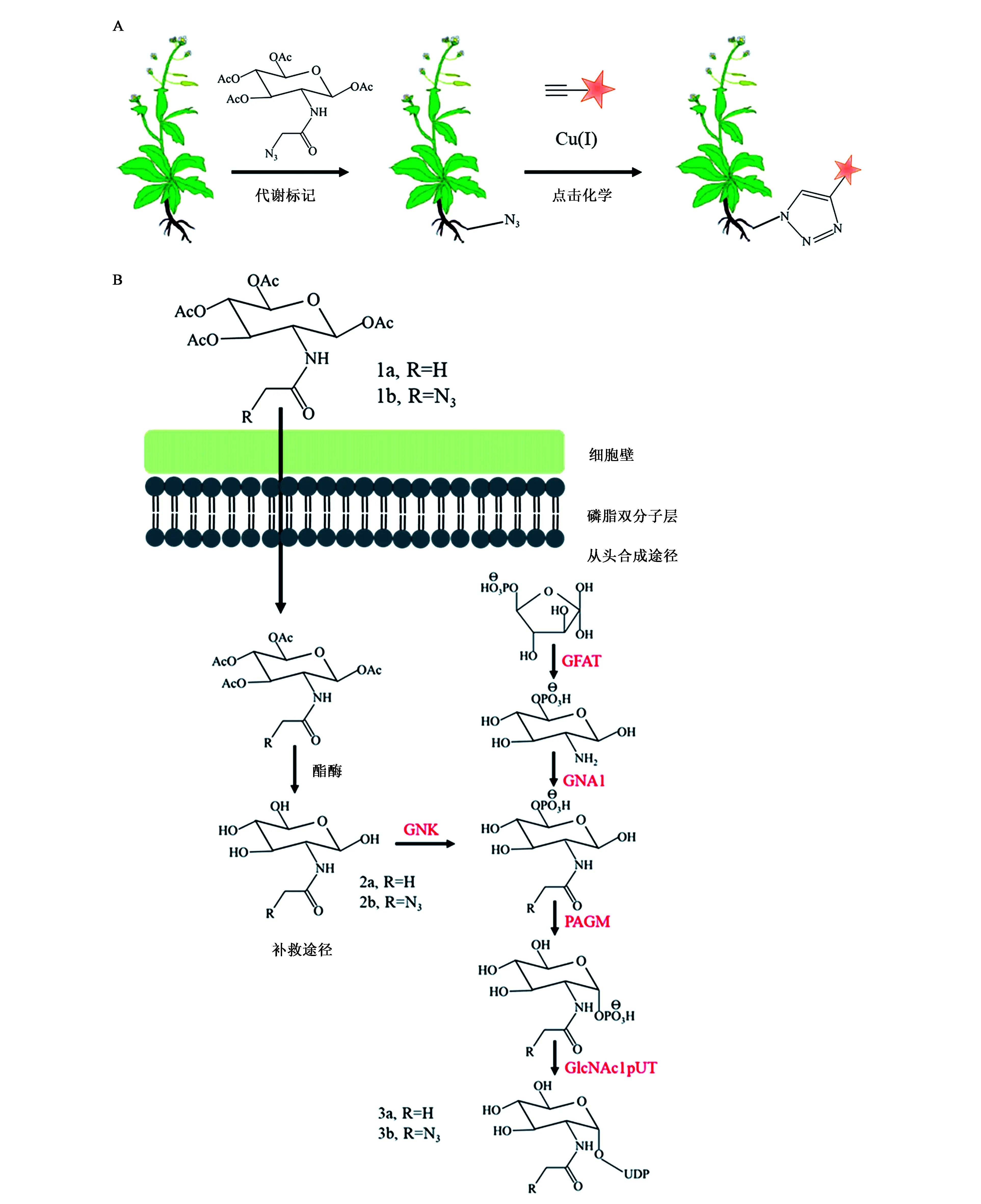
图4 拟南芥N-糖链的Ac4GlcNAz代谢标记[61]Fig.4 Metabolic labeling of Arabidopsis N-glycans with Ac4GlcNAz[61].注:A:Ac4GlcNAz标记N-糖链的基本流程;B:Ac4GlcNAz标记N-糖链所涉及的代谢途径。GNK:GlcNAc激酶;GFAT:谷氨酰胺:果糖-6-磷酸氨基转移酶;GNA:GlcN-6-磷酸乙酰转移酶;PAGM:乙酰葡糖胺磷酸变位酶;GlcNAc1-pUT:GlcNAc-1-磷酸尿苷酰基转移酶。

