传统发酵乳制品中植物乳杆菌的种群结构及遗传进化
,,,,,
(东北农业大学食品学院,乳品科学教育部重点实验室,黑龙江哈尔滨 150030)
乳酸菌因其潜在的益生特性而被广泛认可。植物乳杆菌是乳酸菌的一种,其兼性厌氧,来源安全,广泛分布于乳、肉、蔬菜等食材及其发酵制品中[1-2],此外,还存在于人和动物的消化道中[3]。植物乳杆菌通过发酵可转化生鲜食品中的营养成分[4],提升营养价值,对食品工业和人体健康有着多重有益的功效,具有一定的商业利用价值。
近年来,多种分子分型方法被广泛应用,包括脉冲场凝胶电泳、限制性片段长度多态性、扩增片段长度多态性、随机扩增多态性DNA等。但这些方法针对相似营养需求或相近生态位的菌株,其得到的图谱可能是不准确的[5]。多位点序列分型技术(multilocus sequence typing,MLST)是菌株种内分型的强有力工具[6]。它衍生自多位点酶电泳法,基于部分持家基因,可有效探索菌株的系统发育关系、遗传进化以及种群结构[7-8]。持家基因是维持细胞基本生命活动所必须的基因,其在所有细胞中均稳定表达。在MLST研究中,所选择的持家基因需满足以下条件:具有保守的编码蛋白的能力;均匀分布于整个基因组;单拷贝,长度不短于1 kb[7]。MLST技术首先应用于流行病学领域,用于致病菌的进化和种群生物学研究,而后拓展至乳酸菌的多样性和系统进化分析。乳酸菌中利用此法完成分析的菌株包括干酪乳杆菌、德氏乳杆菌、发酵乳杆菌、旧金山乳杆菌、乳明串珠菌等[7,9-12]。对于植物乳杆菌,已有学者对分离自水果、蔬菜、青贮等材料的菌株进行MLST分析[13-14]。
本文利用MLST方法,选取7个持家基因,对分离自内蒙古和西藏自治区传统发酵乳制品中的植物乳杆菌的种群结构和遗传进化进行分析,从而在基因水平上为菌株的分型和工业食品发酵剂的筛选提供参考,为后续益生菌发酵产品的开发提供菌种资源。
1 材料与方法
1.1 材料与仪器
61株植物乳杆菌分离株 56株分离自内蒙古和西藏自治区传统发酵乳制品中,1株模式菌株L.plantarumATCC 14917T,4株参考菌株L.plantarumWCFS1、L.plantarumP-8、L.plantarumZJ316和L.plantarumJDM1;MRS肉汤培养基 Sigma公司;磷酸盐缓冲液 天津市瑞金特化学品有限公司;2×Taq PCR MasterMix 北京天根生物技术有限公司;细菌基因组DNA提取试剂盒 北京天根生化科技有限公司。
YQX-II型厌氧培养箱 上海龙跃仪器设备有限公司;高压蒸汽灭菌锅 上海博讯实业有限公司医疗设备厂;Bcn1360型生物超净工作台 北京东联哈尔仪器制造有限公司;高速冷冻离心机 上海离心机械研究所;7000 PCR扩增仪 美国Applied Biosystems公司;NanoDrop 2000超微量分光光度计 美国Thermo分光光度计公司;涡旋震荡器 Mo-Bio公司。
1.2 实验方法
1.2.1 持家基因的选择 本研究采用7个持家基因,其中4个基因(pheS、groEL、murC、murE)及其引物是引用Xu等[15]的研究,其余3个基因(recA、recG、ileS)则是选自德氏乳杆菌的MLST研究[9],并以L.plantarumWCFS1的持家基因的基因片段为模板,利用Primer Premier 5.0软件进行引物设计。所选择的持家基因及其引物信息见表1。
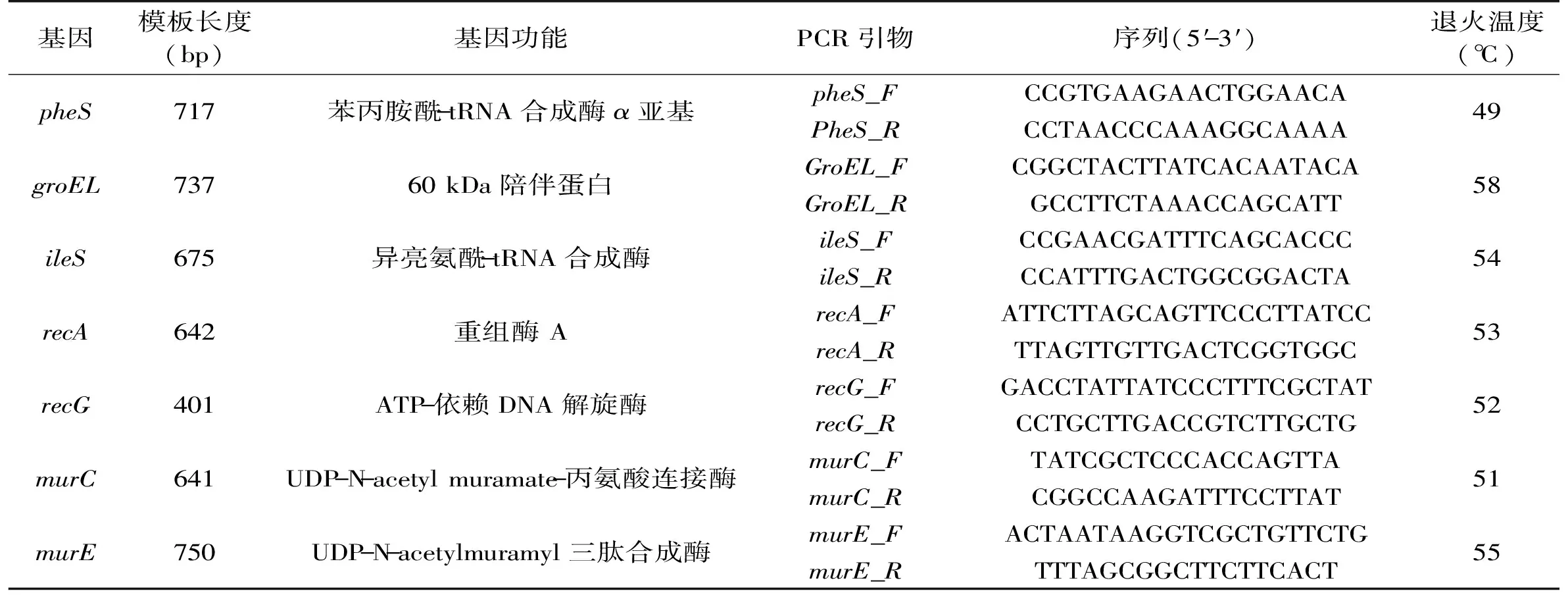
表1 植物乳杆菌多位点序列分型的持家基因及其引物Table 1 The information of housekeeping genes and primers for MLST of L. plantarum
1.2.2 菌株的培养 将甘油冻存的植物乳杆菌分离株,接种至MRS肉汤培养基,置于厌氧培养箱中37 ℃培养24 h,经传2代培养至对数期,取2 mL菌液离心后倒掉上清,待用。
1.2.3 DNA的提取、扩增及测序 采用细菌基因组DNA提取试剂盒提取总基因组DNA,具体步骤按照说明书进行操作。使用表1中的引物进行PCR扩增,扩增体系为50 μL,包括16 μL 2×Taq PCR MasterMix,各1 μL引物,4 μL DNA模板,以双蒸水补足体积。扩增条件为:94 ℃预变性5 min;94 ℃变性30 s,适宜退火温度(表1)30 s,72 ℃延伸30 s,30个循环;72 ℃末端延伸5 min。扩增后的产物经1%的凝胶电泳进行检测,将条带匹配的产物送至北京诺赛生物科技有限公司,进行纯化及双向测序。模式菌株和参考菌株的持家基因序列直接从NCBI数据库下载获得。
1.3 数据分析
一个独特的核酸序列分配一个等位基因号,7个持家基因的不同等位基因号组合构成一个独特的等位基因谱,也就是序列型(sequence type,ST)。利用MEGA 7.0软件对序列进行修剪和比对,进而得出等位基因图谱。利用DnaSP和START软件计算多态位点数、Tajima’s D值、G+C含量(%)、核酸多态性(π)、非同义突变与同义突变比率(dN/dS)以及IA(index of association)。采用eBURST软件对ST型进行克隆复合体归类分析。用MEGA和STRUCTURE对分离株进行系统发育和祖先种群结构探索。采用SplitTree分析菌株的重组情况及遗传进化关系。
2 结果与分析
2.1 核酸多样性分析
等位基因遗传多样性相关系数如表2所示。7个位点的等位基因数处于3~10之间,pheS基因多样性较丰富。多肽位点数在3(recG)~12(pheS)之间,π值处在0.00219(groEL)至0.00720(ileS)之间,显示出较低的位点突变率。Tajima’s D值在-1.33698(groEL)和-0.11553(pheS)之间,均小于0,表明基因序列在进化过程中受定向选择。G+C含量是细菌遗传进化研究中的重要指标,本研究中7个基因序列的G+C含量在42.86%(groEL)和50.99%(murE)之间,平均含量为44.88%,高于模式菌株L.plantarumATCC 14917T的G+C含量(44.50%),表明持家基因的保守性较好。同义突变指核苷酸的变异不会引起所编码的氨基酸的改变,非同义突变指核苷酸的变异会导致氨基酸的改变,本研究中dN/dS均小于1,位于0.021(ileS)~0.927(murC)之间,表明基因变异不会引起氨基酸的变化,其非同义突变受纯化选择,此7个持家基因可用于植物乳杆菌的MLST研究。以上遗传多样性相关系数揭示了这些基因处于稳定化选择[10,12]。
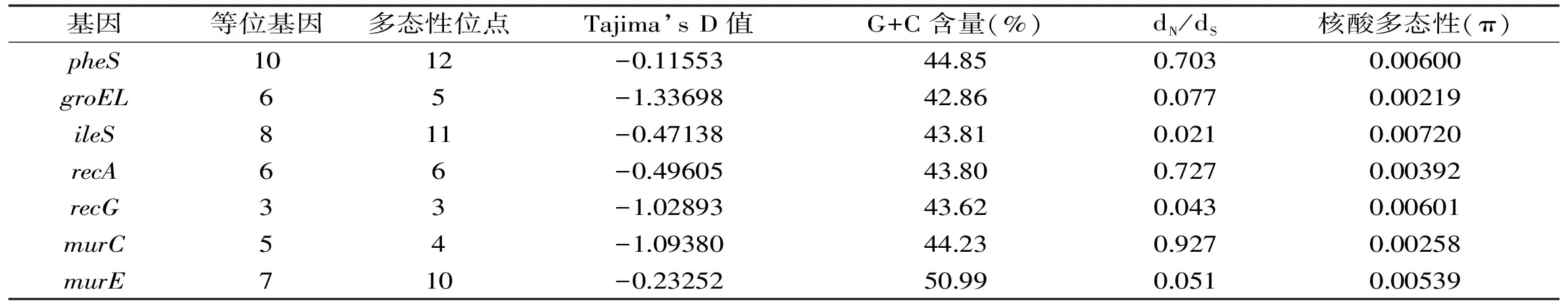
表2 持家基因的遗传多样性Table 2 Genetic diversity at L. plantarum loci
2.2 序列型和克隆复合体
不同等位基因的不同组合共分成27个ST型,其中的20个ST型只对应单一菌株。根据生成的等位基因图谱,利用eBURST软件规划克隆复合体来探索菌株的遗传进化关系。图1 eBURST图谱中,每个点代表一个ST型,数字代表该点所对应的ST型编号,点的大小与所对应ST型的菌株数量成正比。7个等位基因中,不少于5个相同等位基因号的ST型划分为一个克隆复合体(clonal complex,CC)。27个ST型共归为6个CCs和6个独特型(singleton)。CC1、CC2、CC4、CC9、CC10和CC23包含90.9%的分离株。在CC1中,ST-1占据中心位置,包含最多菌株数(28株),被认为是CC1的原始序列型。CC1中的多数菌株来自于西藏自治区,而在CC3中则多是分离自内蒙古自治区。ST-10包含两株分离自发酵乳制品的菌株以及参考菌株L.plantarumP-8,表明来自同一原料的菌株是极为相近的。分离自青贮的参考菌株L.plantarumJDM1同分离自发酵乳制品中的菌株ST-17以单位点差异而共居于CC4,可能是因为它们生活在邻近的生态位。源自人唾液的L.plantarumWCFS1,健康婴儿粪便的L.plantarumZJ316以及模式菌株L.plantarumATCC 14917T均是独特型。综上分析,可认为相同分离源、分离地的菌株具有相似的核酸多样性,换言之,不同原料、不同加工方式会影响植物乳杆菌的遗传进化,加速其对特殊生存环境的适应性。在旧金山乳杆菌和干酪乳杆菌的MLST研究中,提出了加工环境是细菌进化过程中影响其生存适应性的重要因素[7,11]。然而,也有研究学者表示进化和菌株分离源之间并没有关系[10,13]。而本研究中的菌株是采样于相对较窄的生态位,因此得出的结论还需进一步验证。针对这个问题,未来的研究方向会关注于更多样的分离源、分离地域。
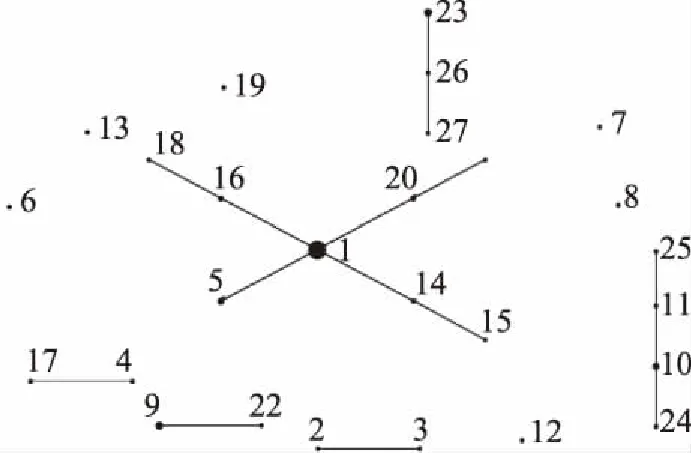
图1 基于等位基因图谱的eBURST分析Fig.1 eBURST analysis based on allelic profiles
2.3 种群结构
通过MEGA软件,采用邻接法对串联的序列片段进行聚类分析的结果与利用STRUCTURE评估的种群结构具有较好的一致性(图2),27个ST型共分成4个遗传谱系。2B图中每行代表一个ST型,不同的颜色代表不同的祖先来源。Cluster 1包含13个ST型,其中的菌株多数源自内蒙古发酵乳制品,其中还包含参考菌株L.plantarumP-8、L.plantarumZJ316和L.plantarumATCC 14917T。Cluster 2仅含有1个ST型,包含单一菌株L.plantarumWCFS1。从图2B中可以看出,超过半数的菌株是单一祖先来源,即每个横线单一颜色有至少85%的占比。剩余的ST型则是由混合的祖先群体构成,在遗传重组过程中,每一个颜色所代表的祖先群体既可以是捐赠者也可以是获得者,因而分离株中呈现了异质的祖先群体结构。Xu等也得出了相同的结论,重组可导致遗传信息的异质性[15]。
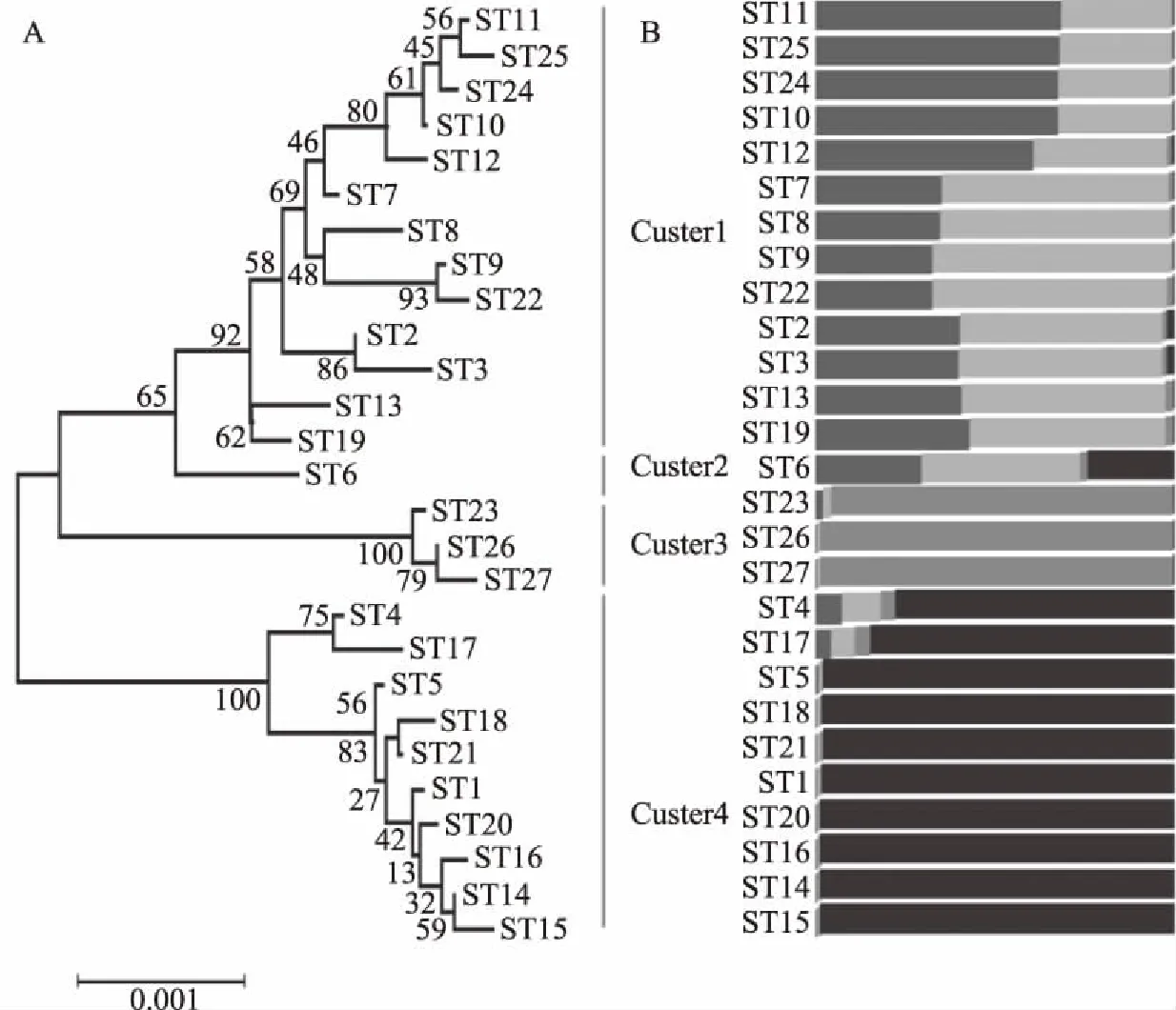
图2 进化树及种群结构Fig.2 Evolutionary tree and population structure注:A:基于串联基因序列获得的N-J树;B:由STRUCTURE获得的祖先来源谱系。
2.4 重组分析
利用IA对植物乳杆菌分离株进行连锁失衡分析,若IA的值显著不同于0,则存在克隆结构,期望值为1,反之则存在重组导致的连锁平衡,期望值为0。本研究中IA和IAS的值分别为1.4364和0.2394(p=0.000),表现出强烈的连锁失衡,表明这些基因中存在克隆结构。此现象也发生在其他乳酸菌的MLST研究中,如乳酸乳球菌、乳明串珠菌等[12,16]。同样,在地衣芽孢杆菌和嗜热链球菌中也已被证实存在[17-18]。
采用重组分解分析来探究分离株的重组现象,在分解分裂图上,若出现相互交错的网状结构或平行四边形结构则说明重组参与其进化。若出现树状图形,则说明该谱系是克隆结构。在单基因的分解分裂图上,pheS出现了平行四边形结构,而其余6个基因则是树状结构,说明pheS基因在进化过程中存在重组,剩余6个基因则无明显的重组现象。针对于pheS基因,在乳明串珠菌的多位点序列分型研究中也发现了重组现象[12]。对串联的持家基因序列进行重组分解分析,在分解分裂图上可以明显的看到网状结构(图3),证明植物乳杆菌分离株在遗传进化过程中受重组影响。并且在植物乳杆菌基因组中存在质粒和一些移动元件,也可能有益于重组的发生[19]。27个ST型分成4组,与进化树及种群结构谱系的结果相一致。

图3 重组分解分析Fig.3 Split-decomposition analysis
3 结论
MLST技术是一种有效且分辨率较高的分型方法,被广泛用于探究菌株的基因多样性、系统发育关系、遗传进化及种群结构。本文基于7个持家基因序列,对分离自内蒙古和西藏自治区传统发酵乳制品中的61株分离菌进行遗传进化和种群结构探索,在这些分离株的基因上,具有较高的遗传多样性,存在克隆结构,且重组影响其遗传进化。本研究从基因水平为菌株的分型和工业食品发酵剂的筛选提供了新的理论与技术支持,且通过分离得到的植物乳杆菌纯化菌株可作为潜在乳酸菌发酵剂,为后续益生菌发酵产品的开发提供菌种资源。

