高分辨率熔解曲线在CRISPR基因编辑检测和基因型鉴定中的应用
贺 文, 吴婉婉, 陆 平, 薛志刚, 盛哲津, 祝献民
(1. 同济大学医学院,上海 200092; 2. 同济大学生命科学与技术学院,上海 200092)
成簇的、规律间隔的短回文重复序列技术(clu-stered regularly interspaced short palindromic repeats, CRISPR)是近年来发展起来的一种高效的基因编辑工具,已经广泛的应用在基因治疗、基因功能研究、农作物品种改良、动物模型制作、靶向药物筛选和材料生物学等领域[1]。它能够快速、高效地对特异的基因组位点进行切割或置换,而且特异性高,细胞毒性低[2]。然而基于核酸内切酶的CRISPR基因编辑检测存在灵敏度低,特异性差等问题[3]。基因型鉴定分析是遗传学和生物医学领域最常见的分析手段,但高通量快速鉴定基因型仍然是亟待解决的问题[4]。综上,快速和高效地检测CRISPR基因编辑产物以及生物样本的基因型具有非常重大的应用前景。高分辨率熔解曲线(high-resolution melting curve, HRM)分析是近年来发展起来的一种新型基因分析技术,通过区分不同核酸组成的熔解曲线差异,能够鉴定基因组突变和单核苷酸多态性[5]。HRM技术具有高特异性、高灵敏性、高通量、低成本、速度快等优点,因而在临床诊断及基因分析上得到迅速的应用与发展,现已成为生命科学研究中的热点技术[6-7]。每种DNA具有特定的熔解曲线[8],因此HRM可以应用于CRISPR技术切割产物以及基因型的快速鉴定。然而目前HRM分析通常依赖PCR平台匹配的收费软件,由于版权和价格问题,不利于在实验室推广。
本研究将在线HRM分析软件与PCR技术进行结合,提高了CRISPR技术体外切割产物验证及不同基因型鉴定的效率。运用该方法准确验证了CRISPR系统在非洲猴肾细胞COS7的视网膜色素上皮65(retinal pigment epithelium 65, RPE65)基因处的切割效率,以及对瘦素(leptin, Lep)突变的SD大鼠的3种子代基因型进行快速的鉴定,并且以CRISPR所产生的不同切割产物和不同基因型PCR扩增产物的熔解曲线为例,详述了HRM分析软件的使用方法。
1 材料与方法
1.1 材料
1.1.1 样本来源 本研究使用非洲猴肾细胞COS7和斯泼累格·多雷(Sprague Dawley, SD)大鼠。COS7细胞体外转染了gRNA和Cas9。鉴定基因型的SD大鼠为Lep敲除的杂合子大鼠(LepΔI14/-)的子代[9],分别为野生型(WT)大鼠、杂合突变(LepΔI14/-)和纯合突变(LepΔI14/ΔI14)大鼠。
1.1.2 主要试剂和仪器 细胞DNA提取试剂盒购自北京天根生化科技有限公司;鼠尾基因组DNA提取试剂盒购自美国Bimake公司;Taq DNA酶,DNA荧光染料EvaGreen购自上海翊圣生物科技有限公司;Q5酶购自美国NEB公司;PCR引物由苏州金唯智生物科技有限公司合成;内参染料ROX购自大连宝生物工程有限公司;QuantStudio7 PCR仪购自美国美国应用生物系统公司;电泳仪购自上海天能科技有限公司。
1.2 基因组DNA提取
使用细胞DNA提取试剂盒提取COS7细胞基因组;使用鼠尾基因组DNA提取试剂盒提取Lep敲除的杂合子大鼠子代基因组,均置于-20℃保存备用。
1.3 引物设计
分别根据食蟹猴RPE65(AGTTTCTTGTAAC-CACCAGC)和大鼠Lep突变位点[9]的序列信息,使用在线引物设计软件Primer3(http:∥primer3.ut.ee/)设计RPE65和Lep突变位点所对应的引物。
1.4 PCR扩增
为了富集突变位点序列,首先扩增300~600bp目的片段[10]。反应体系为20μL,包括10μL缓冲液,7μL水,1μL上游引物,1μL下游引物和1μL DNA。反应条件为: 94℃预变性5min,94℃变性20s,58℃退火30s,72℃延伸20s,共35个循环,最后72℃ 5min。用ddH2O将PCR产物稀释10000倍,作为第2次PCR模板。第2步PCR产物长度范围为45~150bp。反应体系为20μL,包括11μL水,4μL Q5缓冲液,0.4μL dNTP,1μL上游引物,1μL下游引物,0.2μL Q5酶,1μL 20× EvaGreen染料,0.4μL 50× Rox和1μL模板DNA。PCR反应条件为: 98℃预变性30s,98℃变性10s,60℃退火15s,72℃延伸10s,共50个循环,随后继续95℃加热解链2min,25℃ 2min,最后4℃ 2min。
1.5 HRMA检测
本次研究采用384孔,每孔转入10μL PCR产物。在实时荧光定量PCR仪(QuantStudio7)的参数设置中,分别选择“Melt curve”和“Sybr Green”,连续荧光的读数间隔为0.05s。实验结束,使用QuantStudioTMReal-Time PCR软件获得熔解曲线图。
1.6 在线软件HRM Analyzer数据分析
QuantStudio7所得温度和荧光读数等数据整理成.txt文件格式,将所得文件上传至在线软件HRM Analyzer(http:∥www.flyrnai.org/hrma),得到标准化图和聚类图,最后进行HRM分析。
2 结 果
2.1 引物特异性检测
本研究根据相应的基因组信息分别设计了针对食蟹猴RPE65基因和大鼠Lep基因的PCR引物,引物序列如表1所示。为了验证PCR引物的特异性,本研究对PCR产物进行了琼脂糖凝胶电泳。两步PCR的产物条带都非常均一,没有杂带。这说明所设计的引物能够特异性识别模板,可用于HRM分析,见图1。

表1 PCR引物Tab.1 The PCR primers
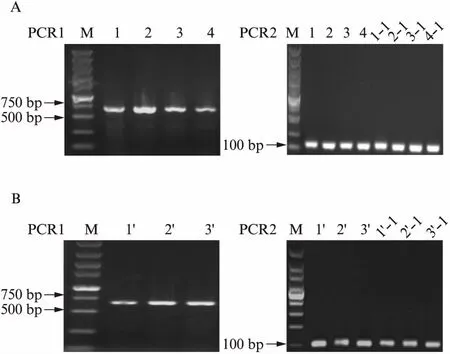
图1 两步PCR琼脂糖凝胶电泳图Fig.1 Agarose gel electrophoresis of the PCR productsA: RPE65基因两步PCR扩增条带;1、2、3、4分别为阳性对照、阴性对照、MOI值为100和20的慢病毒转染COS7细胞;1-1、2-1、3-1、4-1为重复实验;B: Lep基因两步PCR扩增条带;1’、2’和3’分别为野生型、杂合突变(LepΔI14/-)大鼠和纯合突变(LepΔI14/ΔI14)大鼠;1’-1、2’-1、3’-1为重复实验
2.2 CRISPR基因编辑检测
本研究利用慢病毒将CRISPR系统导入COS7细胞,对RPE65基因进行编辑。为了检测基因编辑效率,共选取4种样品,分别为未转染COS7细胞(阴性对照)、RPE65基因已被CRISPR系统编辑的COS7细胞(阳性对照),MOI值分别为100和20的慢病毒转染的COS7细胞(实验组)。细胞基因组提取之后置于QuantStudio7 PCR仪中得到熔解曲线。阳性对照与其他3个样品有明显区别,由此可以初步说明CRISPR系统在被转染的细胞中发挥了编辑作用,见图2A。为验证这一结果,将数据整理并上传至在线软件HRM Analyzer进行分析。阳性对照和其他样品可以区分开来,见图2B、C;同时实验组和阴性对照聚类,表明CRISPR系统在慢病毒感染的COS7细胞中并未编辑RPE65基因,该结果与T7EI检测结果一致,见图2D。与阳性对照相比,实验组未出现3条带,COS7细胞中RPE65基因没有被编辑。
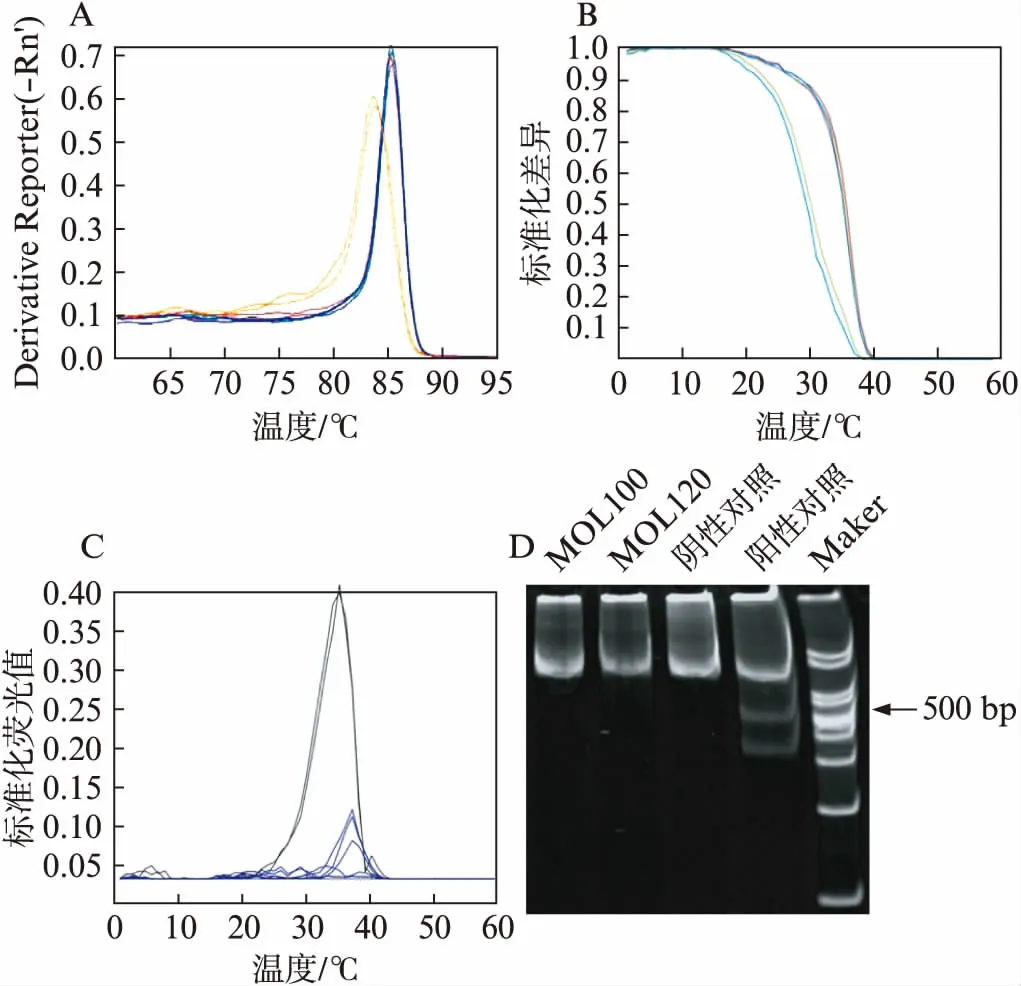
图2 HRM和T7E1分析COS7细胞RPE65基因编辑结果Fig.2 HRM and T7E1 analyses of RPE65 gene editing in COS7 cellsA: QuantStudioTM Real-Time PCR软件所得PCR产物熔解曲线,阳性对照为图中黄色显示曲线;B、C: HRM Analyzer所得标准化图与聚类图,阳性对照分别是图B中位于上方和图C中位于下方的曲线;D: T7酶切验证结果,与阳性对照相比实验组未出现三条带
2.3 基因型鉴定
纯合突变(LepΔI14/ΔI14)大鼠不育,繁殖Lep突变的大鼠需要杂合子[9]。为鉴定子代基因型,本研究使用HRM进行鉴定。同样在PCR之后,将所得的数据传至软件QuantStudio Real-Time PCR Software,实验结果显示杂合突变大鼠与纯合大鼠熔解曲线只有微小差别,见图3A。为验证这一实验结果,将数据上传至在线软件HRM Analyzer进行分析验证。杂合子对应曲线聚为一类,而纯合子和野生型大鼠对应曲线聚为一类,见图3B、C。HRM分析得到的结果与DNA测序结果一致。这说明HRM可将纯合和杂合突变基因区分开,却未能区分纯合突变和野生型大鼠。
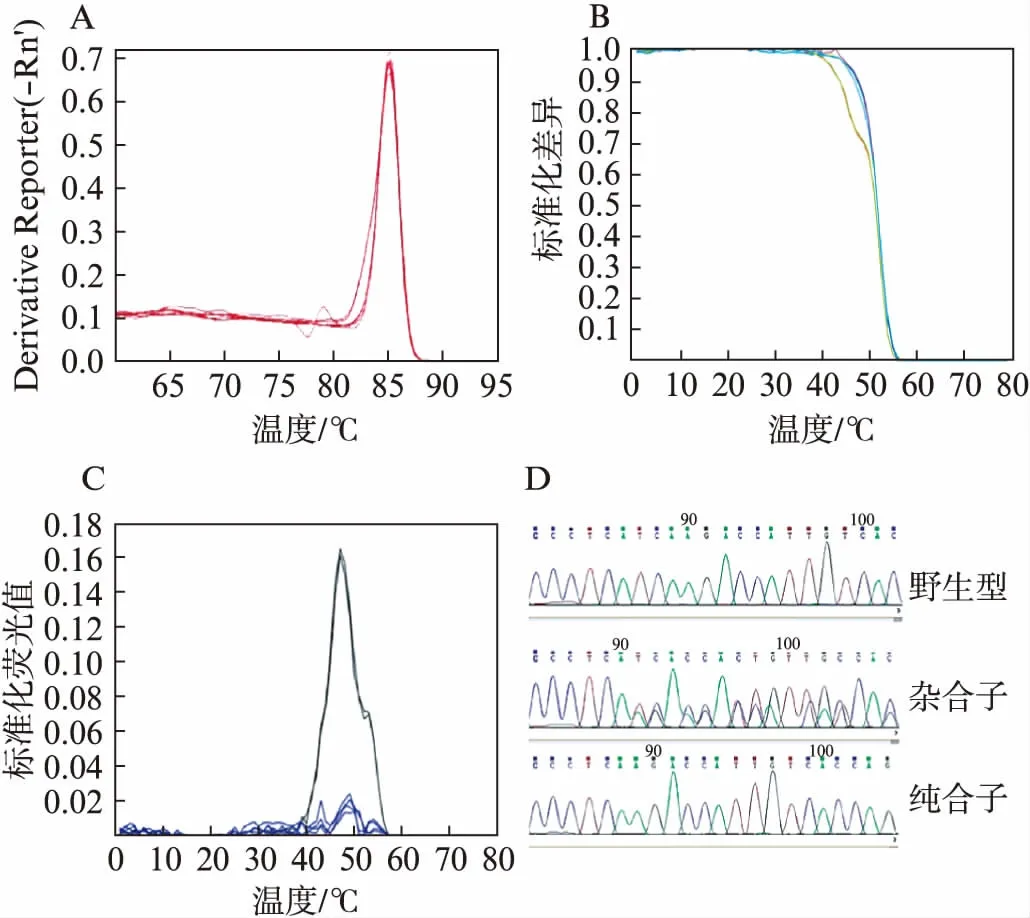
图3 HRM与DNA测序分析Leptin杂合大鼠(LepΔI14/-)子代基因型结果Fig.3 Genotyping of LepΔI14/- rat progeny by HRM and DNA sequencing analysisA: QuantStudio Real-Time PCR软件所得PCR产物熔解曲线,杂合子为左边突出的曲线;B、C: HRMAnalyzer所得标准化图和聚类图,杂合子分别是图B位于下方和图C位于上方的曲线;D: DNA测序分析Lep突变大鼠3种子代基因型
3 讨 论
HRM是近年来开发的用来基因分型和突变扫描的新型遗传学分析方法,可以用来检测目的片段基因组中的序列变化。DNA双链结合饱和荧光染料进行变性升温,然后利用荧光读取DNA解链的过程中的特异熔解曲线而达到鉴定不同DNA的目的[11]。与传统的PCR方法及常规的测序手段相比较,这种方法可在进行两步普通的PCR反应中实现,无需电泳鉴定,具有快速、简便、经济、可重复、污染低等优点。一旦PCR模板中基因组序列发生了改变,HRM分析软件在标准化与聚类后就能准确检测到相应的熔解曲线的变化。因此,这种方法比传统的PCR和测序手段更为便捷和简单,并且更为经济和环保[12-13]。最近,Housden等[10]在果蝇中使用HRM检测了CRISPR技术编辑后的产物,与此同时,将这项技术应用在CRISPR在细胞和哺乳动物水平的检测,对HRM分析手段进行了拓展与延伸。
CRISPR技术的诞生为科学研究和人类疾病的治疗提供了有力的手段和良好的平台,同时也为精准医疗的实现提供了新的策略和方向[14]。但由于其基因编辑效率低,且存在较大的脱靶效应,因此,该种技术手段在基因编辑之后可能会存在多种作用产物。Xu等[9]的研究中对CRISPR-Cas9技术切割产物的验证使用T7EI核酸内切酶进行验证,和本文所阐述的HRM的方法相比较,T7EI核酸内切酶验证的方法不灵敏,非特异性结合比较大,且产物采用电泳实验后进行肉眼观察,可能会引入人为的误差,导致检测结果产生偏差[15]。相比之下,HRM方法使用荧光染料进行检测,灵敏度高,且产物均在原管中进行反应,不会引入外界污染而造成不必要的误差;在两步PCR反应结束后,只需10~20min进行熔解曲线的检测,再通过在线HRM软件分析,更为方便、经济和快捷。
如上所述,HRM分析可以快速鉴定CRISPR编辑和区分基因型。然而在使用在线软件分析HRM时,也发现了软件本身的一些不足。例如: 生成标准化图之前,每组数据之间的上下阈值需要多次手工调试才能生成理想的图像。而且,这种方法不能定量的分析CRISPR的编辑效率;针对大鼠Lep基因时,不能区分纯合的野生型与突变基因型。因此,HRM分析在实际应用中可以根据需要增加其他辅助方法,例如在基因型鉴定中可以对不能分型的纯合子直接进行DNA测序,同样能减少成本和时间,快速达到实验目的。
本研究对CRISPR技术编辑的COS7细胞中RPE65基因进行验证,通过使用在线软件分析,发现经过基因编辑的RPE65基因的熔解曲线与阴性对照显著不同。同样,在Lep突变的杂合子大鼠的子代基因型中,发现HRM可以明显区分杂合子和纯合子。尽管HRM分析还存在某些不足,但是经过进一步的改良,HRM分析技术必将在CRISPR基因编辑检测和基因型鉴定等研究领域展现更广阔的应用前景。

