基于黄褐棉导入系的抗黄萎病QTL鉴定分析
邓 琳, 刘 静, 张 咪, 黄 芳, 周水娟, 刘 倩, 梅 艳, 王 泽, 王 凯, 汪保华
[1.南通大学生命科学学院,江苏南通 226019; 2.福建省海峡植物应用系统生物学重点实验室(福建农林大学),福建福州 350002]
棉花是纺织工业的重要原料,也是重要的油料作物[1]。棉属(GossypiumL.)包含4个栽培种,即草棉(G.herbaceum)、亚洲棉(G.arboreum)、陆地棉(G.hirsutum)、海岛棉(G.barbadense),其中陆地棉栽培对棉花产量贡献最大。经过长期的引种驯化,现代陆地棉栽培种已具备高产优质等诸多优良特性;然而,人类的选育也对遗传多样性造成一定的影响,并直接导致棉花对生物及非生物胁迫的抗性降低[2]。其中,棉花黄萎病病菌导致的土传真菌维管束病害,即棉花黄萎病,在生产中极具毁灭性且难以防治[3]。在棉花育种实践中,利用野生资源拓宽棉花遗传多样性,发掘野生棉一些新的基因位点对陆地棉改良有明显的作用[4]。原产于巴西的黄褐棉(G.mustelinum)[5]在野生异源四倍体棉种里与陆地棉遗传距离较远[6],且具有抗黄萎病的优良特性,其优异的等位基因对改良陆地棉抗黄萎病具备潜在的重要价值。
Tanksley等提出的回交高世代数量性状基因座(advanced backcross quantitative trait locus,简称AB-QTL)分析法,解决了从野生种导入优良基因到栽培品种的问题[7]。Paterson等提出了利用导入系(introgression line,简称IL)作图群体进行QTL精细定位,并取得了良好效果[8]。张星星等在BC3F3种子中鉴定出来自加拿大优质强筋小麦品种Glenlea的Glu-A1a、Glu-B1al、Glu-D1d位点不同组合的7种导入系材料,研究表明,DLGlu1可用于Glu-1 位点近等导入系的快速创制,对Glu-1位点功能研究和改良具有重要价值[9]。张蕾等研究的由小麦和中间偃麦草Z1141杂交、回交而来的高代导入系CH7124,在苗期高度耐受盐胁迫环境,后续用100个简单重复序列(simple sequence repeats,简称SSR)标记对亲本CH7124和绵阳11以及F2群体的耐盐/敏感池进行筛选,结果获得2个多态性标记Xwmc175、Xgwm213[10]。潘晓雪等以死苗率为鉴定指标,对糯稻89-1导入系群体(BC1F9)进行苗期耐寒性鉴定试验,筛选出强抗寒性株系并对筛选出的株系进行了生理生化分析[11]。
为揭示棉花抗病的遗传基础,许多研究者已经进行了独立的研究,并在一些定位群体中检测出许多与棉花抗病性相关联的QTL。Guo等利用2个黄萎病抗性品系5026、60182鉴定了25个与抗病相关的QTL,4个优良QTL分别位于5号、6号、8号、13号染色体上[12]。Zhao等基于疾病苗圃和温室环境,通过关联作图鉴定了与黄萎病抗性相关的42个QTL,广泛分布在15条染色体中,16号染色体上的抗性QTL簇也在其研究中得到证实,这与该染色体的紧密连锁具有一致性[13]。Ning等在抗性Acala-Prema和易感栽培种86-1之间的杂交群体中检测到7个位于1号、3号、5号、7号染色体上的抗性QTL[14]。Fang等构建了易感Sure-Grow 747(陆地棉)与抗性Pima S-7(海岛棉)杂交的回交高世代群体,检测到在4号、19号等染色体上的共42个QTL,增效亲本大多来自抗性Pima S-7[15]。Jiang等利用抗性棉花材料60182与易感栽培品种Junmian 1杂交,在7号和9号染色体上同时筛选出具有高贡献率的QTL簇,这对改善棉花育种的计划有所促进[16]。Bolek等利用高度耐受的棉花品系Pima S-7(海岛棉)和易感的Acala 44(陆地棉)杂交,通过具有明显连锁关联的15个标记,共检测出9个分布于10号、11号、12号、25号染色体的QTL[17]。
针对棉花抗黄萎病相关表达序列标签(expressed sequence tag,简称EST)资源,笔者所在团队已开发106对有效、非冗余的抗黄萎病SSR标记,并对所开发的引物进行评价,同时进行棉属种间转移性研究,成效显著[18]。本研究在利用Wang等将陆地棉与黄褐棉杂交而构建的回交高世代群体[19]的基础上,分子标记辅助选择抗病表现优异的近等基因系,并结合病圃的表型差异,在构建的棉花导入系群体中对抗病QTL进行鉴定和分析,以期为抗病QTL精细定位及抗病分子育种奠定基础。
1 材料与方法
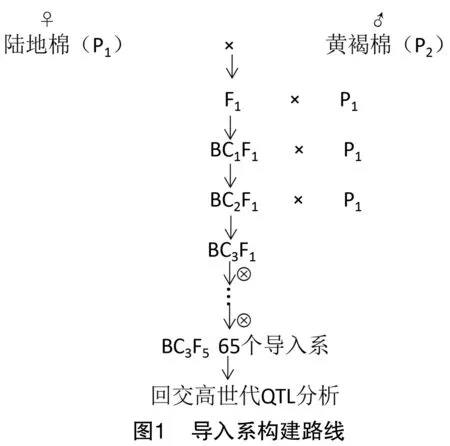
1.1 用于QTL定位的黄褐棉导入系群体的培育
前期研究中以陆地棉PD94042(P1)为受体亲本、以黄褐棉(G.mustelinum)(P2)为供体亲本进行杂交,并以陆地棉PD94042(P1)为轮回亲本构建了回交高世代群体[19],从中选育出1组覆盖棉花全基因组的导入系(共计65份材料),其过程主要包括亲本杂交获得F1代,然后与陆地棉回交3次获得回交3代,再进行自交,获得试验需要的导入系群体(图1)。
1.2 导入系群体黄萎病抗性鉴定
导入系群体及2个亲本分别在2011年种于江苏省南通市(环境1)、盐城市(环境2)两地,各家系按行种植,随机区组设计,2次重复,每个重复每行材料种植5株。2013年12月份鉴定发生黄萎病劈秆的病情指数,病级划分参照吴征彬的方法[20](表1),病情指数计算公式如下:
式中:R是某一家系的黄萎病病情指数;X是病级(0~4);Y是某一病级的单株数;α是所调查家系的棉花单株总数。

表1 棉花黄萎病病级划分标准
1.3 DNA提取、PCR扩增和电泳检测
对各材料的基因组DNA采用十六烷基三甲基溴化铵(cetyltriethyl ammnonium bromide,简称CTAB)法提取,用紫外分光光度计检测其浓度[21]。
PCR反应体系总体积为10 μL,具体包括1.0 μL 10×Reaction buffer(不含Mg2+),1.0 μL Mg2+(25 mol/L),1.0 μL dNTP(10 mmol/L),各0.5 μL上、下游引物(10 μmol/L),0.2 μLTaqDNA聚合酶(2.5 U/μL),1.0 μL模板DNA(50 ng/μL),4.8 μL ddH2O,上覆10.0 μL液体石蜡油。
PCR反应程序:94 ℃预变性5 min;94 ℃变性40 s,56 ℃退火45 s,72 ℃延伸60 s,共35个循环;72 ℃延伸7 min;最后在4 ℃冷却保存。
聚丙烯酰胺凝胶电泳(polyacrylamide gel electrophoresis,简称PAGE)方法:用10%聚丙烯酰胺凝胶进行电泳检测,在扩增产物中加入3 μL溴酚蓝上样缓冲液混匀,从中取2.5 μL加入点样孔,并用50 bp DNA Ladder进行标记。在电泳缓冲液为1×Tris-硼酸,150 V恒压环境下电泳 1.5 h。银染分析:在He等的方法[22-23]基础上稍加改良,经固定、银染、显色、水洗等步骤后,拍照保存。
1.4 导入系群体的数据统计分析
1.4.1 基因型数据统计 通过聚丙烯酰胺凝胶电泳,参考DNA Ladder标准记载,对比条带显示结果,记录亲本及子代SSR标记的类型,共显性标记记录中,与亲本P1相同的纯合带型记为“1”,与亲本P2相同的纯合带型记为“2”,两亲本的杂合带型记为“3”;当亲本P1为显性、亲本P2为隐性时,P1、F1的带型记为“5”,P2的带型记为“2”;当亲本P2为显性、亲本P1为隐性时,P2、F1的带型记为“4”,P1的带型记为“1”;模糊或缺失数据记为“0”。将每个标记的数据输入Excel表格。
1.4.2 表型数据统计 将病圃的表型数据进行相应的统计分析,计算其相应的病情指数。
1.5 黄萎病抗性QTL定位
综合导入系群体的基因型数据,首先对于标记之间的连锁关系用MapMaker 3.0软件进行分析,构建分子标记的遗传连锁图谱。利用Win QTL Cart 2.5软件将病圃的表型数据结合基因型数据开展黄萎病抗性QTL定位,检测QTL最低连锁系数(likelihood of odds,简称LOD)值为2.5,最后对于目标QTL在染色体或连锁图谱上的位置用MapChart绘图软件进行绘制。
2 结果与分析
2.1 抗病表型分析
病情指数越低,抗性越高。由图2可知,陆地棉绝大部分为易感病材料,黄褐棉为抗病材料。根据统计结果可以证明该导入系群体中存在一定量抗病相关的黄褐棉渐渗片段,从而使得该导入系群体抗病性得到提高。
2.2 抗病QTL分析
由表2可知,在2种环境下共检测到10个抗病性QTL,其中,在环境1下检测到2个QTL,qVW-5-1定位在5号染色体60.09 cM处,侧邻标记为CICR016-CICR186,LOD值为3.4,解释17.6%的表型变异率,病指增效基因贡献亲本为陆地棉PD94042;qVW-9-1定位在9号染色体14.99 cM处,侧邻标记为CICR658-BNL3779,LOD值为2.7,解释 14.6% 的表型变异率,增效基因贡献亲本为陆地棉PD94042;在环境2下检测到8个QTL,qVW-4-1定位在4号染色体0.01 cM处,侧邻标记为DPL0085-BNL530,LOD值为 2.7,解释14.0%的表型变异率,增效基因贡献亲本为陆地棉PD94042;qVW-10-1定位在10号染色体0.01 cM处,侧邻标记为BNL1438-CICR002a,LOD值为2.8,解释14.0%的表型变异率,增效基因贡献亲本为黄褐棉;qVW-16-1定位在16号染色体79.67 cM处,侧邻标记为CICR166b-BNL1604,LOD值为2.6,解释19.1%的表型变异率,增效基因贡献亲本为黄褐棉;qVW-18-1定位在18号染色体 49.51 cM 处,侧邻标记为BNL1721-MUSS603,LOD值为 2.7,解释17.9%的表型变异率,增效基因贡献亲本为黄褐棉;定位于19号染色体的抗病QTL有3个,其中qVW-19-1定位在19号染色体312.67 cM处,侧邻标记为BNL3029-DPL0444,LOD值为2.9,解释18.6%的表型变异率,增效基因贡献亲本为黄褐棉;另外qVW-19-2定位在19号染色体33.52 cM处,侧邻标记为CICR524-CICR056,LOD值为2.6,解释21.9%的表型变异率,增效基因贡献亲本为陆地棉PD94042;qVW-19-3定位在19号染色体70.39 cM处,侧邻标记为BNL3811-MUSS118,LOD值为3.0,解释18.7%的表型变异率,增效基因贡献亲本为陆地棉PD94042。特别地,位于22号染色体上的QTLqVW-22-1可解释39.3%的表型变异率,且LOD值高达4.8,其病情指数增效基因来自陆地棉,即黄褐棉等位基因具有抗病作用,有必要开展深入研究。

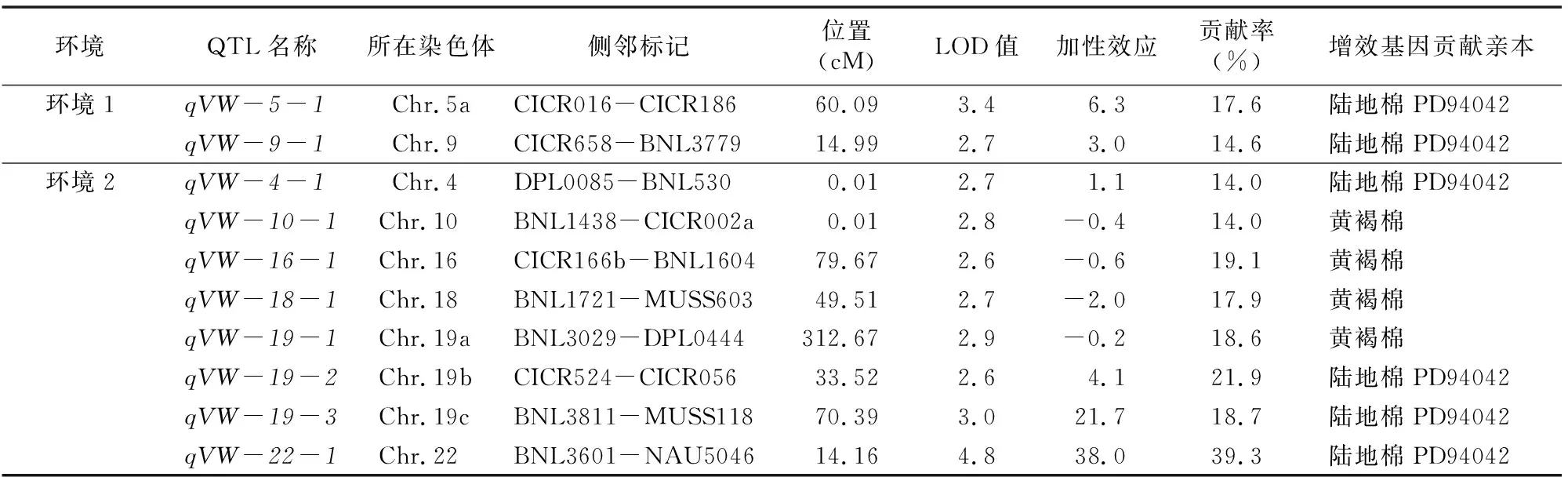
表2 QTL定位结果
用绘图软件MapChart绘制目标QTL在染色体或连锁图谱上的位置,如图3所示:图中为qVW-22-1主效QTL的遗传图谱分析,图上划线标记的是QTL的多态性位点,显示的是可能存在于该区间的抗病QTL,线性标注即是该区间的范围,而中间的矩形实心区域是可能性较大的QTL位置,这表明更为精细的位点可能在此区域;右边的矩形方框是根据LOD值所画的散点图。
3 讨论
在众多作物(水稻[24]、大豆[25]、棉花[26]等)中均有报道通过高世代回交和分子标记辅助选择相结合来构建染色体片段代换系,但具有黄褐棉染色体片段代换系的报道较少。本研究所用亲本材料中的PD94042是感病陆地棉品系,另一个亲本是高抗黄萎病的黄褐棉品系,两亲本在抗黄萎病性状方面差异明显,因此利于进行抗病QTL研究。
前人研究中,已报道许多棉花黄萎病抗性QTL[27-28]。刘剑光等构建的一个以棉花抗黄萎病品系苏抗045和感黄萎病品系苏研116为亲本的含有237个F2单株的群体中,在NAU2970-MUSS138区间内检测到1个定位于5号染色体上的抗黄萎病QTL,可解释9.69%的表型变异率[29]。Shi等构建了来自高抗性品系Hai1(海岛棉)和易感品种CCRI36(陆地棉)作为轮回亲本的BC1F1、BC1S1、BC2F1群体,在全棉基因组中使用来自BC1F1群体的高密度简单重复序列遗传连锁图谱检测与抗病抗性相关的QTL共48个,其中定位于19号染色体上的抗病QTL侧邻标记NAU5489与笔者所在实验室鉴定的qVW-19-3侧邻标记相距甚小,可能有一定的相关性,位于22号染色体上的QTL侧邻标记CICR0438也出现在本研究的图谱上;同时也检测出来自5号、9号、10号染色体上的许多QTL,但由于缺乏共同标记,难以比较[30]。Yang等利用耐受品种Hai7124(海岛棉)与易感品种Junmian 1(陆地棉)进行杂交,检测到定位于4号染色体上的抗病QTL侧邻标记NAU3437也在笔者所在实验室的检测范围内,与笔者所在实验室鉴定的qVW-4-1可能具有一定的关联[31]。
本研究检测到的抗病QTL大多离散地分布在多条染色体上, 而在19号染色体连锁群上有3个QTL,这些QTL主要集中在标记DPL0444、CICR056、MUSS118附近,这种QTL成簇分布现象在其他研究中也有报道,在棉花基因组中可能存在着对棉花生长发育具有更大作用的高度重复的基因富集区,QTL能发挥多重功能以弥补数量上的不足[32]。后续的研究将对一些重点QTL开展精细定位,同时进行标记辅助选择,以期为棉花抗黄萎病育种实践服务。


