改良CTAB方法提取果树不同组织的RNA
关 玲, 赵密珍, 王庆莲, 吴伟民, 钱亚明, 巫建华
(1.江苏省农业科学院果树研究所/江苏省高效园艺作物遗传改良重点实验室,江苏南京 210014;2.江苏现代园艺工程技术中心,江苏句容 212400)
果树作为重要的园艺作物,在国民生产中占有重要地位。分子生物学研究作为有效的试验工具,被广泛应用于果树研究中[1-3]。同时,随着果树基因组数据的逐渐丰富,越来越多的果树物种基因组信息得到了注释,因此,开展果树分子生物学研究,对于挖掘功能基因以及通过基因工程等手段实现为果树育种服务的目标被越来越多的果树育种工作者接受[4-6]。
RNA的提取作为分子生物学研究的基础步骤,其成熟度较为重要。果树作为多年生木本或草本植物,因其多年生的特点,多富含多糖、多酚等次生代谢物质,使其RNA的提取及纯化工作较为困难[7]。生物试剂盒对于分子生物学研究具有快速、简便的优点,但当今市场上尚没有针对果树物种RNA提取的试剂盒,并且利用广谱的植物RNA提取试剂盒提取果树物种RNA具有不稳定的特点。常用的传统RNA提取方法有TRIzol方法、苯酚法、氯化锂沉淀法、异硫氰酸胍法及十二烷基硫酸钠(sodium dodecyl sulfate,简称SDS)/酚抽提方法[8-11],以上方法具有价格昂贵、费时费力的特点,且得到的果树RNA产量较少[12]。此外,笔者通过试验对比发现,TRIzol方法多适用于草本植物,不适用于木本植物材料的RNA提取。本试验通过对十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,简称CTAB)法的进一步改良,得到通用于提取果树不同代表物种如草莓(草本)、苹果(木本)以及葡萄(藤本)不同组织RNA的方法。本研究以期通过对比不同RNA提取方法和进一步改进已有的CTAB提取方法,为果树分子生物学研究提供基础、准确的RNA提取方法,从而方便果树研究者进行后续研究。
1 材料与方法
1.1 试验材料
本试验所用材料草莓、葡萄均取自江苏省农业科学院园艺研究所试验田,草莓(Fragaria×ananassaDuch.)品种为宁玉[13],定植时期为2014年9月初;葡萄(VitisL.)的组培苗、大田苗品种皆为红宝石无核,其中组培苗为胚挽救后代种苗,大田苗为5年生植株;苹果材料采自山东省临沂市平邑县一年生苹果(Malusdomestica)砧木M9。用于RNA提取的草莓、苹果和葡萄的组培苗材料均采集根、茎、叶(图1-A、图1-B、图1-C)3种组织;于葡萄大田苗上采集根、茎、叶、果皮和果肉(图1-D)5种组织,所采集的样品应尽量呈幼嫩状态并立即置于液氮中带回实验室,放置于-70 ℃冰箱中待用。
1.2 试剂与仪器
1.2.1 改进后的CTAB裂解液母液的配制成分 (1)5 mol/L NaCl(使用浓度为1.4 mol/L)。(2)0.5 mol/L EDTA·2Na(pH值为8.0,使用浓度为20 mmol/L)。用磁力搅拌器剧烈搅拌,加入NaOH调节pH值到8.0时,EDTA·2Na方可溶解。(3)1 mol/L Tris-HCl(pH值为8.0,使用浓度为 0.1 mol/L),用HCl调节pH值至8.0。(4)2%(质量浓度)CTAB。(5)2%(质量浓度)聚乙烯吡咯烷酮K-30(polyvinylpyrrolidone K-30,简称PVP K-30)。以100 mL为例,用适量体积的上述各组分母液配制CTAB裂解液:28 mL NaCl,4 mL EDTA·2Na,1 mL Tris-HCl,2 g CTAB,2 g PVP,用RNase free ddH2O定容。
1.2.2 其他组分及试剂 (1)10 mol/L LiCl(LiCl易吸潮,需快速操作,注意溶解放热烫);(2)3 mol/L NaAc(pH值5.2);(3)β-巯基乙醇,使用浓度为1%;(4)70%(体积分数)乙醇;(5)三氯甲烷+异戊醇(体积比24 ∶1);(6)水饱和酚(pH值4.5);(7)无水乙醇;(8)ddH2O(RNase free)。
配制各组分试剂均使用ddH2O,配制完毕后加入外源RNA酶清除剂处理24 h后待用。
1.3 试验方法
1.3.1 果树RNA提取的操作步骤 (1)取待提取RNA试材(除果实外的组织取材质量约为0.5~1.0 g,果实质量为 1.5~2.0 g),置于液氮中迅速研磨成粉末,将1 000 μL CTAB裂解液和20 μLβ-巯基乙醇依次加入2 mL离心管中,于 65 ℃ 水浴30 min(水浴过程中每隔10 min将离心管上下颠倒约30次,以保证待提取试材与CTAB裂解液充分接触);(2)加入等体积的三氯甲烷+异戊醇(体积比24 ∶1),旋涡混匀,8 000g、4 ℃离心10 min;(3)取800 μL上清,加入1/2体积的水饱和酚(pH值4.5),混匀,静置5 min,再加入1/2体积的三氯甲烷+异戊醇(体积比24 ∶1),旋涡混匀;8 000g、4 ℃ 离心10 min;(4)取600 μL上清(在1.5 mL离心管中操作),加入1/30体积的NaAc(3 mol/L,pH值5.2)和1/10体积的无水乙醇,混匀后静置5 min,8 000g、4 ℃离心10 min;(5)取450 μL上清,加入1/3体积的LiCl(10 mol/L),-20 ℃ 静置2~3 h;8 000g、4 ℃离心15 min;(6)弃上清,用70%乙醇洗涤沉淀2次,再用无水乙醇洗涤沉淀1次,室温干燥,将清洗后的沉淀溶于ddH2O(RNase free)中,于-70 ℃保存备用;(7)取2 μL总RNA,用1%琼脂糖凝胶电泳进行检测(电泳仪电压不宜设置过大,否则会因产热引起RNA样品降解导致检测结果不准确);(8)将RNA样品上Biophotometer核酸仪检测,体系为1 μL RNA样品加入49 μL ddH2O(RNase free),RNA浓度、D280 nm/D260 nm值和D260 nm/D230 nm值直接从仪器上读取。
1.3.2 基因组DNA的消化及反转录 利用上述方法提取的RNA样品中,存在少许基因组DNA,其消化及cDNA的合成按照TaKaRa公司的PrimerScriptTMRT reagent Kit(TaKaRa,货号:RR047A)说明书进行。为确保试验结果的可靠性,消化及反转录试验需在冰上进行。
2 结果与分析
2.1 RNA琼脂糖电泳检测
RNA产物使用1.0%琼脂糖凝胶电泳检测,图2结果显示,经过改进的CTAB法提取多年生草本、木本及藤本果树材料均可获得清晰的3条RNA条带,如图2分别是28.0S rRNA、18.0S rRNA、5.8S rRNA及少量gDNA(基因组DNA)。凝胶结果显示点样孔清晰、无污染。
2.2 RNA纯度检测
利用Biophotometer核酸仪对所提取样品RNA的吸光度及产量进行检测。 结果表明, RNA样品的D260 nm/D230 nm值均高于2.0,具体为2.03~2.27(表1)。说明利用改进后的CTAB法抽提获得的RNA所含多糖、酚类及其他小分子物质较少。草莓、苹果大田苗及葡萄组培苗的根、茎、叶片RNA的D260 nm/D280 nm值范围为1.80~1.93,葡萄大田苗各组织RNA的D260 nm/D280 nm值为1.80~1.91,表明利用本方法抽提获得的果树RNA中杂质蛋白质污染较少。不同组织的RNA产量情况如下:根器官RNA产量为600.15~712.75 ng/μL;茎RNA产量为530.61~694.54 ng/μL;叶片RNA产量为617.54~777.53 ng/μL;果皮RNA产量为561.23 ng/μL;果肉RNA产量为40.23 ng/μL。
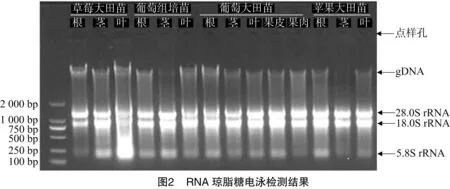
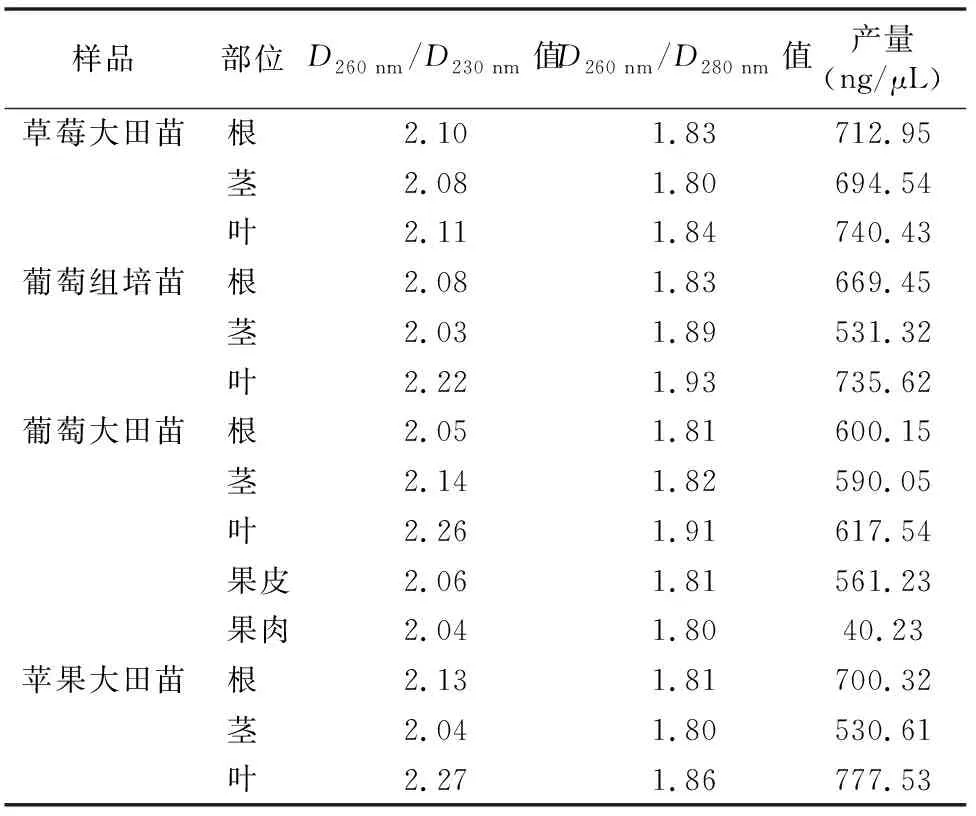
表1 分光光度法检测RNA质量及产量
注:吸光度比值为3个样品重复的平均值。
综合上述4种试材不同组织的D260 nm/D230 nm值及所得RNA产量情况,可以得出草莓、苹果以及葡萄RNA的提取在根、茎、叶3种营养器官中出现一致的难易度趋势,即叶片较根容易提取,两者皆较茎容易提取得到RNA;葡萄大田苗生殖器官的RNA提取难易程度表现为果皮较果肉容易。
2.3 RNA提取后的质量检测
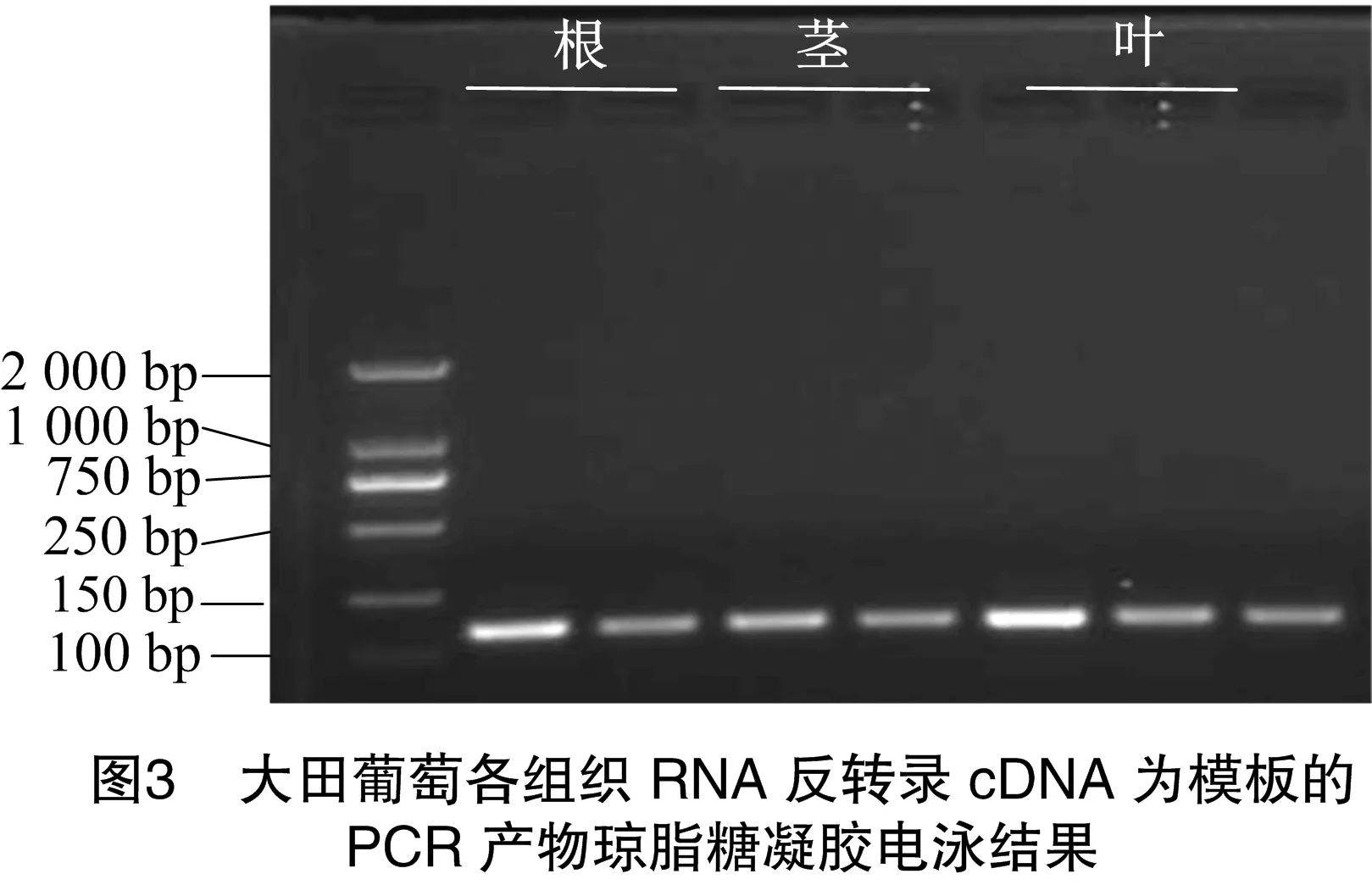
为检测RNA质量是否满足RT-PCR试验,设计草莓的内参基因FvMSI1的引物如下:F,5′-TCCCCACACCTTTGATT GCCA-3′;R,5′-ACACCATCAGTCTCCTGCCAAG-3′。苹果内参基因18S rRNA引物如下:F,5′-GTTACTTTTAGGACT CCGC C-3′;R,5′-TTCCTTTAAGTTTCAGCCTTG-3′。葡萄内参基因为UBI,F:5′-GCTCGCTGTTTTGCAGTTCTAC-3′;R:5′-AACATAGGTGAGGCCGCACTT-3′。使用所提取的果树RNA进行反转录得到cDNA,利用上述引物经PCR扩增后,将产物进行琼脂糖电泳检测(图3),分析结果可知,利用该方法获得的RNA均可用于后续的RT-PCR试验。
3 讨论与结论
3.1 RNA提取前的准备工作
RNA提取的关键是抑制细胞中的RNA分解和防止操作环境中的RNA酶污染。因此在试验中应采取以下措施:(1)使用70%乙醇清洁试验操作台;(2)使用RNA操作专用试验台,或者利用外源RNA酶清除剂处理台面及操作用到的所有器具(研钵、镊子、药匙等);(3)水浴锅提前预热、离心机提前预冷。通过上述方法主要防止外界环境、空气或者试验者唾液中RNA酶的污染。
3.2 改良CTAB法的优点
3.2.1 提取液改进PVP用量 本试验在RNA的提取过程中改进了PVP的用量,不仅能去除果树各组织中的酚类物质和多糖,还能去除一些其他色素和脂类物质,同时可以消除泡沫,保护胶体,可以提取得到更完整和纯度更高的RNA产物。同时与传统的CTAB法相比[12,14],本方法改进了各组分的用量,如CTAB提取液的用量(1 mL)、β-巯基乙醇的使用浓度(1%,体积分数)和NaAc浓度(3 mol/L),一方面可以节约成本,另一方面可以减少如β-巯基乙醇等物质在操作过程中的毒害作用。进一步分析可知,所提取的RNA与传统方法相比,拥有高纯度(D260nm/D230 nm值>2.0,D260 nm/D280 nm值>1.8)的特点,同时RNA产量较高(除果肉RNA产量为 40.23 ng/μL 外,其余材料的产量范围为530.61~777.53 ng/μL),其质量及产量均满足后续的RT-PCR试验要求。
3.2.2 高效稳定 相对于生物公司销售的成品RNA提取试剂盒,本方法具有操作简便、试验结果可靠性高及节本的特点。笔者所在课题组曾尝试使用多种成品试剂盒提取果树不同组织中的RNA,发现效果均不理想。尤其是提取木质化程度较高的果树材料及次生代谢物质含量较高的果树生殖器官RNA,如葡萄的果皮和果肉组织。分析其原因可能是在成品试剂盒中,针对木本果树特有的多酚、多糖的去除技术尚未成熟。同时考虑到果树物种的季节性较强,样品采集的重复性较差,应用高效稳定的RNA提取方法对于果树分子生物学研究十分重要。笔者通过多次试验,发现经本研究改进的CTAB法具有重复性好、可靠性高的特点,尤其适用于木本果树的RNA提取。本研究分别选取草本、木本及藤本果树的不同营养器官及生殖器官开展研究,进一步验证了改进后的CTAB法可用于不同类型果树各组织RNA的提取,从而为果树分子生物学研究提供了高效稳定的RNA提取方法。
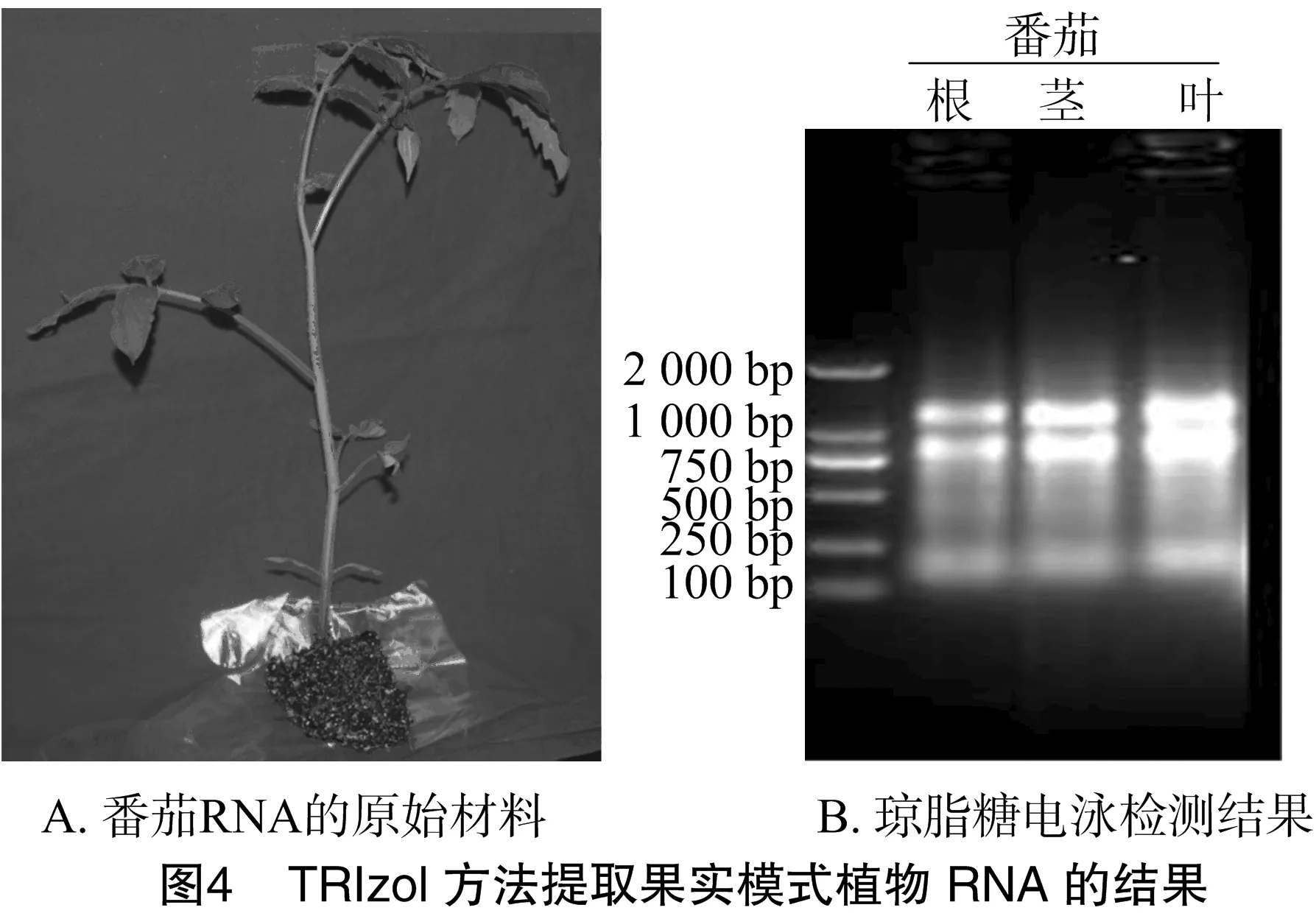
3.2.3 CTAB法与TRIzol方法的比较 笔者曾利用灵敏且快捷的TRIzol方法对苹果材料不同组织的RNA进行提取,结果均不理想(只有幼嫩的叶片材料可获得少量的RNA,其他木质化程度较高的材料均无法获得RNA)。利用TRIzol方法提取拟南芥以及果树研究中的果实模式植物——番茄却可以得到理想结果(图4),但成品TRIzol试剂价格昂贵。笔者分析其原因,可能是木本果树中的多酚等次生代谢物质阻碍了TRIzol试剂对果树组织内RNA的分离,但是应用TRIzol方法提取其他草本植物RNA仍不失为一种优良方法。基于此,笔者提出,本研究中改进的CTAB法适用于木本果树材料,同时对于珍稀材料或者采集重复性较差的试验样品使用该方法可提高稳定性,减少材料损失。
3.2.4 外源RNA酶清除剂替代焦碳酸二乙酯(diethy pyrocarbonate,简称DEPC)处理 利用上述外源RNA酶清除剂可以较快速地去除所用试剂、器具及RNA提取环境中的RNA酶,与传统的DEPC处理方法[15]相比,可以省去繁琐的高温灭菌步骤,且外源RNA酶清除剂的使用,具有快速、操作简便及无毒的特点。
3.3 果树RNA提取过程中可能出现的问题及对策
3.3.1 琼脂糖凝胶点样孔模糊或D260 nm/D230 nm值非正常范围 正常提取所得RNA的D260 nm/D230 nm值≥2,若该值偏小或琼脂糖凝胶点样孔模糊,说明所提取的RNA样品中有杂质多糖、多酚或其他盐类小分子物质。本试验材料的D260 nm/D230 nm值范围为2.03~2.27。可见利用该方法在提取液中加入较高质量浓度的PVP,能够有效地去除果树组织中的酚类、多糖等次生代谢物质,同时提高RNA的完整性和纯度。
3.3.2 提取样品RNA呈现褐色 本研究提取的试材不新鲜,存在褐化,或由于研磨过程中未能保持样品处于液氮中,抑或由于研磨所得的样品在装入离心管之前未在管中及时添加β-巯基乙醇,导致研磨样品与空气中的氧气接触,从而使试材中的酚类物质氧化成醌类物质而呈现褐色。解决对策有以下2个:一是保证提取试材新鲜并处于超低温环境;二是保证研磨所得样品在装入离心管前加入β-巯基乙醇,以防止样品褐化。此外,整个研磨及装管过程需要操作迅速。
3.3.3D260 nm/D280 nm值及产量D260 nm/D280 nm值正常范围为1.8~2.0,其值偏小说明RNA样品中有杂质蛋白质污染;其值偏大说明RNA降解严重。苯酚在低pH值的情况下可促进水相中的蛋白质和DNA向有机相分配,从而最大限度地去除总RNA中的蛋白质和DNA,因此在抽提过程中需要加入水饱和酚(pH值4.5)。本试验中葡萄大田苗生殖器官的RNA提取难易程度为果肉远难于果皮,分析其原因,可能是果肉中的水分含量较高。操作者在研磨时为降低杂质出现的概率,会加入较少量的葡萄果肉材料,导致最终RNA产量较少。因此,对于葡萄果实的提取,可以适当增加试材的研磨量(1.5~2.0 g),以提高果实的RNA产量,从而满足后续试验要求。
3.3.4 LiCl选择性地沉淀RNA 利用该法可以充分去除RNA中的DNA污染。若样品量允许,可以进行2次LiCl沉淀。注意使用70%(体积分数)乙醇至少洗涤2次,避免RNA中因含有LiCl(金属Li+)而影响反转录的效果。本研究通过反复试验,比较经典草本植物RNA提取的TRIzol方法以及改进的CTAB方法,摸索出更加成熟的可通用于草本、木本果树不同组织RNA提取的新方法,经检测,所提取得到的RNA可以直接用于后续研究。

