动物源细菌耐药性研究现状与对策
孙坚,刘雅红,冯友军,2
冯友军 浙江大学医学院/动科院双聘教授。2014年,冯教授入选国家千人计划青年项目,近年来围绕脂肪酸代谢与致病性/耐药性开展了较为系统的研究。在国际知名刊物、、等发表了SCI论文100余篇,引用次数500余次。同时担任和等多家国际知名期刊的编委。并荣获2014年度国家科技进步二等奖和2016–2017年度“浙江大学十大进展”的提名奖。

刘雅红 华南农业大学兽医学院教授,博士生导师。长期从事兽医病原微生物耐药性的风险评估与控制、兽药残留检测与生态毒理学研究和新兽药的研制与临床评价等方面的研究,是国家“杰出青年科学基金”获得者,教育部“长江学者奖励计划”特聘教授,并获得国家“百千万人才工程有突出贡献中青年专家”称号。主持国家杰出青年科学基金、十三五国家重点研发计划专项等科研项目40多项,在、和等国际知名期刊发表SCI论文120余篇。获国家二类新兽药证书2个、省部级以上科技奖励7项,获授权发明专利10项。并组建了国家兽医微生物耐药性风险评估实验室等多个科研平台。
动物源细菌耐药性研究现状与对策
孙坚1,刘雅红1,冯友军1,2
1 华南农业大学 兽医学院 国家兽医微生物耐药性风险评估实验室,广东 广州 510642 2 浙江大学 动物科学学院/医学院,浙江 杭州 310058

孙坚, 刘雅红, 冯友军. 动物源细菌耐药性研究现状与对策.生物工程学报, 2018, 34(8): 1246–1258.Sun J, Liu YH, Feng YJ. Towards understanding antibiotic resistance in animals-borne bacterial pathogens. Chin J Biotech, 2018, 34(8): 1246–1258.
目前我国动物源细菌耐药现象十分普遍,多重耐药甚至泛耐药的菌株不断出现,给公共卫生和食品安全造成了重大威胁。文中从我国动物源耐药性研究的主要问题、细菌耐药性形成和传播机制以及耐药性防控策略等几个方面进行综述,以期为耐药性的研究和防控提供参考。
动物源细菌,耐药基因,传播机制,耐药性防控策略
细菌耐药性是一种古老而自然发生的现象,是细菌间相互竞争过程中随着抗生素的出现而出现的必然产物。抗菌药物在医疗系统、农业或工业过程中大量使用,施加的选择压力有利于耐药菌的存活,进一步加剧了耐药性的出现和扩散。有研究表明人或动物肠道等生态系统中耐药菌的检出率与使用抗菌药物的种类、数量、接触的频率和持续时间有关。在动物生产中,长期使用低剂量的抗微生物药物作为生长促进剂在许多国家包括中国仍然是常见的做法,这在增加经济效益的同时也隐藏着巨大的风险,不断使用的抗菌剂促进并加剧耐药菌的出现。由于人类、动物和环境相互依存且密切相关,评估动物生产过程中使用抗菌药对动物、人以及环境中细菌的耐药性的影响仍然是一个巨大的挑战。
耐药性是一个世界性的难题,面对耐药性的威胁,尽管各个世界组织和国家已经展开了一些卓有成效的工作,在很大程度上减缓了耐药性的产生,但是到目前为止仍然缺乏有效根治耐药性问题的灵丹妙药。未来的研究需要整合多个学科 (例如“一个健康”) 的方法,将人医临床、农业以及环境和社会科学综合在一起来研究耐药性的问题[1],才能在最大程度上遏制耐药性的发展,为人类留下抗生素这份宝贵的资源。
1 我国动物源细菌耐药性面临的主要问题
细菌耐药性被认为是对全球公共卫生和粮食安全的重大威胁。2015年6月,联合国粮食及农业组织 (粮农组织) 在其理事会上通过了一项关于抗微生物药物耐药性的决议。这是继2015年5月通过了世界动物卫生组织 (OIE) 和世界卫生组织 (WHO) 的抗微生物药物耐药性决议后的又一重大事件,标志着三大组织共同努力,在全球抗击抗微生物药物耐药性的开始。
过度或者是不恰当使用抗菌药被认为是抗微生物药物耐药性出现和传播的主要驱动因素。抗微生物药物以治疗和非治疗目的广泛用于食品动物,并且在挽救人类和动物的生命方面发挥重要作用。在过去的十年中,全球畜牧业生产一直在快速增长,并越来越多地转向集约化生产,其中抗菌药的使用是生产的一个组成部分,预计抗菌药的使用在未来增长的三分之二将用于动物生产[2]。尽管近年来发达国家抗菌药在动物促进生长、预防和治疗性预防 (即治疗健康和感染动物的混合群体以控制疾病暴发) 中的使用大幅度减少,但数据显示由于中低收入国家对动物蛋白的需求不断增长,畜牧业抗菌药的使用在未来几十年将继续在中低收入国家增加[2]。因此,这些国家和地区对常用抗生素的耐药性可能会相应增加,这为人类和动物感染的治疗和管理带来威胁。
尽管细菌耐药性问题具有重要的公共卫生意义并且引起了全球的广泛关注,但是其涉及的许多环节和重要问题仍然未被阐明。耐药性的有效监测是实现耐药性控制的前提条件,但是目前几乎所有的监测计划都是自上而下的政府行为,监测细菌的种类和数量有限,而且往往由于客观条件的限制,监测过程缺乏时效性和持续性,由此造成监测结果并没法反映临床真实的数据,监测结果也无法给兽医临床提供用药指导。究其原因,兽医临床不同于人医临床,基层的设备条件有限,加之从业人员专业能力的限制,阻碍了兽医临床耐药性检测的开展。
2 动物源细菌耐药性形成和传播机制
抗生素 (包括化学合成抗菌药) 是人类抵御各种感染性疾病的主要武器。进入21世纪以来,多种病原菌出现了耐药株 (Drug resistant strain)、多重耐药株 (Muti-drug resistant strain) 甚至泛耐药株 (Pan-drug resistant strain,又称全耐药株),给人医和兽医临床带来巨大挑战[3]。动物病原菌是各类耐药基因的主要储库之一,并且会源源不断地传播给人类,成为一个危害公共安全的重大隐患[4]。目前涉及到动物源重要耐药菌的耐药机制主要有3种:灭活酶、靶位修饰、外排泵。这些耐药机制都可由耐药基因介导,随着水平移动元件的转移或者细菌的克隆传播进行扩散。
近10年来我国细菌耐药性到了一个高速发展的时期,从临床抗革兰氏阴性菌感染的一线抗菌药物氟喹诺酮类和第三代头孢菌素类抗菌药物,到抗感染最后的防线碳青霉烯类抗菌药物和黏菌素,细菌都从自然界中找到了最好的应对方法,即各种各样的耐药基因。在众多的耐药机制中,碳青霉烯类药物和黏菌素的耐药性是当下研究的热点,下文将主要描述这两类药物的传播、流行及耐药机制。
2.1 碳青霉烯酶的流行和传播机制
新德里金属β-内酰胺酶 (NDM) 是目前最受关注、研究最热的MBLs,携带NDM的细菌被称为“超级细菌”[5]。2008年首先从印度新德里的克雷伯菌中发现NDM-1[6],之后仅仅几年就检出22种变异型[7]。流行性调查结果发现NDM的传播速度十分惊人,主要报道该基因在人医和兽医临床肠杆菌科细菌 (特别是大肠杆菌和克雷伯氏菌) 中传播流行[8-9],NDM大部分位于接合质粒上[10],且同时携带其他耐药基因,从而呈现多重耐药[11-12]。VIM和IMP是另外两种在动物源细菌中常见的MBLs,在中国的养殖动物中有零星报道[13-14]。编码碳青霉烯酶的基因在全球动物源细菌的流行与分布情况如表1所示。

表1 动物源碳青霉烯耐药菌株流行与分布情况
这几年有关动物源NDM阳性菌的报道越来越多,涉及到鸡和猪等食品动物、伴侣动物及野生动物[15]。2011年国内首次从养殖动物体内检出了携带NDM的“超级细菌”[16],2013年张万江等在广东省发现一株猪源NDM阳性鲍曼不动杆菌[17];2015年张荣民等又在广东省某兽医院分离出3株NDM阳性大肠杆菌,这是首次直接从养殖动物体内分离出NDM阳性肠杆菌科细菌[18]。2016年杨润时等在广东省某地一鸭子体内首次分离到两株同时携带NDM-5与的大肠杆菌,且NDM-5均位于大小相似、约46 kb大小的IncX3型质粒上,这意味着该质粒在番鸭体内能够在不同菌株之间发生转移,对人们的公共卫生安全造成了潜在的威胁[19]。2017年刘宝涛等在山东省某鸡场中分离到37株对美罗培南耐药的大肠杆菌,有34株携带NDM基因,其中21株同时携带基因,研究发现NDM基因以NDM-1和NDM-9为主,且多位于IncB/O型质粒上[20]。2017年孔令汉等在四川省某猪场采集的105份肛拭子中分离到16株不同克隆的大肠杆菌,且均同时携带NDM-5和,这意味着猪场可能是这些耐药基因的重要储库[21]。同年,刘宝涛等在同一动物中发现了携带NDM-9的大肠杆菌以及携带的阪崎肠杆菌[22]。2018年王若冰等从山东省的猪和肉鸡中检出产NDM的大肠杆菌和肺炎克雷伯菌,其中以NDM-9为主要的亚型[23]。与此同时,猪源大肠杆菌和鸡源大肠杆菌中分别发现了NDM-17[24]与NDM-20[25]两种亚型,这表明食品动物已成为NDM的细菌储库,这有驱动NDM演变以增强对某些或所有β-内酰胺酶活性的风险。2013年印度的加尔各答市还从牛奶样品分离出8株NDM阳性大肠杆菌[26],同年美国和德国科学家分别从宠物和野鸟中分离出NDM阳性大肠杆菌和沙门氏菌[27-28]。2017年对鸡产业链中NDM流行情况进行调查,表明NDM阳性大肠杆菌来源于商品鸡场的环境因素,并可以传播至下游生产链条;并可在商品鸡场的鸡、家燕、人、狗和苍蝇等不同环境因素之间传播,部分大肠杆菌能同时携带NDM和两种耐药基因[29]。何涛等在奶牛粪便中也检测到携带NDM-5的大肠杆菌和肺炎克雷伯菌[8,30]。另外,还在生牛乳中检测到携带该基因的肺炎克雷伯菌[8]。2016年孙坚等报道了一株分离自猫中的大肠杆菌,其对美罗培南和黏菌素同时表现为耐药,进一步研究显示该菌株同时携带NDM和,且位于一个48.35 kb大小的IncX3-X4型杂合质粒上[31]。2017年崔兰卿等首次报道了国内伴侣犬中携带含有NDM-1的大肠杆菌,这表明携带碳青霉烯耐药基因的伴侣动物会对公共卫生安全造成潜在威胁,鉴于伴侣动物与人的关系日益密切,应对伴侣动物中耐碳青霉烯菌株进行持续监控[32]。
VIM是MBLs的典型代表[33],2013年德国首先从猪体内分离出VIM阳性沙门氏菌,2017年我国也从鸡场及其养殖环境中分离出VIM阳性假单胞菌,并且发现了一个新的VIM基因亚型,命名为VIM-48[7]。2013年中国从宠物体内分离出IMP-45阳性铜绿假单胞菌,IMP-45基因环境和人医来源的极为相似,推测该菌株来源于人医临床[13]。
2.2 黏菌素耐药基因mcr-like的流行和传播机制
可转移黏菌素耐药基因首次于2015年在我国上海猪源大肠杆菌中发现,其编码的磷酸乙醇胺转移酶能通过修饰细胞膜上的脂质A而介导细菌对黏菌素耐药[34-38]。随后,、、、和(注:无功能假基因) 以及的多种突变体也相继被发现。与一样,可通过磷酸乙醇胺转移酶催化磷酸乙醇胺添加到脂质A4位上的磷酸基团导致黏菌素耐药[35,39]。可转移黏菌素耐药基因在全球动物源细菌中的流行与分布情况如表2所示。
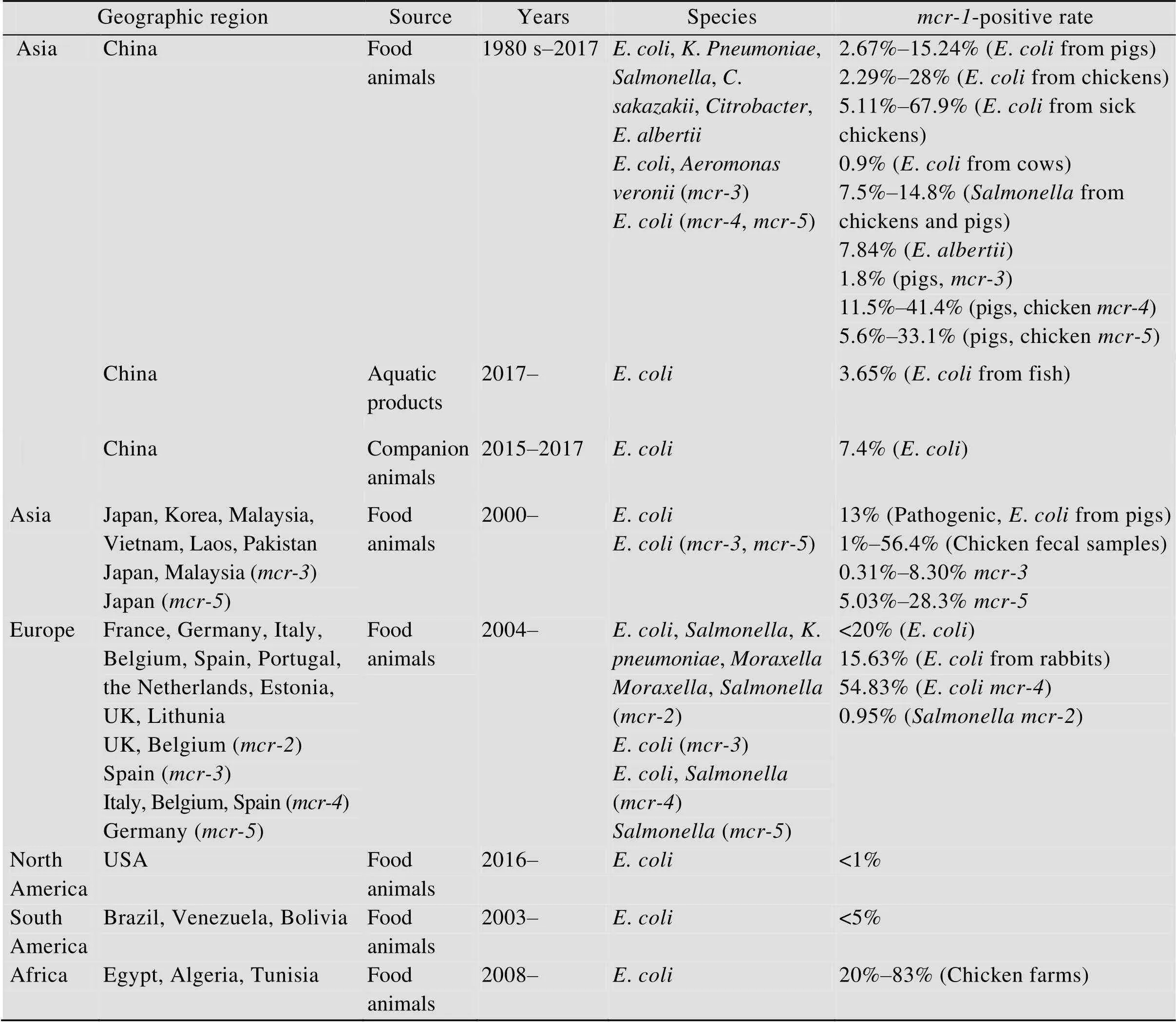
表2 动物源mcr-like阳性菌株流行与分布情况
的传播最为广泛,迄今为止,该基因在全球六大洲四十多国家中流行[40-41],在人、动物和环境等都有检出[42-48]。可在大肠杆菌之间传播,亦可传到肺炎克雷伯菌、沙门菌和柠檬酸菌等,这些结果证明了该质粒介导的耐药性可以跨种属快速传播[49-51]。在首次报道时,检测到我国生肉和屠宰前动物大肠杆菌阳性率分别为15%和21%,并指出已在动物源肠杆菌中广泛传播[34]。随后在鸡源大肠杆菌的回顾性调查中,发现基因的存在最早可以追溯到20世纪80年代。在1980年的3株菌中检测到了基因,而随后20年里并没有检测到基因,直到2009年才再一次检测到了基因阳性大肠杆菌,从2009–2014年,每年基因的检出率从5.2%增长至30.0%,这个持续性的增长趋势与这几年间大肠杆菌中的黏菌素耐药菌增多的趋势相一致[52]。2016年从中国13个省的鸡源大肠杆菌中发现5.11%存在[53]。2017年从697份来自于农场,屠宰场和市场的鸡和猪的样品中分离到193株沙门氏菌,其中22株对黏菌素耐药且它们全部携带[54]。同年,从零售淡水鱼中分离到7株阳性菌株 (3.65%),且分别位于3种质粒和染色体上[55]。基因在食品动物的生产加工销售的全过程均有发现,说明蔬菜、肉制品和水产品的生产和贸易流程,可能构成了一种耐药性的传播途径。不仅在中国被广泛报道,在其他一些东南亚国家如越南、老挝、巴基斯坦检出率也偏高,最高达到了56.4%[56],而在北美及南美一些国家检出率不到5%,这可能与这些国家禁止使用黏菌素添加到饲料中用作生长促进剂有关[40-41]。
随着的广泛检出,发现多个可移动元件包括质粒、转座子、插入序列都与的转移密切相关[57]。携带的质粒类型主要包括IncI2、IncX4、IncHI2等,IncX3-IncX4和IncI2-IncFIB等杂合质粒也参与其中[58],甚至在染色体上也有诸多报道[59]。此外,携带的质粒非常稳定,即使没有黏菌素的选择性压力,也会稳定存在并广泛传播。以IncX4质粒为主线,系统分析了的基因环境,发现的转座与IS密切相关[60]。通过对GenBank中携带序列进行分析,研究者发现转座子Tn在的传播过程中发挥重要作用[61]。
大量研究发现,耐药基因可以和CTX-M、KPC、NDM、VIM等耐药基因共存于菌体内,从而形成名副其实的“超级细菌”[62-64]。尤其值得关注的是,和NDM共存的动物源菌株多有报道,但两种耐药基因在多数情况下位于不同的可接合转移型质粒上[65]。2017年报道了在中国山东省农村地区井水中分离到了多重耐药产ESBL大肠杆菌,同时携带KPC-2、CTX-M和基因[66]。另外,从肉鸡产业链中分离的161株碳青霉烯耐药大肠杆菌中MCR-1阳性率高达22.3% (34/161),但未发现两种耐药基因定位在同一质粒上。
2.3 细菌的耐受与持留
细菌的耐受性是指细菌群体不改变其MIC而能够在致死浓度的抗生素环境下存活的能力。而持留菌被认为是一种普遍存在于各种细菌种群中的一小部分对抗生素不敏感的细菌。持留菌本身并不含有导致耐药的突变,而是处于低代谢的生长状态以使得细菌对抗生素不敏感和耐受。细菌的耐受和持留往往导致抗生素治疗的失败,感染的反复发生,并且会引起抗生素耐药性的产生[67-68]。
降低细菌的生长速率能够使细菌对许多抗生素产生耐受性,其原因是这些抗生素是对生长中的细菌起作用,例如β-内酰胺类抗生素[69]以及氟喹诺酮类抗生素)[70]等。持留菌具有高度的异质性[71-73],其形成的机制也是多种多样的。毒素-抗毒素系统[74]、能量代谢[72]、反式翻译相关基因[75]、生物被膜[76]等因素都与持留菌的形成有关。另外,群体感应使细菌通过检测特定的信号分子来检测其细胞密度,与铜绿假单胞菌群体生长阶段持留形成有关[77]。不过,群体感应系统通过何种途径调控持留菌形成的机制尚不明确。
目前,我国对于细菌持留机制的研究较少。持留菌形成的机制十分复杂,以往的研究多是利用基因突变菌株或基因敲除菌株来研究个别基因对于持留形成的影响,利用二代测序与蛋白质质谱等组学技术研究者们可以从全局研究持留菌的基因表达变化。另外,持留菌具有高度的异质性,目前的研究都是把持留菌当作一个群体来研究,而形成持留菌的单个细菌的生存与代谢状态还尚不明确。
3 动物源细菌耐药性防控策略
我国已成为动物源细菌耐药性最严重的国家之一,而且“耐药性出现、过量使用或滥用抗菌药物、耐药谱或耐药水平加重”的恶性循环仍在继续。因此,如何有效防控动物源细菌耐药性已经迫在眉睫。2017年6月,农业部制定了《全国遏制动物源细菌耐药行动计划》(2017–2020年),提出了实施“监测行动”、完善动物源细菌耐药性监测网的方针(http://www.moa.gov.cn/govpublic/SYJ/ 201706/t20170623_5726086.htm)。建立有效的兽医耐药性监测网络,是获得耐药性基础数据的前提,也是耐药性风险评估以及防控的科学依据,同时,网络的交流平台还可传播耐药性理论、检测标准和技术服务并融入到养殖业控制体系。其次,合理的用药,尤其是PK/PD同步模型,在推荐合理的剂量、优化抗菌药物的临床给药方案以及降低或避免细菌耐药性的产生方面具有重要的指导作用,是防控细菌耐药性的重要手段。最后,开展畜禽养殖重要病原菌的动物专用抗菌药物和针对重要耐药病原菌开发耐药逆转剂、生物制剂以及针对细菌致病力的抑制剂的研究,获得与现有抗菌药物联用或单独使用有效治疗耐药病病原菌感染和消减病原菌耐药性的候选药物以及新兽药,也是控制细菌耐药性的重要策略。
20世纪90年代欧美地区的发达国家意识到细菌耐药性的潜在危害,先后成立了国家耐药性监测系统,如丹麦的DANMAP[78]。我国在动物源细菌耐药性方面的研究相对较晚,动物源细菌耐药性监测网络组建始于2008年,迄今已经连续监测10年,监测范围覆盖全国22个省 (自治区) 和4个直辖市,监测菌种包括食源性病原菌大肠杆菌、沙门氏菌、金黄色葡萄球菌、弯曲杆菌和肠球菌,以及畜禽重要病原菌 (包括猪链球菌、副猪嗜血杆菌和巴氏杆菌等)。此外,2016年1月,山东省农业科学院与山东省畜牧兽医局共建“山东省兽医抗药性监测网”启动,采集收藏了自2007年至今6 000株耐药性菌株及其耐药信息。对耐药菌株的持续监测有助于我们更好地了解耐药性的发生和发展变化,从而为指导临床用药提供理论基础。例如可转移的黏菌素耐药的发现和广泛传播直接影响到了国内和国际上黏菌素在畜禽中使用政策的调整,农业部根据此项研究,已将黏菌素禁用于所有动物的饲料添加中,这对维护人类健康和保障食品安全有着重要的意义。
通过PK-PD (药动/药效学) 模型,优化抗菌药物的临床给药方案,是目前降低或避免细菌耐药性产生的一项重要举措。国外主要是半体内模型研究,即体内药动学和体外药效学数据的同步拟合,相关研究集中于氟喹诺酮类药物在牛、羊的细菌感染模型[79-81]。国内兽医领域,通过体外动态模型、半体内模型、体内组织笼模型以及小鼠模型等主要开展了动物专用药头孢喹肟、马波沙星以及沃尼妙林对牛、猪和鸡巴氏杆菌、大肠杆菌、金黄色葡萄菌、支原体、链球菌感染等体内药动药效同步模型研究[82-88]。近年来,华南农业大学兽医学院药理教研室开展了大量基于PK/PD同步模型的优化兽用抗菌药物合理用药的研究,分别建立和完善了从体外动态模型,到半体内及体内感染的各类PK/PD同步模型,研究了抗菌药物不同给药策略对畜禽重要病原菌抗菌效果的影响,以优化兽用抗菌药物的给药方案,达到临床中治愈疾病的同时又能抑制细菌耐药性的产生的理想目标,为进一步控制耐药菌株的产生和选择性富集提供了数据支撑。
在开发新兽药上,专用的抗菌药物的开发是研发的方向。近年来,国内外学者通过在截短侧耳素母核的C12和C14支链上的改造,发现C12衍化为12-epi-pleuromutilins具有广泛的抗菌活性,包括革兰氏阴性菌,还获得了C14支链上连接的一系列截短侧耳素类化合物,其中一些具有良好的抗革兰氏阳性菌尤其是金黄色葡萄球菌作用[89-90]。在针对耐药菌逆转耐药性的技术上,开发β-内酰胺酶抑制剂 (克拉维酸等);筛选针对耐药菌的主要外输泵 (NorA和AcrAB-TolC等) 天然抑制剂 (5-甲氧基大风子品-D以及青蒿琥酯)[91-92];筛选增加细胞膜通透性提高抗菌药物摄入的小分子 (黏菌素提高利福平和万古霉素抗革兰氏阴性菌活性)[93-94];改变代谢环境的小分子抗生素佐剂 (葡萄糖或丙氨酸小分子提高卡那霉素抗菌活性)[95];质粒消除剂,包括抑制质粒的复制(安普霉素可抑制IncB质粒的复制)[96],利用毒素-抗毒素 (TA) 系统 (利福平、氯霉素、壮观霉素、三甲氧苄啶与磺胺药等抗菌药物可引起细菌发生TA系统依赖性程序性死亡)[97],抑制质粒的接合转移 (二磷酸化合物以及长链不饱和脂肪酸)[98]。在生物制剂遏制抗生素耐药性上,主要是噬菌体和抗菌疫苗,噬菌体产品尚处在研究阶段,而疫苗主要起预防作用[99-101]。在筛选细菌致病力抑制剂方面,包括针对群体感应系统[102]、生物被膜[103]以及细菌毒力[104-105]等抑制剂的研发。在兽医领域,也有以SPF微生态体理论指导建立动物病原菌耐药性防控体系。还有基于SPF微生态体理论,通过SPF鸡粪菌移植,重新构建雏鸡微生态体,建立动物病原菌耐药性防控体系。
尽管近年来,我国在动物源耐药性监测和控制上动作频频,而且取得了长足的进步,但是考虑到我国目前耐药性的严峻形势,仍然急需新型监测和防控策略。比如,亟待建立自下而上的耐药性监测网,实时获取病原菌耐药性数据,同时,需建立动物源病原菌-耐药谱型-耐药基因型一体的网络化的耐药性数据库以及全国动物源病原菌耐药性研究协作网络和共享技术平台。在PK-PD模型优化给药方案上,需要将生理药动学 (PBPK)和群体药动学 (PPK) 等新型药动学模型有机地整合到PK/PD模型中;其次,需进一步研究防止抗菌药物耐药突变选择的给药方案;最后,还需在兽医领域构建基于机制的PK/PD模型,将机体免疫系统、细菌生长繁殖、药物作用机制等因素进一步整合进入药效学影响因素,使PK/PD模型接近药物、机体、细菌相互作用真实情况。在开发新兽药上,针对耐药菌的主要耐药途径筛选的抑制剂以及抗细菌毒力的抑制剂,多处于实验室研究阶段,缺乏相应的产品。在细菌耐药性逆转剂上,基于抗生素联合对多重耐药质粒的消除、特定酶(NDM) 或外排泵(AcrAB-tolC以及OptrA) 的抑制以及基于功能代谢组学的耐药性重编分子上将是研究的热点。此外,针对耐药菌和耐药基因在人和动物间传递,开发新型的动物专用抗菌药物,切断人类和动物之间的耐药性联系,也是遏制细菌耐药性的重要策略。
[1] Sun J, Liao XP, Liu YH, et al. One Health: an approach to resolving antibiotic resistance. Infect Dis Transl Med, 2016, 2(3): 122–124.
[2] van Boeckel TP, Brower C, Gilbert M, et al. Global trends in antimicrobial use in food animals. Proc Natl Acad Sci USA, 2015, 112(18): 5649–5654.
[3] Arias CA, Murray BE. Antibiotic-resistant bugs in the 21st century—a clinical super-challenge. N Engl J Med, 2009, 360: 439–443.
[4] Carattoli A. Animal reservoirs for extended spectrum β-lactamase producers. Clin Microbiol Infect, 2008, 14(S1): 117–123.
[5] Woodford N, Wareham DW, Guerra B, et al. Carbapenemase-producing Enterobacteriaceae and non-Enterobacteriaceae from animals and the environment: an emerging public health risk of our own making? J Antimicrob Chemother, 2014, 69(2): 287–291.
[6] Yong D, Toleman MA, Giske CG, et al. Characterization of a new metallo-β-lactamase gene,NDM-1, and a novel erythromycin esterase gene carried on a unique genetic structure insequence type 14 from India. Antimicrob Agents Chemother, 2009, 53(12): 5046–5054.
[7] Kobayashi K, Hayashi I, Kouda S, et al. Identification and characterization of a novelassociated with theIMP-1-integron in a multidrug-resistant. PLoS ONE, 2013, 8(8): e70557.
[8] He T, Wang Y, Sun LC, et al. Occurrence and characterization of blaNDM-5-positiveisolates from dairy cows in Jiangsu, China. J Antimicrob Chemother, 2016, 72(1): 90–94.
[9] Martínez-Martínez L, González-López JJ. Carbapenemases in Enterobacteriaceae: types and molecular epidemiology. Enferm Infecc Microbiol Clín, 2014, 32(S4): 4–9.
[10] Carattoli A. Plasmids and the spread of resistance. Int J Med Microbiol, 2013, 303(6/7): 298–304.
[11] Johnson AP, Woodford N. Global spread of antibiotic resistance: the example of New Delhi metallo-β-lactamase (NDM)-mediated carbapenem resistance. J Med Microbiol, 2013, 62(4): 499–513.
[12] Poirel L, Lagrutta E, Taylor P, et al. Emergence of metallo-β-Lactamase NDM-1-producing multidrug- resistantin Australia. Antimicrob Agents Chemother, 2010, 54(11): 4914–4916.
[13] Wang Y, Wang X, Schwarz S, et al. IMP-45-producing multidrug-resistantof canine origin. J Antimicrob Chemother, 2014, 69(9): 2579–2581.
[14] Zhang RM, Liu ZH, Li JY, et al. Presence of VIM-positivespecies in chickens and their surrounding environment. Antimicrob Agents Chemother, 2017, 61(7): e00167-17.
[15] Madec JY, Haenni M, Nordmann P, et al. Extended-spectrum β-lactamase/AmpCand carbapenemase-producing Enterobacteriaceae in animals: a threat for humans? Clin Microbiol Infect, 2017, 23(11): 826–833.
[16] Wang Y, Wu CM, Zhang QJ, et al. Identification of New Delhi metallo-β-lactamase 1 inof food animal origin. PLoS ONE, 2012, 7(5): e37152.
[17] Zhang WJ, Lu ZL, Schwarz S, et al. Complete sequence of theNDM-1-carrying plasmid pNDM-AB fromof food animal origin. J Antimicrob Chemother, 2013, 68(7): 1681–1682.
[18] Zhang RM, Wang Y, Liu ZH, et al. Characterization of NDM-1-producing carbapenemase inspp. andisolates from diseased pigs. Front Agr Sci Eng, 2015, 2(3): 223–229.
[19] Yang YQ, Zhang AY, Ma SZ, et al. Co-occurrence ofand ESBL on a single plasmid in. J Antimicrob Chemother, 2016, 71(8): 2336–2338.
[20] Liu BT, Song FJ, Zou M, et al. High incidence of Escherichia coli strains coharboringandNDMfrom chickens. Antimicrob Agents Chemother, 2017, 61(3): e02347–16.
[21] Kong LH, Lei CW, Ma SZ, et al. Various sequence types of Escherichia coli isolates coharboringNDM-5andgenes from a commercial swine farm in China. Antimicrob Agents Chemother, 2017, 61(3): e02167–16.
[22] Liu BT, Song FJ, Zou M, et al. Emergence of colistin resistance geneinproducing NDM-9 and infrom the same animal. Antimicrob Agents Chemother, 2017, 61(2): e01444–16.
[23] Wang RB, Liu YQ, Zhang Q, et al. The prevalence of colistin resistance inandisolated from food animals in China: coexistence ofandNDMwith low fitness cost. Int J Antimicrob Agents, 2018, 51(5): 739–744.
[24] Liu ZH, Wang Y, Walsh TR, et al. Plasmid-mediated novelNDM-17gene encoding a carbapenemase with enhanced activity in a sequence type 48strain. Antimicrob Agents Chemother, 2017, 61(5): e02233–16.
[25] Liu ZH, Li JY, Wang XM, et al. Novel variant of new delhi metallo-β-lactamase, NDM-20, in. Front Microbiol, 2018, 9: 248.
[26] Ghatak S, Singha A, Sen A, et al. Detection of new delhi metallo-beta-Lactamase and Extended-Spectrum beta-Lactamase Genes inisolated from mastitic milk samples. Transbound Emerg Dis, 2013, 60(5): 385–389.
[27] Fischer J, Schmoger S, Jahn S, et al. NDM-1 carbapenemase-producingsubsp. enterica serovar Corvallis isolated from a wild bird in Germany. J Antimicrob Chemother, 2013, 68(12): 2954–2956.
[28] Shaheen BW, Nayak R, Boothe DM. Emergence of a New Delhi metallo-β-lactamase (NDM-1)-encoding gene in clinicalisolates recovered from companion animals in the United States. Antimicrob Agents Chemother, 2013, 57(6): 2902–2903.
[29] Wang Y, Zhang RM, Li JY, et al. Comprehensive resistome analysis reveals the prevalence of NDM andin Chinese poultry production. Nat Microbiol, 2017, 2: 16260, doi: 10.1038/nmicrobiol. 2016.260.
[30] He T, Wei RC, Zhang LL, et al. Characterization of NDM-5-positive extensively resistantisolates from dairy cows. Vet Microbiol, 2017, 207: 153–158.
[31] Sun J, Yang RS, Zhang QJ, et al. Co-transfer ofNDM-5andby an IncX3-X4 hybrid plasmid in. Nat Microbiol, 2016, 1: 16176.
[32] Cui LQ, Lei L, Lv Y, et al.NDM-1-producing multidrug-resistantisolated from a companion dog in China. J Glob Antimicrob Resist, 2017, 13: 24–27.
[33] Lauretti L, Riccio ML, Mazzariol A, et al. Cloning and characterization ofVIM, a new integron-borne metallo-β-lactamase gene from a Pseudomonas aeruginosa clinical isolate. Antimicrob Agents Chemother, 1999, 43(7): 1584–1590.
[34] Liu YY, Wang Y, Walsh TR, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: a microbiological and molecular biological study. Lancet Infect Dis, 2016, 16(2): 161–168.
[35] Gao RS, Hu YF, Li ZC, et al. Dissemination and mechanism for the MCR-1 colistin resistance. PLoS Pathog, 2016, 12(11): e1005957.
[36] Wei WH, Srinivas S, Lin JX, et al. Defining ICR-Mo, an intrinsic colistin resistance determinant from. PLoS Genet, 2018, 14(5): e1007389.
[37] Xu YC, Lin JX, Cui T, et al. Mechanistic insights into transferable polymyxin resistance among gut bacteria. J Biol Chem, 2018, 293(12): 4350–4365.
[38] Xu YC, Wei WH, Lei S, et al. An evolutionarily conserved mechanism for intrinsic and transferable polymyxin resistance. mBio, 2018, 9(2): e02317–17.
[39] Sun J, Xu YC, Gao RS, et al. Deciphering MCR-2 Colistin Resistance. mBio, 2017, 8(3): e00625–17.
[40] Sun J, Zhang HM, Liu YH, et al. Towards understanding MCR-like colistin resistance. Trends Microbiol, 2018, doi: 10.1016/j.tim.2018.02.006.
[41] Feng YJ. Transferability of MCR-1/2 polymyxin resistance: Complex dissemination and genetic mechanism. ACS Infect Dis, 2018, 4(3): 291–300.
[42] Wang XN, Zhang HM, Sun J, et al. The MCR-1 colistin resistance: A new challenge to global public health. Chin Sci Bull, 2017, 62(10): 1018–1029.
[43] Lv J, Mohsin M, Lei S, et al. Discovery of a-bearing plasmid in commensal colistin-resistantfrom healthy broilers in Faisalabad, Pakistan. Virulence, 2018, doi: 10.1080/21505594. 2018.1462060.
[44] Gao RS, Wang QJ, Li P, et al. Genome sequence and characteristics of plasmid pWH12, a variant of the-harbouring plasmid pHNSHP45, from the multi-drug resistant. Virulence, 2016, 7(6): 732–735.
[45] Li ZC, Tan C, Lin JX, et al. Diversified variants of the-carrying plasmid reservoir in the swine lung microbiota. Sci China Life Sci, 2016, 59(9): 971–973.
[46] Gao RS, Li Y, Lin JX, et al. Unexpected complexity of multidrug resistance in the-harbouring. Sci China Life Sci, 2016, 59(7): 732–734.
[47] Zhang HM, Seward CH, Wu ZW, et al. Genomic insights into the ESBL and MCR-1-producing ST648with multi-drug resistance. Sci Bull, 2016, 61(11): 875–878.
[48] Wang QJ, Li ZC, Lin JX, et al. Complex dissemination of the diversifiedharbouring plasmids inof different sequence types. Oncotarget, 2016, 7(50): 82112–82122.
[49] Schwarz S, Johnson AP. Transferable resistance to colistin: a new but old threat. J Antimicrob Chemother, 2016, 71(8): 2066–2070.
[50] Li XP, Fang LX, Song JQ, et al. Clonal spread ofin PMQR-carrying ST34isolates from animals in China. Sci Rep, 2016, 6: 38511.
[51] Li XP, Fang LX, Jiang P, et al. Emergence of the colistin resistance genein. Int J Antimicrob Agents, 2017, 49(6): 786–787.
[52] Shen ZQ, Wang Y, Shen YB, et al. Early emergence ofinfrom food-producing animals. Lancet Infect Dis, 2016, 16(3): 293.
[53] Yang YQ, Li YX, Song T, et al. Colistin resistance geneand its variant inisolates from chickens in China. Antimicrob Agents Chemother, 2017, 61(5): e01204–16.
[54] Ma SZ, Lei CW, Kong LH, et al. Prevalence, antimicrobial resistance, and relatedness of salmonella isolated from chickens and pigs on farms, abattoirs, and markets in Sichuan province, China. Foodborne Pathog Dis, 2017, 14(11): 667–677.
[55] Lv L, Cao Y, Yu P, et al. Detection ofgene amongisolates from farmed fish and characterization of-bearing incp plasmids. Antimicrob Agents Chemother, 2018, 62(3): e02378–17.
[56] Trung NV, Matamoros S, Carrique-Mas JJ, et al. Zoonotic transmission ofcolistin resistance gene from small-scale poultry farms, Vietnam. Emerg Infect Dis, 2017, 23(3): 529–532.
[57] Wang RB, van Dorp L, Shaw LP, et al. The global distribution and spread of the mobilized colistin resistance gene. Nat Commun, 2018, 9(1): 1179.
[58] Wang QQ, Sun J, Li J, et al. Expanding landscapes of the diversified-bearing plasmid reservoirs. Microbiome, 2017, 5: 70.
[59] Sun J, Li XP, Fang LX, et al. Co-occurrence ofin the chromosome and on an IncHI2 plasmid: persistence of colistin resistance in. Int J Antimicrob Agents, 2018, 51(6): 842–847.
[60] Sun J, Fang LX, Wu ZW, et al. Genetic analysis of the IncX4 plasmids: implications for a unique pattern in theacquisition. Sci Rep, 2017, 7: 424.
[61] Snesrud E, McGann P, Chandler M. The birth and demise of the IS--IScomposite transposon: the vehicle for transferable colistin resistance. mBio, 2018, 9(1): e02381–17.
[62] Poirel L, Kieffer N, Liassine N, et al. Plasmid-mediated carbapenem and colistin resistance in a clinical isolate of. Lancet Infect Dis, 2016, 16(3): 281.
[63] Sun J, Li XP, Yang RS, et al. Complete nucleotide sequence of an IncI2 plasmid coharboringCTX-M-55and. Antimicrob Agents Chemother, 2016, 60(8): 5014–5017.
[64] Sun J, Zeng X, Li XP, Liao, X. P, et al. Plasmid-mediated colistin resistance in animals: current status and future directions. Animal Health Res Rev, 2017, 18(2): 136–152.
[65] Zhong LL, Zhang YF, Doi Y, et al. Coproduction of MCR-1 and NDM-1 by colistin-resistantisolated from a healthy individual. Antimicrob Agents Chemother, 2017, 61(1): e01962–16.
[66] Sun P, Bi ZW, Nilsson M, et al. Occurrence ofKPC-2,CTX-Mandin Enterobacteriaceae from well water in rural China. Antimicrob Agents Chemother, 2017, 61(4): e02569–16.
[67] Cohen NR, Lobritz MA, Collins JJ. Microbial persistence and the road to drug resistance. Cell Host Microbe, 2013, 13(6): 632–642.
[68] Levin-Reisman I, Ronin I, Gefen O, et al. Antibiotic tolerance facilitates the evolution of resistance. Science, 2017, 355(6327): 826–830.
[69] Horne D, Tomasz A. Tolerant response ofto Beta-Lactams and other cell wall inhibitors. Antimicrob AgentsChemother, 1977, 11(5): 888–896.
[70] Evans DJ, Allison DG, Brown MRW, et al. Susceptibility ofandbiofilms towards ciprofloxacin: effect of specific growth rate. J Antimicrob Chemother, 1991, 27(2): 177–184.
[71] Li YF, Zhang Y. PhoU is a persistence switch involved in persister formation and tolerance to multiple antibiotics and stresses in. Antimicrob Agents Chemother, 2007, 51(6): 2092–2099.
[72] Ma C, Sim S, Shi WL, et al. Energy production genesandare involved in persister survival and tolerance to multiple antibiotics and stresses in. FEMS Microbiol Lett, 2010, 303(1): 33–40.
[73] Allison KR, Brynildsen MP, Collins JJ. Heterogeneous bacterial persisters and engineering approaches to eliminate them. Curr Opin Microbiol, 2011, 14(5): 593–598.
[74] Harrison JJ, Wade WD, Akierman S, et al. The chromosomal toxin geneis a determinant of multidrug tolerance forgrowing in a biofilm. Antimicrob Agents Chemother, 2009, 53(6): 2253–2258.
[75] Li JH, Ji L, Shi WL, et al. Trans-translation mediates tolerance to multiple antibiotics and stresses in. J Antimicrob Chemother, 2013, 68(11): 2477–2481.
[76] Olson ME, Ceri H, Morck DW, et al. Biofilm bacteria: formation and comparative susceptibility to antibiotics. Canad J Vet Res, 2002, 66(2): 86–92.
[77] Möker N, Dean CR, Tao JS.increases formation of multidrug-tolerant persister cells in response to quorum-sensing signaling molecules. J Bacteriol, 2010, 192(7): 1946–1955.
[78] Hammerum AM, Heuer OE, Emborg HD, et al. Danish integrated antimicrobial resistance monitoring and research program. Emerg Infect Dis, 2007, 13(11): 1632–1639.
[79] Paulin A, Schneider M, Dron F, et al. Pharmacokinetic/pharmacodynamic evaluation of marbofloxacin as a single injection for Pasteurellaceae respiratory infections in cattle using population pharmacokinetics and Monte Carlo simulations. J Vet Pharmacol Ther, 2017, 41(1): 39–50.
[80] Vallé M, Schneider M, Galland D, et al. Pharmacokinetic and pharmacodynamic testing of marbofloxacin administered as a single injection for the treatment of bovine respiratory disease. J Vet Pharmacol Ther, 2012, 35(6): 519–528.
[81] Landoni MF, Comas W, Mucci N, et al. Enantiospecific pharmacokinetics and pharmacodynamics of ketoprofen in sheep. J Vet Pharmacol Ther, 1999, 22(6): 349–359.
[82] Xiao X, Sun J, Yang T, et al. Pharmacokinetic/pharmacodynamic profiles of tiamulin in an experimental intratracheal infection model of. Front Vet Sci, 2016, 3: 75.
[83] Guo CN, Liao XP, Wang MR, et al.Pharmacodynamics of cefquinome in a neutropenic mouse thigh model ofserotype 2 at varied initial inoculum sizes. Antimicrob Agents Chemother, 2016, 60(2): 1114–1120.
[84] Yu Y, Zhou YF, Chen MR, et al.pharmacokinetics/pharmacodynamics of cefquinome in an experimental mouse model ofmastitis following intramammary infusion. PLoS ONE, 2016, 11(5): e0156273.
[85] Zhou YF, Shi W, Yu Y, et al. Pharmacokinetic/pharmacodynamic correlation of cefquinome against experimental catheter-associated biofilm infection due to. Front Microbiol, 2015, 6: 1513.
[86] Zhou YF, Tao MT, He YZ, et al.bioluminescent monitoring of therapeutic efficacy and pharmacodynamic target assessment of antofloxacin againstin a neutropenic murine thigh infection model. Antimicrob Agents Chemother, 2017, 62(1): e01281–17. .
[87] Zhao DH, Zhou YF, Yu Y, et al. Integration of pharmacokinetic and pharmacodynamic indices of valnemulin in broiler chickens after a single intravenous and intramuscular administration. Vet J, 2014, 201(1): 109–115.
[88] Shan Q, Wang J, Yang F, et al. Pharmacokinetic/pharmacodynamic relationship of marbofloxacin againstin a tissue-cage model in yellow cattle. J Vet Pharmacol Ther, 2014, 37(3): 222–230.
[89] Luo J, Yang QE, Yang YY, et al. Design, synthesis, and structure-activity relationship studies of novel pleuromutilin derivatives having a piperazine ring. Chem Biol Drug Des, 2016, 88(5): 699–709.
[90] Murphy SK, Zeng MS, Herzon SB. A modular and enantioselective synthesis of the pleuromutilin antibiotics. Science, 2017, 356(6341): 956–959.
[91] Lowrence RC, Raman T, Makala HV, et al. Dithiazole thione derivative as competitive NorA efflux pump inhibitor to curtail multi drug resistant clinical isolate of MRSA in a zebrafish infection model. Appl Microbiol Biotechnol, 2016, 100(21): 9265–9281.
[92] Mahmood HY, Jamshidi S, Sutton JM, et al. Current advances in developing inhibitors of bacterial multidrug efflux pumps. Curr Med Chem, 2016, 23(10): 1062–1081.
[93] Aoki N, Tateda K, Kikuchi Y, et al. Efficacy of colistin combination therapy in a mouse model of pneumonia caused by multidrug-resistant. J Antimicrob Chemother, 2009, 63(3): 534–542.
[94] Gordon NC, Png K, Wareham DW. Potent synergy and sustained bactericidal activity of a vancomycin-colistin combination versus multidrug-resistant strains of. Antimicrob Agents Chemother, 2010, 54(12): 5316–5322.
[95] Peng B, Su YB, Li H, et al. Exogenous alanine and/or glucose plus kanamycin kills antibiotic-resistant bacteria. Cell Metab, 2015, 21(2): 249–261.
[96] DeNap JCB, Thomas JR, Musk DJ, et al. Combating drug-resistant bacteria: small molecule mimics of plasmid incompatibility as antiplasmid compounds. J Am Chem Soc, 2004, 126(47): 15402–15404.
[97] Moritz EM, Hergenrother PJ. Toxin-antitoxin systems are ubiquitous and plasmid-encoded in vancomycin-resistant enterococci. Proc Natl Acad Sci USA, 2007, 104(1): 311–316.
[98] Fernandez-Lopez R, Machón C, Longshaw CM, et al. Unsaturated fatty acids are inhibitors of bacterial conjugation. Microbiology, 2005, 151(11): 3517–3526.
[99] Jamal M, Hussain T, Rajanna Das C, et al. Isolation and characterization of aMJ1 bacteriophage against multi-drug resistant3. Jundishapur J Microbiol, 2015, 8(11): e25917.
[100] Francis DH, Willgohs JA. Evaluation of a live avirulentvaccine for K88+, LT+ enterotoxigenic colibacillosis in weaned pigs. Am J Vet Res, 1991, 52(7): 1051–1055.
[101] Ruan XS, Liu M, Casey TA, et al. A tripartite fusion, FaeG-FedF-LT192A2:B, of enterotoxigenic(ETEC) elicits antibodies that neutralize cholera toxin, inhibit adherence of K88 (F4) and F18 fimbriae, and protect pigs against K88ac/heat-labile toxin infection. Clin Vaccine Immunol, 2011, 18(10): 1593–1599.
[102] Kalia VC, Purohit HJ. Quenching the quorum sensing system: potential antibacterial drug targets. Crit Rev Microbiol, 2011, 37(2): 121–140.
[103] Resch A, Rosenstein R, Nerz C, et al. Differential gene expression profiling ofcultivated under biofilm and planktonic conditions. Appl Environ Microbiol, 2005, 71(5): 2663–2676.
[104] Feng HH, Xiang H, Zhang JY, et al. Genome-wide transcriptional profiling of the response ofto cryptotanshinone. J Biomed Biotechnol, 2009, 2009: 617509.
[105] Qiu JZ, Xiang H, Hu C, et al. Subinhibitory concentrations of farrerol reduce α-toxin expression in. FEMS Microbiol Lett, 2011, 315(2): 129–133.
(本文责编 郝丽芳)
Towards understanding antibiotic resistance in animals-borne bacterial pathogens
Jian Sun1, Yahong Liu1, and Youjun Feng1,2
1 National Risk Assessment Laboratory for Antimicrobial Resistance of Animal Original Bacteria, South China Agricultural University, Guangzhou 510642, Guangdong, China 2 College of Animal Sciences/School of Medicine, Zhejiang University, Hangzhou 310058, Zhejiang, China
Antimicrobial resistance is a common problem in both veterinary and human medicine. The increasing incidence of multi-drug resistance and even pan-drug resistance is posing serious threat to public health and food safety. This review aimed to present an updated overview on antimicrobial resistance, covering the transmission mechanisms and its potential controlling strategies.
bacteria of animal origin, resistance gene, transmission mechanism, strategy to counter antimicrobial resistance
10.13345/j.cjb.180162
April 25, 2018;
June 20, 2018
National Key Research and Development Program of China (Nos. 2017YFD0500202, 2016YFD0501300).
Yahong Liu. Tel/Fax: +86-20-85280006; E-mail: lyh@scau.edu.cn
Youjun Feng. Tel/Fax: +86-571-88208524; E-mail: fengyj@zju.edu.cn
国家重点研发计划 (Nos. 2017YFD0500202,2016YFD0501300) 资助。

