东山岛星座短腹海鞘共附生微生物多样性研究
乔玉宝, 田晓清, 唐莹莹, 樊成奇, 马丽艳, 陆亚男
(1中国水产科学研究院东海水产研究所,农业部东海与远洋渔业资源开发利用重点实验室,上海 200090; 2上海海洋大学,上海 201306)
星座短腹海鞘(Aplidiumconstellatum,也称星座褶胃海鞘),原名星座美洲海鞘(AmarouciumconstellatumVerrill,也称星座列精海鞘 ),属脊索动物门(Chordata)尾索动物亚门(Urochordata)海鞘纲(Asciacea)三段海鞘科(Polyclinidae)短腹海鞘属(或称褶胃海鞘属),为复海鞘,在渤海、黄海、东海海域均有分布,是我国海鞘常见优势物种之一[1-2]。此前已有文献报道从该属海鞘中分离出了具有多种骨架类型的生物碱、酚醌、环肽、大环内酯等成分,并显示出抗菌、抗肿瘤等多种生物活性[3-4],提示其可能具有药学或保健方面的应用价值。但迄今为止,尚未有关于星座短腹海鞘共附生微生物及其活性物质的报道。前期对星座短腹海鞘脂溶性提取物进行了研究,首次发现其具有降血脂功效,提示其可能具有进一步的开发利用价值,并开展了其脂溶性提取物提取工艺的初步研究[5-6]。
已有研究表明,海洋生物次生代谢产物的类型与多样性与其共附生微生物的次生代谢产物类型与多样性具有较强的相关性。海鞘中蕴含着丰富的微生物资源,其共附生微生物中可能存在着与海鞘生物中相类似的活性天然产物[7-8]。为进一步研究该海鞘及其共附生微生物的次生代谢产物,本文以福建东山岛采集的星座短腹海鞘为研究材料,通过免培养微生物高通量测序技术和纯培养技术对其细菌多样性进行分析,一方面通过高通量测序技术首次解析了其共附生菌群的种类、丰度及多样性信息,另一方面也积累了丰富的该种海鞘共附生微生物菌种资源,以期为进一步探索海鞘与微生物之间的共生、附生关系,并开展海鞘降血脂活性物质的开发利用奠定基础。
1 材料与方法
1.1 实验材料
星座短腹海鞘于2017年5月采自福建漳州东山岛海域。采集后用大量海水冲洗并快速置于4 ℃冷藏保存,24 h内运回实验室进行分析。
Ezup柱式基因组DNA抽提试剂盒(上海生工生物有限公司);16 s引物(上海生工生物有限公司);PCR扩增体系(北京康为世纪)。
1.2 实验方法
1.2.1 高通量测序
星座短腹海鞘(20 g)样品用无菌海水清洗3次,清洗干净表面的泥沙,将清洗好的样品用灭菌研钵研磨成浆,然后用适量无菌海水进行稀释,备用。
DNA抽提:使用 Stool DNA Kit 试剂盒(OMEGA,美国)提取星座短腹海鞘样品微生物的DNA。所有样品的DNA 提取步骤均参照Stool DNA Kit试剂盒说明书。对提取到的样品基因组DNA 用质量分数为1% 的琼脂糖凝胶电泳检测质量。
PCR扩增:针对细菌16S rRNA基因V3 + V4 区设计含 barcode 的特异引物338F /806R,引物序列为:338F:5′-ACTCCTACGGGAGGCAGCAG - 3′,806R:5′-GGACTACHVGGGTWTCTAAT- 3′。PCR扩增采用TransGen AP221-02:TransStart FastPfu DNA Polymerase,20 μL反应体系:5×FastPfu Buffer 4 μL,2.5 mM dNTPs 2 μL,Forward Primer(5 μM) 0.8 μL,Reverse Primer(5 μM) 0.8 μL,FastPfu Polymerase 0.4 μL,BSA 0.2 μL,Template DNA 10 ng,补ddH2O至20 μL。PCR条件:95 ℃变性3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,27个循环;最后72 ℃ 10 min延伸(PCR仪:ABI Gene Amp® 9700型)。然后将获得PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物,Tris-HCl洗脱; 2%琼脂糖电泳检测。
Miseq文库构建及Miseq测序:构建测序文库后在 Illumina Miseq平台进行高通量测序(美吉生物公司,上海)[9]。
数据质控:MiSeq测序得到的是双端序列数据,首先根据PE reads之间的overlap关系,将成对的reads拼接(merge)成一条序列,同时对reads的质量和merge的效果进行质控过滤,根据序列首尾两端的barcode和引物序列区分样品得到有效序列,并校正序列方向。数据去杂方法和参数:1)过滤reads尾部质量值20以下的碱基,设置50bp的窗口,如果窗口内的平均质量值低于20,从窗口开始截去后端碱基,过滤质控后50bp以下的reads,去除含N碱基的reads;2)根据PE reads之间的overlap关系,将成对reads拼接(merge)成一条序列,最小overlap长度为10 bp;3)拼接序列的overlap区允许的最大错配比率为0.2,筛选不符合序列;4)根据序列首尾两端的barcode和引物区分样品,并调整序列方向,barcode允许的错配数为0,最大引物错配数为2;使用软件:FLASH、Trimmomatic。
数据分析:按照97%相似性对非重复序列(不含单序列)进行OTU聚类,得到OTU的代表序列;将所有优化序列map至OTU代表序列,选出与代表序列相似性在97%以上的序列,生成OTU表格。采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,并分别在各个分类水平:domain(域),kingdom(界),phylum(门),class(纲),order(目),family(科),genus(属),species(种)统计各样本的群落组成。16S细菌比对数据库:Silva、RDP、Greengene。
软件及算法: Qiime平台, RDP Classifier,置信度阈值为0.7。
1.2.2 可培养共附生菌的分离及鉴定
可培养共附生菌的分离:将1.2.1中剩余海鞘研磨样品用适量无菌海水稀释,置4 ℃冰箱过夜,次日取上清液用无菌海水进行梯度稀释至10-1、10-2、10-3、10-4,分别涂布于2216E培养基,28 ℃培养10 d。待平板菌落长成,挑取不同形状、不同颜色的菌落平板划线进行纯化,最后挑取划线平板上单菌落接到斜面上,保种备用。
可培养共附生菌的鉴定:1)细菌基因组DNA的制备:将已生长到对数期的斜面菌落,接种到5 mL 2216液体培养基中,置于摇床28 ℃培养3~5 d,根据细菌基因组DNA提取试剂盒说明书步骤提取目标基因组DNA。2)引物设计:海洋细菌16S rRNA基因扩增选用通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGTTACCTTGTTACGACTT-3′)进行PCR扩增。3)PCR扩增体系为50 μL∶2×TaqPCR MasterMix 25 μL,引物各1 μL,模板DNA 3 μL,重蒸水20 μL。扩增程序:94 ℃预变性5 min,94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸10 min[10]。PCR产物进行1%琼脂糖凝胶电泳[11]。4)DNA测序和系统发生学分析:委托上海美吉生物公司完成测序。获得的16S rRNA基因序列提交GenBank用BLAST 软件与已知序列进行对比,利用MEGA 5.1软件绘制系统发育进化树。
2 结果与分析
2.1 样品信息统计
星座短腹海鞘所获得的37 911条序列长度分布在293~480(表1),420~440范围内序列最多,为30 325条,其次是441~460,为7 502条(图1)。

图1 序列长度分布Fig.1 Length distribution of trimmed sequences
从稀释曲线来看,随着测序量的增加,稀释曲线斜率逐渐降低,曲线趋向平坦,说明测序数据量合理,更多的数据量只会产生少量的新的物种(图2)。

图2 稀释曲线Fig.2 Rarefaction curve
2.2 物种注释与评估
可操作分类单元(OTU,operational taxonomic units) 是在系统发生学Greengene或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系,种,属,分组等)设置的统一标志。要了解一个样本测序结果中的菌种、菌属等数目信息,就需要对序列进行聚类。通过聚类操作,对各个样品的有效序列进行系谱分类,以 97%相似性水平为标准划分OTU[11],使得星座短腹海鞘共附生微生物所获得的37 911条序列分为310个OTUs,注释到24个门,41个纲,84个目,124个科,197个属,249个种。
从多样性指数表来看,ace值和chao值分别为310.327和310.091,说明所测样品中所含OTU数目为310;Simpson值为0.373,Shannon值为2.494,说明所测样品群落多样性较高;样本覆盖率(Coverage)为99.99%,说明本次测序结果可以代表样本中微生物的真实情况(表2)。
2.3 物种组成分析
从门的水平来看(图4),星座短腹海鞘共附生微生物优势门有7个,分别为变形菌门(Proteobacteria,78.86%)、放线菌门(Actinobacteria,6.01%)、拟杆菌门(Bacteroidetes,4.67%)、蓝藻门(Cyanobacteria,2.62%)、厚壁菌门(Firmicutes,1.79%)、柔膜菌门(Tenericutes,1.76%)、螺旋菌门(Spirochaetae,1.33%),其它的17个门占2.95%。

表1 样本信息统计表Tab.1 Sample information statistics
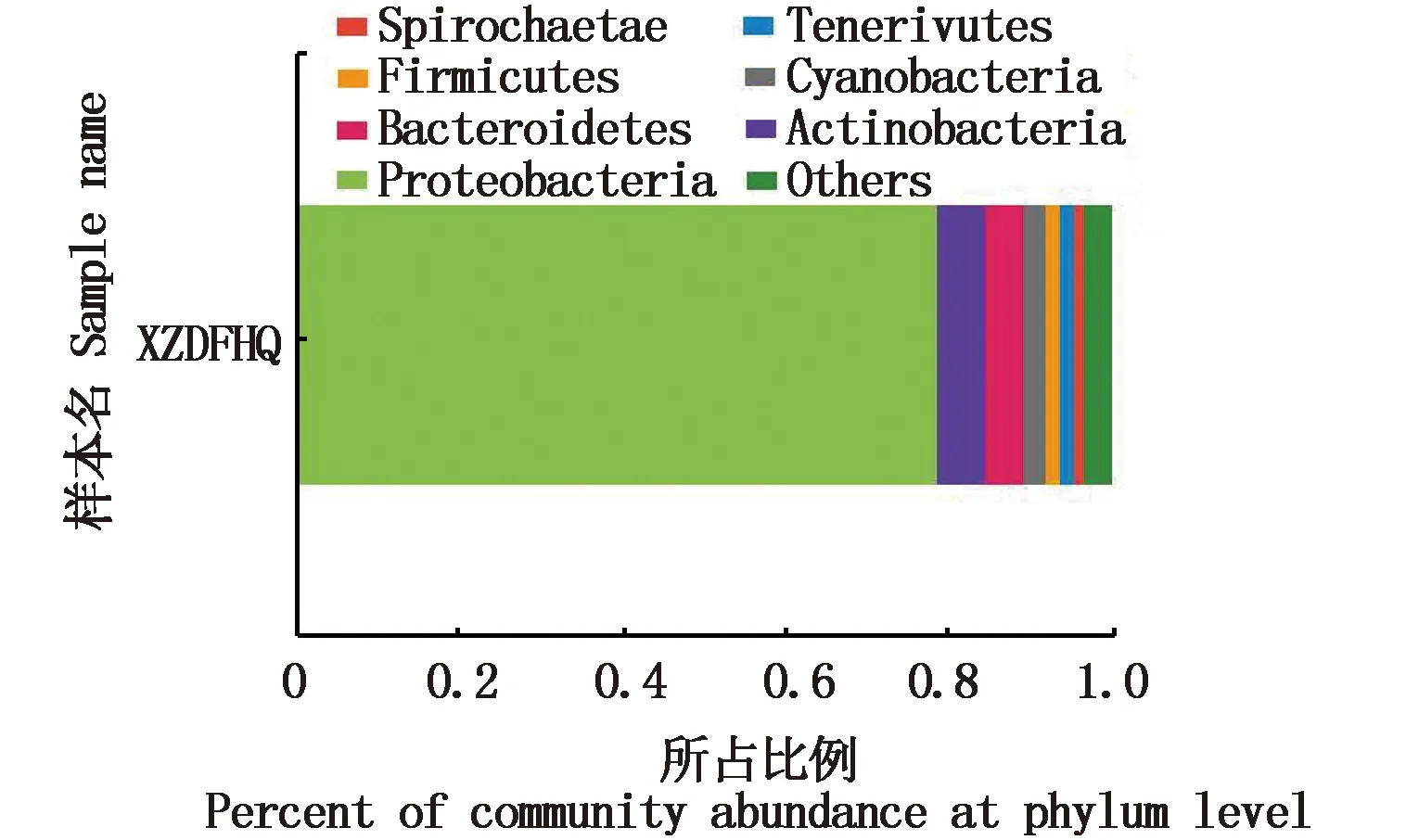
图3 星座短腹海鞘共附生菌门水平分布图Fig.3 The phylum level distribution of Aplidium constellatum associated bacteria
从属的水平来看(图3),星座短腹海鞘共附生微生物分属于197个属,优势属有10个,分别为Ruegeria(60.83%)、SAR116_clade科下未知属(3.27%)、Rhodococcus(2.40%)、Cyanobacteria纲下未知属(2.26%)、Candidatus_Hepatoplasma(1.76%)、PeM15目下未知属(1.61%)、Spirochaetaceae科下未分类属(1.33%)、Thalassobius(1.26%)、Rhodobacteraceae科下未分类属(1.23%)、Rhodospirillaceae科下未分类属(1.08%)。
2.4 星座短腹海鞘可培养微生物类群的多样性
从星座短腹海鞘中共分离获得15株可培养共附生菌,对这15株可培养共附生菌16S rRNA基因进行PCR扩增及测序,所得序列与GenBank数据库进行同源性比对(表3),结果表明,15株可培养共附生菌分属于8个属,分别为弧菌属Vibrio(7株)、希瓦氏菌属Shewanella(2株),其余的属Pseudomonas、Pseudovibrio、Thalassobius、Exiguobacterium、Flavobacterium、Thalassospira各1株。从相似度来看,15株可培养共生菌与GenBank数据库中菌种相似度在98%以上。基于16S rRNA基因的系统发育分析见图 5。

表2 多样性指数表Tab.2 Table of alpha diversity indices

图4 星座短腹海鞘共附生菌属平分布图Fig.4 The genus level distribution of Aplidium constellatum associated bacteria

属 Genus菌株编号 Strain No.相近模式菌株 Similar strain相似度/% SimilarityVibrio XZDF-10Vibrio tasmaniensis LMG 21574T (AJ514912)99.24XZDF-11Vibrio atlanticus Vb 11.11T (EF599163)99.17XZDF-16Vibrio neocaledonicus NC 470T (JQ934828)99.22XZDF-20Vibrio hyugaensis 090810aT (LC004912)99.86XZDF-24Vibrio crassostreae LGP 7T (CCJW01000022)99.31XZDF-25Vibrio chagasii R-3712T (AJ316199)98.80XZDF-7Vibrio cyclitrophicus P-2P44T (U57919)98.34ShewanellaXZDF-14Shewanella loihica PV-4T (CP000606)98.07XZDF-23Shewanella aquimarina SW-120T (AY485225)99.10PseudomonasXZDF-8-1Pseudomonas zhaodongensis NEAU-ST5-21T(JQ762275)98.93PseudovibrioXZDF-8-2Pseudovibrio ascidiaceicola DSM 16392T (AB175663)99.57ThalassobiusXZDF-12Thalassobius aestuarii DSM 15283T (AY442178)99.12ExiguobacteriumXZDF-13Exiguobacterium aestuarii TF-16T (AY594264)98.90FlavobacteriumXZDF-15Flavobacterium rakeshii FCS-5T (JF830803)99.51ThalassospiraXZDF-26Thalassospira xiamenensis M-5T (CP004388)98.71
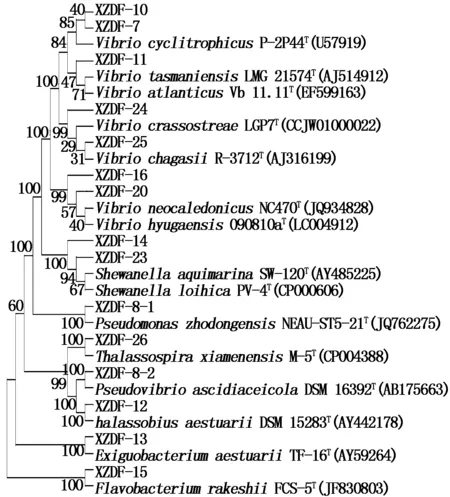
图5 基于16S rRNA序列构建的星座短腹海鞘可培养共附生菌的系统发育进化树Fig.5 Phylogenetic tree based on neighbor-joining analysis of 16S rRNA sequences
3 讨论
本文通过高通量测序技术首次解析了星座短腹海鞘共附生微生物种类、丰度及多样性信息,此结果对于深化对海鞘共附生多样性的认识,指导其可培养共附生微生物的分离具有重要意义。
从研究结果发现,通过纯培养技术所得到的可培养共附生微生物种属数量较少,可能是由于本实验只使用了2216E一种海洋培养基的缘故。以往的研究结果表明,使用不同的培养基对于微生物的多样性发现有较大影响。因此在后续研究中应当增加分离培养基的种类,以提高共附生微生物的分离效果[8]。
研究结果显示,从星座短腹海鞘中分离出的细菌中有弧菌、希瓦氏菌这两种致病菌,这可能与本实验采集海鞘的地点为当地主要海水养殖区有关,样本可能在生长过程中受到养殖水产病害的污染。虽然这两种致病菌并不属于优势菌株,但仍然提示在后期的海鞘开发利用过程中,应注意原料来源;在原料采集或共附生微生物的规模化培养中,要注意监测水产致病菌的存在情况,避免可能带来的安全隐患。
以往对海鞘共附生微生物的研究多采用纯培养方式,本实验采用了高通量测序方法。这主要是由于海洋中有许多微生物是不可培养的,只靠纯培养技术不能全面了解微生物多样性,而Illumina MiSeq高通量测序技术打破了纯培养技术的限制,使得人们可以更快捷、全面了解微生物多样性。因此,在以后的共附生微生物多样性的研究中,结合高通量测序技术与纯培养方法来探讨海洋微生物多样性,将更有助于深化人们对微生物资源的认识。
参考文献:
[1] 郑成兴. 中国沿海海鞘的物种多样性[J].生物多样性, 1995, 3(4): 201-205.
ZHENG C X. Species diversity of Ascidian in the coastal China Seas[J]. Chinese Biodiversity, 1995, 3(4): 201-205.
[2] 黄宗国. 中国海洋生物种类与分布:增订版[M]. 北京:海洋出版社,2008: 727.
HUANG Z G. Species and distribution of marine organisms in China: enlarged edition[M]. Beijing:Ocean Press, 2008: 727.
[3] EVA Z, MARIA J O, JAVIER S. Natural products chemistry in marine ascidians of the genusAplidium[J]. Mini-Reviews in Organic Chemistry, 2005, 2(4): 389.
[4] HAN Q H, FAN C Q, LU Y N,etal. Chemical constituents from the ascidianAplidiumconstellatum[J]. Biochemical Systematics and Ecology, 2013(48): 6-8.
[5] 张建同, 陆亚男, 杨 桥, 等. 星座短腹海鞘脂溶性成分的提取工艺[J]. 海洋渔业, 2015, 37(3): 264-269.
ZHANG J T, LU Y N,YANG Q,etal. On the extraction of liposoluble components fromAplidiumconstellatum[J]. Marine Fisheries, 2015, 37(3): 264-269.
[6] 樊成奇, 赵树明, 田晓清, 等. 两种复海鞘油脂中的脂肪酸成分分析[J]. 海洋渔业, 2012, 34(1): 117-120.
FAN C Q, ZHAO S M, TIAN X Q,etal. On fatty acids in oils from two colonial ascidians[J]. Marine Fisheries, 2012, 34(1): 117-120.
[7] 崔 迪. 威海海域海鞘来源可培养细菌多样性和活性菌株筛选[D]. 哈尔滨:哈尔滨工业大学, 2014.
CUI D. Diversity of bacteria associated with ascidians from Weihai and screening of strains with bioactivities[D]. Harbin: Harbin Institute of Technology, 2014.
[8] 符常明. 两种海鞘相关可培养细菌多样性和抗肿瘤活性筛选[D]. 哈尔滨: 哈尔滨工业大学, 2015.
CHANG F M. Bacterial diversity associated with two species of ascidians and screening of cytotoxic activity of the strains[D]. Harbin: Harbin Institute of Technology, 2015.
[9] JIANG X T, PENG X, DENG G H,etal. Illumina sequencing of 16S rRNA tag revealed spatial variations of bacterial communities in a mangrove wetland[J]. Microbial Ecology, 2013(66): 96-104.
[10] 丁 慧, 王能飞, 臧家业, 等. 北极黄河站地区不同基底中真菌的分离培养及初步鉴定[J]. 海洋学报(中文版), 2014(10): 124-130.
DING H, WANG N F, ZANG J Y,etal. Isolation and preliminary identification of fungi in different substrate from the Arctic Yellow River Station[J]. Acta Oceanologica Sinica, 2014(10): 124-130.
[11] BRUNATI M, LUIS R J, SPONGA F,etal. Diversity and pharmaceutical screening of fungi from benthic mats of Antarctic lakes[J]. Marine Genomics, 2009(1): 43-50.

