梅花鹿茸生长中心软骨组织差异基因表达分析
刘美欣 赵 雨 张 梅 刘宇昕 胡耀中 幺宝金*
(1.长春中医药大学中医药与生物工程研究开发中心,长春,130117;2.长春中医药大学创新实践中心,长春,130117)
鹿茸为鹿科动物未骨化密生茸毛的幼角,鹿茸惊人的生长速度一直是本领域研究的热点,在快速生长期(生茸后 60 d),鹿茸的生长速度可达 2 cm/d[1-4]。鹿茸的生长主要由鹿茸顶端生长中心驱动和控制,通过软骨内骨化过程实现,生长中心主要包括茸皮、间充质和软骨组织[5-7]。因此,鹿茸可以作为研究软骨生长和发育机制的良好模型。鹿茸生长中心软骨组织中富含大量血管组织,与正常的软骨组织存在巨大差异,这与鹿茸具有惊人的生长速度并且可以修复再生必然存在联系[8-10]。因此,研究不同生长时期鹿茸软骨组织基因表达变化,对于揭示鹿茸快速生长机制具有重要意义。同时,对于软骨组织损伤修复和骨关节退行性疾病的相关研究具有重要的指导作用。
近年来,随着高通量测序技术的不断发展,以转录组学为代表的组学研究在基因表达分析研究中发挥重要作用。转录组指特定组织或细胞在某个发育阶段或某种特定生理病理条件下,转录出来的全部RNA的总和。转录组测序(RNA-seq)是指利用高通量测序技术对RNA进行测序,通过生物信息学分析方法,研究特定组织或细胞的全部转录本情况,揭示其基因表达水平与变化,进而揭示其具体分子作用机制。与传统测序方法如Sanger法测序、基因芯片等相比较,具有样品用量少、分辨率和准确度高、价格低和应用范围广等优势[11-15]。同时,可以实现对无参考基因组物种进行从头测序[16-19]。本研究采用 RNA-seq技术对快速生长期和骨化期鹿茸生长中心软骨组织进行转录组测序,通过Trinity软件[20]对测序结果进行从头组装和拼接,通过生物信息学分析方法筛选不同生长时期鹿茸软骨组织差异表达基因,这些研究结果对于揭示鹿茸快速生长机制具有重要意义。
1 材料与方法
1.1 材料
随机选取4岁龄健康的东北梅花鹿(Cervus nippon hortulorum)6只,分别于生茸后60 d(快速生长期)和90 d(骨化期)时采集两侧的鹿茸顶端生长组织(距离顶端长度5 cm)。按照Li等[7]的实验方法分离鹿茸顶端软骨组织,置于纯化水中反复漂洗后切成小块,迅速置于液氮罐中保存。
1.2 方法
1.2.1 鹿茸软骨组织总RNA提取制备
采用TRIzol(Invitrogen,USA)法从鹿茸生长中心软骨组织中提取总RNA,通过Bioanalyzer 2100系统(Agilent Technologies,USA)来评估RNA完整性。
1.2.2 测序文库构建
采用 TruSeq Stranded mRNA试剂盒(Illumina,USA)进行测序文库构建。具体方法为:采用oligo(dT)偶联磁珠(Life technologies,USA)从总RNA中纯化富集mRNA,并进行mRNA片段化;经逆转录酶和随机引物合成双链cDNA,经核糖核酸酶处理除去剩余mRNA;经末端修复和接头连接后,通过PCR扩增获得最终的测序文库。
1.2.3 文库测序和生物信息学分析
在Illumina Hiseq 2000平台(Illumina,USA)上进行文库测序。采用Illumina HCS 1.1软件进行数据转换,去除低质量序列和载体序列。采用Trinity软件进行从头组装。通过BLASTX比对程序,以E值<10-5为评价标准,将测序组装数据与nr、Swiss-Prot蛋白数据库进行比对。采用RPKM法[21]进行基因表达量分析,采用DEGseq软件包[22]对快速生长期和骨化期软骨组织进行差异基因表达分析,评价标准为:差异表达倍数在2倍以上(即log2[Fold change]≥1或≤-1,Fold change=骨化期 RPKM/快速生长期RPKM),且假发现率(FDR)≤0.001。
2 结果
2.1 测序和组装
通过转录组测序,从快速生长期和骨化期鹿茸软骨组织中分别获得4.47和4.45 Gb的测序数据。其中,raw reads分别为45423156和45422448条,去除载体序列和低质量序列后,分别获得44715694和44535200条clean reads,测序质量值Q20值分别为98.44和98.05。通过 Trinity软件组装,分别获得39377和37331条组装序列(Unigenes),平均长度分别为1138和1143 nt。Unigenes长度分布见图1。
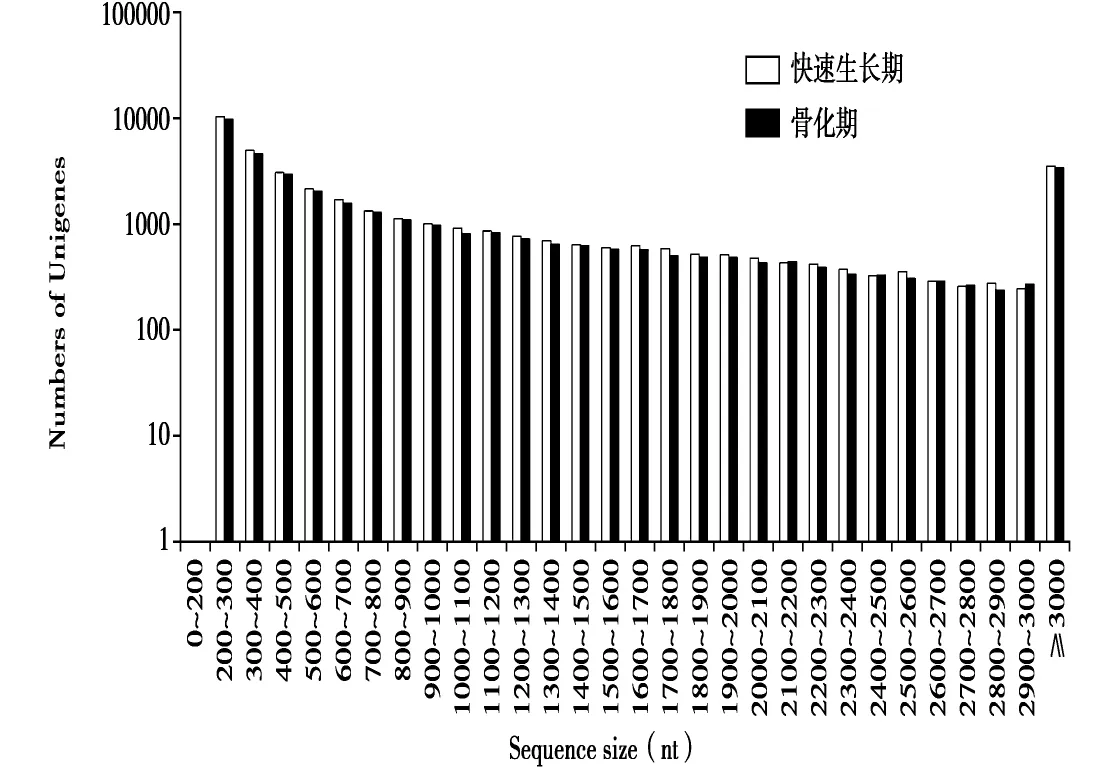
图1 快速生长期和骨化期鹿茸软骨组织Unigenes长度分布Fig.1 Unigenes length distribution of antler cartilage tissue at rapid growth and ossification stages
2.2 蛋白数据库比对及差异基因表达分析
通过BLASTX比对程序,将Unigenes与nr、Swiss-Prot蛋白数据库进行比对,以E值<10-5为评价标准,同时与2个数据库比对上的Unigenes共有20551条。针对这些Unigenes,采用RPKM法进行基因表达量分析,采用DEG-seq软件包进行差异基因表达分析,共筛选出4422条差异表达基因(log2[Fold change]≥1或≤-1;FDR≤0.001)。与快速生长期比较,骨化期显著上调的差异表达基因有2493条,显著下调的差异表达基因有1929条。我们针对这些差异表达基因进行进一步的筛选,找出与软骨生长和骨化相关的差异表达基因,这些基因主要包括生长因子、转录因子和胶原类成分。
2.2.1 生长因子(growth factor)类差异表达基因
在快速生长期和骨化期鹿茸软骨组织差异表达基因中,共筛选出7种生长因子(表1)。其中,与快速生长期比较,骨化期鹿茸软骨组织中表达量显著下调的生长因子有6种,分别为血管内皮生长因子VEGFD、表皮生长因子样蛋白EGFL6、胰岛素样生长因子IGF1、肝细胞生长因子HGF、成纤维细胞生长因子FGF7和FGF11;血管内皮生长因子VEGF-C表达量显著上调。
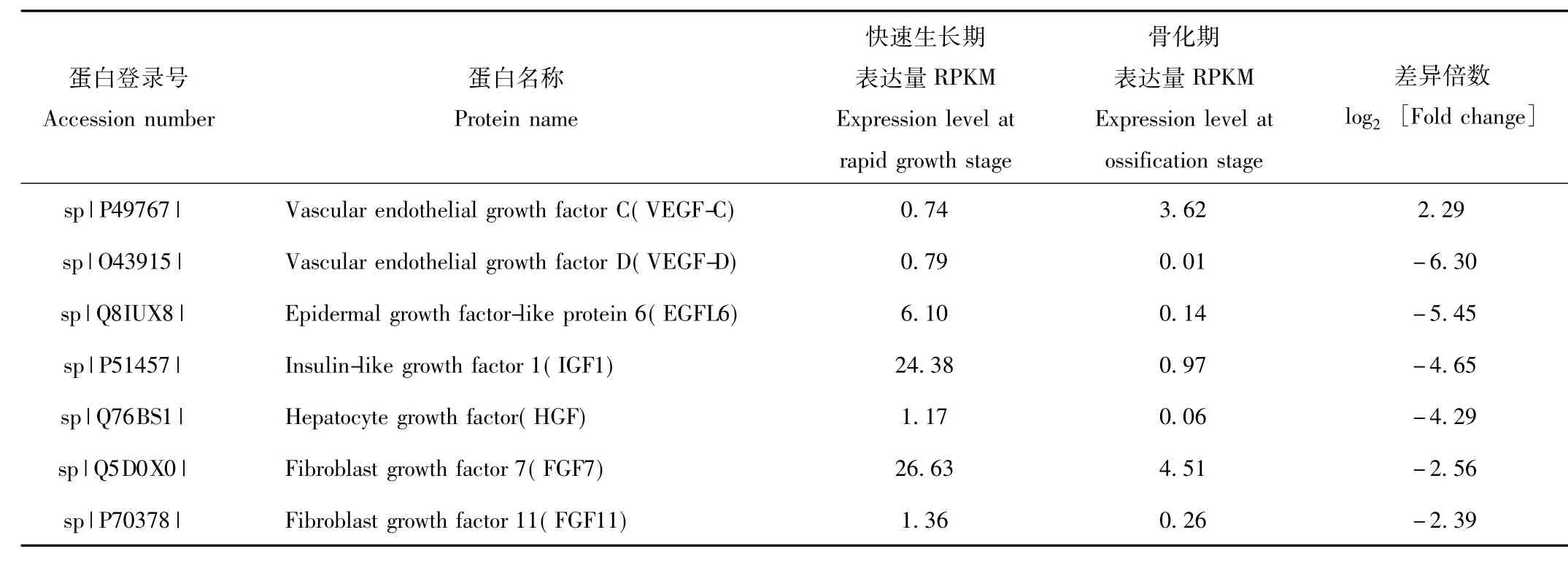
表1 不同生长时期鹿茸生长中心软骨组织生长因子类差异表达基因Tab.1 Differential growth factors of antler growth center cartilage at different growth stages
2.2.2 转录因子(transcription factor)类差异表达基因
在快速生长期和骨化期鹿茸软骨组织差异表达基因中,共筛选出12种转录因子(表2)。其中,与快速生长期比较,骨化期鹿茸软骨组织中表达量显著下调的转录因子有 7种,分别为转录因子 ATF6β、SOX12、ELF2、ELF4、RUNX1、MAFK和 TCF4;表达量显著上调的转录因子有5种,分别为转录因子SOX9、GTF3A、SOX8、ATF3和 MAFF。
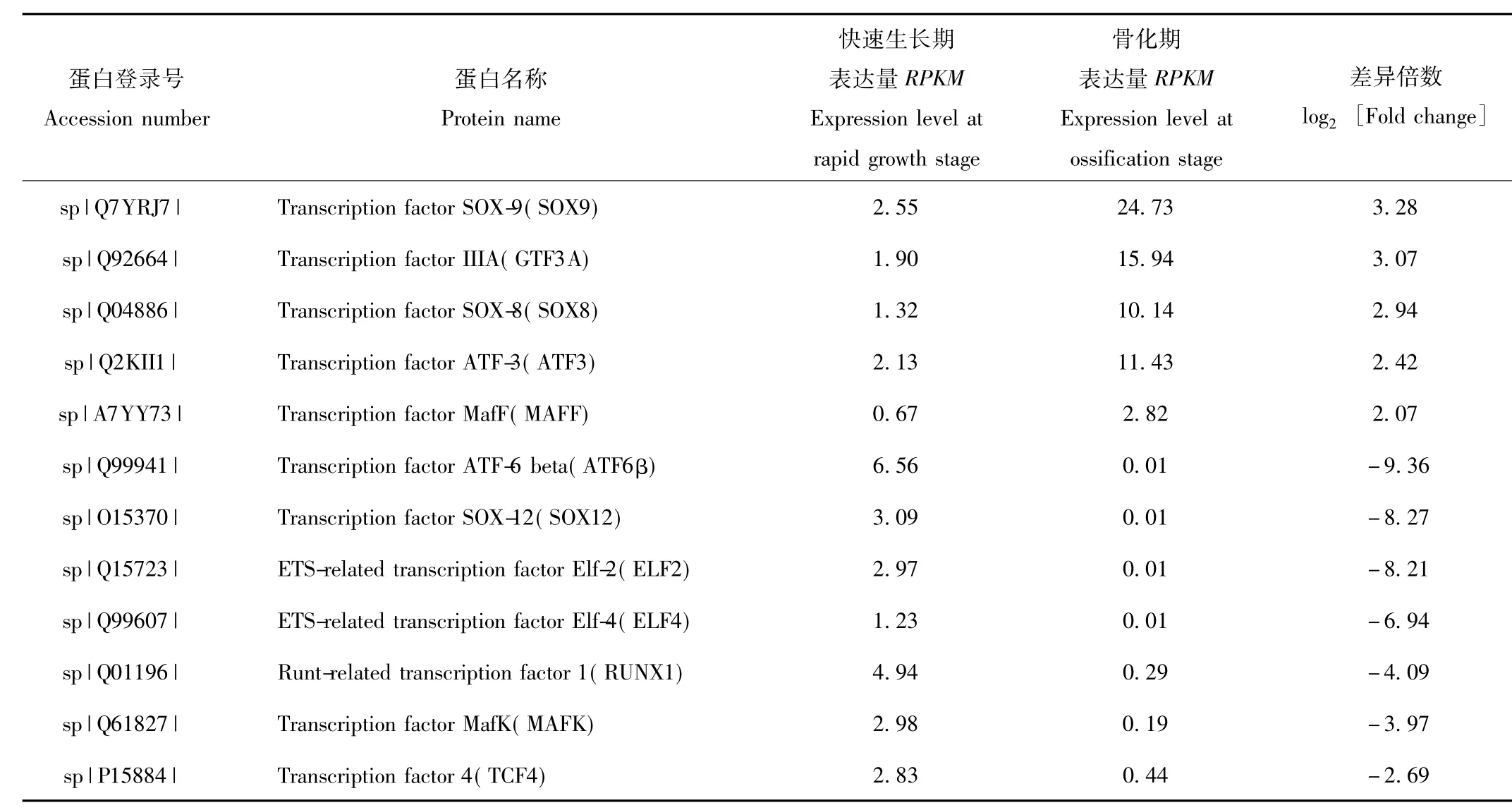
表2 不同生长时期鹿茸生长中心软骨组织转录因子类差异表达基因Tab.2 Differential transcription factors of antler growth center cartilage at different growth stages
2.2.3 胶原(collagen)类差异表达基因
在快速生长期和骨化期鹿茸软骨组织差异表达基因中,共筛选出8种类型胶原(表3)。其中,与快速生长期比较,骨化期鹿茸软骨组织中表达量显著下调的胶原有3种,分别为胶原COL16A1、COL8A1和COL14A1;表达量显著上调的胶原有5种,分别为胶原 COL10A1、COL27A1、COL9A1、COL2A1 和COL25A1。
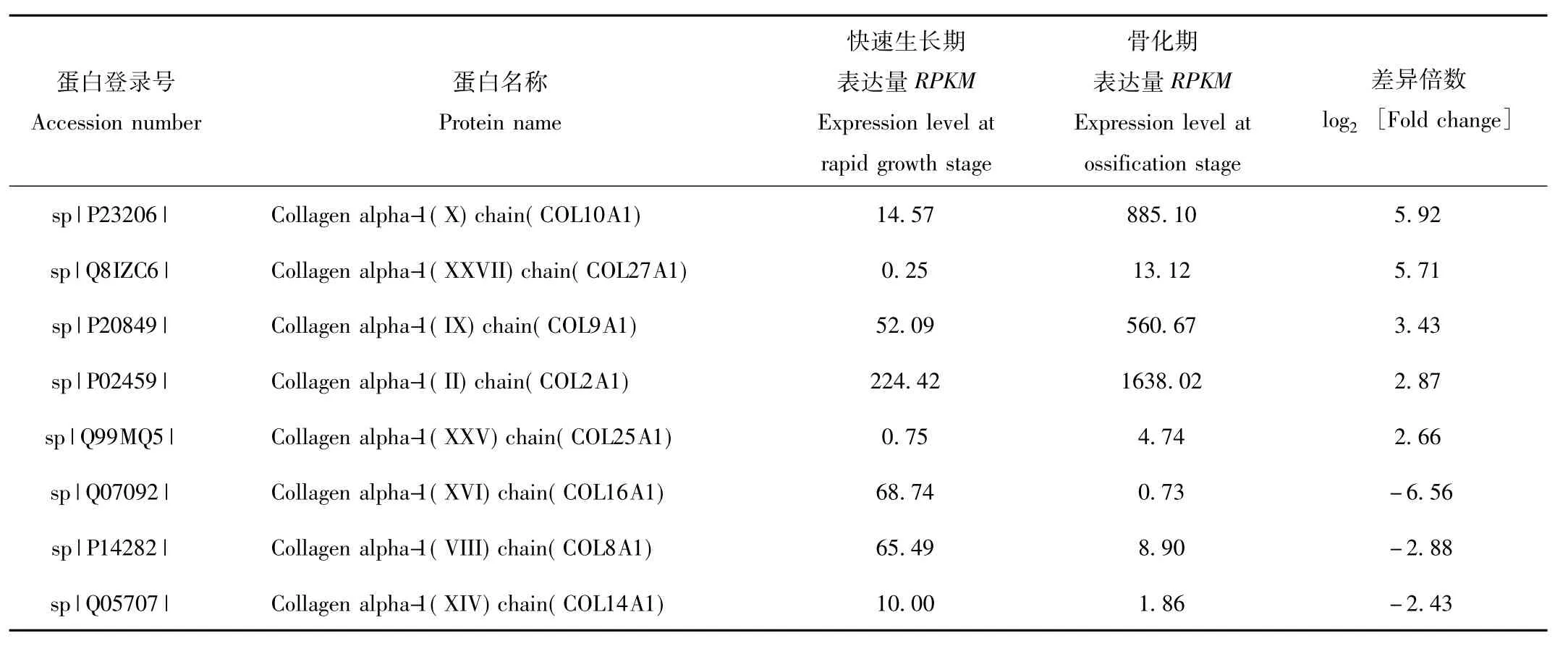
表3 不同生长时期鹿茸生长中心软骨组织胶原类差异表达基因Tab.3 Differential collagens of antler growth center cartilage at different growth stages
3 讨论
鹿茸的快速生长机制一直是本领域的研究热点。要系统和全面地揭示这一独特的生长机制,就必须对鹿茸生长过程中的基因表达变化进行深入分析。基于Illumina HiSeqTM2000高通量测序平台的转录组测序技术具有快速、高效、低成本等特点,可以对任意物种的组织或细胞的全转录本进行检测,提供全面的转录组信息,尤其适合于研究缺少基因组信息的非模式生物的转录组情况。本课题组在前期研究工作中针对快速生长期和骨化期鹿茸顶端生长中心的转录组进行较为深入的分析,筛选出多种调控鹿茸快速生长和骨化的差异表达基因[23-25]。然而,这些研究结果无法反映鹿茸生长过程中生长中心的软骨组织的基因表达变化。本研究针对快速生长期和骨化期鹿茸生长中心软骨组织转录组进行深度测序,采用生物信息学方法对测序结果进行组装、拼接、比对和差异基因表达分析,筛选出7种差异表达生长因子,12种差异表达转录因子和8种差异表达胶原。
在快速生长期和骨化期鹿茸转录组差异表达生长因子中,血管内皮生长因子VEGF-D、表皮生长因子样蛋白EGFL6、胰岛素样生长因子IGF1、肝细胞生长因子HGF、成纤维细胞生长因子FGF7和FGF11在快速生长期表达量较高,在骨化期表达量显著下调,而血管内皮生长因子VEGF-C在快速生长期表达量较低,在骨化期表达量显著上调。这些研究结果表明鹿茸的快速生长受到这些生长因子的协同调控作用。VEGF-D和VEGF-C是2种血管内皮细胞因子亚型,具有促进血管和淋巴管形成等功能[26]。表皮生长因子样蛋白EGFL6的功能与血管内皮生长因子相似,具有促血管生成作用,尤其在肿瘤组织中表达量较高[27]。这些研究结果提示我们这3种生长因子对鹿茸生长过程中软骨组织中血管的生成具有重要的调控作用。胰岛素样生长因子IGF1是一种多功能生长因子,对于细胞的存活、增殖和分化具有重要调节作用。IGF1软骨特异性基因敲除小鼠出现软骨细胞增殖减少,分化和肥大延迟,细胞凋亡增加,软骨内骨化进程迟缓等现象[28]。肝细胞生长因子HGF、成纤维细胞生长因子FGF7和FGF11三种生长因子在组织损伤修复和再生过程中起着关键的调节作用[29-31]。这些研究结果表明鹿茸快速生长受到 IGF1、HGF、VEGF和EGF6等多种生长因子的协同调控。
在快速生长期和骨化期鹿茸转录组差异表达转录因子中,SOX9、GTF3A、SOX8、ATF3和MAFF的表达量在骨化期表达量显著上调,而ATF6β、SOX12、ELF2、ELF4、RUNX1、MAFK和TCF4的表达量在骨化期表达量显著下调。这些转录因子主要集中为SOX家族、ATF家族和ELF家族。其中,SOX家族成员包括SOX8、SOX9和SOX12,这3种转录因子在胚胎发育、神经系统发育、软骨及多种组织器官形成和发育过程中发挥重要作用[32]。ATF家族成员包括 ATF3、ATF6β,这2个转录因子对于应激条件下软骨细胞存活和凋亡具有重要调控作用[33]。除此之外,RUNX1也是软骨生长过程中重要的调控因子[34]。这些研究结果表明鹿茸快速生长受到SOX8、SOX9、SOX12、ATF3、ATF6β和RUNX1等多种转录因子的协同调控。
在不同生长时期鹿茸软骨组织转录组数据中,我们也发现了许多表达量存在显著差异的胶原类成分,与快速生长期比较,骨化期鹿茸软骨组织中表达量显著下调的胶原有 3种,分别为胶原 COL16A1、COL8A1和COL14A1;表达量显著上调的胶原有5种,分别为胶原 COL10A1、COL27A1、COL9A1、COL2A1和 COL25A1。其中,COL10A1、COL9A1和COL2A1广泛存在于软骨组织中,是重要的软骨细胞外基质成分,在软骨形成和生长发育过程中发挥重要作用[35]。这些研究结果表明鹿茸快速生长和骨化过程中胶原表达量发生巨大改变,有关这些胶原基因表达调控机制仍然有待于进一步研究。
4 结论
本研究采用Illumina高通量测序技术,对快速生长期和骨化期东北梅花鹿茸生长中心软骨组织进行转录组测序和生物信息学分析。通过差异基因表达分析筛选出7种差异表达生长因子,12种差异表达转录因子和8种差异表达胶原。这些差异表达基因对于鹿茸的快速生长和骨化具有重要的调节作用,本研究结果为鹿茸快速生长及骨化机制研究提供理论基础。
[1] Chu W,Zhao H,Li J,et al.Custom-built tools for the study of deer antler biology[J].Frontiers in Bioscience(Landmark Ed),2017,22:1622-1633.
[2] Sui Zhigang,Weng Yejing,Zhao Qun,et al.Ionic liquid-based method for direct proteome characterization of velvet antler cartilage[J].Talanta,2016,161:541-546.
[3] Park J,Jeon B,Kang S,et al.Study on the changes in enzyme and insulin-like growth factor-1 concentrations in blood serum and growth characteristics of velvet antler during the antler growth period in sika deer(Cervus nippon) [J].Asian-Australasian Journal of Animal Sciences,2015,28(9):1303 -1308.
[4] Hu Wei,Meng Xingyu,Lu Tiancheng,et al.MicroRNA-1 inhibits the proliferation of Chinese sika deer-derived cartilage cells by binding to the 3'-untranslated region of IGF-1[J].Molecular Medicine Reports,2013,8(2):523 -528.
[5] Price J S,Allen S,Faucheux C,et al.Deer antlers:a zoological curiosity or the key to understanding organ regeneration in mammals[J].Journal of Anatomy,2005,207(5):603 -618.
[6] Price J,Allen S.Exploring the mechanisms regulating regeneration of deer antlers[J].Philosophical Transactions of the Royal Society B:Biological Sciences,2004,359(1445):809 -822.
[7] Li C,Clark D E,Lord E A,et al.Sampling technique to discriminate the different tissue layers of growing antler tips for gene discovery[J].The Anatomical Record,2002,268(2):125-130.
[8] Clark D E,Li C,Wang W,et al.Vascular localization and proliferation in the growing tip of the deer antler[J].The Anatomical Record,2006,288(9):973-981.
[9] Clark D E,Lord E A,Suttie J M.Expression of VEGF and pleiotrophin in deer antler[J].The Anatomical Record,2006,288A(12):1281-1293.
[10] Lai A K W,Hou W L,Verdon D J,et al.The distribution of the growth factors FGF-2 and VEGF,and their receptors,in growing red deer antler[J].Tissue and Cell,2007,39(1):35 -46.
[11] Hrdlickova R,Toloue M,Tian B.RNA-Seq methods for transcriptome analysis[J].Wiley Interdisciplinary Reviews:RNA,2017,8(1):e 1364.
[12] Kukurba K R,Montgomery S B.RNA sequencing and analysis[J].Cold Spring Harbor Protocols,2015(11):951 -969.
[13] Han Y,Gao S,Muegge K,et al.Advanced applications of RNA sequencing and challenges[J].Bioinformatics and Biology Insights,2015,9(S 1):29 -46.
[14] Hoeijmakers W A M,Bártfai R,Stunnenberg H G.Transcriptome analysis using RNA-Seq[M].Malaria Humana Press,2012:221-239.
[15] Mutz K O,Heilkenbrinker A,Lonne M,et al.Transcriptome analysis using next-generation sequencing[J].Current Opinion in Biotechnology,2013,24(1):22 -30.
[16] Lee J H.De Novo gene expression Reconstruction in space[J].Trends in Molecular Medicine,2017,23(7):583-593.
[17] Moreton J,Izquierdo A,Emes R D.Assembly,assessment,and availability of de novo generated eukaryotic aranscriptomes[J].Frontiers in Genetics,2016,6:361.
[18] Unamba C I N,Nag A,Sharma R K.Next generation sequencing technologies:the doorway to the unexplored genomics of non-model plants[J].Frontiers in Plant Science,2015,6:1074.
[19] Góngora-Castillo E,Buell C R.Bioinformatics challenges in de novo transcriptome assembly using short read sequences in the absence of a reference genome sequence [J].Natural Product Reports,2013,30(4):490-500.
[20] Grabherr M G,Haas B J,Yassour M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome [J].Nature Biotechnology,2011,29(7):644-652.
[21] Mortazavi A,Williams B A,Mccue K,et al.Mapping and quantifying mammalian transcriptomes by RNA-Seq [J].Nature Methods,2008,5(7):621-628.
[22] Wang Likun,Feng Zhixing,Wang Xi,et al.DEGseq:an R package for identifying differentially expressed genes from RNA-seq data[J].Bioinformatics,2010,26(1):136-138.
[23] Zhao Yu,Yao Baojin,Zhang Mei,et al.Comparative analysis of differentially expressed genes in sika deer antler at different stages[J].Molecular Biology Reports,2013,40(2):1665 -1676.
[24] Yao Baojin,Zhao Yu,Wang Qun,et al.De novo characterization of the antler tip of Chinese sika deer transcriptome and analysis of gene expression related to rapid growth[J].Molecular and Cellular Biochemistry,2012,364(1/2):93 -100.
[25] Yao Baojin,Zhao Yu,Zhang Haishan,et al.Sequencing and de novo analysis of the Chinese sika deer antler-tip transcriptome during the ossification stage using Illumina RNA-Seq technology[J].Biotechnology Letters,2012,34(5):813 -822.
[26] Hu K,Olsen B R.The roles of vascular endothelial growth factor in bone repair and regeneration[J].Bone,2016,91:30-38.
[27] Wang Xuanchun,Gong Ye,Wang Daijun,et al.Analysis of gene expression profiling in meningioma:deregulated signaling pathways associated with meningioma and EGFL6 overexpression in benign meningioma tissue and serum [J].PLoS One,2012,7(12):e52707.
[28] Wang Yongmei,Bikle D D,Chang Wenhan.Autocrine and paracrine actions of IGF-I signaling in skeletal development[J].Bone Research,2013,1(3):249 -259.
[29] Miyagi H,Thomasy S M,Russell P,et al.The role of hepatocyte growth factor in corneal wound healing[J].Experimental Eye Research,2018,166:49-55.
[30] Seo H S,Lee D J,Chung J H,et al.Hominis placenta facilitates hair re-growth by upregulating cellular proliferation and expression of fibroblast growth factor-7[J].BMC Complementary and Alternative Medicine,2016,16(1):187.
[31] Yang J,Kim W J,Jun H O,et al.Hypoxia-induced fibroblast growth factor 11 stimulates capillary-like endothelial tube formation[J].Oncology Reports,2015,34(5):2745 -2751.
[32] Kamachi Y,Kondoh H.Sox proteins:regulators of cell fate specification and differentiation [J].Development,2013,140(20):4129-4144.
[33] Yuan Xiaoliang,Liu Haiqing,Li Linfu,et al.The roles of endoplasmic reticulum stress in the pathophysiological development of cartilage and chondrocytes [J].CurrentPharmaceuticalDesign,2017,23(11):1693-1704.
[34] LeBlanc K T,Walcott M E,Gaur T A,et al.Runx1 activities in superficial zone chondrocytes,osteoarthritic chondrocyte clones and response to mechanical loading[J].Journal of Cellular Physiology,2015,230(2):440-448.
[35] Stocco E,Barbon S,Radossi P,et al.Autologous chondrocytes as a novel source for neo-chondrogenesis in haemophiliacs[J].Cell and Tissue Research,2016,366(1):51-61.

