傅立叶变换红外光谱技术对阪崎肠杆菌及奇异变形杆菌的分类鉴定
张占林,乔勇升,王萍
(泰州市食品药品检验所,江苏泰州225300)
阪崎肠杆菌(Enterobacter sakazakii)是根据日本微生物学家Riichi Sakazakii名字命名[1]的一种革兰阴性无芽孢杆菌,它寄生于人和动物肠道内,有周生鞭毛、能运动。2008年,Iversen等[2]根据扩增片段长度多态性、16S rRNA全序列、DNA-DNA杂交遗传特征和Biotype100、Biologe细菌鉴定系统的表型特征等综合分析,阪崎肠杆菌被重新分为肠杆菌科1个新属,即克罗诺杆菌属。阪崎肠杆菌能引起新生婴幼儿菌血症、小肠结肠炎和脑膜炎等严重疾病,在一些新生儿阪崎肠杆菌感染事件的调查中发现婴幼儿配方奶粉为其主要感染源[3-4],奶粉中3 CFU/100 g的阪崎肠杆菌污染就能导致感染的发生;此外,还可引起体弱者、孕妇和老年人局部感染和菌血症等。而传统检测法中常有某些肠道细菌如变形杆菌因含有与阪崎肠杆菌相同的α-D-葡萄糖苷酶,或是奶粉中复杂的成分,影响了酶与底物的反应[5],致使阪崎肠杆菌在显色培养基上出现假阳性和假阴性的现象。因此,近年来阪崎肠杆菌的检测给食品安全防治工作带来严峻的挑战[6-7]。
目前常用的阪崎肠杆菌检测方法有生理生化鉴定法和分子生物学法[8-9],基于FT-IR技术高分辨率、高指纹、快速、低成本等优势,我们用FT-IR技术对阪崎肠杆菌标准菌株、能力验证样品中分离得到的阪崎肠杆菌和干扰菌奇异变形杆菌进行了研究,从扫描得到的光谱信息中选取特征光谱区域,运用化学计量学方法分析各菌株间的异同,从而对它们进行更好的分类。
1 材料与方法
1.1 菌株与试剂
标准菌株阪崎肠杆菌CGMCC1.6765:中国普通微生物菌种保藏管理中心;样品阪崎肠杆菌和奇异变形杆菌:江苏泰州市食品药品检验所微生物检验室参加NIFDC-PT-065能力验证中分离保存且经过VITEK 2 Compact鉴定的菌株;营养肉汤和平板计数琼脂:北京陆桥技术有限责任公司;氯化钠(分析纯)、溴化钾(光谱纯):国药集团化学试剂有限公司。
1.2 仪器设备
傅立叶变换红外光谱仪(Thermo is10):美国赛默飞世尔科技公司;冷冻台式高速离心机(Allegra 64R):美国贝克曼库尔特公司;水浴恒温振荡器(SUA-C):常州国华电器有限公司;真空恒温干燥箱(VD23):德国宾德公司;紫外-可见分光光度计(UV-2550PC):日本岛津公司;隔水式恒温培养箱(GHP-9270):上海慧泰仪器制造有限公司。
1.3 操作方法
1.3.1 菌株复苏及处理
从3株菌株保藏管中各挑取一环分别划线接种于平板计数琼脂平板上,36℃培养过夜,活化菌株。分别挑取已活化菌株的单菌落于装10 mL营养肉汤试管中,置36℃隔水式恒温培养箱培养18 h,制成储备培养物。各取一环试管中培养物分别接种于装有100 mL营养肉汤三角瓶中,36℃,160 r/min震荡培养18 h。取10 mL菌悬液并用适量营养肉汤稀释,使3株菌的菌液浓度在 3.5×108CFU/mL~3.8×108CFU/mL 之间;此后将浓度一致的菌悬液进行离心沉淀,离心条件设置为20 ℃,8 000 r/min,5 min;弃去液体,收集菌体细胞,用0.9%的NaCl悬浮,同样条件离心以达到洗涤效果;以同样方式洗涤3次后,置于真空恒温干燥箱中干燥。每个菌株进行8次相同试验,每次设置3个平行重复。为了减少误差,确保每次试验的培养条件及细菌浓度尽可能统一。
1.3.2 菌体光谱信息采集
将干燥的菌体细胞研磨成均匀的菌粉,为减少微生物量的变化对谱图的影响,以3∶100(质量比)加入溴化钾,与菌粉混合研磨,利用压片机制备薄片。将压制好的均匀透明薄片置于傅立叶变换红外光谱仪窗口采集光谱信息,采集条件设置为:自动扣除大气背景,测量范围为 4 000 cm-1~400 cm-1,分辨率为 4 cm-1,扫描32次。
1.3.3 谱图处理及分析
用OMNIC 9.0软件先将各谱图进行透光率与吸光度转化,为了避免原始光谱基线偏离和由于样品量不同而带来的光谱数据偏差,先对光谱进行自动基线校正,纵坐标归一化处理;再对归一化后的谱图进行比较筛选,剔除有较大偏离的异常谱图,然后对筛好的光谱图求平均光谱图;最后将筛选出的光谱图进行CSV数据格式转化,以方便后续用PAST软件对特定波数范围内的光谱数据进行PCA和HCA分析。PAST软件操作方法及原理详见黄冰等[10]文献报道。
2 结果与分析
2.1 谱图结果分析
经过筛选,每株菌株建立了15张谱图,其平均谱图见图1。
图1中4 000 cm-1~400 cm-1波数范围内的3株菌株平均光谱图极为相似,无法用肉眼区分,这可能跟这3株菌株都是革兰氏阴性肠杆菌,它们的结构组成本就相似以及亲缘关系比较相近有关,故对其准确高效的分类还需借助计算机及化学计量学方法。据文献报道[11-12]和本实验室经验[13],光谱信息具有很强的指纹特征,能捕捉到细菌菌体间的细微差别,而这种差别与细菌的结构组成具有特定对应关系,在1 800 cm-1~900 cm-1光谱波数范围内有3个特定吸收的“指纹区域”:酰胺区、混合区(包括脂肪酸、磷酸化合物和蛋白质)和多糖区。因此我们选择1 800.224 cm-1~915.780 6 cm-1波数范围内的光谱数据进一步做聚类分析。
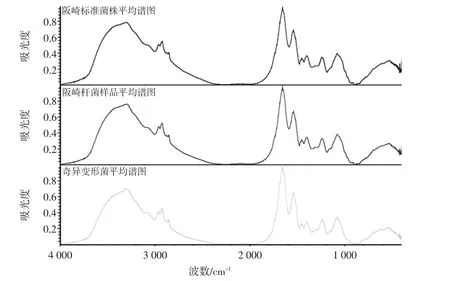
图1 3株菌株的平均FT-IR光谱Fig.1 Average FT-IR spectra of three strains
2.2 PCA分析
主成分是原始变量按一定权重线性组合之后而产生的保持原始数据的特征信息的新变量,代表每一类具有相似特征的变量所体现的综合特点。主成分分析(principal component analysis)是一种基于投影技术的数据分析方法[14],通过PCA分析投影计算处理后得到的第一个主成分PC1,代表原始数据的最大差异。除此,对仍有部分重要信息未被包含在PC1内的数据,沿着与PC1垂直方向寻得次级差异显著的直线,经投影计算形成第二个主成分PC2。在此基础上,PCA对空间的多维数据降维,最终直观地展现出数据的分布散点图,而使数据变得简单易于理解。通过PCA分析不仅可对不同种群之间亲缘关系进行判别,还可对不同属种间的相似性进行研究比较[15]。将3株菌株的光谱数据经PCA分析,结果如图2。
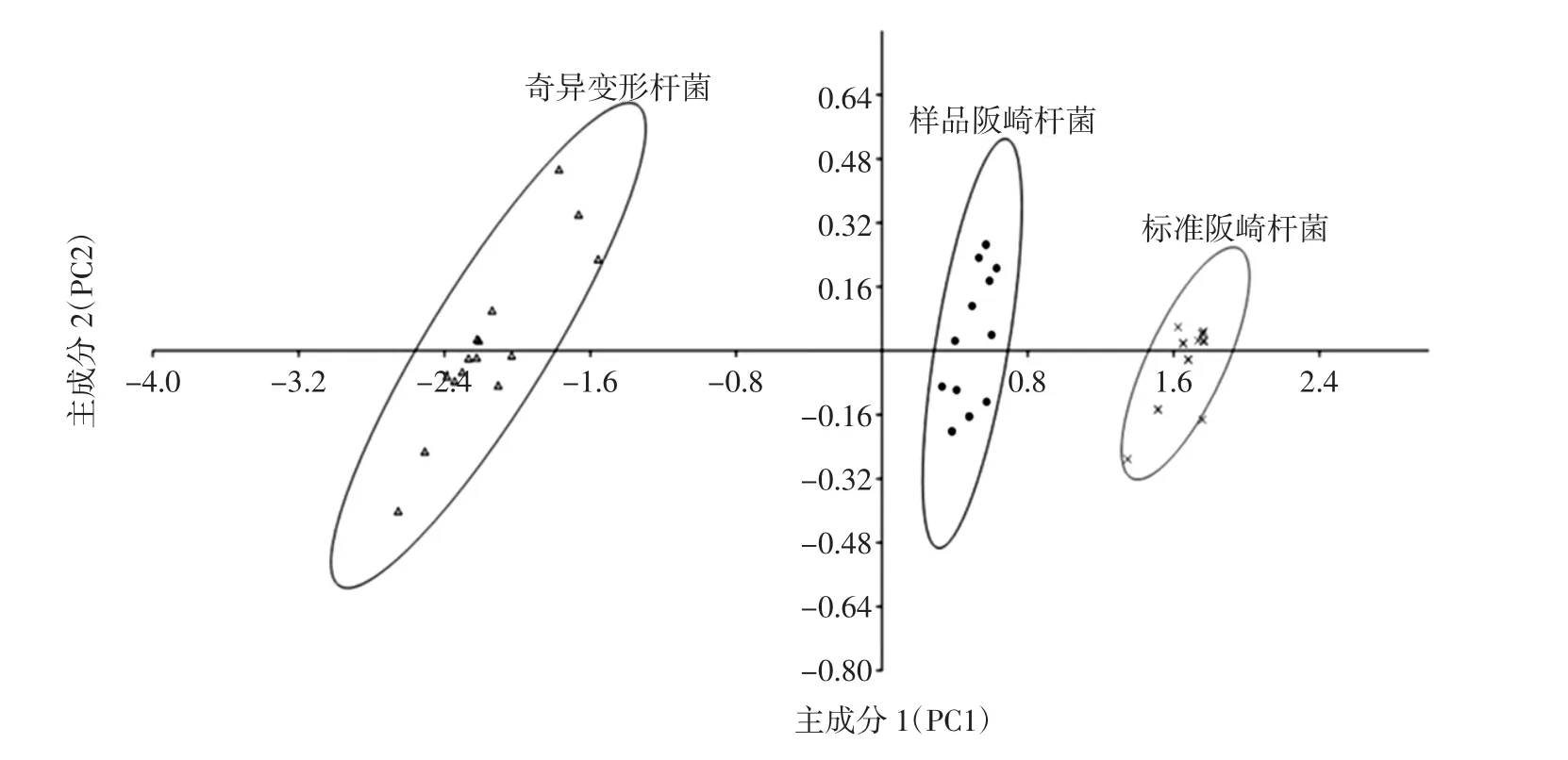
图2 3株菌株的PCA聚类分析图Fig.2 PCA cluster results of three strains
如图2所示,横坐标PC1的贡献率为97.417%,纵坐标PC2的贡献率为1.0366%,两者贡献率共达98.4536%,说明此聚类分析模型具有代表性。图2中可见,3株菌株散点图分布在纵轴左右两个不同的区域,其中标准和样品2株阪崎肠杆菌分在同一区域,但是它们却分别单独聚类;而奇异变形杆菌却被单独分在了另一个区域,这说明FT-IR技术结合PCA分析可以快速地对阪崎肠杆菌和奇异变形杆菌进行分类和鉴定。
2.3 HCA分析
聚类分析(cluster analysis)是一种将研究对象进行多元统计分析,先聚类后再利用判别分析研究各个群体之间的异同,常用来衡量不同数据源间的相似性[15]。用离差平方和法(Ward,s method),欧氏距离(Euclidean)对3株菌株的光谱数据进行HCA分析,结果见图3。
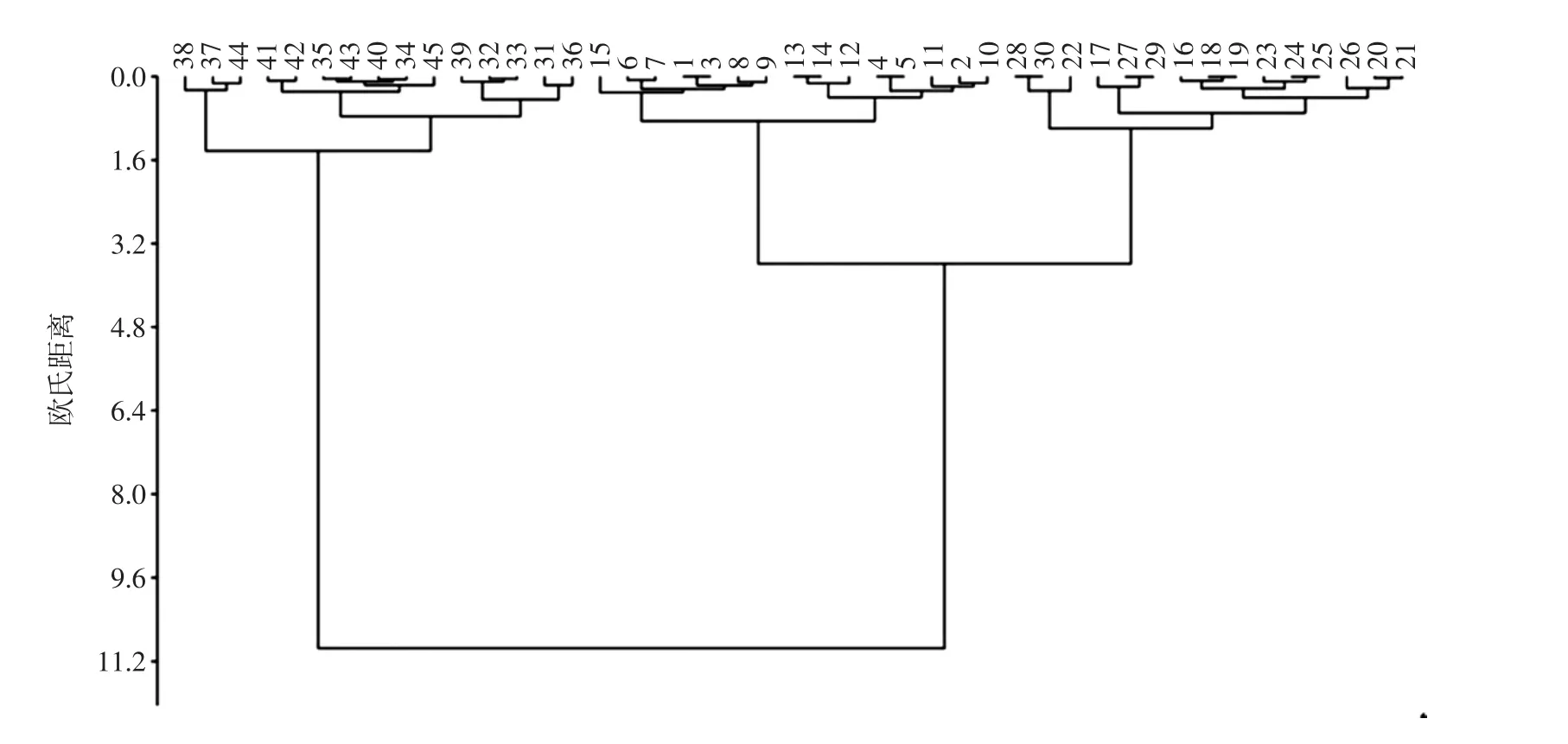
图3 3株菌株的HCA聚类分析图Fig.3 HCA cluster results of three strains
图3中,同一菌株所有实验数据归为1簇,而不同菌株间实验数据各自聚类且无交叉,共聚3簇。同时,标准与样品2株阪崎肠杆菌聚在同一支上,且与奇异变形杆菌分属不同的支类。由此可见,FT-IR技术结合HCA分析可以快速鉴定并区分阪崎肠杆菌和奇异变形杆菌。不同来源的2株阪崎肠杆菌虽然被分在同一支,但是也分别单独聚为一类,与PCA聚类分析一致。
3 讨论与结论
在GB 4789系列食品安全国家标准中对一些食源性致病菌的分离检测主要是靠一些分离培养基及一些显色培养基加以分离培养,而在这些培养基上常常会出现一些与目标菌的形态特征极为相似的干扰菌,因此这就要求检测人员用更多的时间和精力加以辨别区分。阪崎肠杆菌和奇异变形杆菌在显色培养基上分离培养时虽然容易造成混淆,但是文中通过FT-IR技术,使阪崎肠杆菌和奇异变形杆菌得到了很好地区分。不同来源的2株阪崎肠杆菌虽聚到了同一类,但却分别单独聚类而得到明显区分,这可能是两株阪崎肠杆菌虽是同一属却属不同种所致,也有可能是来源、生长环境等其他一些外在的条件所致微生物菌体生化组成成分的稍许改变所致。同时,这也提示我们,为了得到更准确、更完整的光谱数据库,我们在试验过程中应确保每次试验的培养条件、操作过程尽可能做到一致;此外,还应加大不同种的标准菌株信息采集,使光谱数据库信息更全面,更稳健,更具代表性和实用性。
FT-IR技术结合化学计量学方法建立了部分阪崎肠杆菌和奇异变形杆菌的光谱数据库,简化了操作过程,缩短检验周期。PCA分析和HCA分析方法成功地将阪崎肠杆菌和奇异变形杆菌进行各自聚类并加以区分,说明FT-IR技术对阪崎肠杆菌及奇异变形杆菌的分类鉴定提供了一种有效的工具,为其他食源性致病菌的分型研究提供了一种新的思路,也给食品质量安全检测中微生物快速检测的新方法开发提供一定参考。
参考文献:
[1]FRMER III J J,ASBURY M A,HICKMAN F W,et al.The Enterobacteriaceae Study Group.Enterobacter sakazaki:a new species of“Enterobacteriaceae”isolated from clinical specimens[J].International Journal of Systematic and Evolutionary Microbiology,1980,30:569-584
[2]IVERSEN C,MULLANE N,MCCARDELL B,et al.Cronobactergen.nov.,a new genus to accommodate the biogroups of Enterobacter sakazakii,and proposal of Cronobacter sakazakii gen.nov.,comb.nov.,Cronobacter malonaticus sp.nov.,Cronobacter turicensis sp.nov.,Cronobacter muytjensii sp.nov.,Cronobacter dublinensis sp.nov.,Cronobacter genomospedesI,and of three subspedes,Cronobacter dublinensis subsp.dublinensis subsp.nov.,Cronobacter dublinensis subsp.lausannensis subsp.nov.and Cronobacter dublinensis subsp.lactaridi subsp.nov.[J].Intemational Journal of Systematic and Evolutionary Microbiology,2008,58(6):1442-1447
[3]IVERSEN C,FORSYTHE S.Isolation of Enterobacter sakazakii and other Enterobacteriaceae from powdered infant formula milk and related products[J].Food Microbiology,2004,21:771-777
[4]徐湾,姜华,张选飞,等.我国克罗诺杆菌污染现状与预防控制措施研究进展[J].中国病原生物学杂志,2015,10(9):848-851
[5]卢勉飞,蔡芷荷,吴清平,等.阪崎肠杆菌显色培养基的应用研究[J].微生物学通报,2009,36(11):1789-1793
[6]李艳娟,李朝旭,房新平,等.婴幼儿乳粉中阪崎肠杆菌污染防控及检验技术的研究进展[J].中国乳品工业,2015,43(5):38-42
[7]徐励琴,罗泽燕,刘思超,等.2012~2014年惠州市市售食品中食源性致病菌污染状况调查[J].实用预防医学,2015,22(7):812-815
[8]陈万义,任婧,刘振民,等.婴幼儿配方粉中克罗诺杆菌属菌株检测方法研究进展[J].乳业科学与技术,2015(2):23-28
[9]李贞,杨保伟,夏效东,等.婴儿配方乳粉中阪崎克罗诺杆菌的研究进展[J].中国乳品工业,2015,43(7):35-41
[10]黄冰,DAVID A T HARPER,φYVIND HAMMER.定量古生物学软件PAST及其常用功能[J].古生物学报,2013,52(2):161-181
[11]DZIUBA B,BABUCHOWSKI A,NALECZ D,et al.Identification of lactic acid bacterica using FT-IR spectroscopy and cluster analysis[J].International Dairy Journal,2007,17(3):183-189
[12]王静,杨丽君,李兆杰,等.傅立叶变换红外光谱技术对两株大肠杆菌的鉴别[J].微生物学通报,2012,39(12):1844-1850
[13]王萍,张占林,乔勇升.傅立叶变换红外光谱技术对铜绿假单胞菌及其干扰菌的分类鉴定[J].食品科技,2017,42(4):302-305
[14]阿基业.代谢组学数据处理方法--主成分分析[J].中国临床药理学与治疗学,2010,15(5):481-489
[15]AL-QADIRI HM,LIN M,CAVINATO AG,et al.Fourier transform infrared spectroscopy,detection and identification of Escherichia coli O157:H7 and Alicyclobacillus strains in apple juice[J].International Journal of Food Microbiology,2006,111:73-80

