基于CRISPR/Cas9技术的水稻温敏不育基因tms5突变体的构建
黄忠明 周延彪 唐晓丹 赵新辉 周在为 符星学王 凯 史江伟 李艳锋 符辰建2, 杨远柱,2,,,*
基于CRISPR/Cas9技术的水稻温敏不育基因突变体的构建
黄忠明1周延彪2,3,*唐晓丹3赵新辉3周在为3符星学3王 凯3史江伟3李艳锋4符辰建2,3杨远柱1,2,3,4,*
1湖南农业大学农学院, 湖南长沙 410128;2袁隆平农业高科技股份有限公司 / 抗病虫水稻育种湖南省工程实验室, 湖南长沙 410001;3湖南隆平高科种业科学研究院有限公司, 湖南长沙 410119;4湖南师范大学生命科学学院, 湖南长沙 410081
为创新优良温敏不育系, 促进两系杂交稻育种的发展, 我们以水稻温敏不育基因为编辑对象, 设计了由水稻U3启动子驱动、长20 bp的guide RNA (gRNA)靶点以靶向编辑基因的第1个外显子, 将靶点与表达载体pCAMBIA1301连接, 再用农杆菌介导法获得水稻转基因株系。提取T0代转基因株系的基因组DNA, 并对编辑位点附近的DNA片段进行PCR检测及测序分析。结果表明, T0代植株的突变率为63.89%, 其中纯合缺失突变率为34.78%。对T1代纯合缺失突变体的不育起点温度和农艺性状调查分析结果表明, 28℃是突变体花粉育性的转换温度。大田试验结果表明, 与野生型相比,突变体的结实率和单株重均显著降低。粳稻突变体的获得为培育粳稻不育系奠定了材料基础。
水稻; 基因编辑; CRISPR/Cas9; TMS5; 温敏不育
水稻是我国最重要的粮食作物之一, 针对紧缺的自然资源和有限的耕地面积, 粮食的供需变得越来越不平衡, 培育高产稳产的优质作物品种是确保粮食安全的根本途径。杂交水稻的产量比常规水稻提高20%~30%, 因此发展杂交水稻是解决粮食短缺问题的重要方法[1]。
杂交水稻的育种方法主要包括两系法和三系法。三系即保持系、不育系和恢复系, 主要是通过细胞质雄性不育系和恢复系杂交产生杂交种子, 利用保持系和不育系杂交繁殖细胞质雄性不育系。两系即不育系和恢复系, 主要是利用光/温敏雄性核不育系与恢复系杂交产生杂交种子。光/温敏不育系在长日/高温条件下花粉败育, 而在短日/低温条件下能恢复花粉育性, 故可一系两用。光/温敏雄性核不育系的育性由核基因控制, 无恢保关系限制, 配组自由, 选出优良组合机率高[2]。因此, 光/温敏不育系在杂交水稻育种中潜力巨大, 被广泛应用。杂交水稻的品种选育, 从三系发展到两系, 如何进一步突破产量潜力, 实现高产与优质高效结合, 是目前的迫切需求。2014年, Zhou等[3]克隆了安农S-1和株1S的温敏不育基因, 首次揭示了水稻温敏雄性不育的分子机制。序列分析结果显示, 安农S-1和株1S中编码区第71位碱基均由C突变成A, 形成了一个提前的终止密码子, 导致TMS5蛋白不能正确合成。
培育优良的杂交水稻组合, 首要要实现不育系的突破。常规杂交转育是培育两系不育系较常用的方法。但常规杂交育种有选育周期漫长和一些优良性状会丢失等缺点, 越来越难以实现日益提高的育种目标。目前, CRISPR/Cas9 (clustered regularly interspaced short palindromic repeats/CRISPR- associated protein 9)基因编辑技术可对作物内源基因进行定向改造, 并且具有成本低廉、技术门槛低、操作简单、试验周期短等优点, 为作物遗传改良提供了新的途径[4-6]。CRISPR/Cas9 系统主要包括 Cas9核酸酶和 sgRNA 两个重要组分。这两个组分形成复合体, 切割与sgRNA上的间隔序列(spacers)互补的基因组DNA, 引起DNA双链断裂, 并通过体内的非同源末端连接(non-homologous end joining, NHEJ)进行修复形成碱基缺失、插入、替换等突变类型[7-8]。2013年, Shan等[9]利用CRISPR/Cas9技术对水稻基因进行编辑, 获得及突变体, 可以用于水稻香米株系和耐冷性株系的创建。同年, 冯争艳等[10]对水稻基因进行突变, 可以用于水稻卷叶育种及糯性育种。2016 年, 中国科学院遗传与发育生物学研究所高彩霞研究组和李家洋研究组合作利用在修复途径中占主导地位的非同源末端连接(NHEJ)修复方式在水稻中建立了基于 CRISPR/Cas9 技术的基因替换以及基因定点插入体系, 并通过碱基替换策略获得抗草甘膦的水稻材料[11]。CRISPR/Cas9 基因编辑技术的发展使其在水稻遗传改良中的应用越来越广泛[12-13]。本研究利用CRISPR/Cas9技术定点编辑温敏不育基因, 获得了粳稻F197的突变体, 并对其不育起点温度进行了研究, 为利用基因创制新型的温敏不育新种质提供了材料基础。
1 材料与方法
1.1 靶位点设计
设计(LOC_Os02g12290)靶点序列参照Xie等的方法[14]。在第1外显子上, 靠近编码蛋白质的N’端, 设计长度为20 bp的靶位点序列, PAM序列为CGG (图1)。靶点序列正义链和反义链的5¢端分别添加TGGC和AAAC, 使其与中间载体18T-Cas9经I酶切后形成黏性互补末端。
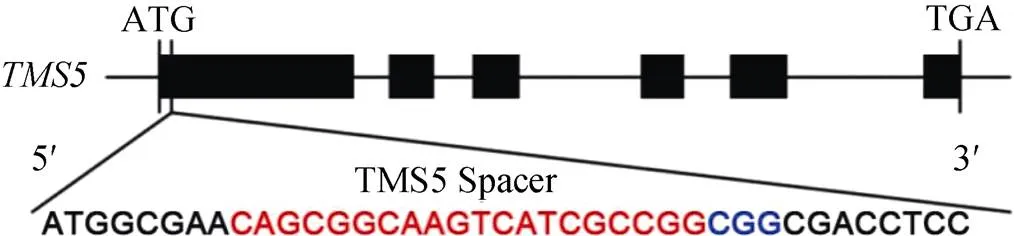
图1 TMS5靶点位置
红色的字母为靶点序列; 蓝色的字母为PAM序列。
The red letters are the target genome sequences; The blue letters are the protospacer adjacent motif (PAM) sequences.
1.2 CRISPR/Cas9表达载体的构建
利用合成的靶点序列引物Target-1 (表1), 经过98℃处理30 s, 再室温放置5 min制成互补双链DNA, 用于载体构建。用I酶切18T-Cas9载体、酶切体系包括18T-Cas9载体2 μg、10×FastDigest buffer 2 μL、I 0.1 μL (10 U μL–1), 加ddH2O至20 μL。对酶切后的18T-Cas9质粒进行胶回收, 然后用T4 DNA连接酶将的接头与回收后的18T-Cas9载体连接。连接产物转化大肠杆菌DH5a, 用18T-Cas9载体上的引物M13F与靶点序列引物Target-1R进行菌液PCR检测, 挑选阳性菌株送公司测序, 筛选正确的重组中间载体18T-Cas9-TMS5- gRNA, 然后分别用I和d III酶切植物表达载体pCAMBIA1301和重组的中间载体, 回收酶切产物, 再通过T4 DNA连接酶连接, 构建pCAMBIA1301-Cas9-TMS5-gRNA表达载体, 再通过酶切鉴定确认植物表达载体构建成功后转化农杆菌EHA105, 载体构建的具体流程见图2。
1.3 转基因阳性植株的获得
将构建好的阳性农杆菌EHA105转化粳稻F197的愈伤组织, 用潮霉素筛选获得T0代植株。提取T0代植株的DNA, 用载体上的特异引物GUS-JC-F/ GUS-JC-R (表1)进行PCR扩增, 扩增产物大小为718 bp, 能扩增出目的大小片段的植株为阳性转基因植株。同时, 因为pCAMBIA1301载体上有GUS报告基因, 因此可以通过GUS组织化学染色[15]的方法快速鉴定阳性转基因植株。
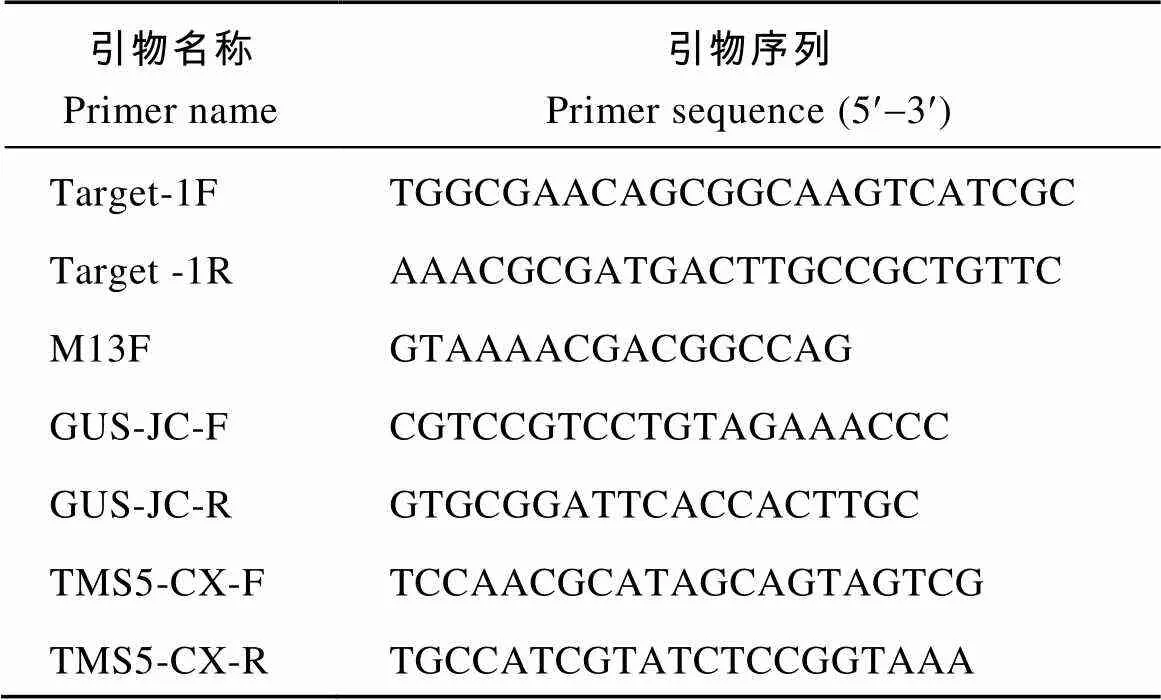
表1 本研究所用的引物
1.4 测序检测靶位点
为了检测靶位点的突变情况, 在的靶位点两端分别设计测序引物TMS5-CX-F和TMS5- CX-R (表1), 扩增产物大小为974 bp。将测序引物扩增的目的片段送测序公司测序。同时, 以野生型F197的基因组DNA为模板, 将测序引物扩增目的片段的测序结果作为对照序列。
1.5 T1代无选择标记基因突变株的获得
对T1代植株的叶片进行GUS组织化学染色, 不能染上蓝色的为无选择标记基因的植株, 以野生型叶片的染色为对照。再以无选择标记基因植株的基因组为模板用TMS5-JC-F和TMS5-JC-R引物扩增, 将目的条带测序, 根据测序结果分析是否有突变。
1.6 碘-碘化钾染色检测花粉育性
对照F197和T1代无转基因元件且功能缺失的突变株在主茎幼穗分化到六期的时候进行人工控光温处理, 分别用24℃、28℃和32℃处理14 d (12 h光照/12 h黑夜)后, 在自然条件下生长, 用碘-碘化钾染色[16]观察除主茎外的其他茎节上花粉的育性, 并调查成熟期F197和突变体的结实率。
1.7 大田农艺性状调查
参照Zhou等的方法[17], 以F197为对照, 于2017年5月至10月在湖南长沙自然条件下调查突变体的田间农艺性状。5月25日播种, 8月11日至20日为突变体幼穗敏感期, 这期间日平均气温为27.8℃。在成熟期选择F197和突变体各20株, 分别考察株高、千粒重、有效穗、结实率和单株产量, 采用检验统计分析数据。
2 结果与分析
2.1 CRISPR/Cas9表达载体的构建
为了获得粳稻F197的基因敲除的突变体, 根据CRISPR/Cas9技术原理, 结合生物信息学网站http://www.genome.arizona.edu/crispr/, 在第1外显子区域设计20 bp的靶点序列Target-1 (表1和图1)。退火形成的双链互补靶序列与中间载体18T-Cas9连接形成重组载体18T-Cas9-Rice-TMS5- gRNA (图2-A)。重组载体经I和d III双酶切, 将CRISPR/Cas9元件连接到植物表达载体pCAMBIA1301中形成重组载体pCAMBIA1301- Cas9-TMS5-gRNA (图2-B, C)。通过酶切鉴定, 说明pCAMBIA1301-Cas9-TMS5-gRNA表达载体构建成功(图3-A)。将构建成功的重组载体通过电击转化农杆菌EHA105, 再用pCAMBIA1301载体上的报告基因GUS的检测引物GUS-JC-F/GUS-JC-R (表1)进行菌液PCR, 扩增产物大小为718 bp (图3-B), 能扩增出目的片段大小的菌液为阳性菌株, 将阳性的农杆菌菌株用于转化粳稻F197。
2.2 转基因植株的鉴定及突变位点测序分析
采用CTAB法提取T0代水稻植株叶片的基因组DNA, 用GUS报告基因的特异引物GUS-JC-F/GUS- JC-R进行PCR, 扩增目的片段大小为718 bp的植株为阳性转基因植株(图4-A)。另外, 对T0代植株进行GUS组织化学染色可以快速地鉴定转基因植株, 能染上蓝色的为阳性转基因植株(图4-B)。在F197中共获得57株组培苗, 其中有36株阳性转基因植株。为了分析T0代阳性转基因植株的突变情况, 在靶点附近设计测序引物TMS5-CX-F/ TMS5-CX-R。PCR测序结果表明, 在36株阳性转基因植株中, 检测到23株发生突变, 其中纯合突变有8株, 纯合突变率为34.78%, 其余15株的靶序列突变位点附近出现双峰。将出现双峰的测序文件通过在线工具DSDecode (http://skl.scau.edu.cn/ dsdecode/)自动解码[18], 结果显示均为杂合突变(附图)。纯合突变主要来自PAM前第3和第4碱基之间有单碱基A、T和G的插入, 分别占62.5%、25.0%和12.5%, 而没有检测到单碱基C的插入(图5)。
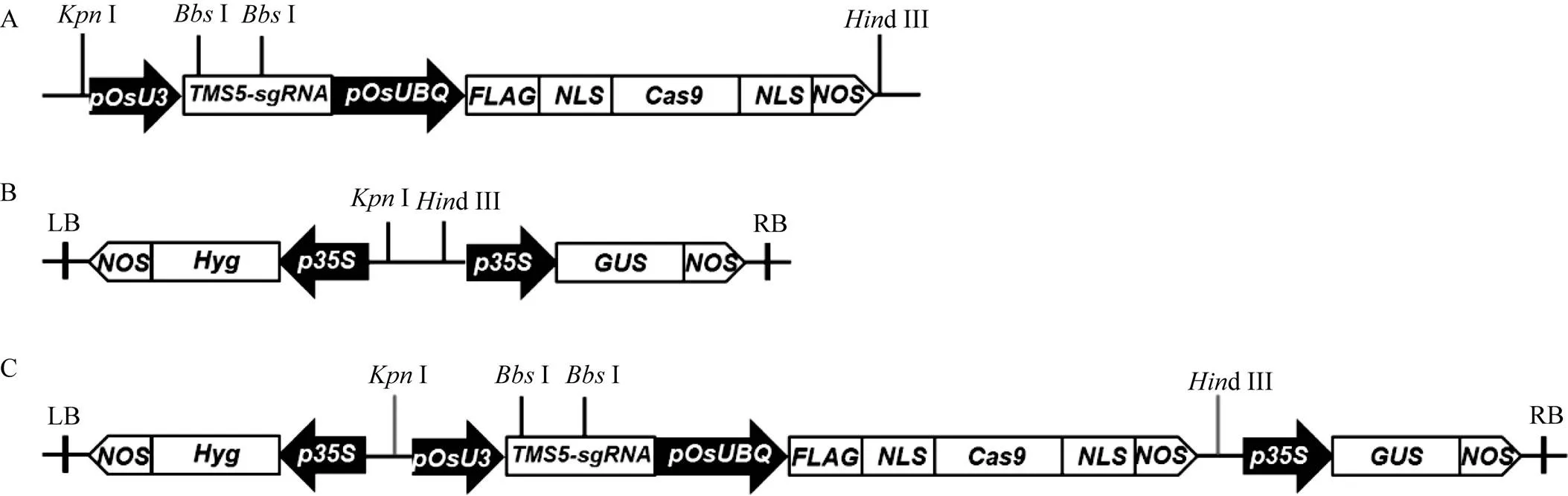
图2 CRISPR/Cas9-TMS5载体构建示意图
A: 18T-Cas9-TMS5-gRNA重组载体结构示意图; B: pCAMBIA1301载体的T-DNA片段; C: pCAMBIA1301-Cas9-TMS5-gRNA重组载体结构示意图。LB: 左边界; RB: 右边界。
A: schematic diagram of the recombinant vector of 18T-Cas9-TMS5-gRNA; B: T-DNA fragment of pCAMBIA1301 vector; C: schematic diagram of the pCAMBIA1301-Cas9-TMS5-gRNA construct. LB: left border; RB: right border.
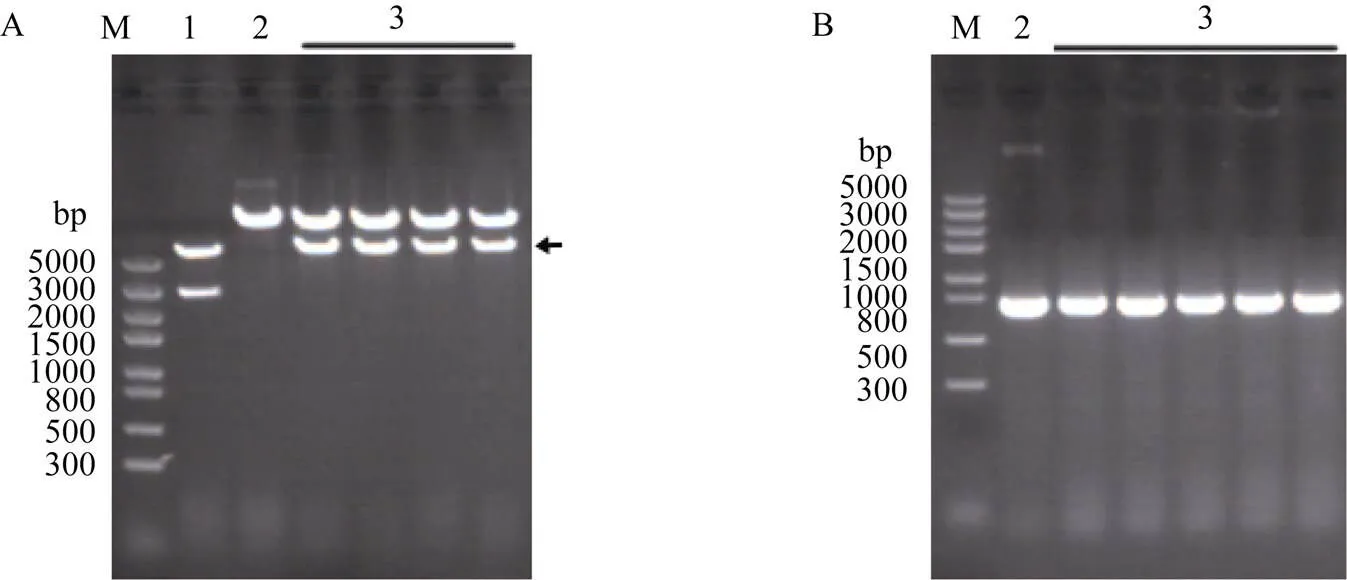
图3 酶切和PCR鉴定pCAMBIA1301-Cas9-TMS5-gRNA载体
A:I和d III酶切鉴定pCAMBIA1301-Cas9-TMS5-gRNA载体。箭头所示为CRISPR/Cas9系统元件的片段; B: 利用GUS-JC-F和GUS-JC-R引物PCR鉴定农杆菌中的pCAMBIA1301-Cas9-TMS5-gRNA载体。M: DL2000; 1: 18T-Cas9-TMS5-gRNA; 2: pCAMBIA1301; 3: pCAMBIA1301-Cas9-TMS5-gRNA。
A: identification of the pCAMBIA1301-Cas9-TMS5-gRNA plasmid digested withI andd III; B: identification of the pCAMBIA1301-Cas9-TMS5-gRNA plasmid inby PCR with primers of GUS-JC-F and GUS-JC-R. M: DL2000; 1: 18T-Cas9-TMS5-gRNA; 2: pCAMBIA1301; 3: pCAMBIA1301-Cas9-TMS5-gRNA.
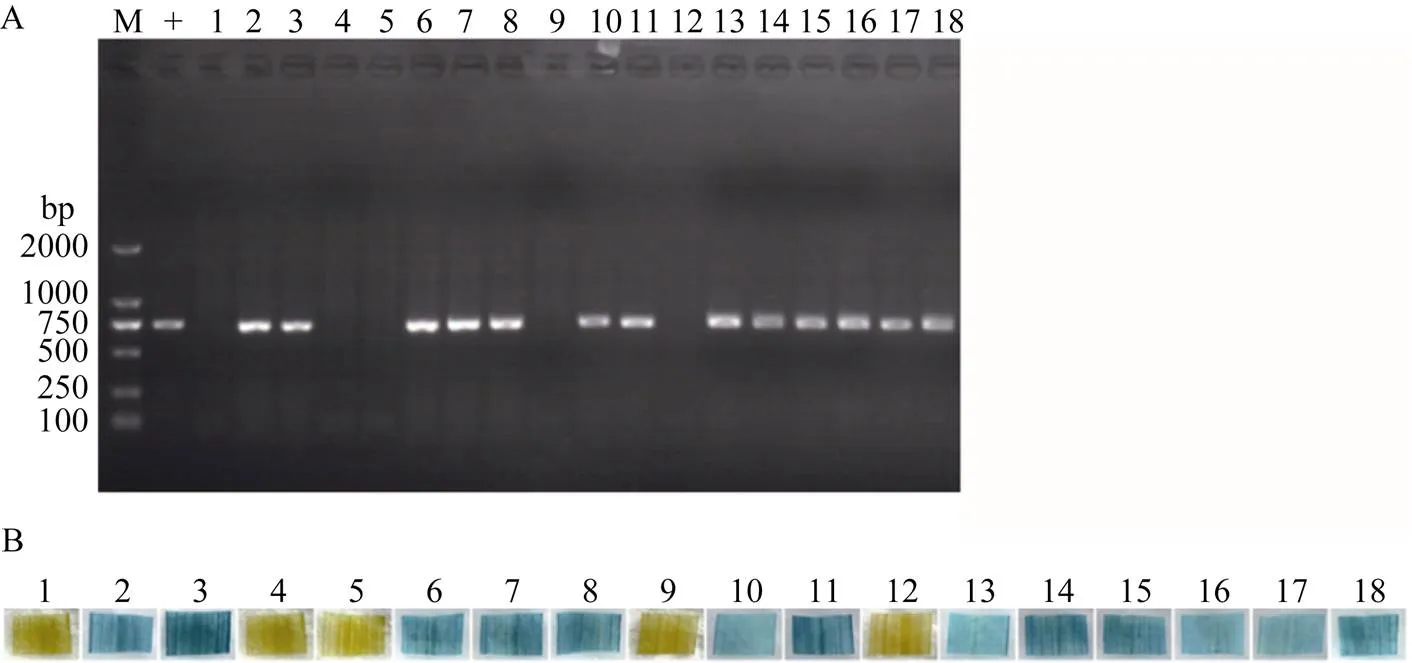
图4 转基因植株的鉴定
A: PCR检测阳性转基因植株; B: GUS组织化学染色检测阳性转基因植株。M: DL2000; +: 阳性对照; 1–18: 转基因植株。
A: identification of the positive transgenic plants by PCR; B: identification of the positive transgenic plants by GUS histochemical staining. M: DL2000; +: positive control; 1–18: transgenic plants.

图5 T0代转基因植株TMS5纯合突变类型和频率
A: 纯合突变类型序列比对; B: 纯合突变类型不同碱基插入的频率。红色字母为靶点序列; 蓝色字母为PAM序列; 绿色的字母为插入的碱基。
A: Sequence alignment of homozygous mutation types; B: Frequency of different base inserts in homozygous mutation types. The red letters are the target genome sequences; The blue letters are PAM; The green letters are the insert base.
2.3 T1代无选择标记基因突变株的获得及突变株不育起点温度的鉴定
将上述单碱基A或T或G插入的纯合突变单株自交, 结实后得到T1代种子, 将其播种成苗后进行GUS染色, 没有染上蓝色的为不含转基因成分的单株, 然后提取非转基因植株叶片的基因组DNA进行PCR测序, 分别得到单碱基A或T或G插入的不含转基因成分的单株, 编号分别为用于不育起点温度的鉴定。
当野生型(WT)和突变体主茎的幼穗处于六期的时候, 将WT和突变体置于24℃、28℃和32℃的恒温培养箱分别培养14 d, 然后对除主茎外的其他茎节上的花粉进行镜检, 并考察成熟期结实率。结果表明, 在24℃培养下, WT和突变体的花粉都是可育的, 在成熟期WT和突变体的结实率没有显著变化; 28℃培养下,突变体的花粉大部分是不育的, 而WT的花粉是可育的, 在成熟期突变体的结实率显著低于WT的; 32℃培养下,突变体的花粉是完全不育的, 而野生型的花粉是可育的, 在成熟期突变体的结实率为0 (图6)。因此, 28℃是突变株花粉育性的转换温度。

图6 野生型和tms5突变体在不同温度处理条件下的花粉育性和结实率
数据表示平均值±方差。**表示突变体与WT之间存在显著差异(< 0.01)。
Data are presented as average values ± standard error of the mean. **, significant difference betweenmutant and WT (< 0.01).
2.4 T1代植株农艺性状分析
为了研究F197的基因缺失后对植株农艺性状的影响。我们调查了野生型和突变体的株高、千粒重、结实率、有效穗和单株重。结果表明突变体的结实率和单株重比野生型明显降低(<0.01), 而株高、千粒重、有效穗与野生型相比均无显著差异(表2)。
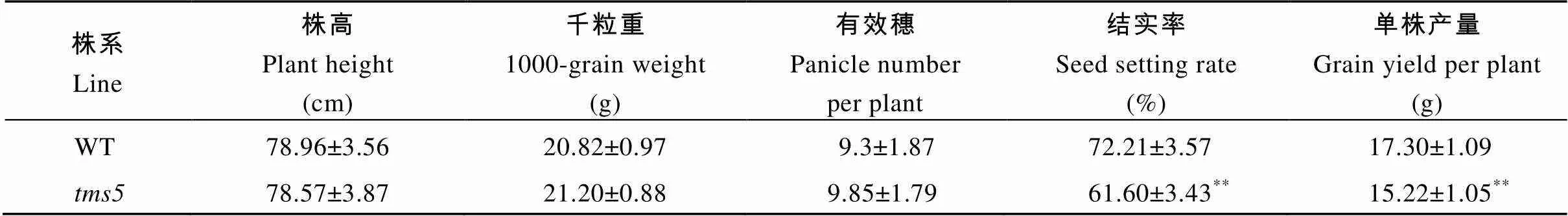
表2 野生型(WT)和tms5突变体的农艺性状
数据表示平均值±标准误(=20)。**表示突变体与WT之间存在显著差异(< 0.01)。
Data are presented as average values ± standard error (=20).**, significant difference betweenmutant and WT (< 0.01)
3 讨论
CRISPR/Cas9基因编辑技术是一种新兴的基因组定点编辑技术, 具有操作简单、编辑效率高、成本低廉等特点。由于插入位置与作用位点分离, 在转基因植物的后代中通过自交等手段可将转基因成分从基因组中分离出去, 从而最小范围地特异性地改变植物基因组。因此, CRISPR/Cas9基因组编辑技术可以帮助育种工作者更加快速、准确地定向改造作物品种。目前该技术已经在许多作物中广泛应用, 如水稻[19-20]、玉米[21-22]、小麦[23-24]、大豆[25-26]等。2014年, Zhou等[3]克隆了安农S-1和株1S的温敏不育基因, 首次揭示了水稻温敏雄性不育的分子机制。本研究为探讨基因功能缺失在粳稻中的效应, 利用CRISPR/Cas9基因编辑技术在粳稻品种F197中敲除基因, 成功获得了功能缺失的突变体, 并在T1代功能缺失的非转基因材料中考察了花粉育性的起点温度和农艺性状。
CRISPR/Cas9系统中的Cas9核酸酶以PAM上游第3碱基处为切割中心进行突变[27]。其中, 超过一半的突变为单碱基插入, 其中又以A 或T插入为主[28-29]。本研究中,基因的纯合突变发生在PAM上游第3碱基和第4碱基之间, 并且是单碱基A或T或G的插入, 其中单碱基A插入占62.5%, 单碱基T插入占25%, 单碱基G插入占12.5%。基因的突变情况与前人的研究结果类似。
研究显示我国25个主要使用的两用光/温敏核不育系中有24个均含有基因, 表明基因是控制我国温敏型不育系的主要不育基因[1]。Zhou等[3]利用CRISPR/Cas9技术对粳稻品种中花11、GAZ和籼稻品种粤晶丝苗、泰丰B、五山丝苗、ReB、珍汕97B的基因进行敲除, 并分别获得了这些品种的功能缺失的突变体。粳稻品种中花11和GAZ的突变体在28℃培养下花粉表现为完全不育, 而籼稻品种粤晶丝苗、泰丰B、五山丝苗、ReB、珍汕97B的突变体分别在24℃、26℃、26℃、26℃、28℃培养下花粉表现为完全不育[18]。同时, 水稻广亲和温敏不育系籼稻株1S的不育起点温度为22.6℃[30-31]。因此, 可以看出籼稻品种的功能缺失突变体的不育起点温度较粳稻品种低, 这种结果可能是粳稻和籼稻的遗传背景不同造成的, 同时也说明基因功能缺失与不育起点温度无关。本研究中, 粳稻F197的突变体花粉在24℃培养下完全可育, 28℃培养下大部分不育, 32℃培养下完全不育(图6)。因此, 28℃可能是突变体花粉育性的转换温度。
产量是作物最重要的性状之一, 由多个产量相关性状共同决定, 并且易受外界环境的影响。产量的决定因素主要有千粒重、结实率和有效穗数[32]。编码一个短版的RNAase Z同源蛋白RNAase ZS1, 通过对UbmRNA的加工调控水稻温敏雄性不育[1,3]。本试验中粳稻品种F197的突变体由于花粉育性降低, 导致其结实率比野生型的低, 但是突变体的株高、千粒重和有效穗数与野生型相比没有明显变化。
4 结论
利用CRISPR/Cas9技术获得了敲除温敏不育基因的粳稻品种F197的突变体, 为培育水稻温敏雄性核不育系提供了重要材料基础。28℃是突变体的育性转换温度, 并且在32℃表现为完全不育, 这为敲除的粳稻品种不育起点温度的研究提供了重要参考。
[1] 周海, 周明, 杨远柱, 曹晓风, 庄楚雄. RNase ZS1加工UbL40 mRNA控制水稻温敏雄性核不育. 遗传, 2014, 36: 1274 Zhou H, Zhou M, Yang Y Z, Cao X F, Zhuang C X. RNase ZS1processesUbmRNAs and controls thermosensitive genic male sterility in rice.(Beijing), 2014, 36: 1274 (in Chinese)
[2] 袁隆平. 我国两系法杂交水稻研究的形势、任务和发展前景. 农业现代化研究, 1997, 18(1): 1–3 Yuan L P. Current status and developing prospects in two-line hybrid rice research in China., 1997, 18(1): 1–3 (in Chinese with English abstract)
[3] Zhou H, Zhou M, Yang Y Z, Li J, Zhu L Y, Jiang D G, Dong J F, Liu Q J, Gu L F, Zhou L Y, Feng M J, Qin P, Hu X C, Song C L, Shi J F, Song X W, Ni E D, Wu X J, Deng Q Y, Liu Z L, Chen M S, Liu Y G, Cao X F, Zhuang C X. RNase ZS1processesUbmRNAs and controls thermosensitive genic male sterility in rice., 2014, 5: 4884
[4] Belhaj K, Chaparro-Garcia A, Kamoun S, Patron N J, Nekrasov V. Editing plant genomes with CRISPR/Cas9., 2015, 32: 76–84
[5] Baltes N J, Voytas D F. Enabling plant synthetic biology through genome engineering., 2015, 33: 120–131
[6] 瞿礼嘉, 郭冬姝, 张金喆, 秦跟基. CRISPR/Cas系统在植物基因组编辑中的应用. 生命科学, 2015, (1): 64–70 Qu L J, Guo D S, Zhang J Z, Qin G J. The application of CRISPR/Cas system in plant genome editing., 2015, (1): 64–70 (in Chinese with English abstract)
[7] Wiedenheft B, Sternberg S H, Doudna J A. RNA-guided genetic silencing systems in bacteria and archaea., 2012, 482: 331–338
[8] Symington L S, Gautier J. Double-strand break end resection and repair pathway choice., 2011, 45: 247–271
[9] Shan Q W, Wang Y P, Li J, Zhang Y, Chen K L, Liang Z, Zhang K, Liu J X, X J J, Qiu J L, Gao C X. Targeted genome modification of crop plants using a CRISPR-Cas system., 2013, 31: 686–688
[10] Feng Z Y, Zhang B T, Ding W N, Liu X D, Yang D L, Wei P L, Cao F Q, Zhu S H, Zhang F, Mao Y F, Zhu J K. Efficient genome editing in plants using a CRISPR/Cas system., 2013, 23: 1229–1232
[11] Li J, Meng X B, Zong Y, Chen K L, Zhang H W, Liu J X, Li J Y, Gao C X. Gene replacements and insertions in rice by intron targeting using CRISPR-Cas9., 2016, 2: 16139
[12] Li J Y, Sun Y W, Du J L, Zhao Y D, Xia L Q. Generation of targeted point mutations in rice by a modified CRISPR/CAS9 system., 2017, 10: 526–529
[13] Lu Y, Zhu J K. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system., 2017, 10: 523–525
[14] Xie K, Zhang J, Yang Y. Genome-wide prediction of highly specific guide RNA spacers for CRISPR-Cas9-mediated genome editing in model plants and major crops., 2014, 7: 923–926
[15] Lin J Z, Zhou B, Yang Y Z, Mei J, Zhao X Y, Guo X H, Huang X Q, Tang D Y, Liu X M. Piercing and vacuum infiltration of the mature embryo: a simplified method for Agrobacterium-mediated transformation ofrice., 2009, 28: 1065–1074
[16] Zhang H, Xu C X, He Y, Zong J, Yang X J, Si H M, Sun Z X, Hu J P, Liang W Q, Zhang D B. Mutation in CSA creates a new photoperiod-sensitive genic male sterile line applicable for hybrid rice seed production., 2013, 110: 76–81
[17] Zhou Y B, Liu H, Zhou X C, Yan Y Z, Du C Q, Li Y X, Liu D R, Zhang C S, Deng X L, Tang D Y, Zhao X Y, Zhu Y H, Lin J Z, Liu X M. Over-expression of a fungal NADP(H)-dependent glutamate dehydrogenaseGDH improves nitrogen assimilation and growth quality in rice., 2014, 34: 335–349
[18] Liu W, Xie X, Ma X, Li J, Chen J, Liu Y G. DSDecode: a web-based tool for decoding of sequencing chromatograms for genotyping of targeted mutations., 2015, 8: 1431–1433
[19] Zhou H, He M, Li J, Chen L, Huang Z F, Zheng S Y, Zhu L Y, Ni E D, Jiang D G, Zhao B R, Zhuang C X. Development of commercial thermo-sensitive genic male sterile rice accelerates hybrid rice breeding using the CRISPR/Cas9-mediated TMS5 editing system., 2016, 6: 37395
[20] Li M R, Li X X, Zhou Z J, Wu P Z, Fang M C, Pan X P, Lin Q P, Luo W B, Wu G J, Li H Q. Reassessment of the four yield-related genes,,, andin rice using a CRISPR/Cas9 system., 2016, 7: 377
[21] Svitashev S, Schwartz C, Lenderts B, Young J K, Ciqan A M. Genome editing in maize directed by CRISPR-Cas9 ribonucleoprotein complexes., 2016, 7: 13274
[22] Feng C, Yuan J, Wang R, Liu Y, Birchler J A, Han F P. Efficient targeted genome modification in maize using CRISPR/Cas9 system., 2016, 43: 37–43
[23] Shan Q W, Wang Y P, Li J, Gao C X. Genome editing in rice and wheat using the CRISPR/Cas system., 2014, 9: 2395–2410
[24] Wang Y P, Cheng X, Shan Q W, Zhang Y, Liu J X, Gao C X, Qiu J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew., 2014, 32: 947–951
[25] Cai Y P, Chen L, Liu X J, Sun S, Wu C X, Jiang B J, Han T F, Hou W S. CRISPR/Cas9-mediated genome editing in soybean hairy roots., 2015, 10: e0136064
[26] Jacobs T B, LaFayette P R, Schmitz R J, Parrott W A. Targeted genome modifications in soybean with CRISPR/Cas9., 2015, 15: 16
[27] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna J A, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337: 816–821
[28] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R, Xie Y Y, Shen R X, Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants., 2015, 8: 1274–1284
[29] Zhang H, Zhang J S, Wei P L, Zhang B T, Gou F, Feng Z Y, Mao Y F, Yang L, Zhang H, Xu N F, Zhu J K. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation., 2014, 12: 797–807
[30] 杨远柱, 符辰建, 胡小淳, 张章, 周永祥, 宋永帮. 株1S温敏核不育基因的发现及超级杂交早稻育种研究. 中国稻米, 2007, (6): 17–22 Yang Y Z, Fu C J, Hu X C, Zhang Z, Zhou Y X, Song Y B. Discovery of thermo-sensitive genic male sterile genes of Zhu1S and study on super hybrid early rice breeding., 2007, (6): 17–22 (in Chinese)
[31] 李必湖, 吴厚雄, 徐孟亮, 梁满中, 张振华, 陈良碧. 温敏核不育水稻育性对低温持续时间的敏感性差异比较研究. 作物学报, 2003, 29: 930–936 Li B H, Wu H X, Xu M L, Liang M Z, Zhang Z H, Chen L B. Comparative studies on the sensitivity of fertility of TGMS rice lines to low temperature of consecutive time., 2003, 29: 930–936 (in Chinese with English abstract)
[32] Xing Y Z, Zhang Q F. Genetic and molecular bases of rice yield., 2010, 61: 421–442

附图1 靶位点突变序列分析
Supplementary Fig. 1 Analysis of mutation sequences of target
红色的字母为靶点序列; 蓝色的字母为PAM序列; 绿色的字母为插入的碱基; +表示插入; WT表示野生型; Ho表示纯合突变; He表示杂合突变。
The red letters are the target genome sequences; the blue letters are PAM; the green letters are the insert base; +: Insertion; WT: Wild-type; Ho: homozygous mutation; He: heterozygous mutation.
Construction ofMutants in Rice Based on CRISPR/Cas9 Technology
HUANG Zhong-Ming1, ZHOU Yan-Biao2,3,*, TANG Xiao-Dan3, ZHAO Xin-Hui3, ZHOU Zai-Wei3, FU Xing-Xue3, WANG Kai3, SHI Jiang-Wei3, LI Yan-Feng4, FU Chen-Jian2,3, and YANG Yuan-Zhu1,2,3,4,*
1College of Agronomy, Hunan Agricultural University, Changsha 410128, Hunan, China;2Yuan Longping High-Tech Agriculture Co. Ltd., Hunan / Hunan Engineering Laboratory for Disease and Pest Resistance Rice Breeding, Changsha, 410001, Hunan, China;3Hunan Longping High-Tech Seeds Science Academy Co. Ltd., Changsha 410119, Hunan, China;4College of Life Sciences, Hunan Normal University, Changsha 410081, Hunan, China
In order to create excellent temperature sensitive sterile line and promote the development of two line hybrid rice breeding. a 20 bp guide RNA (gRNA) was targeted to the first exon ofa thermo-sensitive genic male sterile geneas the editable object in present study, and driven by the rice U3 promoter. The target site of gRNA was ligated into the vector pCAMBIA1301, and its transgenic lines were obtained via-mediated transformation. The genomic DNA of T0transgenic lines was then extracted, and thelocus was detected by PCR and sequenced. The mutagenesis frequency forwas 63.89%, of which the frequency of homozygous deletion mutation was 34.78%. Furthermore, the fertility transition temperature and agronomic traits of T1homozygous deletion mutants were investigated, showing that the fertility transition temperature ofmutants was 28°C. Both seed setting rate and grain yields inmutants were significantly decreased compared with those in wild-type plants in paddy field condition. Together, the successful construction ofmutants lays a material foundation for breeding the thermo-sensitive male sterile line ofrice.
rice; genome editing; CRISPR/Cas9; TMS5; thermo-sensitive genic male sterile
2017-10-25;
2018-03-26;
2018-04-08.
10.3724/SP.J.1006.2018.00844
杨远柱, E-mail: yzhuyah@163.com; 周延彪, E-mail: zhouyb@lpht.com.cn
E-mail: 604654239@qq.com
本研究由国家转基因生物新品种培育重大专项(2016ZX08001-004)和湖南省重点研发计划项目(2016JC2026)资助。
This study was supported by the Major Project of China on New Varieties of GMO Cultivation (2016ZX08001-004) and the Key Research Program of Hunan Province (2016JC2026).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20180408.1014.010.html

