PUF蛋白的研究进展
张 静,王文珍,蒲首丞,孙梅好
(浙江师范大学化学与生命科学学院,浙江金华321000)
细胞能够精准地控制mRNA在恰当的时候在特定的位置产生定量的蛋白质,而RNA结合蛋白(RNA-binding proteins,RBPs) 和小 RNA(miRNA)控制着这些进程。它们结合特定的mRNAs,从而控制mRNA的稳定性、翻译过程和定位[1]。一个RNA结合蛋白能够结合许多的RNA,从而建造一个庞大的RNA网络来调控特定的生物学功能。蛋白质结合RNA有多种多样的方法,并且通常都难以预料是以怎样的方式结合。目前,存在一些与RNA作用并对其产生影响的工具的使用,类似于短干扰RNA和小分子RNA的使用,但这些工具的使用会降低目标RNA的多样性和在细胞中的表达量[2]。因此,能够被设计的工程性RNA结合蛋白是十分具有吸引力的,因为它们能与任何想要的效应结构域相结合,能够选择性结合一种特定的RNA目标,从而来研究或者控制某一方面的新陈代谢作用。最早在果蝇D.melanogaster中和线虫C.elegans中分别发现的PUMILIO和FBF,并以此来命名的PUF蛋白就是这样一类蛋白质[3-4]。PUF蛋白普遍存在于真核生物中,从酵母、果蝇到小鼠、人类都存在同源基因,并且在细胞分裂、分化及生殖发育方面有着非常重要的作用。PUF家族蛋白通过结合于目的mRNA的3′UTR的特定序列,并聚集其他促进mRNA降解或影响其翻译表达的蛋白质来调节其表达。PUF蛋白家族大致能够分为4个分支,其中2个分支是细胞质蛋白[5]。啤酒酵母(Saccharomyces cerevisiae)中的PUF3p,PUF4p和PUF5p是细胞质中PUF蛋白的代表,人类中的PUM1也属于胞质类[4]。
果蝇PUMILIO是最原始的一种PUF蛋白,其识别的RNA序列特异性较为简单,并且可被预测。而FBF显示出与PUMILIO不同的RNA识别特性。Hunchback(hb)mRNA是其中最早发现的一个可被PUF蛋白识别的目标,在果蝇中被PUMILIO识别,其包含一连串的纳米响应原件(Nanos Response Element,NRE),NRE 序列在 hb mRNA 的表达过程和胚胎极性建立的过程中是必不可少的[6]。hb NRE的核心序列(5′-U1G2U3A4-U/C5-A6U7A8-3′)是 PUF 蛋白识别目标位点的模型。所有的PUF蛋白都包含一个特异性序列,即RNA结合域,也被称为Pumilio同源域(Pumiliohomology domain,PUM-HD)或PUF结构域。研究发现[7-8],PUM-HD包含8个串联的α-螺旋PUF重复单元,整体呈月牙型。PUF蛋白的RNA识别序列起始于5′-UGUR(R代表嘌呤),紧接着是一些可变的3′序列,其中可能包含着一些保守元素[4]。人类PUMILIO1(PUM1)结合于hb RNA的PUM-HD的晶体结构显示了PUF重复单元识别RNA序列的大致原理[9]。RNA结合于PUM1的内凹面,每个PUF重复通过3个保守侧链识别一个RNA碱基,其中,2个侧链与RNA的碱基边缘形成氢键或者分子间作用力,第3个侧链堆叠在同一碱基或前一个碱基。RNA与蛋白质之间“反向平行”,核酸链的1~8位与蛋白质的8~1位分别相互识别(图1-a)[10]。这种简单的一个PUF重复识别一个碱基的识别模式就是典型PUF蛋白识别RNA的基本原理。
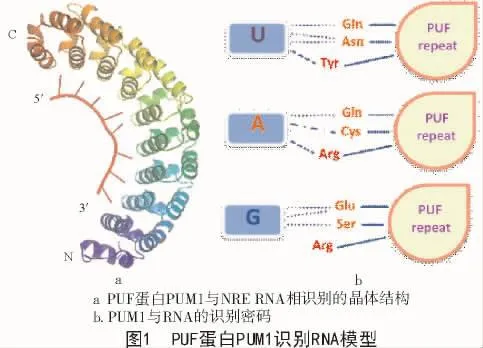
1 PUF蛋白的研究现状
1.1 结构研究
人类PUM1与RNA复合体的晶体结构说明特定的PUF重复识别特定的碱基。这种简洁的识别模式暗示着PUF蛋白结合序列的碱基特异性可被定点诱变。这种思路已被用于连接效应器结构域或荧光分子与MS2外壳蛋白,但这需要在目的RNA中插入MS2发卡序列[11]。OPPERMAN[12]研究发现,蠕虫中的2种PUF蛋白可以识别不同长度的核心序列。其中PUF-8识别8个碱基的序列,与hb NRE序列相似,而另一种PUF蛋白FBF更倾向于识别9个碱基的序列,与hb NRE序列相似,但在其第4,5个碱基之间还含有一个额外的碱基。一些研究已经证明,自然环境可以改变PUF蛋白所识别的RNA的特异性。STUMPF等[13]的试验结果显示,在线虫中RNA特异性极其相似的PUF5和PUF6蛋白都含有10个碱基的核心序列,以5′UGU开始3′UGU结束,而这2个蛋白都只含有8个重复序列,说明这些蛋白不是采用一个重复结合一个RNA碱基的模式,其碱基特异性已经被环境所改变。同样,在啤酒酵母中也存在类似的证据。酵母共表达6种PUF蛋白,这些蛋白调节不同的目的RNA[14-15],基因组学已经发现,其中的PUF4和PUF5分别识别9个和10个碱基的RNA序列。结构生物化学研究进一步阐释了PUF蛋白的特异性如何适应更长的序列。MILLER等[16]用酵母PUF4与其识别RNA序列的晶体结构,揭示了9个碱基的RNA序列如何被8个PUF重复所识别。有了PUF4的晶体结构,就知道了它是如何结合9个碱基的目的序列的,而修饰对特异性的改变是否有影响,相同的原理是否适用于其他PUF蛋白呢?对此,OPPERMAN[12]通过构建嵌合蛋白,证明了PUF蛋白的特异性可以通过改变嵌入碱基附近的蛋白质组分而得到转变。GUPTA[17]用另一种方式研究PUF蛋白对RNA识别序列的适应性,通过研究PUM1结合非同源RNA序列,结果显示,PUM1与2条非同源的RNA序列有相对较高的亲和力,证明了PUF蛋白具有通过游离碱基而优化识别目标的能力,也暗示着PUF蛋白可以进化,从而识别更长的RNA序列。
目前,PUF蛋白与RNA识别特点越来越清晰,我们也更清楚地知道,RNA结合模型是如何识别比从PUM1结构中预期的更大范围的序列。单个重复的适应性和通过把额外的核苷酸游离于RNA结合面以外的适应能力揭示了为什么PUF蛋白间的RNA结合序列是高度保守。
1.2 功能研究
随着PUF蛋白与RNA识别模式的深入研究,更多人把研究重点放在了PUF蛋白的作用上。不同的生物体间编码PUF基因的数量差异非常大,它们在细胞水平上的功能也不尽相同,目前研究的功能已经包括了细胞分化和发育[18-19]、生殖细胞[20]、神经功能与记忆[21-23]、细胞周期[21]以及线粒体生物合成[23]。一直以来,关于PUF的研究都认为,PUF蛋白的典型作用是作为转录后抑制子[4],除了这个作用,在不同的生物体中,PUF蛋白表现出不同的作用。有试验表明,PUF蛋白还有助于mRNA的激活表达[24-27]和亚细胞定位[28-30]。
WICKENS等[4]首次阐述了PUF蛋白抑制mRNA表达的机制,研究显示,酵母Puf5特异性地直接结合于Ccr4-Pop2-NOTmRNA腺嘌呤酶复合物的Pop2亚基,从而引导腺嘌呤酶到mRNA,这种细胞质核酸外切酶缩短mRNA poly(A)尾巴[31],对mRNA的稳定性和翻译都产生影响。PUF蛋白的激活子功能是一个新的概念,但在不同的生物体中有越来越多的证据表明PUF蛋白具有该功能[24-27]。目前,基于PUF蛋白的激活机制还没有很明确的定义。有证据显示[24-27],PUF蛋白对于mRMA的激活作用可能是直接的,PUF蛋白依赖性的调节作用取决于mRNA 3′UTR上的PUF结合位点。果蝇中的FBF被提到可激活另一个mRNA gel4,但其机制还不清楚,很有可能是通过PUF蛋白和microRNA共同合作实现的[26]。非洲爪蟾蜍也是PUF激活转录的一个例子,在其卵母细胞中,Pum结合元件(Pum-binding element,PBE)能促进转录激活,由细胞质聚腺嘌呤基化(cytoplasmic polyadenylation element-binding,CPEB)和其同源的细胞质聚腺苷酸化元件(cytoplasmic polyadenylation element,CPE)介导[24]。但其转录激活的要求非常严格,如果CPE是非典型的,或CPEB出现二聚体,其激活都会失败,这说明Pum的激活作用可能是因为协同作用,从而使CPEB与转录子结合更稳定[24]。动质体目原生动物的寄生虫布氏锥虫(Trypanosoma brucei)中也存在基于PUF蛋白的mRNA激活[27]。Puf9能够在其细胞周期的S期稳定mRNA,Puf9的消耗会减少与其相互作用mRNA的多样性,说明Puf9能够稳定其目标,但若突变Puf9的结合区会导致mRNA的稳定性增加[27]。对此最简单的解释是Puf9与转录抑制子相互竞争目标mRMA 3′UTR序列[27]。对于是否所有的PUF蛋白都是激活子和抑制子,或者仅限定于某些PUF蛋白,这仍然是一个值得研究的问题。
PUF蛋白还具有mRNA定位的功能,对表达的空间控制具有一定的作用。目前,大多数关于PUF蛋白的定位功能都来自于酵母。其Puf3将mRNA定位到线粒体[22,32],Puf6对ASH1转移到酵母出芽过程中能够促进其不对称定位[15],Puf5与过氧化物酶体影响PEX14 mRNA的定位[29]。此外,在果蝇的嗅觉神经元中FBF能够激活周围胞体和感觉纤毛egl-4 mRNA的翻译[26],在哺乳动物中Pum2可能参与定位神经元中mRNA翻译[30,33]。有科学家研究了酵母中Puf6和Puf3的作用机制,研究发现,Puf6调节ASH1 mRNA的翻译和定位[15],ASH1编码一个只在子细胞中表达的转录抑制子,并不在酵母母细胞中表达。这是由于非对称和编码的mRNA的表达[15]。Puf6结合于ASH1的3′-UTR,通过与翻译起始因子elF5B/Fun12相互作用,从而抑制其在转移到胚芽过程中的翻译[28]。当ASH1 mRNA到达了胚芽的目标位置,Puf6被CK2激酶磷酸化,这影响了Puf6与mRNA的结合,从而开启了ASH1抑制。Puf3与编码线粒体蛋白的mRNA相互作用[14],并促进其在线粒体中的定位[22]。有试验证明了Puf3在线粒体中的作用,Puf3与线粒体定位mRNA发生免疫共沉淀[14]、puf3诱变体中会出现mRNA的错误定位[23]、Puf3与线粒体的结合是通过其与ERMES(ER-Mitochondria Encounter Structure)的亚基Mdm12相互作用[32]。除了有助于mRNA的定位,Puf3还会抑制其mRNA并使其脱腺苷化[32,34-35],目前,还不清楚 Puf3在mRNA定位和抑制间有什么联系。
1.3 在植物中的研究进展
目前,大多数基于PUF蛋白的研究都集中在酵母、果蝇等模式生物中,也有科学家研究了拟南芥和水稻中的PUF基因家族,发现这2种模式植物的PUF基因家族成员比其他模式物种多得多,可能是由于整个基因组的复制导致了大量的PUF基因[4]。这些大量的PUF蛋白家族成员暗示着它们在细胞中对相关RNA的稳定和翻译是非常重要的。植物PUF蛋白早期只在关于植物发展史形成的文献中稍有提及[4,36-38],也有个别文献[39]提到拟南芥中PUF蛋白潜在目标mRNA的鉴定。近年来,有文献[40-41]针对植物中PUF蛋白的作用进行了研究。而植物中由于PUF基因家族数目的庞大,其功能涉及广泛,目前还未研究透彻。
2 结论
最近证据显示,PUF是多功能的mRNA调节子,可以作为抑制子、激活子以及mRNA定位因子。基于这些调节子对细胞分化、发育以及干细胞维持等过程的影响,可以认为,PUF蛋白家族采用多种机制来调节目标RNA,从而行使不同的细胞功能。PUF家族对于RNA定位的广泛作用和其识别作用与mRNA的抑制和激活是如何相互协调的都还不是很清楚。所以,目前需要解决的是PUF蛋白是如何调节众多特定的目标mRNA,是激活还是抑制,或者二者都有?PUF依赖的调节对于细胞内外的信号又是怎样变化的?更多的是,我们应该形成一种mRNA靶向功能的共同认识,这就能更清晰地知道PUF目标mRNA的亚细胞定位以及这种定位是如何依赖于PUF的,并且有助于启发我们去探索基因的时空表达与PUF作用的相互联系。
参考文献:
[1]PORTERDF.Target selection by natural and redesigned PUF proteins[J].Proceedings of the National Academy of Sciences of the U-nited States of America,2015,112(52):15868.
[2]BEITZINGERM,MEISTERG.MicroRNAs:Fromdecay todecoy[J].Cell,2010,140(5):612-614.
[3]WHARTONRP,AGGARWAL A K.mRNA regulation by Puf domain proteins[J].Science S Stke,2006,2006:e37.
[4]WICKENS M,AL E.A PUF family portrait:3′UTRregulation as a way of life[J].Trends in Genetics,2002,18:150-157.
[5]KERNERP.Evolution of RNA-binding proteins in animals:insights from genome-wide analysis in the sponge Amphimedon queenslandica[J].Molecular Biology&Evolution,2011,28(8):2289.
[6] MURATA Y,WHARTON R P.Binding of pumilio to maternal hunchback mRNA is required for posterior patterning in Drosophila embryos[J].Cell,1995,80(5):747-756.
[7] EDWARDS T,PYLE S,WHARTON R,et al.Structure of Pumilio reveals similarity between RNA and peptide binding motifs[J].Cell,2001,105(2):281.
[8] WANG X,ZAMORE P,HALL T E.Crystal structure of a pumilio homology domain[J].Molecular Cell,2001,7(4):855.
[9]WANGX.Modular recognition of RNA by a human pumilio-homology domain[J].Cell,2002,110(4):501-512.
[10]LUG,DOLGNERS J,HALL TMT.Understanding and engineering RNA sequence specificity of PUF proteins[J].Current Opinion in Structural Biology,2009,19(1):110-115.
[11] BERTRAND E.Localization of ASH1 mRNA particles in living yeast[J].Molecular Cell,1998,2(4):437.
[12] OPPERMANL.A single spacer nucleotide determines the specificities of two mRNA regulatory proteins[J].Nature Structural&Molecular Biology,2005,12:945-951.
[13]STUMPF CR,KIMBLE J,WICKENS M.A Caenorhabditis elegans PUF protein family with distinct RNA binding specificity[J].Rna-a Publication of the Rna Society,2008,14(8):1550-1557.
[14]GERBERA P,HERSCHLAGD,BROWNP O.Extensive association of functionally and cytotopically related mRNAs with Puf family rNA-binding proteins in yeast[J].Plos Biology,2012,2(3):E79.
[15] GU W.A new yeast PUF family protein,Puf6p,represses ASH1 mRNA translation and is required for its localization[J].Genes&Development,2004,18(12):1452-1465.
[16] MILLER M T,HIGGIN J J,HALL T M T.Basis of altered RNA-binding specificity by PUF proteins revealed by crystal structures of yeast Puf4p[J].Nature Structural&Molecular Biology,2008,15(4):397.
[17]GUPTA Y K.Structures of human Pumilio with noncognate RNAs reveal molecular mechanisms for binding promiscuity[J].Structure,2008,16(4):549-557.
[18] CHOP F.Cap-dependent translational inhibition establishes two opposing morphogen gradients in drosophila embryos[J].Current Biology Cb,2006,16(20):2035-2041.
[19] PRINZ S.Control of signaling in a MAP-kinase pathway by an RNA-binding protein[J].PloS One,2007,2(2):e249.
[20]KADYROVA L Y.Translational control of maternal Cyclin B mRNA by nanosin the drosophila germline [J].Development,2007,134(8):1519.
[21] TRAVEN A.The yeast PUF protein puf5 has Pop2-independent roles in response to DNA replication stress[J].PloS One,2009,5(5):e10651.
[22]ELIYAHU.Tom20 mediates localization of mRNAs tomitochondria in a translation-dependent manner[J].Molecular&Cellular Biology,2010,30(1):284-294.
[23] KERSHNER A M,KIMBLE J.Genome-wide analysis of mRNA targets for Caenorhabditis elegans FBF,a conserved stem cell regulator[J].Proceedings of the National Academy of Sciences of the United States of America,2010,107(8):3936-3941.
[24]PIQUÉ M.A combinatorial code for CPE-mediated translational control[J].Cell,2008,132(3):434.
[25] SUH N Y.FBF and its dual control of gld-1 expression in the caenorhabditis elegans germline [J].Genetics,2009,181(4):1249-1260.
[26]KAYE J A.A 3′UTRPumilio-binding element directs translational activation in olfactory sensory neurons[J].Neuron,2009,61(1):57.
[27] ARCHER S K.Trypanosoma brucei PUF9 regulates mRNAs for proteins involved in replicative processes over the cell cycle[J].PloS Pathogens,2009,5(8):e1000565.
[28]DENGY,SINGERRH,GUW.Translation of ASH1 mRNA is repressed by Puf6p-Fun12p/eIF5B interaction and released by CK2 phosphorylation [J]. Genes& Development,2008,22 (8):1037-1050.
[29]ZIPORG.Localization of mRNAs coding for peroxisomal proteins in the yeast,Saccharomyces cerevisiae[J].Proceedings of the National Academy of Sciences of the United States of America,2009,106(47):19848-19853.
[30]VESSEYJ P.Mammalian Pumilio2 regulates dendrite morphogenesis and synaptic function[J].Proceedings of the National Academy of Sciences of the United States of America,2010,107(7):3222-3227.
[31] TUCKER M.The transcription factor associated Ccr4 and Caf1 proteins are components of the major cytoplasmic mRNA deadenylase in Saccharomyces cerevisiae[J].Cell,2001,104(3):377-386.
[32]GARCÍARODRÍGUEZL J,GAY A C,PONL A.Puf3p,a Pumilio family RNA binding protein,localizes to mitochondria and regulates mitochondrial biogenesis and motility in budding yeast[J].Journal of Cell Biology,2007,176(2):197-207.
[33] VESSEY J P.Dendritic localization of the translational repressor Pumilio2 and its contribution todendritic stress granules[J].Journal of Neuroscience,2006,26(24):6496-6508.
[34]LEE D.PUF3 acceleration of deadenylation in vivocan operate independently of CCR4 activity,possibly involving effects on the PAB1-mRNP structure[J].Journal of Molecular Biology,2010,399(4):562.
[35]OLIVAS W,PARKERR.The Puf3 protein is a transcript-specific regulator of mRNA degradation in yeast[J].EmboJournal,2000,19(23):6602.
[36]GERBER A P.Genome-wide identification of mRNAs associated with the translational regulator PUMILIO in Drosophila melanogaster[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(12):4487-4492.
[37] SPASSOV D S,JURECIC R.The PUF family of RNA-binding proteins:does evolutionarily conserved structure equal conserved function?[J].Iubmb Life,2003,55(7):359.
[38] SPASSOV D S,JURECIC R.Cloning and comparative sequence analysis of PUM1 and PUM2 genes,human members of the Pumilio family of RNA-binding proteins[J].Gene,2002,299(1/2):195.
[39]FRANCISCHINI CW,QUAGGIORB.Molecular characterization of Arabidopsis thaliana PUF proteins--binding specificity and target candidates[J].Febs Journal,2009,276(19):5456.
[40]TAMP P.The Puf family of RNA-binding proteins in plants:phylogeny,structural modeling,activity and subcellular localization[J].Bmc Plant Biology,2010,10(1):44.
[41] HUANG T,KERSTETTER R A,IRISH V F.APUM23,a PUF family protein,functions in leaf development and organ polarity inArabidopsis[J].Journal of Experimental Botany,2014,65(4):1181-1191.

