复合菌剂发酵玉米秸秆对绵羊瘤胃液细菌多样性的影响
郭 威,郭晓军,周 贤,李术娜,朱宝成*
(1. 河北农业大学生命科学学院,保定 071000; 2. 河北众邦生物技术有限公司,保定 071000)
我国每年产各类农作物秸秆近8亿吨,但其饲料转化利用率不足30%。秸秆主要由纤维素、半纤维素、木质素组成,传统的秸秆加工方法不能改变秸秆的组织结构,牲畜消化吸收率低。为了将秸秆加工转化成优质饲草,提高消化吸收利用率,研究者利用微生物发酵农作物秸秆加工处理技术,有效改变了秸秆的细胞组织结构,降低了木质纤维素含量,同时生产了大量的微生物菌体蛋白,经过大量的饲喂试验显示,饲料转化率显著提高,牲畜生长速度明显加快,养殖效益增加[1-2]。李红亚等[3-4]利用傅立叶红外光谱(FTIR)和气质联用色谱(GC/MS)验证了功能菌株对木质纤维素的降解作用。李术娜等[5]研究发现,饲喂发酵玉米秸秆可显著提高育肥羊能量代谢水平,促进能量沉积与利用,提高净能。慧小双[6]用发酵玉米秸秆饲喂新吉细毛羊,不仅能够提高试羊的生长性能和瘤胃内总挥发性脂肪酸浓度,同时还可以降低甲烷排放量。
纤维素是反刍动物一种必需的营养素,瘤胃发酵产生的挥发性脂肪酸能为反刍动物提供能量需要的65%~75%[7],但是20%~70%的纤维可能不能被动物直接降解利用,绝大多数归功于瘤胃微生物的帮助[8]。瘤胃微生物数量庞大、种类繁多,共同参与反刍动物体内纤维、淀粉、蛋白质等营养物质的代谢,其中瘤胃细菌是瘤胃微生物中最大和最重要的群体[9-10]。高通量测序技术(又称第二代测序技术),具有测序通量高、准确度高、性价比高等优点,已被广泛应用于反刍动物瘤胃微生物的研究[11-16]。然而前人研究多集中在不同饲草来源和饲粮精粗比对瘤胃微生物的影响,很少考虑同种纤维来源的饲草经过不同处理后不同营养组分对瘤胃微生物的影响。
本试验选用去穗玉米秸秆为粗饲料来源,分别进行青贮和利用复合微生物菌剂发酵,借助16S rDNA高通量测序技术研究青贮和发酵玉米秸秆对绵羊瘤胃细菌群落结构多样性的影响。
1 材料与方法
1.1 玉米秸秆发酵菌剂
玉米秸秆发酵菌剂由枯草芽胞杆菌MZS-3-6、枯草芽胞杆菌XWS-8和酵母菌JM-1组成。其中枯草芽胞杆菌MZS-3-6、XWS-8具有降解纤维素和木质素的功能[3-4],枯草芽胞杆菌MZS-3-6、XWS-8和酵母菌JM-1均具有利用无机氮源生长转化为菌体蛋白的功能[1-2]。
1.2 发酵玉米秸秆的制作
取去穗青玉米秸秆,添加河北众邦生物技术有限公司生产的复合微生物发酵菌剂(有效活菌数1.0×1010CFU·g-1,100 g/T)、硫酸铵(3.0%/T),混合均匀后装袋厌氧发酵60 d。青贮不加发酵菌剂。青贮、发酵玉米秸秆的主要营养组分见表1。
1.3 试验动物和样品采集
选用装有瘤胃瘘管的南非肉用美利奴羊(♂)×东北细毛羊(♀)的F1代成年公羊12只,随机分为两组,每组6只。日粮参考美国NRC(1985)绵羊饲养标准配制,营养水平见表2。分别在饲喂前1 d,饲喂第7 天、第21 天晨饲后6 h取瘤胃液,每组同一时间所取样品混合均匀,分别命名为青贮组(YD0、YD7、YD21)和发酵组(YS0、YS7、YS21)。
表1发酵及青贮玉米秸秆的主要营养成分比较(干物质基础)
Table1Themainnutrientcomponentsoffermentedandsilagecornstalk(DMbasis) %

表2试验饲粮组成及营养水平(干物质基础)
Table2Compositionandnutrientlevelsofexperimentaldiets(DMbasis) %
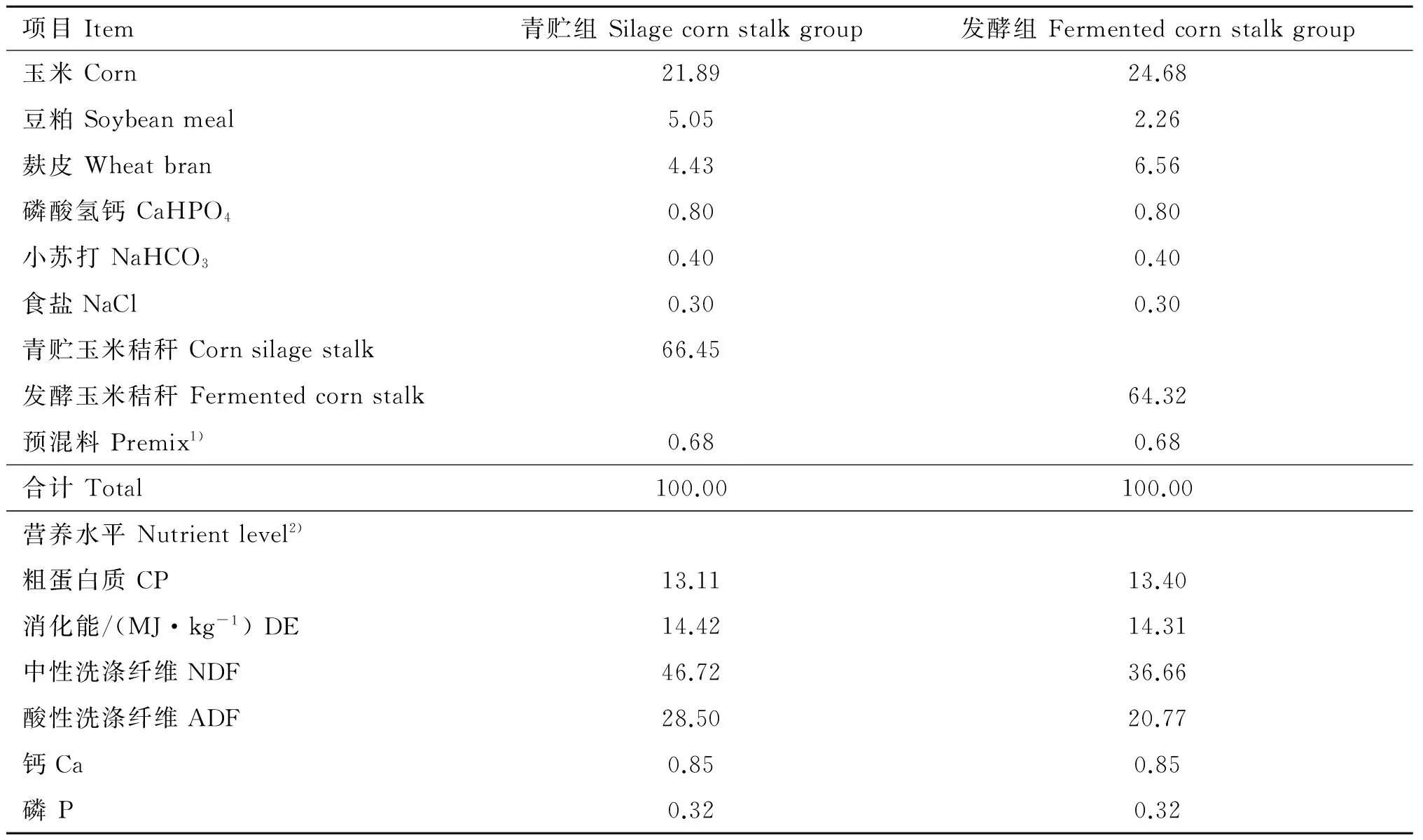
1). 预混料为每千克饲粮提供:VA 15 000 IU,VD 5 000 IU,VE 50 mg,Fe 90 mg,Cu 12.5 mg,Mn 50 mg,Zn 100 mg,I 0.8 mg,Co 0.5 mg;2). CP、Ca、P为实测值,DE、NDF、ADF为计算值
1). The premix provided the following per kg of diets:VA 15 000 IU,VD 5 000 IU,VE 50 mg,Fe 90 mg,Cu 12.5 mg,Mn 50 mg,Zn 100 mg,I 0.8 mg,Co 0.5 mg;2). CP, Ca and P were measured values, while DE, NDF and ADF were calculated values
1.4 DNA提取和扩增
每个样品用4层绵纱布过滤,滤液经12 000 r·min-1离心15 min,取其沉淀。用DNA提取试剂盒(TIANGEN)提取瘤胃液细菌样本。每个样本提取两次DNA,将两次提取的DNA混合均匀后作为16S rDNA和序列分析的模板。DNA的质量和纯度用Nanodrop 2000检测。
1.5 高通量测序
利用Hiseq测序装置对各样品的16S rDNA基因V3~V4区进行扩增。引物选用340F(5′-CCTACGGGNBGCASCAG-3′)和805R(5′-GACTACNVGGGTATCTAATCC-3′),其中引物340F的5′尾端带有条形码标记。PCR反应体系(50 μL):5 μL 10×Buffer A,1 μL dNTP(10 mmol·L-1),2 μL Primer R,2 μL Primer F,0.2 μL KAPA Taq,1 μL gDNA和38.8 μL ddH2O。PCR程序:95 ℃预变性3 min;95 ℃变性30 s,50 ℃退火30 s,72 ℃延伸60 s,30个循环;72 ℃保温7 min。PCR产物用1.5%琼脂糖凝胶电泳检测。用NEXTflexTMRapid DNA-Seq工具包构建序列库,并用HiSeq 2500进行测序分析。
1.6 数据处理
16S测序分析采用QIIME[17]进行质量控制。质控后的序列按照97%的相似性分别聚到一个操作分类单元(OTU)。此外,利用RDP Classifier[18]对OTUs进行分类学注释。将具有代表性的序列系统分类到界、门、纲、目、科、属、种的水平。
Alpha丰富度和多样性分析包括ChaoI、ACE指数、Coverage、Shannon、Simpson指数。Beta多样性分析,选取属层次的菌落丰度结果,对样本在属
层次进行聚类,绘制Heatmap图。
2 结 果
2.1 绵羊瘤胃液细菌OTU数量和Alpha多样性分析
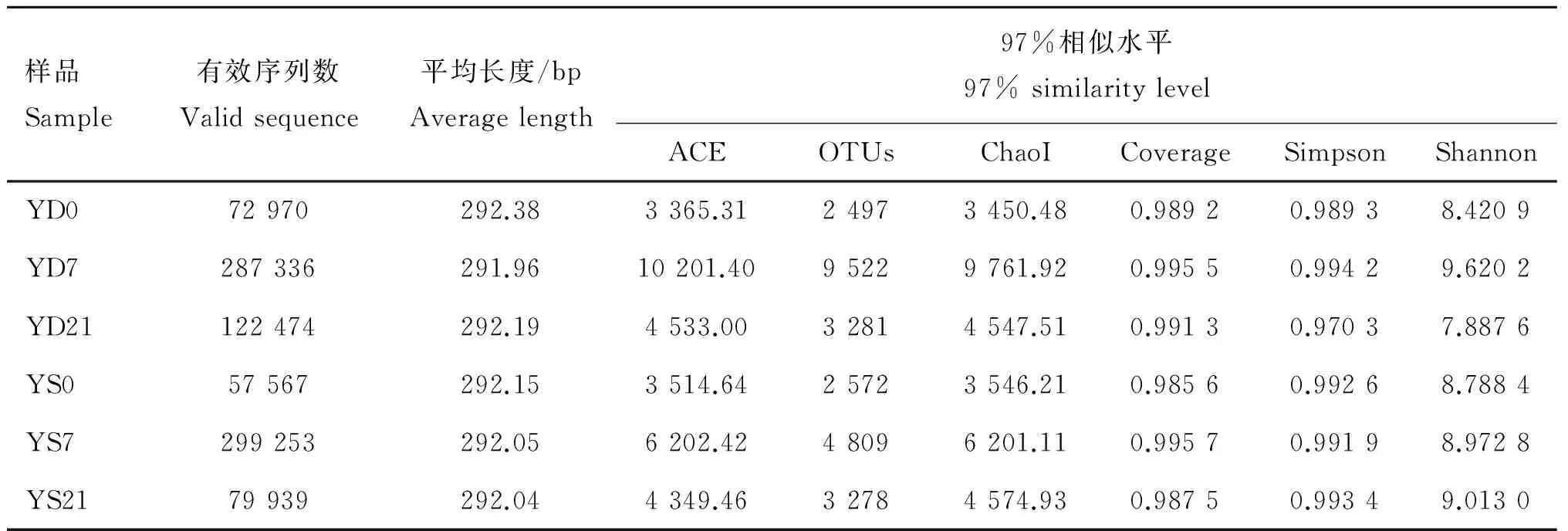
绵羊瘤胃液细菌OTU数目和Alpha多样性指数见表3。本试验共得到919 539条有效序列,其中青贮组482 780条,发酵组436 759条;平均每个样品有153 256条有效序列(57 567~299 253条不等)。所有序列按照97%相似性水平划分成不同的聚类单元(OTU),各个样品得到的OTU数量差别很大(2 497~9 522个OTU不等),平均序列长为292.04 bp。其中YD7的OTU数量最多(9 522),其Alpha多样性亦最高(ACE和ChaoI指数分别为10 201.40 和9 761.92);YD0的OTU数量最少(2 497),Alpha多样性亦最低(ACE和ChaoI指数分别为3 365.31和3 450.48)。YS21与YD21的OTU数量相近,但YS21的Alpha多样性指数更高。样品的覆盖率(Coverage)均在0.98以上,表明能够较好地反映每个样本中瘤胃细菌群落的组成和多样性。
表3样本OTU数量和Alpha多样性
Table3OTUnumberandAlphadiversityofsamples
样品Sample有效序列数Validsequence平均长度/bpAveragelength97%相似水平97%similaritylevelACEOTUsChaoICoverageSimpsonShannonYD072970292.383365.3124973450.480.98920.98938.4209YD7287336291.9610201.4095229761.920.99550.99429.6202YD21122474292.194533.0032814547.510.99130.97037.8876YS057567292.153514.6425723546.210.98560.99268.7884YS7299253292.056202.4248096201.110.99570.99198.9728YS2179939292.044349.4632784574.930.98750.99349.0130
2.2 绵羊瘤胃液细菌分类学组成
经过分类学比对,6个样品共鉴定得到29个门,74个纲,135个目,215个科,428个属。
所有样本均由拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、纤维杆菌门(Fibrobacteres)、黏胶球形菌门(Lentisphaerae)4个优势菌门主导,其中others是丰度排名位于前20名以外的分类比例(图1)。青贮组以拟杆菌门为优势菌群,其次为黏胶球形菌门、厚壁菌门和纤维杆菌门。发酵组亦以拟杆菌门为优势菌群,其次为厚壁菌门、黏胶球形菌门和纤维杆菌门。经过21 d的饲喂,优势菌门在各组的丰度有所变化。青贮组的拟杆菌门从饲喂前的39.63%提高到42.46%,发酵组由42.20%提高到55.41%而后又回降到46.13%。作为第二优势菌门,黏胶球形菌门在青贮组增加了37.14%(由17.05%增加到23.38%),在发酵组增加了22.59%(由11.38%增加到13.95%)。青贮组厚壁菌门从开始到试验结束降低了36.04%,而发酵组变化甚微。拟杆菌门与厚壁菌门丰度之和在青贮组降低了4.66%(由54.53%下降到51.99%),而在发酵组升高了6.92%(由56.64%提高到70.81%而后又降到60.56%)。纤维杆菌门丰度在青贮组增加了85.92%,而在发酵组只增加了30.75%,青贮组增加量大于发酵组。说明用发酵玉米秸秆替代青贮玉米秸秆饲喂绵羊对瘤胃微生物组成结构具有很大影响。
其他菌门占所有序列的比例较低,但其中有些菌门也在饲料降解过程中起着重要的作用。如螺旋体门(Spirochaetae)可以有效降解纤维素、果胶和磷酸酯,发酵形成挥发性脂肪酸,为动物机体提供能量。赵圣国[19]研究发现,65.25%的脲酶是由瘤胃中变形门(Proteobacteria)类细菌产生,田雨佳[20]也进一步证实了瘤胃可以降解一些非蛋白氮源的研究结论。螺旋体门所占比例在青贮组降低了40.38%,而在发酵组中增加了13.07%。本研究发酵组YS21中变形菌门较青贮组YD21丰度更高,表明发酵过程中残留的非蛋白氮可能较好地被利用了。

图1 全样本在门层次的菌落结构Fig.1 Community structure histogram of all samples at phylum level
在属水平上,共检测到428个属。其中,拟杆菌门理研菌科RC9肠道菌体属(RikenellaceaeRC9 gut group)和普雷沃氏菌(Prevotella)、黏胶球形菌门中的RFP12肠道菌属(LentisphaeraeRFP12 gut group)、纤维杆菌属(Fibrobacter)和食物谷菌属(Victivallis)是所有样品的优势菌属(图2)。Prevotella降解底物宽泛,饲喂21 d后,青贮组绵羊瘤胃液中Prevotella的丰度较饲喂前降低了69.77%,发酵组较饲喂前升高了68.05%,而Victivallis的丰度在两组中的变化趋势恰恰相反。LentisphaeraeRFP12 gut group和Fibrobacter的丰度在两组中都有所增加,但青贮组增加的幅度大于发酵组。
除分解利用木质纤维素外,多数瘤胃细菌还有降解粗蛋白的能力。瘤胃中主要的蛋白降解菌包括嗜淀粉瘤胃杆菌(Ruminobacteramylophilus)、栖瘤胃普雷沃氏菌(Prevotella)和溶纤维丁酸弧菌(Butyrivibriofibrisolvens)。Prevotella是瘤胃中数量最多的蛋白分解菌。本研究中,Prevotella在发酵组YS7(相对丰度达到15.56%)时成为最优势菌属,而在青贮组大大降低(相对丰度由10.48% 降低到3.17%)。而作为主要的瘤胃蛋白分解菌,丁酸弧菌属(Butyrivibrio)在两组绵羊瘤胃液中丰度均小于1%。此外,厚壁菌门中的反刍兽新月形单胞菌属(Selenomonas)在瘤胃除具有代谢乳酸的作用[21]外,同时还具有一定的蛋白降解活性。但其在两组绵羊瘤胃液中的丰度均小于0.1%。
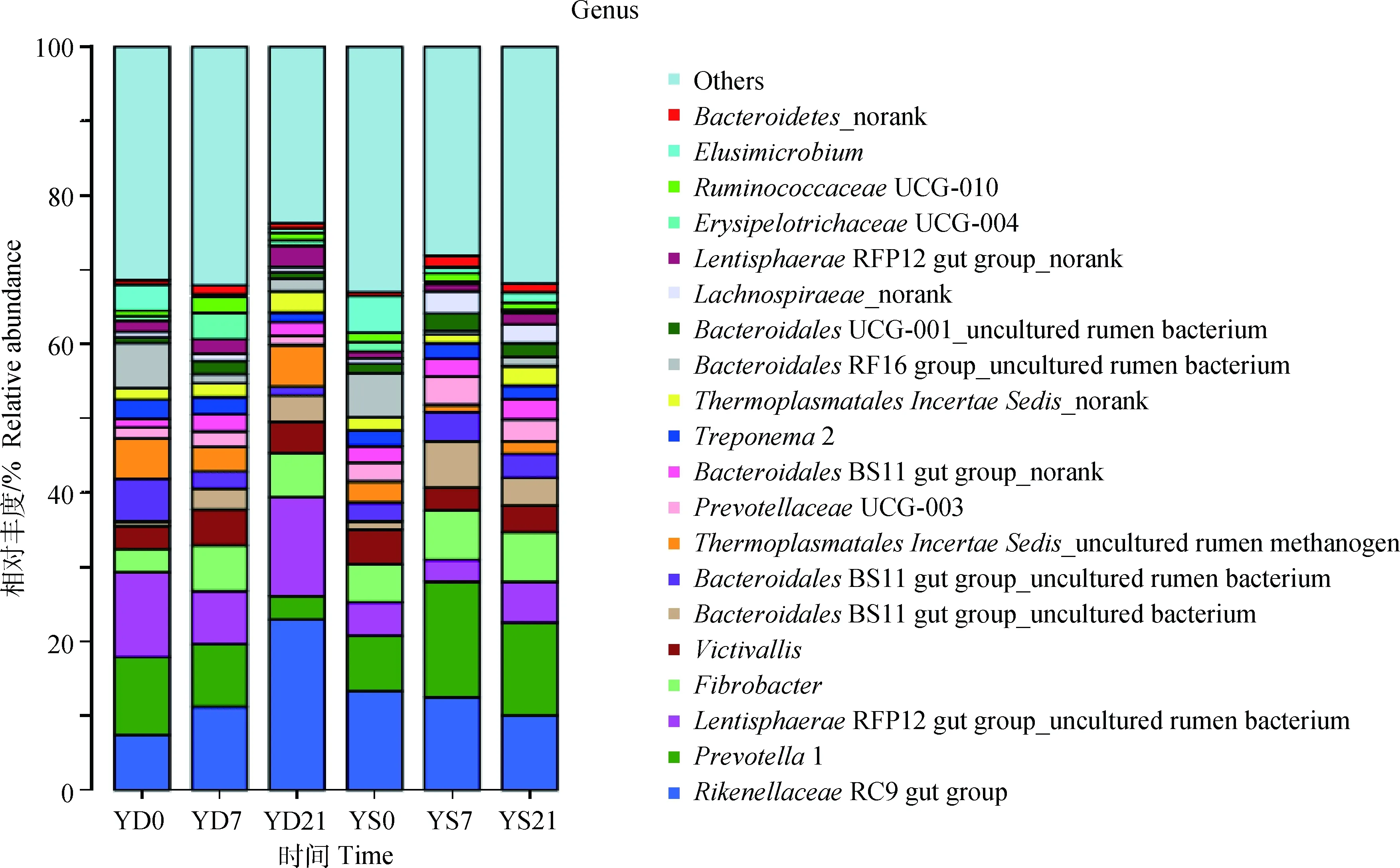
图2 全样本在属层次的菌落结构Fig.2 Community structure histogram of all samples at genus level
2.3 OTU组成分析
图3为样本基于前50个属菌落丰度的Heatmap图。Heatmap图体现了6个样品间的相似性和差异性。Prevotella在YS7的相对丰度最大,YD7和YS21次之,而在YD21的相对丰度最小。6个 样品分成两大簇,左边4个样品(YS7、YS21、YD7、YS21)聚成一大簇,右边2个样品(YD0和YS0)聚成一簇。其中,YS7和YS21、YD7和YD21各自聚成一小簇。结果证明,饲喂7 d后绵羊瘤胃内细菌群落趋于稳定,而饲喂不同营养组分的青贮和发酵玉米秸秆对绵羊瘤胃液的细菌群落多样性的影响有一定的差异。
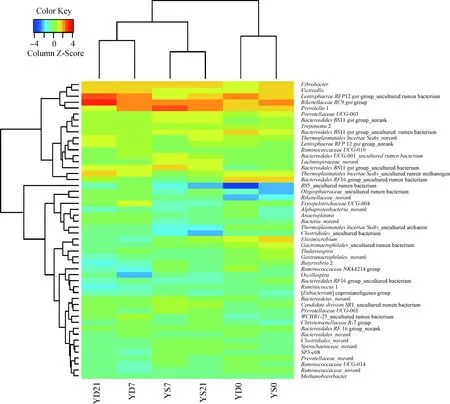
图3 样本在属水平的菌落丰度Fig.3 Community abundance heatmap of samples at genus level
3 讨 论
M.L.Sogin等[22]首次应用16S rDNA高通量测序技术对深海微生物群落进行了分析,近几年该技术逐渐被应用到反刍动物瘤胃微生物群落结构[23-25]的研究中。本试验利用16S rDNA高通量测序技术对饲喂发酵玉米秸秆的绵羊瘤胃细菌多样性进行研究。通过质控共得到919 539条有效序列用于后续多样性组成分析。各样本瘤胃液稀释曲线趋于平缓,表明测序深度已经达到多样性分析的要求,且样本的覆盖率均在0.98以上,表明焦磷酸测序库能够代表大部分的OTUs。两组样本Alpha多样性差异较大,表明样本间细菌数量及组成结构上差异较大,且发酵组YS21较青贮组YD21拥有更高的Shannon和Simpson指数,以上结果均表明饲喂发酵玉米秸秆对瘤胃细菌的多样性影响更大。
大多数研究报道,在门水平上拟杆菌门和厚壁菌门是反刍动物瘤胃内的主要微生物菌群[26-27],表明两菌门在反刍动物瘤胃内食物代谢的过程中具有重要作用。本研究发酵组得到了相同的结果,其中发酵组优势菌群是拟杆菌门,其次是厚壁菌门,这与R.Y.Zhang等[28]采用高通量测序技术研究不同饲草对奶牛瘤胃细菌组成和多样性的结果一致。但本研究结果与S.E.Hook等[29]用高谷物饲粮饲喂奶牛瘤胃中厚壁菌门细菌占主导地位、拟杆菌门次之的结果相反,究其原因可能是我们采用的是高粗料饲粮。两菌门丰度之和在发酵组呈升高趋势,而在青贮组有所降低,表明低NDF粗料日粮能够提高拟杆菌门和厚壁菌门在所有细菌菌群中的比例。本试验青贮组黏胶球形菌门丰度远高于发酵组,据报道黏胶球形菌门与纤维二糖的降解有关[30-31],高含量的黏胶球形菌门可能归因于青贮玉米秸秆中NDF等纤维类物质含量高的缘故,这与R.Y.Zhang等[28]研究结果一致。与纤维降解有关的纤维杆菌门有着与其相似的变化趋势,其在青贮组增加幅度远超发酵组,这与D.W.Pitta等[16]报道的随着日粮中纤维含量的增加纤维杆菌门的丰度亦增加的结果相一致。
在属水平上,本试验的差异主要是RikenellaceaeRC9 gut group(在青贮组增加了2.1倍)、Prevotella(发酵组增加了68%)、LentisphaeraeRFP12 gut group和Fibrobacter。Prevotella是瘤胃内数量最为丰富的一类细菌[13,15],可达瘤胃微生物总量的60%~70%[32],能够高效降解半纤维素[33],有效利用植物非纤维多糖和蛋白质[34],并对淀粉、木聚糖和果胶的降解起重要作用[35]。D.J.Grilli等[36]在研究山羊从饲草到精料的转变中瘤胃细菌多样性的变化时发现,在饲喂高饲草的山羊瘤胃中Prevotella的丰度更高。据报道,D.W.Pitta等[16]用高粗蛋白的小麦秸秆饲喂肉牛发现,肉牛瘤胃液中Prevotella含量显著增加。本研究中,Prevotella的相对丰度在发酵组更高,这与 I.S.Cunha等[37]报道的山羊瘤胃中Prevotella为优势菌属结果相一致。
发酵玉米秸秆中的NDF等纤维素类物质较青贮玉米秸秆显著降低,故推断青贮组绵羊瘤胃液中的纤维降解菌含量及丰度应更高,本研究恰恰证实了这一观点。属水平的群落结构图显示,青贮组中具有纤维分解功能的纤维杆菌属丰度升高幅度大于发酵组。但与刘大程等[38]研究发现的随着粗饲料品质的升高产琥珀酸丝状杆菌数量有增加趋势的结果相反。很可能与纤维降解有关的细菌,不仅与纤维降解菌有关还与其他菌有关,S.Koike等[39]就曾报道,纤维的降解在纤维降解菌与非纤维降解菌的相互作用下会加速。此外,高比例的未知分类的菌属使饲草的评价很难进行,也体现了我们在绵羊瘤胃细菌组成知识方面的欠缺。但是,我们的结果基本上支持核心微生物组的概念。在本研究得到的29个门里,有14个门(相对丰度之和达到了99.5%以上)在所有样本中被检测到,这意味着他们在瘤胃内饲料的降解中占有特殊的生态地位。
Heatmap图证实了两组样本细菌组成的不同,这可能通过简单的分类学注释和Alpha多样性不能明显地比较出来。基于前50个属的菌落丰度Heatmap图显示,饲喂7和21 d后同组样品聚在一起,然而不同细菌属在不同样本中呈现不同的丰度。说明饲喂7 d后,绵羊瘤胃液细菌群落结构趋于稳定,而饲喂等能等氮但NDF含量不同的日粮对绵羊瘤胃细菌区系具有较大影响。
4 结 论
4.1饲喂发酵玉米秸秆与饲喂青贮玉米秸秆对绵羊瘤胃细菌多样性影响具有较大差异,且饲喂发酵玉米秸秆的瘤胃细菌多样性更高。
4.2饲喂发酵玉米秸秆绵羊瘤胃液的拟杆菌门和厚壁菌门的丰度下降,而黏胶球形菌门和纤维杆菌门的菌群数量增加。
参考文献(References):
[1] 朱宝成, 王来福. 秸秆发酵生产高蛋白饲草技术[J]. 中国畜牧业, 2014(10): 60-62.
ZHU B C, WANG L F. Technology of straw fermentation producing high protein forage[J].ChinaAnimalIndustry, 2014(10): 60-62. (in Chinese)
[2] 朱宝成. 超级人工体外瘤胃——秸秆发酵生产高蛋白饲草技术[J]. 北方牧业, 2014(17): 14-16.
ZHU B C. Super artificialinvitrorumen——technology of straw fermentation producing high protein forage[J].TheAnimalHusbandry, 2014(17): 14-16. (in Chinese)
[3] 李红亚, 李术娜, 王树香, 等. 产芽孢木质素降解菌MN-8的筛选及其对木质素的降解[J]. 中国农业科学, 2014, 47(2): 324-333.
LI H Y, LI S N, WANG S X, et al. Screening, identification of lignin-degradatingBacillusMN-8 and its characteristics in degradation of maize straw lignin[J].ScientiaAgriculturaSinica, 2014, 47(2): 324-333. (in Chinese)
[4] 李红亚, 李术娜, 王树香,等. 解淀粉芽孢杆菌MN-8对玉米秸秆木质纤维素的降解[J]. 应用生态学报, 2015, 26(5): 1404-1410.
LI H Y, LI S N, WANG S X, et al. Degradation of lignocellulose in the corn straw byBacillusamyloliquefaciensMN-8[J].ChineseJournalofAppliedEcology, 2015, 26(5): 1404-1410. (in Chinese)
[5] 李术娜, 惠小双, 李红亚, 等. 多功能复合菌剂发酵玉米秸秆对育肥羊能量代谢的影响[J]. 动物营养学报, 2015, 27(7): 2231-2240.
LI S N, HUI X S, LI H Y,et al.Effects of multifunctional complex microbial agent fermented corn straw on energy metabolism of fattening sheep[J].ChineseJournalofAnimalNutrition, 2015, 27(7): 2231-2240. (in Chinese)
[6] 慧小双. 发酵玉米秸秆饲料对育肥羊氮、碳及能量代谢影响的研究[D]. 保定: 河北农业大学, 2013.
HUI X S.Effects of fermented corn straw on performance,energy metabolism and nitrogen, carbon deposition of fattening sheep[D]. Baoding: Hebei Agricultural University, 2013. (in Chinese)
[7] 王尧悦, 赵钊艳, 王兴涛, 等. 日粮营养水平对150~180日龄滩羊瘤胃相关微生物菌群数量、pH和VFA含量的影响[J]. 畜牧兽医学报, 2016, 47(10): 2060-2070.
WANG Y Y, ZHAO Z Y, WANG X T, et al. Effect of dietary nutrient levels on the number of related microbes, pH and VFA levels in rumen of tan sheep aged from 150 to 180 days[J].ActaVeterinariaetZootechnicaSinica, 2016, 47(10): 2060-2070. (in Chinese)
[8] KOIKE S, YOSHITANI S, KOBAYASHI Y,et al.Phylogenetic analysis of fiber-associated rumen bacterial community and PCR detection of uncultured bacteria[J].FEMSMicrobiolLett, 2003, 229(1): 23-30.
[9] KAMRA D N. Rumen microbial ecosystem[J].CurrSci, 2005, 89(1): 124-135.
[10] 廖 奇, 刘旭川, 李 清, 等. RNA-Seq技术在瘤胃微生物研究中的应用进展[J]. 动物营养学报, 2015, 27(4): 1061-1067.
LIAO Q, LIU X C, LI Q, et al. RNA-Seq technology and its application in rumen microbes[J].ChineseJournalofAnimalNutrition, 2015, 27(4): 1061-1067. (in Chinese)
[11] 李岚捷, 成述儒, 刁其玉, 等. 不同NFC/NDF水平饲粮对犊牛瘤胃发酵参数和微生物区系多样性的影响[J]. 畜牧兽医学报, 2017, 48(12): 2347-2357.
LI L J, CHENG S R, DIAO Q Y, et al. Effects of diets with different NFC/NDF levels on the rumen fermentation parameters and bacterial community in male calves[J].ActaVeterinariaetZootechnicaSinica, 2017, 48(12): 2347-2357. (in Chinese)
[12] HESS M, SCZYRB A, EGAN R,et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen[J].Science, 2011, 331(6016): 463-467.
[13] KIM M, MORRISON M, YU Z T. Status of the phylogenetic diversity census of ruminal microbiomes[J].FEMSMicrobiolEcol, 2011, 76(1): 49-63.
[14] THOETKIATTIKUL H, MHUANTONG W,LAOTHANACHAREON T,et al. Comparative analysis of microbial profiles in cow rumen fed with different dietary fiber by tagged 16S rRNA gene pyrosequencing[J].CurrMicrobiol, 2013, 67(2): 130-137.
[15] BEKELE A Z, KOIKE S, KOBAYASHI Y. Genetic diversity and diet specificity of ruminalPrevotellarevealed by 16S rRNA gene-based analysis[J].FEMSMicrobiolLett, 2010, 305(1): 49-57.
[16] PITTA D W, KUMAR S,VEICCHARELLI B,et al. Bacterial diversity associated with feeding dry forage at different dietary concentrations in the rumen contents of Mehshana buffalo (Bubalusbubalis) using 16S pyrotags[J].Anaerobe, 2014, 25: 31-41.
[17] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J].NatMethods, 2010, 7(5): 335-336.
[18] WANG Q, GARRITY G M,TIEDJE J M, et al. Naïve bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].ApplEnvironMicrobiol, 2007, 73(16): 5261-5267.
[19] 赵圣国. 牛瘤胃脲酶基因多样性分析与酶活性调控[D]. 北京: 中国农业科学院, 2012.
ZHAO S G. Diversity of urease gene and regulation of urease activity in the rumen of cattle[D]. Beijing: Chinese Academy of Agricultural Sciences, 2012. (in Chinese)
[20] 田雨佳. 苜蓿干草与苜蓿青贮对奶牛瘤胃蛋白质利用的影响及作用机制的研究[D]. 北京: 中国农业大学, 2014.
TIAN Y J.Study on mechanism of nitrogen utilization in rumen of dairy cows with alfalfa as hay or silage[D]. Beijing: China Agricultural University, 2014. (in Chinese)
[21] 毛胜勇, 龙黎明, 朱伟云. 体外研究反刍兽新月形单胞菌及与酵母联用对瘤胃微生物发酵的影响[J]. 草业学报, 2010, 19(4): 176-186.
MAO S Y, LONG L M,ZHU W Y.Effect ofSelenomonasruminantiumalone, or in combination with yeast cultures, oninvitrorumen bacterial fermentation[J].ActaPrataculturaeSinica, 2010, 19(4): 176-186. (in Chinese)
[22] SOGIN M L, MORRISON H G, HUBER J A, et al. Microbial diversity in the deep sea and the underexplored “rare biosphere”[J].ProcNatlAcadSciUSA, 2006, 103(32): 12115-12120.
[23] MAO S Y, ZHANG R Y, WANG D S, et al. Impact of subacute ruminal acidosis (SARA) adaptation on rumen microbiota in dairy cattle using pyrosequencing[J].Anaerobe, 2013, 24: 12-19.
[24] MYER P R, KIM M, FREETLY H C, et al. Evaluation of 16S rRNA amplicon sequencing using two next-generation sequencing technologies for phylogenetic analysis of the rumen bacterial community in steers[J].JMicrobiolMethods, 2016, 127: 132-140.
[25] GHARECHAHI J, ZAHIRI H S, NOGHABI K A, et al. In-depth diversity analysis of the bacterial community resident in the camel rumen[J].SystApplMicrobiol, 2015, 38(1): 67-76.
[26] SINGH K M, AHIR V B, TRIPATHI A K,et al.Metagenomic analysis of Surti buffalo (Bubalusbubalis) rumen: a preliminary study[J].MolBiolRep, 2012, 39(4): 4841-4848.
[27] DE OLIVEIRA M N, JEWELL K A, FREITAS F S, et al. Characterizing the microbiota across the gastrointestinal tract of a Brazilian Nelore steer[J].VetMicrobiol, 2013, 164(3-4): 307-314.
[28] ZHANG R Y, ZHU W Y, ZHU W, et al. Effect of dietary forage sources on rumen microbiota, rumen fermentation and biogenic amines in dairy cows[J].JSciFoodAgric, 2014, 94(9): 1886-1895.
[29] HOOK S E, STEELE M A, NORTHWOOD K S, et al. Impact of subacute ruminal acidosis (SARA) adaptation and recovery on the density and diversity of bacteria in the rumen of dairy cows[J].FEMSMicrobiolEcol, 2011, 78(2): 275-284.
[30] ZOETENDAL E G, PLUGGE C M, AKKERMANS A D L, et al.Victivallisvadensisgen. nov., sp. nov., a sugar-fermenting anaerobe from human faeces[J].IntJSystEvolMicrobiol, 2003,53(Pt 1): 211-215.
[31] LIMAM R D, BOUCHEZ T, CHOUARI R, et al. Detection of WWE2-relatedLentisphaeraeby 16S rRNA gene sequencing and fluorescenceinsituhybridization in landfill leachate[J].CanJMicrobiol, 2010, 56(10): 846-852.
[32] MEYER M, STENZEL U,HOFREITER M.Parallel tagged sequencing on the 454 platform[J].NatProtoc, 2008, 3(2): 267-278.
[33] MATSUI H, OGATA K, TAJIMA K, et al. Phenotypic characterization of polysaccharidases produced by fourPrevotellatype strains[J].CurrMicrobiol, 2000, 41(1): 45-49.
[34] BEKELE A Z, KOIKE S, KOBAYASHI Y. Phylogenetic diversity and dietary association of rumenTreponemarevealed using group-specific 16S rRNA gene-based analysis[J].FEMSMicrobiolLett, 2011,316(1):51-60.
[35] PURUSHE J, FOUTS D E, MORRISON M,et al. Comparative genome analysis ofPrevotellaruminicolaandPrevotellabryantii: insights into their environmental niche[J].MicrobEcol, 2010, 60(4): 721-729.
[37] CUNHA I S, BARRETO C C, COSTA O Y A, et al. Bacteria and Archaea community structure in the rumen microbiome of goats (Caprahircus) from the semiarid region of Brazil[J].Anaerobe, 2011, 17(3): 118-124.
[38] 刘大程, 卢德勋, 侯先志, 等. 不同品质粗饲料日粮对瘤胃发酵及主要纤维分解菌的影响[J]. 中国农业科学, 2008, 41(4): 1199-1206.
LIU D C, LU D X, HOU X Z, et al. Effect of dietary forage of different qualities on rumen fermentation and predominant fibrolytic bacterial populations[J].ScientiaAgriculturaSinica, 2008, 41(4): 1199-1206. (in Chinese)
[39] KOIKE S, PAN J, KOBAYASHI Y,et al. Kinetics of in Sacco fiber-attachment of representative ruminal cellulolytic bacteria monitored by competitive PCR[J].JDairySci, 2003, 86(4): 1429-1435.

