小鼠miR-125a超表达载体的构建
,,,,,( , 712100)
精子发生是指精子在睾丸内产生的全过程,包括精细管上皮的生精细胞由精原细胞经过精母细胞到精子细胞的増殖分化过程和精子形成过程。哺乳动物中,雄性生殖细胞发育成为精子的过程受到外因和内因的调控[1]。据估计大约有1 000个基因参与人精子发生的过程,其中仅有351个在生殖细胞中表达,这些基因在转录水平以及翻译水平调控精子发生[2]。这些基因功能的发挥需要大量的microRNAs对其在转录水平和翻译水平上进行调控。
microRNAs (miRNAs)是一类长度约为22个核苷酸、在进化上高度保守的非编码单链RNA分子。在人类基因组中有2%~3%的基因编码miRNAs[3]。目前已经有大量报道miRNAs的表达具有组织特异性,并且已经证明miRNAs在调节细胞的增殖和分化过程中起重要作用[4-6]。同时,越来越多的研究表明miRNAs对于调节精子发生及维持雄性生育是必需的;如缺乏形成miRNAs必需的Dicer酶小鼠,产生异常伸长的精子,并且呈现雄性不育[7];生殖干细胞的分裂及干细胞通过G1/S关卡需要miRNAs的参与[8]。Mmu-miR-125a35p(后文统称为miR-125a)位于小鼠基因组第17号染色体上,与mmu-miR-99b和mmu-let-7e成簇分布。研究发现通过抑制miR-125a的表达能够促进人胚胎干细胞向神经前体细胞的转变[9],然而关于miR-125a在哺乳动物雄性生殖细胞中的研究还未见报道。
为了探明miR-125a在精子发生中的功能,本研究构建了miR-125a的超表达载体,并初步验证其超表达效果,为进一步研究miR-125a的功能及其作用机制奠定了基础。
1 材料与方法
1.1 试验动物与主要试剂
7日龄和7周龄公猪采购于陕西杨凌某猪场;GC-1spg细胞系由上海交通大学何祖平教授惠赠,由本试验室传代冻存;大肠杆菌DH5α及质粒载体pcDNA3.1(+)为本试验室保存;DMEM高糖培养基、胎牛血清和0.25%胰酶购于Gibico公司;反转录试剂盒(RR047A)、荧光定量检测试剂盒(DRR820S)、Taq DNA聚合酶、DNA限制性内切酶(HindⅢ、BamHⅠ)和T4 DNA连接酶购于大连宝生物工程有限公司;胶回收试剂盒和质粒提取试剂盒购于OMEGA公司; Trizol,Lipofectamine 2000 Reagent,opti-MEM购于Invitrogen公司;小鼠抗GAPDH和山羊抗C-KIT多克隆抗体购于Santa Cruz公司;蛋白裂解液RIPA、PMSF、HRP标记山羊抗小鼠和HRP标记兔抗山羊IgG购于北京博奥森生物技术有限公司;BCA蛋白浓度测定试剂盒购于Thermo公司;引物合成于上海生工公司。
1.2 MiR-125a、Gapdh、C-kit和Pcna的定量检测
1.2.1 引物设计与合成 参照基因库提供的鼠Gapdh(NM_008085.1)、C-kit(NM_001122733.1)和Pcna(NM_011045.2)的完整序列设计引物,引物跨外显子设计;根据Chen文献[10]设计miR-125a的反转录和PCR引物,引物序列和扩增长度如表1。
1.2.2 反转录与实时定量 RNA提取:Trizol试剂常规法抽提组织和细胞总RNA,DEPC水溶解沉淀,核酸蛋白分析仪(ND-1000 Spectrometer, USA)测定RNA浓度和纯度,根据RNA在A260 nm/A280 nm≥1.8及凝胶电泳28S/18SRNA条带比值≥1.5鉴定RNA纯度及完整性。

表1 引物序列Table 1 Primers sequence
miR-125a和内参基因的反转录反应:RNA 1 μg、5×gDNA Eraser Buffer 2 μL、gDNA Eraser 1 μL,DEPC水补足10 μL至一无RNase的PCR管中;42 ℃孵育3 min,4 ℃暂时保存。随后加入5×Prime Script® Buffer 4 μL、Prime Script® RT Enzyme Mix I 1 μL、miR-125a RT引物1 μL(或者RT Primer Mix 4 μL),DEPC水补足20 μL;16 ℃孵育30 min,42 ℃(或者37 ℃)孵育30 min,85 ℃孵育1 min,4 ℃保存。
定量PCR反应:利用SYBR Green 荧光染料法于定量PCR仪(IQ5,BioRad)检测miR-125a、Gapdh、C-kit和Pcna的表达。反应体系如下:cDNA 1 μL、Primers(F+R)2 μL、SYBR GreenI荧光染料10 μL、灭菌水7 μL。反应条件:95 ℃ 10 min;95 ℃ 15 s、60 ℃ 30 s、72 ℃ 30 s,共45个循环;72 ℃ 10 min,每个样品均作三复管PCR反应。数据在Excel中采用2-△△Ct法分析汇总。
1.3 MiR-125a超表达载体的构建与转染
1.3.1 MiR-125a 前体PCR引物设计与合成 在UCSC数据库搜索得到小鼠miR-125a前体序列(pre-miR-125a),并且向其两端侧翼各延伸150 bp得到一段约380 bp的序列,利用Premier 5设计含有酶切位点的引物,引物序列见表1。下划线分别为Hind Ⅲ和BamHⅠ的酶切位点。
1.3.2 MiR-125a 真核表达载体(pcDNA3.1(+)-miR-125a)的构建 PCR反应扩增pre-miR-125a:以C57小鼠基因组DNA为模板,pre-miR-125a-F和pre-miR-125a-R分别为上下游引物,进行PCR扩增。体系如下:模板DNA 1μL、Primers(F+R)2 μL、2×Taq mix 10 μL、灭菌水7 μL。反应条件:95 ℃ 5 min;94 ℃ 30 s、58 ℃ 30 s、72 ℃ 30 s,共34个循环;72 ℃ 10 min。
胶回收、双酶切、连接和鉴定:pcDNA3.1(+)质粒和纯化得到的PCR片段分别进过HindⅢ和BamHⅠ后,在T4 DNA连接酶作用下,16 ℃连接过夜;经过转化和涂板,挑取阳性菌落进行摇菌。将菌液PCR鉴定正确的菌液进行质粒提取纯化,分别用BamHⅠ单酶切,HindⅢ和BamHⅠ双酶切鉴定。经酶切鉴定正确的克隆送北京Invitrogen公司测序进一步证实。
1.3.3 MiR-125a超表达载体的转染 GC-1 spg细胞在含10% FBS的DMEM完全培养基中于37 ℃、5%CO2条件下培养。转染24 h前接种5×105个细胞到6孔板,待细胞融合度达70%左右开始转染。根据Lipofectamine 2000 Reagent说明书,分别转染pcDNA3.1(+)空载体及pcDNA3.1(+)-miR-125a重组质粒,共分3组:未转染组、pcDNA3.1(+)空载体组、pcDNA3.1(+)-miR-125a重组质粒组、每组设3个复孔。转染6 h后,更换为含10% FBS的DMEM完全培养基。继续培养30 h,待细胞密度达到100%时收取细胞进行下步试验。
1.4 目的基因的Western blot检测
细胞的转染按照上步转染进行操作,设置2个处理组:pcDNA3.1(+)空载体组、pcDNA3.1(+)-miR-125a重组质粒组、每组设3个复孔。转染48 h后收集细胞,经RIPA裂解缓冲液裂解后收集上清蛋白;采用BCA法测定蛋白质浓度,蛋白质经过变性处理后取20 μg进行后续试验。蛋白电泳S1:80V,5min;S2: 120V,1.5 h。半干法15V 转膜25 min,5%脱脂奶粉封闭2 h,小鼠抗GAPDH和山羊抗C-KIT多克隆抗体4 ℃孵育过夜,然后分别以HRP标记的山羊抗小鼠和兔抗山羊二抗与之在室温反应1 h;ECL法显色,照相,分析结果。
2 结果与分析
2.1 MiR-125a在7日龄小鼠和7周龄小鼠睾丸组织中的差异表达
qRT-PCR结果显示,miR-125a在小鼠睾丸的发育过程中呈现差异性表达,随着睾丸的发育成熟呈现上升趋势(图1)。此结果暗示miR-125a在成年小鼠睾丸中的高表达可能与精子发生过程中生殖细胞的增殖与分化有关。
2.2 MiR-125a前体片段(pre-miR-125a)的扩增
为了进一步探究miR-125a与小鼠雄性生殖细胞增殖与分化的联系,采用PCR方法将pre-miR-125a从小鼠基因组DNA扩增出来,双酶切后定向插入真核表达载体pcDNA3.1(+),构建miRNA真核表达载体pcDNA3.1(+)-miR-125a,进而转染至GC-1 spg细胞。PCR产物10 g/L琼脂糖凝胶电泳鉴定,在290 bp处有一特异扩增条带,与理论条带长度符合(图2)。
2.3 重组质粒pcDNA3.1(+)-miR-125a的鉴定
随机挑取单个阳性菌落进行菌液PCR扩增,反应条件与miR-125 a前体片段扩增条件一致。产物经10 g/L琼脂糖凝胶电泳鉴定,结果见图3A。经菌液PCR鉴定正确的菌液提取质粒,进行BamHⅠ单酶切,Hind Ⅲ/BamHⅠ双酶切鉴定,分别可以得到5 728 bp,5 428 bp和290 bp的片段,其大小与预期结果相符(图3B)。酶切鉴定正确的质粒送往Invitrogen公司测序鉴定,测序结果显示miR-125a前体片段没有突变,说明质粒pcDNA3.1(+)-miR-125a构建成功。

图1 miR-125a在小鼠睾丸组织中的差异表达Fig.1 Differential expression of miR-125a in testis tissue
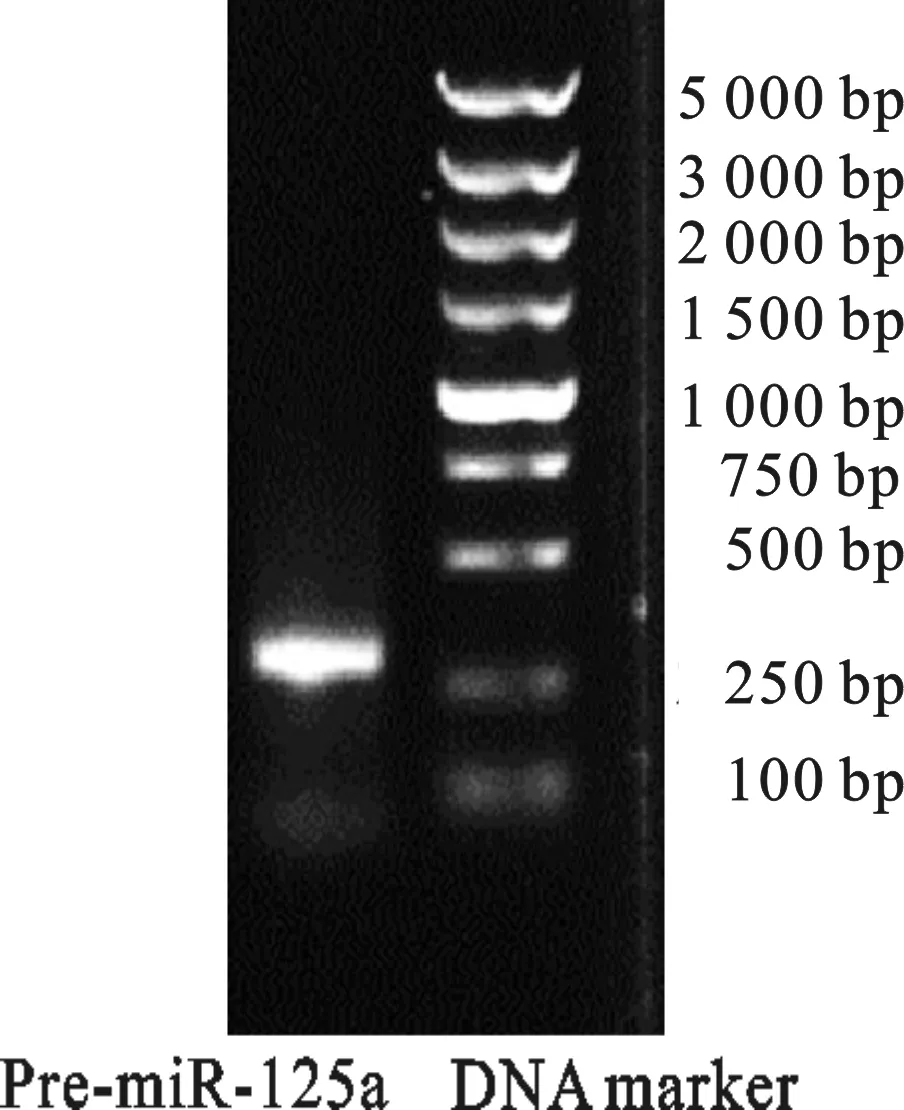
图2 miR-125a前体片段的PCR扩增鉴定Fig.2 PCR verification of pre-miR-125a
2.4 转染质粒pcDNA3.1(+)-miR-125a的细胞中miR-125a的差异表达
将pcDNA3.1(+)-miR-125a质粒和pcDNA3.1(+)对照质粒分别转染至小鼠GC-1 spg细胞,转染36 h后,抽提细胞总RNA;实时定量PCR检测空白对照组、pcDNA3.1(+)空质粒组和pcDNA3.1(+)-miR-125a处理组细胞总RNA中miR-125a的表达。结果显示(图4),空质粒组与空白对照相比,没有明显的差异,表明pcDNA3.1(+)质粒对GC-1spg细胞没有明显的毒副作用;与空白组和空质粒组相比,转染pcDNA3.1(+)-miR-125a质粒的试验组中miR-125a表达水平升高约3.5倍,表明pcDNA3.1(+)-miR-125a能够在GC-1 spg细胞中过表达miR-125a。
2.5 miR-125a对生殖细胞增殖分化的影响
如图5所示,转染了pcDNA3.1(+)-miR-125a质粒与空质粒组相比,与生殖细胞分化密切相关基因C-kit有显著的上升(约4倍),而与生殖细胞增殖密切相关的基因Pcna并无明显的变化。初步表明miR-125a可能在一定程度上促进生殖细胞的分化,但对增殖可能没有明显的促进或者抑制作用。Western Blot结果进一步证实在过表达了miR-125a后C-KIT有明显的上升趋势(约3倍)(图6)。

图3 重组质粒pcDNA3.1(+)-miR-125a的鉴定A. 重组质粒的菌液PCR鉴定;B. 重组质粒的酶切鉴定Fig.3 Verification of the recombinant plasmid(pcDNA3.1(+)-miR-125a)A. PCR detection for bacterial liquid; B. Restriction analysis

图4 重组质粒pcDNA3.1(+)-miR-125a在GC-1 spg细胞系中的过表达效果Fig.4 Differential expression of miR-125a in GC-1 spg cells
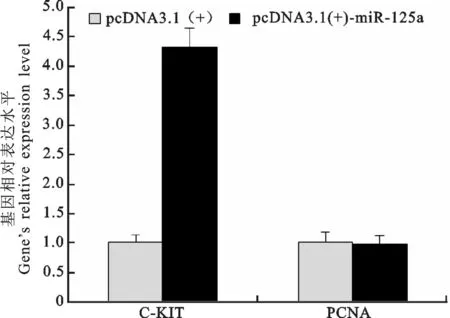
图5 qRT-PCR检测GC-1 spg细胞超表达miR-125a后C-kit和Pcna表达的变化Fig.5 Expression of C-kit and Pcna in GC-1 spg cells detected by qRT-PCR analysis
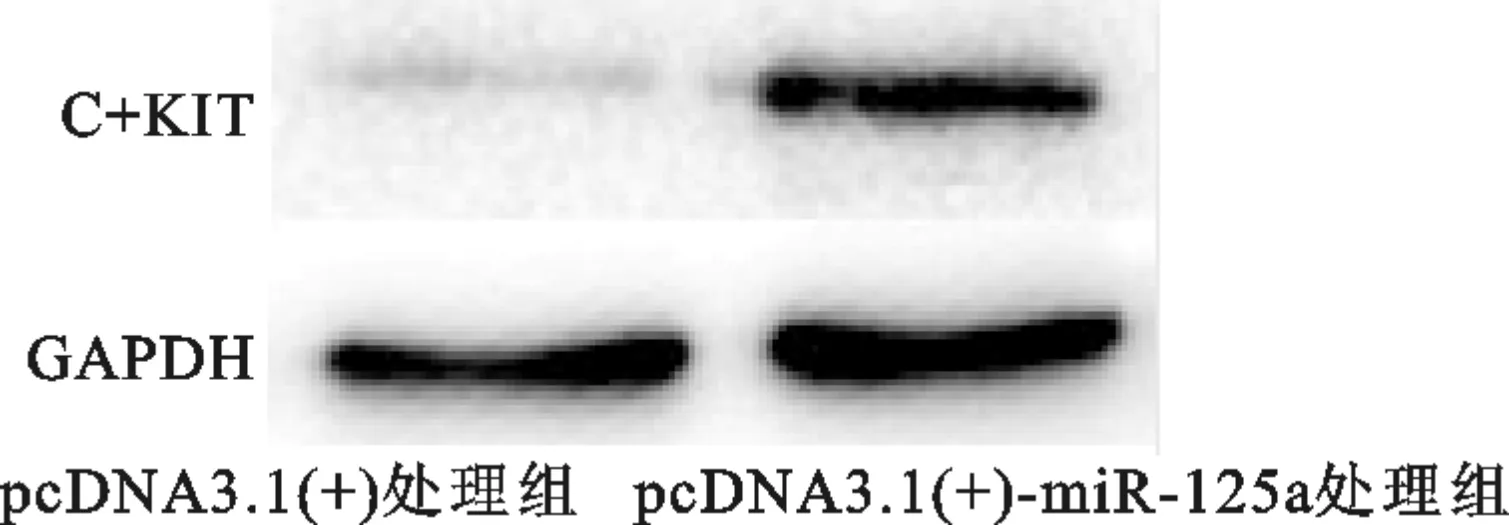
图6 Western blot检测C-KIT在GC-1 spg细胞超表达miR-125a后的表达变化Fig.6 Western blot analysis of C-KIT expression in GC-1 spg cells transfected with pcDNA3.1(+)-miR-125a
3 讨 论
位于小鼠基因组17号染色体上的miR-125a与miR-99b和let-7e成簇分布,是Lagos-Quintana等在小鼠脑组织中克隆到的一个与线虫miRNA-Lin-4具有高度同源性的miRNA[11]。
在胚胎干细胞中,miR-125a在未分化的细胞中表达量很低,但随着分化的进行,表达量逐渐升高[12]。另外,通过抑制miR-125a的表达能够促进体细胞向诱导多能干细胞(induced pluripotent stem cells, iPS)的重编程[13]。此外,miR-125a在果蝇发育过程中的表达具有时序性,仅在果蝇发育到蛹和成虫阶段表达而在胚胎和幼虫发育阶段不表达[14-16]。然而关于miR-125a在哺乳动物雄性生殖细胞中的研究还未见报道。本研究首先分析了miR-125a在小鼠睾丸发育过程中的表达变化,结果显示其在7周龄成年小鼠睾丸中高表达,说明其可能参与生殖细胞的增殖与分化过程。结合文献所报道的miR-125a表达具有时序性,并且在分化的胚胎干细胞中高表达[12],预测其与生殖细胞的分化具有密切的联系。为了进一步探究miR-125a与小鼠雄性生殖细胞分化的关系,通过pcDNA3.1(+)-miR-125a超表达质粒载体在生殖细胞系GC-1 spg中进行了验证。GC-1 spg细胞系是一种介于B型精原细胞和初级精母细胞之间的雄性生殖细胞。本试验中,将miR-125a的前体从小鼠基因组DNA扩增出来,双酶切后定向插入真核表达载体pcDNA3.1(+),构建pcDNA3.1(+)-miR-125a超表达载体。转染细胞后pcDNA3.1(+)-miR-125a在CMV启动子作用下转录为具有"茎环"结构的pre-miR-125a,通过细胞内RNA聚合酶 Ⅲ Dicer的作用剪接加工形成成熟的miR-125a,从而发挥超表达调控作用。试验观察到转染了pcDNA3.1(+)-miR-125a的GC-1spg细胞中C-kit mRNA和蛋白的表达量上升,但与细胞增殖相关的基因Pcna并没有明显的变化。
综上所述,本研究成功构建了miR-125a的超表达载体,并初步验证其超表达效果,这为进一步研究miR-125a的功能及其作用机制奠定了基础。
参考文献:
[1] EDDY E M. Male germ cell gene expression[J]. Recent Prog Horm Res, 2002, 57: 103-28.
[2] SCHLECTH U, DEMOUGIN P, KOCH R, et al. Expression profiling of mammalian male meiosis and gametogenesis identifies novel candidate genes for roles in the regulation of fertility[J]. Mol Biol Cell, 2004, 15(3): 1 031-1 043.
[3] LEWIS B P, BURGE C B, BARTEL D P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets [J]. Cell, 2005, 120(1): 15-20.
[4] BAE Y, YANG T, ZENG H C, et al. miRNA-34c regulates Notch signaling during bone development [J]. Hum Mol Genet, 2012, 21(13): 2 991-3 000.
[5] LAKSHMIPATHY U, DAVILA J, HART R P. miRNA in pluripotent stem cells [J]. Regen Med, 2010, 5(4): 545-555.
[6] VERRIER J D, LAU P, HIDSPN L, et al. Peripheral myelin protein 22 is regulated post-transcriptionally by miRNA-29a [J]. Glia, 2009, 57(12): 1 265-1 279.
[7] MAATOUK D M, LOVELAND K L, MCMANUS M T, et al. Dicer1 is required for differentiation of the mouse male germline [J]. Biology of Reproduction, 2008, 79(4): 696-703.
[8] HATFIELD S D, SHCHERBATA H R, FISHER K A, et al. Stem cell division is regulated by the microRNA pathway [J]. Nature, 2005, 435(7044): 974-978.
[9] BOISSART C, NISSAN X, GIRAUD-TRIBOULT K, et al. miR-125 potentiates early neural specification of human embry-onic stem cells [J]. Development, 2012, 139(7): 1 247-1 257.
[10] CHEN C, RIDZON D A, BROOMER A J, et al. Real-time quantification of microRNAs by stem-loop RT-PCR [J]. Nucleic Acids Research, 2005, 33(20): e179.
[11] LAGOS-QUINTANA M, RAUHUT R, YALCIN A, et al. Identification of tissue-specific microRNAs from mouse [J]. Current Biology, 2002, 12(9): 735-739.
[12] ZHONG X M, LI N, LIANG S, et al. Identification of MicroRNAs Regulating Reprogramming Factor LIN28 in Embryonic Stem Cells and Cancer Cells [J]. Journal of Biological Chemistry, 2010, 285(53): 41 961-41 971.
[13] WILSON K D, VENKATASUBRAHMANYAM S, JIA F J, et al. MicroRNA Profiling of Human-Induced Pluripotent Stem Cells [J]. Stem Cells and Development, 2009, 18(5): 749-757.
[14] PASQUINELLI A E, REUBGART B J, SLACK F, et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA [J]. Nature, 2000, 408(6808): 86-89.
[15] HUTVAGNER G, MCLACHLAN J, PASQUINELLI A E, et al. A cellular function for the RNA-interference enzyme Dicer in the maturation of the let-7 small temporal RNA [J]. Science, 2001, 293(5531): 834-838.
[16] SEMPERE L F, DUBROVSKY E B, DUBROVSKAYA V A, et al. The expression of the let-7 small regulatory RNA is controlled by ecdysone duriniz metamorphosis in Drosophila melanogaster [J]. Developmental Biology, 2002, 244(1): 170-179.

