长链非编码RNA HOXA11-AS表达对头颈部鳞状细胞癌细胞迁移和患者预后的影响
张 莉,伍 靖,李晓宇
(深圳市龙华区人民医院口腔科,广东 深圳 518109)
长链非编码RNA(long non-coding RNA,lncRNA)是一类含200~100 000个核苷酸,无或很少编码蛋白质的RNA分子,其在转录沉默、转录激活、染色质重塑、组蛋白修饰等多个方面参与基因的表达调控[1-2]。起初,lncRNA一直被认为是基因组的“暗物质”,不具有生物学功能。但是,随着研究的深入,人们逐渐认识到lncRNA参与了包括恶性肿瘤在内的多种疾病的发生、发展过程[3-5]。深入研究lncRNA的表达、功能和分子机制,对于理解肿瘤等复杂疾病的发生发展机制,寻找更加合适的诊断和治疗靶点具有重要意义。同源异型盒11反义RNA (homeobox A11 antisense RNA,HOXA11-AS)是一个定位在HOXA11基因反义DNA链上的lncRNA,转录本长度为5.1 kb[6]。近年来,已有多个研究团队发现,HOXA11-AS在包括胃癌、结直肠癌、宫颈癌等肿瘤中差异表达,可以作为这几种肿瘤的潜在生物学标志物[7-9]。但是,截止目前,HOXA11-AS在头颈部鳞状细胞癌中的表达情况及其作用尚无报道。本研究通过Gene Expression Profiling Interactive Analyses(GEPIA)数据库数据挖掘,并检测HOXA11-AS在头颈部鳞状细胞癌组织中的表达,来探究其对头颈部鳞状细胞癌细胞迁移的影响,以阐明HOXA11-AS在头颈部鳞状细胞癌进程中的作用。
1 资料与方法
1.1组织标本及细胞系收集中南大学湘雅二医院口腔颌面外科2016年10月至2017年3月手术切除的24例头颈部鳞状细胞癌组织及癌旁0.5 cm外正常组织(癌旁组织)标本,其中男17例,女7例;年龄26~61(45.83±8.90)岁;舌癌14例,颊癌10例。所有病例经病理科确诊为鳞状细胞癌,并取得患者的知情同意,患者手术前均未接受过放射治疗和化学治疗。人头颈部鳞状细胞癌细胞系Tca8113为中南大学湘雅医院医学科研中心馈赠。
1.3方法
1.3.1GEPIA数据库数据挖掘首先,在GEPIA[10]数据库中选择来源于TCGA数据库的所有头颈部鳞状细胞癌及正常组织的RNA测序数据,以“HOXA11-AS”为检索词,以P<0.01为具有显著性差异,分析其是否存在差异表达。然后以HOXA11-AS的表达从高到低进行排序,前25%的样本为高表达样本,后25%的样本为低表达样本,分析其表达与患者总体生存率和无病生存率的关系。
1.3.2HOXA11-AS表达的检测按照TRIzol试剂说明书上的步骤分别提取头颈部鳞状细胞癌组织、癌旁组织或者头颈部鳞状细胞癌细胞系Tca8113的总RNA。将提取完成的总RNA进行琼脂糖凝胶电泳检测RNA的完整性后,测定总RNA浓度。然后,采用qRT-PCR技术检测HOXA11-AS的表达(根据反转录及qRT-PCR试剂盒说明书进行),以β-Actin为内参,相对表达量采用2-ΔΔCt法计算。
1.3.3细胞接种、转染及分组取头颈部鳞状细胞癌Tca8113细胞接种于含有体积分数10%胎牛血清和双抗的RPMI-1640培养基中,在37 ℃、含体积分数5% CO2的恒温培养箱中培养。
取处于对数生长期的Tca8113细胞,以每孔5×105个细胞接种于6孔板中。待细胞生长至 60%~70%融合时,分为实验组和对照组。实验组细胞加HOXA11-AS siRNA进行转染(按照Hiperfect脂质体转染试剂说明书进行),对照组加阴性对照siRNA进行转染,然后将细胞置于含体积分数5% CO2的细胞培养箱中培养48 h,然后采用qRT-PCR法检测 2组细胞中HOXA11-AS的表达。
1.3.4Transwell小室迁移实验待Tca8113细胞转染siRNA达到24 h后,胰蛋白酶消化细胞,无血清培养基洗涤细胞2次,并调整细胞浓度至10×107L-1。向Transwell小室下腔室中加入800 μL含体积分数12%胎牛血清的培养基,上室中加入 200 μL 上述细胞悬液,置于培养箱中培养48 h。取出Transwell小室,生理盐水中清洗小室2遍,甲醛溶液固定细胞30 min,清水洗涤小室3遍。加入 1 g·L-1结晶紫室温染色5 min,再用生理盐水洗去多余结晶紫,倒置显微镜下观察细胞染色情况,每个孔拍照5张并统计细胞数目。
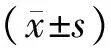
3)在搭建无线网络链路时要合理选择基站位置,避免建造难度过大。对于较长链路,要勘查优化,绘制链路纵断面图,分析净空,确保信号传输无遮挡,保障传输质量。还要结合周围环境考虑,避免与区域内通讯、输电设施相互干扰。
2 结果
2.1HOXA11-AS在头颈部鳞状细胞癌中的表达及其与患者预后的相关性数据库分析结果显示,HOXA11-AS在头颈部鳞状细胞癌组织(n=519)中的表达较正常组织(n=44)中显著增高,是正常组织的1.21倍,差异有统计学意义(P<0.01),见图1。HOXA11-AS表达与患者的总体生存率和无病生存率相关,风险比(hazard ratio,HR)分别为1.5和1.6,P值分别为0.043和0.039,见图2和图3。
2.2HOXA11-AS在24例头颈部鳞状细胞癌中的表达HOXA11-AS在头颈部鳞状细胞癌组织和癌旁组织中的表达水平分别为2.58±0.37、0.95±0.29,头颈部鳞状细胞癌组织中的表达高于癌旁组织,差异有统计学意义(P<0.05)。

图1头颈部鳞状细胞癌组织及正常组织中HOXA11-AS表达
Fig.1ExpressionofHOXA11-ASinthenormaltissuesandheadandnecksquamouscellcarcinoma
2.3siRNA对HOXA11-AS的沉默效果HOXA11-AS在实验组和对照组Tca8113细胞中的表达水平分别为0.43±0.05,1.08± 0.07,实验组Tca8113细胞中HOXA11-AS表达低于对照组,差异有统计学意义(P<0.05)。

图2HOXA11-AS表达与头颈部鳞状细胞癌患者总体生存率的关系
Fig.2RelationshipbetweentheexpressionofHOXA11-ASandoverallsurvivalofheadandnecksquamouscellcarcinomapatients

图3HOXA11-AS表达与头颈部鳞状细胞癌患者无病生存率的关系
Fig.3RelationshipbetweentheexpressionofHOXA11-ASanddiseasefreesurvivalofheadandnecksquamouscellcarcinomapatients
2.4沉默HOXA11-AS表达对头颈部鳞状细胞癌细胞迁移能力的影响对照组和实验组穿过Transwell小室的细胞数目分别为212±15、113±9个。实验组穿过Transwell小室的细胞数目显著少于对照组,差异有统计学意义(P<0.05),见图4。

A:对照组;B:实验组。
图4HOXA11-AS表达对Tca8113细胞迁移能力的影响
Fig.4EffectofHOXA11-ASonTca8113cellsmigrationability
3 讨论
lncRNA是一组内源性、长度超过200 nt、缺少特异完整的开放阅读框、无或很少有蛋白编码功能的RNA分子。其可作为功能蛋白质的信号、诱导、诱饵或支架分子等,在表观遗传水平、转录水平、翻译及翻译后水平等多层面调控基因的表达,参与肿瘤的发生、发展过程[11-12]。在头颈部肿瘤中以鼻咽癌中的功能性lncRNA报道较为多见。早在2002年,TAN等[13]发现,鼻咽癌相关lncRNA NAG7能够影响鼻咽癌细胞的细胞周期分布和凋亡水平。此后,又有多个lncRNA被发现在鼻咽癌中差异表达且具有重要的生物功能[14-17]。然而,还有大量与头颈部鳞状细胞癌发生、发展和转归相关的lncRNA表达、功能及分子机制尚不清楚。
本研究通过对GEPIA数据库的TCGA进行数据挖掘,发现HOXA11-AS在头颈部鳞状细胞癌中高表达,且其与患者的预后密切相关;继而利用qRT-PCR技术在24例头颈部鳞状细胞癌中验证了HOXA11-AS的高表达;最后在利用RNAi技术干扰了HOXA11-AS的表达后,检测了其对头颈部鳞状细胞癌细胞迁移能力的影响。本研究首次证实了HOXA11-AS在头颈部鳞状细胞癌中高表达,沉默HOXA11-AS表达能够在体外抑制人头颈部鳞状细胞癌细胞系Tca8113的迁移能力。这极大地丰富了lncRNA参与头颈部鳞状细胞癌发生、发展过程的机制。但是,HOXA11-AS影响头颈部鳞状细胞癌细胞迁移的具体分子机制以及其在头颈部鳞状细胞癌中是否还具有其他的生物功能,目前尚不十分清楚,还有待深入研究。
综上所述,HOXA11-AS在头颈部鳞状细胞癌中发挥着类似致瘤基因的功能,能够显著促进头颈部鳞状细胞癌细胞的迁移。深入理解其作用机制,将对未来头颈部鳞状细胞癌的生物标志物开发和治疗靶点确证具有较大的意义。
参考文献:
[1] GONG Z,ZHANG S,ZHANG W,etal.Long non-coding RNAs in cancer[J].SciChinaLifeSci,2012,55(12):1120-1124.
[2] LAU E.Non-coding RNA:zooming in on lncRNA functions[J].NatRevGenet,2014,15(9):574-575.
[3] QUAGLIATA L,MATTER M S,PISCUOGLIO S,etal.Long noncoding RNA HOTTIP/HOXA13 expression is associated with disease pro-gression and predicts outcome in hepatocellular carcinoma patients[J].Hepatology,2014,59(3):911-923.
[4] YANG X,SONG J H,CHENG Y,etal.Long non-coding RNA HNF1A-AS1 regulates proliferation and migration in oesophageal adenocarcinoma cells[J].Gut,2014,63(6):881-890.
[5] LU M H,TANG B,ZENG S,etal.Long non-coding RNA BC032469,a novel compet-ing endogenous RNA,upregulateshTERT expression by sponging miR-1207-5p and promotes prolifera-tion in gastric cancer[J].Oncogene,2016,35(27):3524-3534.
[6] ZHANG Y,HE R Q,DANG Y W,etal.Comprehensive analysis of the long noncoding RNA HOXA11-AS gene interaction regulatory network in NSCLC cells[J].CancerCellInt,2016,16:89.
[7] LIU Z,CHEN Z,FAN R,etal.Over-expressed long noncoding RNA HOXA11-AS promotes cell cycle progression and metastasis in gastric cancer[J].MolCancer,2017,16(1):82.
[8] LI T,XU C,CAI B,etal.Expression and clinicopathological significance of the lncRNA HOXA11-AS in colorectal cancer[J].OncolLett,2016,12(5):4155-4160.
[9] KIM H J,EOH K J,KIM L K,etal.The long noncoding RNA HOXA11 antisense induces tumor progression and stemness maintenance in cervical cancer[J].Oncotarget,2016,7(50):83001-83016.
[10] TANG Z,LI C,KANG B,etal.GEPIA:a web server for cancer and normal gene expression profiling and interactive analyses[J].NucleicAcidsRes,2017,45(W1):W98-W102.
[11] LATOS P A,PAULER F M,KOERNER M V,etal.Airn transcriptional overlap,but not its lncRNA products,induces imprinted Igf2r silencing[J].Science,2012,338(6113):1469-1472.
[12] GUPTA R A,SHAH N,WANG K C,etal.Long non-coding RNA HOTAIR reprograms chro-matin state to promote cancer metastasis[J].Nature,2010,464(7291):1071-1076.
[13] 谭琛,彭聪,黄宇琛,等.鼻咽癌相关基因NAG7对鼻咽癌细胞周期及凋亡的影响[J].癌症,2002,21(5):449-455.
[14] GONG Z,ZHANG S,ZENG Z,etal.LOC401317,a p53-regulated long non-coding RNA,inhibits cell proliferation and induces apoptosis in the nasopharyngeal carcinoma cell line HNE2[J].PLoSOne,2014(11):e110674
[15] BO H,GONG Z,ZHANG W,etal.Upregulated long non-coding RNA AFAP1-AS1 expression is associated with progression and poor prognosis of nasopharyngeal carcinoma[J].Oncotarget,2015,6(24):20404-20418.
[16] WANG Q,ZHANG W,HAO S.LncRNA CCAT1 modulates the sensitivity of paclitaxel in nasopharynx cancers cells via miR-181a/CPEB2 axis[J].CellCycle,2017,16(8):795-801.
[17] CHAK W P,LUNG R W,TONG J H,etal.Downregulation of long non-coding RNA MEG3 in nasopharyngeal carcinoma[J].MolCarcinog,2017,56(3):1041-1054

