肺炎克雷伯菌对复方新诺明的耐药性及其整合子分布差异的研究
支晓阳 毕文姿 吴庆 李佳慧 董通雨 张艺之 董郭枫 周铁丽
肺炎克雷伯菌是造成医院获得性感染的重要病原菌,可引起人体多部位感染[1]。随着抗菌药物的广泛使用甚至滥用,多重耐药和广泛耐药肺炎克雷伯菌检出率不断增加,尤其对碳青霉烯类抗菌药物耐药菌株的大量出现,使临床抗感染治疗面临“束手无策”的局面[2]。复方新诺明为甲氧苄啶和磺胺甲恶唑的复方制剂,对多种革兰阴性菌、革兰阳性菌具有协同抑菌、杀菌作用,且对多重耐药菌、耐甲氧西林金黄色葡萄球菌感染具有良好的治疗效果[3]。研究表明,肺炎克雷伯菌临床分离株对复方新诺明的逐年耐药率较平稳,且耐碳青霉烯类肺炎克雷伯菌对复方新诺明仍保持一定程度的敏感性[4]。整合子是多种抗菌药物耐药基因水平传播的重要因素,可捕获、重排和表达耐药基因,与复方新诺明等多种抗菌药物的耐药性相关[5]。目前尚缺乏关于肺炎克雷伯菌内整合子分布及其与耐药表型相关性的系统研究。为探究肺炎克雷伯菌对复方新诺明的耐药性、临床菌株内整合子分布及其与耐药表型的相关性,笔者对812株临床分离肺炎克雷伯菌进行分析,为临床耐药基因的水平传播、耐药菌株的控制提供理论依据。
1 材料和方法
1.1 菌株来源 收集2013年1月至2015年5月温州医科大学附属第一医院临床分离的812株肺炎克雷伯菌,剔除了同一例患者相同部位分离的重复菌株。所有菌株经VITEK 2 Compact全自动微生物分析仪鉴定。质控菌株为大肠埃希菌ATCC 25922和铜绿假单胞菌ATCC 27853,购自卫生部临检中心。
1.2 仪器与试剂 VITEK 2 Compact全自动微生物分析仪(法国生物梅里埃公司);S-100TMThermal Cycler PCR仪(美国BIO-RAD公司);Bio-Rad凝胶电泳成像分析仪(美国BIO-RAD公司);Thermo FRESCO21型离心机(美国Thermo Fisher有限公司);DR100型比浊仪(法国Biomerieux有限公司);DYY-5稳压稳流电泳仪(北京六一仪器厂);PCR反应试剂盒(日本TaKaRa公司);PCR引物(上海华大基因科技有限公司);核酸染料Gelred(索莱宝科技有限公司);琼脂糖干粉(法国Biowest有限公司)。
1.3 菌株纯化、鉴定与药敏试验 参照《全国临床检验操作规程》对细菌进行分离纯化。使用VITEK 2 Compact全自动微生物分析仪对细菌进行菌种鉴定。按照操作规程检测肺炎克雷伯菌对临床常用抗菌药物的敏感性。药敏结果参照2015年美国临床和实验室标准协会标准被判读为敏感、中介和耐药[6],其中中介和耐药合并称为非敏感,其所占比率为非敏感率。
1.4 基因组提取 将细菌接种至哥伦比亚血平板,置35℃孵箱培养16~18h后,通过煮沸法粗提肺炎克雷伯菌基因组作为PCR反应模板,具体步骤如下:取新鲜培养的纯菌于含200μl双蒸水的EP管中充分研磨、混匀和溶解,100℃煮沸15min,并快速置于冰上10min,使其充分变性,12000r/min离心2min,吸取上清液(即菌株DNA基因组)于新的EP管中,-20℃保存备用。
1.5 整合子扩增检测 整合子的PCR扩增体系及引物合成参照先前报道[7]。整合子引物序列和目的产物长度见表1。反应条件:预变性94℃ 5min;变性94℃ 30s,退火 55℃ 30s,延伸 72℃ 2min,30个循环;72℃延伸 10 min。PCR产物经1%琼脂糖凝胶电泳,Gelred染色后凝胶成像观察结果,阳性产物送至上海华大基因科技公司进行测序。

表1 PCR引物序列及目的产物长度
1.6 统计学处理 应用SPSS17.0统计软件,计数资料的比较采用χ2检验,P<0.05为差异有统计学意义。
2 结果
2.1 肺炎克雷伯菌对临床常用抗菌药物的耐药性分析 肺炎克雷伯菌临床分离株对头孢吡肟、头孢哌酮/舒巴坦、阿米卡星、亚胺培南、厄他培南耐药率相对较低,对其他抗菌药物3年耐药率呈逐年递增趋势或趋于平稳,但仅对复方新诺明的耐药率呈逐年递减趋势。进一步对115株耐碳青霉烯类肺炎克雷伯菌分析发现,耐亚胺培南(111株)和耐厄他培南(85株)肺炎克雷伯菌对复方新诺明耐药率较低,总体耐药率分别为13.5%、18.8%。详见表2~3。
2.2 肺炎克雷伯菌整合子分布情况 812株肺炎克雷伯菌中,302株携带Ⅰ型整合子,阳性率为37.2%,本研究未检测到Ⅱ、Ⅲ型整合子。按照药敏试验结果,将肺炎克雷伯菌临床分离株分为复方新诺明耐药组(175株)和复方新诺明敏感组(637株),复方新诺明耐药组的整合子阳性率为63.4%(111/175),复方新诺明敏感组的整合子阳性率为30.0%(191/637),复方新诺明耐药组的整合子阳性率明显高于敏感组(P<0.01)。由于样本量较大,选取其中24株Ⅰ型整合子阳性菌株进行图示,其PCR电泳结果如图1。

表2 肺炎克雷伯菌对临床常用抗生素的逐年耐药率(%)
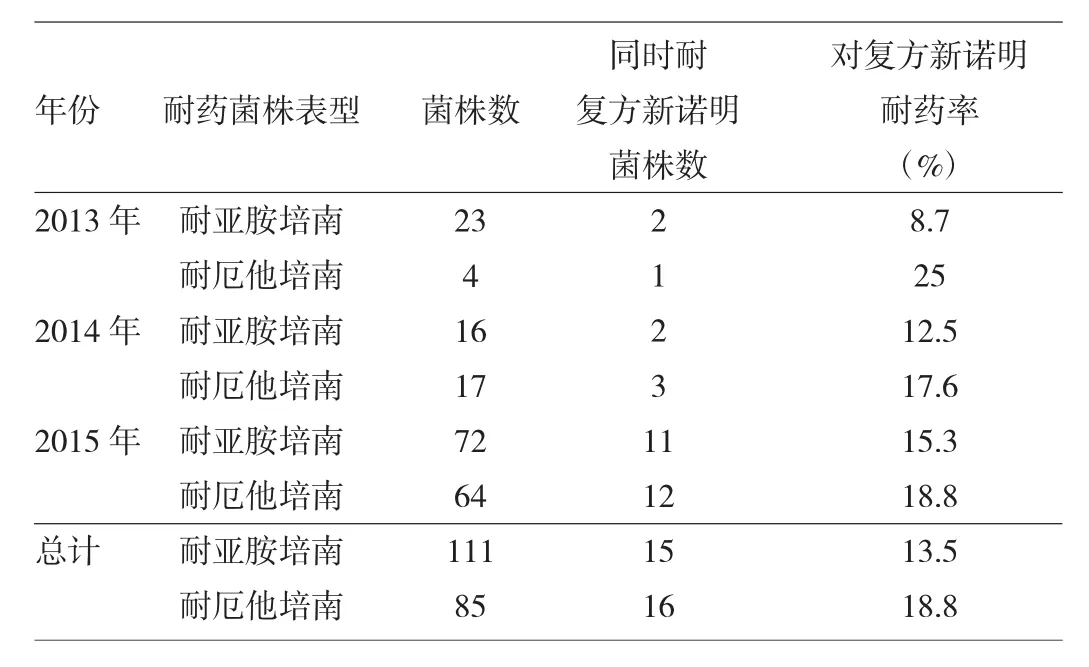
表3 耐碳青霉烯肺炎克雷伯菌对复方新诺明敏感性分析

图1 Ⅰ型整合子阳性菌株PCR电泳图
2.3 整合子阳性/阴性菌株对临床常用抗菌药物耐药性比较 整合子阳性菌株组对头孢哌酮/舒巴坦、氨苄西林/舒巴坦、头孢他啶、头孢吡肟、头孢曲松、庆大霉素、阿米卡星、妥布霉素、环丙沙星、左氧氟沙星的非敏感率均明显高于整合子阴性菌株组,差异均有统计学意义(均P<0.05)。详见图2。
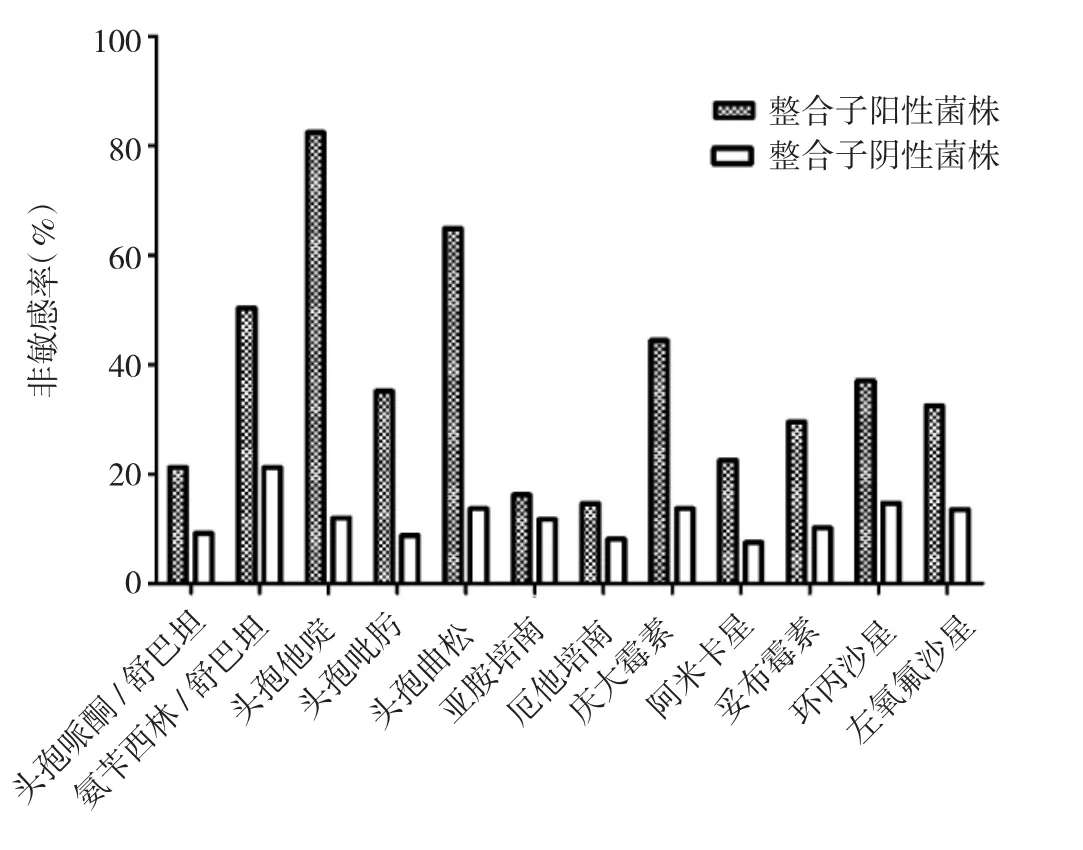
图2 肺炎克雷伯菌整合子阳性/阴性菌株对其他抗生素敏感性分析
2.4 不同耐药表型菌株的整合子分布情况 复方新诺明耐药组中,多重耐药菌株中有92株携带Ⅰ型整合子,阳性率为73.0%(92/126),广泛耐药菌株有20株携带Ⅰ型整合子,阳性率为100.0%(20/20);复方新诺明敏感组中,多重耐药菌株有93株携带Ⅰ型整合子,阳性率为53.1%(93/175),低于复方新诺明耐药组的多重耐药菌株,广泛耐药菌株有79株携带Ⅰ型整合子,阳性率为84.9%(79/93),同样低于复方新诺明耐药组的广泛耐药菌株,各组间差异均有统计学意义(均P<0.01)。详见表4。
3 讨论
肺炎克雷伯菌是引起院内感染的常见条件致病菌之一。多重耐药肺炎克雷伯菌检出率不断增高,导致住院患者出现较高的致死率[8]。随着耐药基因的水平传播,耐碳青霉烯肺炎克雷伯菌分离率不断增高,使临床抗感染治疗更加困难[9]。由于碳青霉烯类抗菌药物的广泛使用,耐碳青霉烯肺炎克雷伯菌数量逐年增加,因此临床亟需寻找其他可替代的抗菌药物。

表4 复方新诺明耐药菌株和敏感菌株内IntⅠ整合子携带情况
复方新诺明抗菌谱广,对大肠埃希菌、肺炎克雷伯菌、金黄色葡萄球菌、志贺菌属等均具有抗菌作用[10]。尽管肺炎克雷伯菌临床分离株对复方新诺明总耐药率达21.6%,但笔者分析发现肺炎克雷伯菌对复方新诺明的耐药率呈逐年下降趋势,这种现象可能与临床用药频度的相对较低以及抗菌药物的合理使用有关[11-12]。Xiao等[13]研究表明产碳青霉烯酶菌株对复方新诺明耐药率高于碳青霉烯酶阴性菌株。而本研究,耐碳青霉烯肺炎克雷伯菌对复方新诺明敏感性良好,亚胺培南和厄他培南耐药菌株对复方新诺明总耐药率分别仅为9.9%、10.2%,提示复方新诺明可作为耐碳青霉烯肺炎克雷伯菌感染治疗良好的药物选择,为临床抗感染治疗提供指导。
整合子是具有位点特异性重组功能的可移动基因元件,其中Ⅰ、Ⅱ、Ⅲ型整合子可导致耐药基因在肠杆菌科细菌间的克隆传播。这种播散可使菌株产生对多种抗菌药物的耐药性,进而导致多重耐药菌株的暴发流行,因此又称为耐药整合子[14-15]。Frank等[16]研究表明,sul与dfrA可分别拮抗磺胺甲恶唑、甲氧苄啶的抗菌机制,且主要由Ⅰ型整合子介导,在不同菌株间进行播散,成为病原菌对复方新诺明的重要耐药机制。本研究发现,复方新诺明耐药组的Ⅰ型整合子检出率明显高于复方新诺明敏感组,且两组中多重耐药菌株和广泛耐药菌在的Ⅰ型整合子阳性率显著高于非多重耐药菌株。此外,整合子阳性菌株对头孢类、氨基糖苷类、喹诺酮类等临床常用抗菌药物耐药率均明显高于整合子阴性菌株。以上结果均说明整合子不仅与复方新诺明耐药性相关,整合子上耐药基因盒的整合与播散也是菌株获得对临床常用抗菌药物耐药性的重要耐药机制,这与Xu等[7]研究结果相契合。
本研究中,肺炎克雷伯菌仅对复方新诺明的耐药率呈现逐年降低趋势,且耐碳青霉烯肺炎克雷伯菌对复方新诺明敏感性良好。据分析,整合子与复方新诺明耐药性、多重耐药表型、广泛耐药表型的出现均密切相关。基于复方新诺明可作为耐碳青霉烯菌株抗感染治疗的另一药物选择,加强感染控制、合理用药、及时监测、有效隔离等措施对保持抗菌药物良好的敏感性、限制整合子耐药基因的水平传播都至关重要。
[1]Hennequin C,Robin F.Correlation between antimicrobial resistance and virulence in Klebsiella pneumonia[J].Eur J Clin Microbiol,2016,35(3):333-341.
[2]许毛宇,吴胜军.2010-2014年杭州邵逸夫医院临床分离肺炎克雷伯菌的药敏分析[J].中华全科医学,2017,15(2):304-307.
[3]Goldberg E,Bishara J.Contemporary unconventional clinical use ofco-trimoxazole[J].Clin MicrobiolInfec,2012,18(1):8-17.
[4]徐安,卓超,苏丹虹,等.2005-2014年CHINET克雷伯菌属细菌耐药性监测[J].中国感染与化疗杂志,2016,16(3):267-274.
[5]Grape M,Farra A,KronvallG,et al.Integrons and gene cassettes in clinical isolates of co-trimoxazole-resistant Gram-negative bacteria[J].Clin MicrobiolInfec,2005,11(3):185-192.
[6]Wayne.Clinical and Laboratory Standards Institute.Performance standards for antimicrobial susceptibility testing;twenty-fifth informationalsupplement[J].Document M00-S25.CLSI,2015.
[7]Xu X,LiX,Luo M,et al.Molecular characterisations of integrons in clinical isolates of Klebsiella pneumoniae in a Chinese tertiary hospital[J].Microbialpathogenesis,2017,104:164-170.
[8]Mezzatesta ML,Caio C,Gona F,et al.Colistin Increases the Cidal Activity of Antibiotic Combinations Against Multidrug-Resistant Klebsiella pneumoniae:An In Vitro Model Comparing Multiple Combination BactericidalTesting at One Peak Serum Concentration and Time-Kill Method[J].Microb Drug Resist,2016,22(5):360-363.
[9]Akturk H,Sutcu M,Somer A,et al.Carbapenem-resistant Klebsiella pneumoniae colonization in pediatric and neonatal intensive care units:risk factors for progression to infection[J].Braz J Infect Dis,2016,20(2):134-140.
[10]Huovinen P,Sundstrom L,Swedberg G,et al.Trimethoprim and sulfonamide resistance[J].Antimicrob Agents Ch,1995,39(2):279-289.
[11]楼晓清,赵锎,徐月萍,等.细菌耐药率及抗菌药物用药频度相关性分析[J].中国解放军药学学报,2016,32(4):357-359.
[12]杨凤娉,王平,王业菊,等.某院细菌耐药率与抗菌药物用药频度相关性分析[J].宜春学院学报,2014,36(6):59-62.
[13]Xiao SZ,Wang S,Wu WM,et al.The Resistance Phenotype and Molecular Epidemiology of Klebsiella pneumoniae in Bloodstream Infections in Shanghai,China,2012-2015[J].Frontiers in microbiol,2017,8:250.
[14]Salles MJ,Zurita J,Mejia C,et al.Resistant gram-negative infections in the outpatient setting in Latin America[J].Epidemiol Infect,2013,141(12):2459-2472.
[15]Canton R,Gonzalez-Alba JM,Galan JC.CTX-M Enzymes:Origin and Diffusion[J].Frontiers in microbiology,2012,3:110.
[16]Frank T,Gautier V,Talarmin A,et al.Characterization of sulphonamide resistance genes and class 1 integron gene cassettes in Enterobacteriaceae,Central African Republic(CAR)[J].The Journal of antimicrobial chemotherapy,2007,59(4):742-745.

