毛果杨CBF/DREB1基因家族生物信息学分析
丁 咚,陈亚娟,崔进荣,魏建华,张玉红,张杰伟*
(1.东北林业大学, 森林植物生态学教育部重点实验室, 黑龙江 哈尔滨 150040;2.北京市农林科学院北京农业生物技术研究中心, 农业基因资源与生物技术北京市重点实验室,北京 100097)
【研究意义】C重复序列结合因子/干旱应答元件结合蛋白(c-repeat binding factor/dehydration-responsive element binding protein 1, CBF/DREB1)是植物特有的一类转录因子,属于AP2/EREBP(apetla2/ehylene responsive element binding protein)类转录因子亚家族。迄今已发现的CBF/DREB1 转录因子基因内均无内含子,且含有一段由约60个氨基酸构成的AP2/ERF DNA结合结构域(AP2结构域),除此之外,在AP2结构域之前含有保守的核定位信号区(nuclear localization signal, NLS):PKK/RPAGRxKFxETRHP(PKKPAGR)序列,之后含有DSAWR序列,这两段氨基酸序列已经成为区分其他AP2/EREBP转录因子的特征序列[1-2]。 植物中CBF/DREB1类转录因子都能通过AP2 结构域特异性的与CRT/DRE (c-repeat/dehydration-responsive element)顺式作用元件(A/GCCGAC)结合,激活响应低温、干旱和高盐等非生物胁迫应答基因,从而提高植物的抗逆性[3-4]。【前人研究进展】 1997年,Stockinger 等利用酵母单杂交方法首次从拟南芥cDNA文库中分离到DRE /CRT结合蛋白并命名为AtCBF1[5]。随后,Gilmour等从低温处理后的拟南芥cDNA 文库中鉴定出与AtCBF1在同一染色体上顺序排列的两个DRE /CRT 结合蛋白AtCBF2/DREB1C 和AtCBF3/DREB1A[6]。后续的研究发现,AtCBF4/DREB1D受干旱和ABA 诱导且不受低温胁迫诱导[7],AtCBF5/DDF2/DREB1E和AtCBF6/DDF1/DREB1F均能够响应高盐胁迫诱导[8-9]。目前,已经从玉米[10]、胡杨[11]、海岛棉[12]等多种植物中分离鉴定出CBF 转录因子。由美国能源部启动并实施的杨树全基因组测序计划的完成[13],为后续通过生物信息学挖掘、鉴定和分析杨树功能基因提供重要的基础。【本研究切入点】本研究从毛果杨基因组数据库出发,重点分析毛果杨CBF基因家族进化关系、编码蛋白的保守结构域和磷酸化位点等,为进一步研究杨树CBF 蛋白的生物学功能以及其磷酸化修饰提供重要信息。
1 材料与方法
1.1 杨树CBF/DREB1基因家族基因组、cDNA和蛋白序列的获得
拟南芥(Arabidopsisthaliana)CBF/DREB1基因及其推导的蛋白序列来自TAIR数据库(http://www.arabidopsis.org);毛果杨(Populustrichocarpa)CBF/DREB1基因及其推导的蛋白序列下载自phytozome 数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Ptrichocarpa);胡杨(P.euphratica)CBF/DREB1基因及其推导的蛋白序列下载自胡杨基因组数据(http://me.lzu.edu.cn/stpd/#main_tabs=0);海岛棉CBF蛋白序列来自NCBI数据库(https://www.ncbi.nlm.nih.gov/)。
分别以拟南芥AtCBF1(AT4G25490)、AtCBF2 (AT4G25470)、AtCBF3(AT4G25480)、AtCBF4 (AT5G51990)、AtCBF5(AT1G63030)和AtCBF6 (AT1G12610)蛋白序列为参比序列,利用phytozome 数据库(杨树)中Blast 程序进行BlastP检索,检索结果的全序列E值大于e-8的舍去,再利用美国国家生物技术信息中心提供的在线CDS(conserved domain search)程序(http://www.ncbi.nlm.nih.gov/cdd)预测这些蛋白有无AP2结构域,同时利用欧洲生物信息研究所在线Clustal Omega程序进行多重序列比对,寻找AP2结构域前后是否含有PKKPAGR和DSAWR序列,同时具有这两个特征的蛋白序列属于CBF/DREB1基因家族。
1.2 植物CBF/DREB1基因家族编码蛋白系统进化树的构建及保守基序分析
利用Clustal X(2.0)[14]软件对鉴定出所有毛果杨CBF蛋白、6个拟南芥CBF蛋白、8个胡杨CBF蛋白[15]和5个海岛棉CBF蛋白[12]进行多重序列比对,使用MEGA 6.0[16]软件,采用邻接(neighbor-joining,NJ)算法构建系统进化树,进行1000 次Bootstrap 抽样。应用在线软件MEME(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)对拟南芥和毛果杨的CBF蛋白保守基序进行分析[17]。参数设置如下:基序的最大发现数目是2 个,其它参数为程序默认值。
1.3 杨树CBF/DREB1基因家族编码蛋白磷酸化位点预测
利用NetPhos 3.1 Server在线软件[18](http://www.cbs.dtu.dk)对鉴定出所有毛果杨CBF蛋白序列进行磷酸化位点预测,所有参数都选用程序默认值。
2 结果与分析
2.1 杨树CBF/DREB1基因家族的鉴定和命名
分别以6个拟南芥CBF蛋白序列为参比序列,利用phytozome 数据库提供的Blast P程序进行检索,共得到11个毛果杨候选CBF基因。利用保守基序在线预测软件MEME进行验证AP2保守结构域的存在,同时在其前后分别含有PKKPAGR和DSAWR序列,初步确定了6个毛果杨CBF基因。毛果杨CBF基因分布在第1、4、9、12和15五条染色体上,根据通用植物基因的命名方法,对鉴定到的6个CBF基因进行了命名,并统计了这6个基因对应的转录本、别名、染色体定位、基因长度以及对应的推导蛋白长度和内含子个数等,推导出这6个基因的蛋白质长度从219个氨基酸到225个氨基酸不等(表1)。
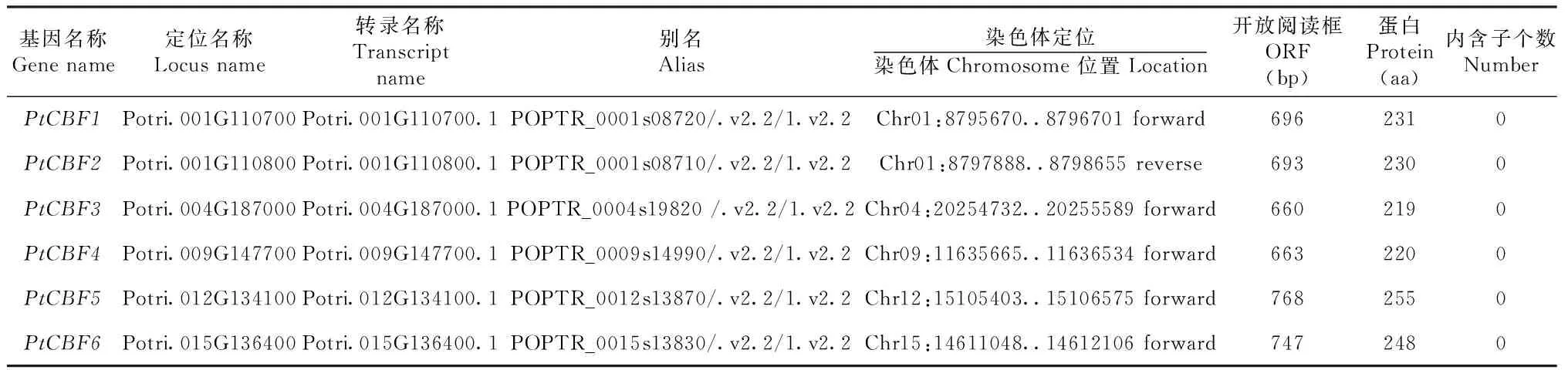
表1 毛果杨CBF/DREB1基因家族的基本特征

图1 拟南芥和毛果杨的CBF/DREB1家族基因编码蛋白保守基序分布Fig.1 Distribution of conserved motifs in Arabidopsis and P. trichocarpa CBF/DREB1 proteins
2.2 杨树CBF/DREB1基因家族保守结构域分析
利用在线软件MEME对6个AtCBF蛋白序列和6个PtCBF蛋白序列保守基序进行分析,结果表明拟南芥和毛果杨CBF基因家族蛋白序列中均包含由基序1(相似度为5.0e-454)和基序2(相似度为8.2e-259)共同构成的AP2保守结构域(图1)。
2.3 杨树CBF/DREB1基因家族蛋白相似性和系统进化分析
利用进化树分析软件MEGA 6.0 对6个拟南芥AtCBF蛋白、5个海岛棉GbCBF蛋白、8个胡杨PeuCBF蛋白和6个毛果杨PtCBF蛋白全蛋白序列进行进化分析。结果显示:这26个CBF按照同源进化关系可以分为3个亚家族,分别含有13、4和8个成员(图2)。第Ⅰ亚家族由拟南芥、海岛棉、胡杨和毛果杨共同组成;第Ⅱ亚家族由胡杨和毛果杨组成,第Ⅲ亚家族中不含有拟南芥成员。毛果杨CBF/DREB1基因家族分布于3个不同的亚家族中,其中毛果杨PtCBF5和PtCBF6、胡杨PeuCBF1和PeuCBF5、拟南芥AtCBF1~6、海岛棉GbCBF1~3聚类于第Ⅰ亚家族;毛果杨PtCBF3和PtCBF4、胡杨PeuCBF4和PeuCBF8聚类
于第Ⅱ亚家族;毛果杨PtCBF1、PtCBF2、胡杨PeuCBF2、PeuCBF3、PeuCBF6、PeuCBF7、海岛棉GbCBF4和GbCBF5聚类于第Ⅲ亚家族。利用欧洲生物信息研究所在线Clustal Omega程序对6个毛果杨的蛋白序列进行多重序列比对,发现他们的氨基酸相似性在46.45 %~81.31 %,其中PtCBF3 和PtCBF4氨基酸序列的相似性达到 81.31 %(表2)。

图2 拟南芥、海岛棉、胡杨和毛果杨的CBF/DREB1蛋白进化树Fig.2 Phylogenetic tree for Arabidopsis,Gossypium barbadense,P. euphratica and P. trichocarpa CBF/DREB1 proteins
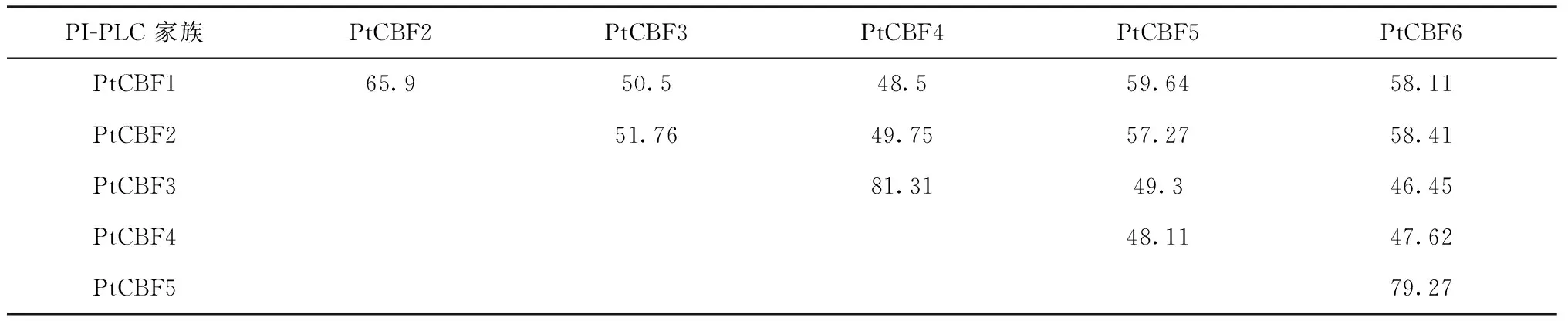
PI-PLC家族PtCBF2PtCBF3PtCBF4PtCBF5PtCBF6PtCBF165.950.548.559.6458.11PtCBF251.7649.7557.2758.41PtCBF381.3149.346.45PtCBF448.1147.62PtCBF579.27
表3毛果杨基因组中CBF/DREB1蛋白潜在磷酸化位点分析
Table 3 Analysis of the CBF/DREB1 proteins potential phosphorylation site inP.trichocarpa

CBF家族丝氨酸Serine苏氨酸Threoine酪氨酸TyrosinePtCBF12141PtCBF21451PtCBF31070PtCBF41573PtCBF51561PtCBF61971
2.4 杨树CBF/DREB1基因家族蛋白磷酸化位点预测
利用在线软件NetPhos 3.1 Serverl对6个毛果杨CBF/DREB1蛋白进行磷酸化位点预测,结果显示PtCBF1~6蛋白中存在着17~27个丝氨酸、苏氨酸及酪氨酸潜在磷酸化位点,预测磷酸化位点主要是以丝氨酸的形式存在,其次是苏氨酸,络氨酸的磷酸化位点最少(表3)。预测PtCBF1潜在磷酸化位点中存在21个丝氨酸、4个苏氨酸和1个酪氨酸位点。PtCBF3潜在蛋白磷酸化位点中不含有酪氨酸(图3)。
3 讨 论
本研究鉴定毛果杨CBF/DREB1基因家族含有6个成员,而已鉴定的拟南芥[18]、水稻[18]、海岛棉[12]和胡杨[15]CBF/DREB1基因家族分别含有6、10、5和8个成员,这说明在植物进化过程中,CBF/DREB1基因可能经历了不断发生谱系的特异扩张和拷贝丢失。毛果杨CBF/DREB1基因家族的6个成员均只含有1个外显子,均含有AP2结构域,且在AP2结构域的前后分别含有PKKPAGR和DSAWR序列,这表明CBF/DREB1基因家族的高度保守性,推测这些转录因子在植物诸多生理过程中起着类似作用。不同植物中CBF/DREB1基因家族含有的成员数目存在差异,推测不同植物的各成员在特定组织响应低温、干旱和高盐等信号后精细调节各生理过程。
进化分析表明,毛果杨CBF/DREB1基因家族分布于不同的亚家族中,PtCBF5、PtCBF6、PeuCBF1、PeuCBF5、AtCBF1~6和GbCBF1~3聚类于第Ⅰ亚家族;PtCBF3、PtCBF4、PeuCBF4和PeuCBF8聚类于第Ⅱ亚家族;PtCBF1、PtCBF2、PeuCBF2、PeuCBF3、PeuCBF6、PeuCBF7、GbCBF4和GbCBF5聚类于第Ⅲ亚家族。目前,CBF/DREB1基因家族各成员确切的功能分析主要集中在拟南芥中。近年,伴随着植物基因组学、生物信息学等新兴学科的兴起,大量的CBF基因从不同植物中克隆出来,部分林木CBF/DREB1基因成功克隆并功能也得到初步分析。第Ⅲ亚家族中毛果杨PtCBF1、PtCBF2和胡杨PeuCBF2、PeuCBF3、PeuCBF6 和PeuCBF7 与并不是一一对应关系,表明经过长期进化选,毛果杨和胡杨CBF/DREB1基因也发生了分离,这与贾会霞[15]等的研究结果一致。胡杨PeuCBF1~3和PeuCBF5~7均在雄花序和雌花序的表达量较高,且胡杨PeuCBF1~3和PeuCBF5~6主要响应低温胁迫。据此推测: 毛果杨PtCBF1和PtCBF2可能也主要在雄花序和雌花序中表达且响应低温胁迫。最近的研究发现,胡杨PeuCBF4过量表达转化三倍体毛白杨,获得的转基因毛白杨的光和效率较对照(野生型和转空载体转基因毛白杨)增加34.7 %~165.7 %、瞬间水利用效率增加48.9 %~103.7 % 、叶绿素荧光fv/fm增加2.14 %~5.89 % ;同时SOD活性增加、MDA含量降低;转基因毛白杨表现出PeuCBF4、PtRCI2A 和PtDI21表达量显著上调同时伴随有植株矮化现象。表明:胡杨PeuCBF4过量表达能够显著增强转基因植株的抗逆性[11]。鉴于毛果杨PtCBF4与PeuCBF4同源性最高,据此推测毛果杨PtCBF4在植物抗逆生理中发挥重要作用。聚类在一起的不同植物的同源基因可能具有不同的生物学功能[19],因此不能简单推测毛果杨CBF/DREB1基因家族各成员的生物学功能,其各成员确切的生物学功能还需要逐一进行功能验证。
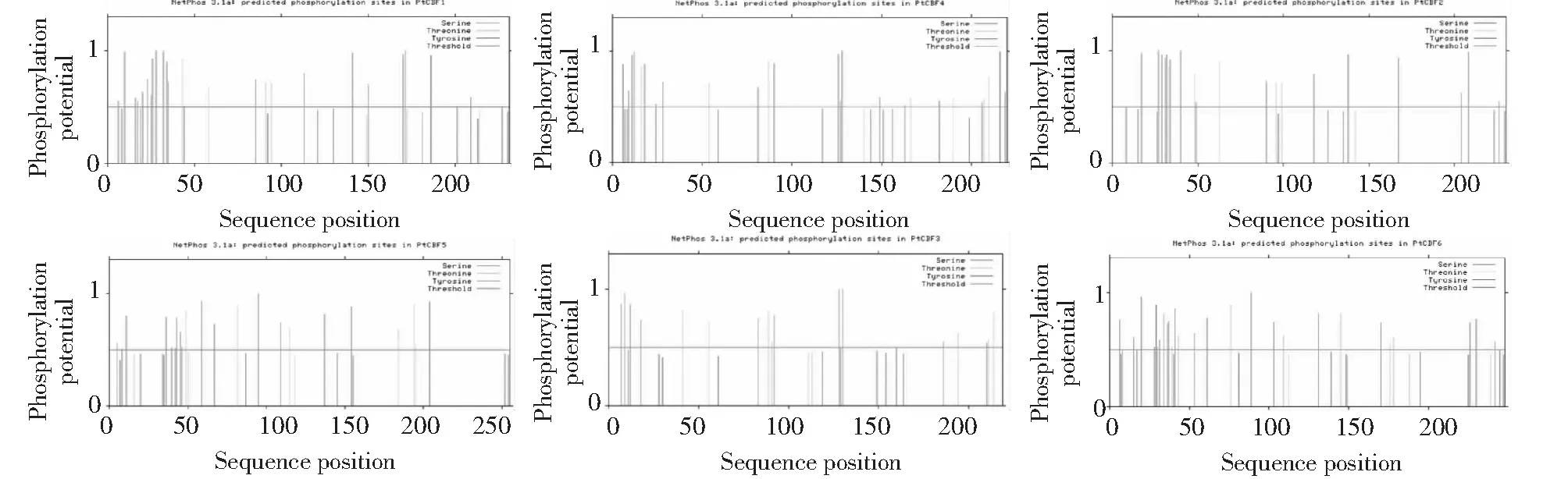
图3 毛果杨CBF/DREB1蛋白磷酸化位点预测Fig.3 Phosphorylation site prediction of CBF/DREB1 proteins in P. trichocarpa
磷酸化分析表明,毛果杨PtCBFs蛋白存在大量潜在磷酸化位点。目前,植物CBF/DREB1 转录因子确切的磷酸化作用尚未见报道。研究发现御谷PgDREB2A 是一个磷酸化蛋白,其体外磷酸化位点是苏氨酸残基。御谷PgDREB2A蛋白磷酸化后不能与RD29A启动子上DRE 元件(ACCGAC)结合,在磷酸酶作用下,PgDREB2A 结合DRE 元件的能力得到了恢复,表明:御谷PgDREB2A蛋白磷酸化作用负调控其对DNA 的结合活性[20]。毛果杨PtCBFs蛋白中潜在的磷酸化位点还需要通过定点突变、体外磷酸化等试验进一步研究和验证。
4 结 论
毛果杨基因组中鉴定出6个CBF/DREB1基因家族成员,均不含有内含子,位于毛果杨的第1、4、9、12和15染色体上。毛果杨CBF/DREB1蛋白均含有AP2结构域及其前后的特征序列:PKKPAGR和DSAWR均含有大量潜在的磷酸化位点。
参考文献:
[1]Jaglo K R, Thomashow M F. Components of theArabidopsisC-repeat/dehydration-responsive element binding factor cold-response pathway are conserved inBrassicanapusand other plant species[J]. Plant Physiology, 2001, 127(3):910-917.
[2]Navarrete-Campos D, Feuvre R L, Balocchi C, et al. Overexpression of three novel CBF transcription factors fromEucalyptusglobulus, improves cold tolerance on transgenicArabidopsisthaliana[J]. Trees, 2017:1-15.
[3]Novillo F, Medina J, Rodríguezfranco M, et al. Genetic analysis reveals a complex regulatory network modulating CBF gene expression andArabidopsisresponse to abiotic stress[J]. Journal of Experimental Botany, 2012, 63(1):293-304.
[4]Zhou M, Hu C, Wei D, et al.ArabidopsisCBF3 and DELLAs positively regulate each other in response to low temperature[J]. Scientific Reports, 2017(7):39819.
[5]Stockinger E J, Gilmour S J, Thomashow M F.ArabidopsisthalianaCBF1 encodes an AP2 domain-containing transcriptional activator that binds to the C-repeat/DRE, acis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(3):1035-1040.
[6]Gilmour S J, Zarka D G, Stockinger E J, et al. Low temperature regulation of theArabidopsisCBF family of AP2 transcriptional activators as an early step in cold-inducedCORgene expression[J]. Plant Journal, 1998, 16(4):433-442.
[7]Haake V, Cook D, Riechmann J, et al. Transcription factor CBF4 is a regulator of drought adaptation inArabidopsis[J]. Plant Physiology, 2002, 130(2):639-648.
[8]Sakuma Y, Liu Q, Dubouzet J G, et al. DNA-binding specificity of the ERF/AP2 domain ofArabidopsisDREBs, transcription factors involved in dehydration-and cold-inducible gene expression[J]. Biochemical and Biophysical Research Communications, 2002, 290(3):998-1009.
[9]Magome H, Yamaguchi S, Hanada A, et al. dwarf and delayed-flowering 1, a novelArabidopsis, mutant deficient in gibberellin biosynthesis because of overexpression of a putative AP2 transcription factor[J]. Plant Journal, 2004, 37(5):720-729.
[10]Liu S, Wang X, Wang H, et al. Genome-wide analysis ofZmDREBgenes and their association with natural variation in drought tolerance at seedling stage ofZeamaysL[J]. Plos Genetics, 2012, 9(9):e1003790.
[11]Tian Q, Chen J, Wang D, et al. Overexpression of aPopuluseuphraticaCBF4 gene in poplar confers tolerance to multiple stresses[J]. Plant Cell Tissue & Organ Culture, 2017, 128(2):391-407.
[12]李 月, 代培红, 刘 超, 等. 海岛棉5个CBF/DREB基因的克隆与表达分析[J]. 棉花学报, 2016, 28(1):42-51.
[13]Tuskan G A, Difazio S, Jansson S, et al. The genome of black cottonwood,Populustrichocarpa(Torr. & Gray)[J]. Science, 2006, 313(5793): 1596-1604.
[14]Larkin M A, Blackshields G, Brown N P, et al. Clustal W and clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948.
[15]贾会霞, 李建波, 孙 佩, 等. 胡杨CBF基因家族的鉴定及表达特性分析[J]. 分子植物育种, 2017, 15(2): 492-500.
[16]Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729.
[17]Bailey T L, Boden M, Buske F A, et al. MEME SUITE: Tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37(Web Server issue): 202-208.
[18]Nakano T, Suzuki K, Fujimura T, et al. Genome-wide analysis of the ERF gene family inArabidopsisand rice[J]. Plant Physiology, 2006, 140(2):411-432.
[19]张杰伟, 任 飞, 张中保, 等. 桃磷酸肌醇特异性磷脂酶C基因家族鉴定与分析[J]. 江苏农业学报, 2017, 33(1):185-190.
[20]Agarwal P, Agarwal P K, Nair S, et al. Stress-inducible DREB2A transcription factor fromPennisetumglaucumis a phosphoprotein and its phosphorylation negatively regulates its DNA-binding activity[J]. Molecular Genetics and Genomics, 2007, 277(2):189-198.

