唐古特虎耳草谱系地理学研究
更吉卓玛,李 彦,贾留坤,夏铭泽,高庆波
(1 中国科学院西北高原生物研究所 中国科学院高山植物适应与进化重点实验室,西宁 810001;2 中国科学院大学,北京 100039;3 中国科学院西北高原生物研究所 青海省作物分子育种重点实验室,西宁 810001)
由于复杂的地理拓扑结构、微环境以及更广泛的生态位,高山地区往往具有较高的生物多样性,如高加索山、安第斯山、阿尔卑斯山等[1-3]。青藏高原及其周边的高山地区包含了3个全球生物多样性热点地区,即东部的横断山区、南部的喜马拉雅山系及西部的阿尔泰和天山山脉[4]。一般认为,与造山运动相关的地理隔离和异域分化是导致青藏高原地区植物多样性较高的原因之一[5-6]。快速辐射分化在青藏高原地区的很多植物类群中被检测到,如马先蒿属(PedicularisL.)[7]、红景天属(RhodiolaL.)[8]、风毛菊属(SaussureaDC.)[9]、虎耳草属(SaxifragaL.)[10-11]、槖吾属-垂头菊属-蟹甲草属复合群(LigulariaCass.-CremanthodiumBenth.-ParasenecioW. W. Sm. & J. Small)[12]等。然而,与较高分类阶元的分化模式不同,青藏高原地区植物种内居群间的分化可能更多的受到第四纪冰期气候波动的影响。更新世晚期以来,青藏高原经历了至少4次规模较大的冰期,其中聂聂雄拉冰期(第二冰期,约720~500 ka)强度最大,冰川扩张最广,此后冰期强度逐渐减弱[13-14]。而全球末次盛冰期时青藏高原冰川的扩张程度非常有限,这与欧洲和北美大陆末次冰期形成统一的冰盖不同[15]。第四纪冰期与间冰期交替所引起的气候波动对于现存生物类群的地理分布格局和遗传结构产生了极大的影响[16-17]。由于复杂的地理地貌特征、未被统一的大冰盖覆盖以及东亚季风气候和印度季风气候的影响,青藏高原地区的植物可能具有更复杂的第四纪冰期进化历史[18-19]。一方面,某些植物,如祁连圆柏(Juniperusprzewalskii)[20]、青海云杉(Piceacrassifolia)[21]等,在第四纪冰期时由高原台面退缩到海拔较低的东南部边缘避难所内,间冰期或冰期后再从边缘避难所回迁到高原台面,从而形成现有的地理分布格局。另一方面,由于第四纪冰期时青藏高原未被统一的大冰盖覆盖,仅有不同程度的山地冰川发育,高原台面可能存在适宜的局部微环境而成为某些耐寒植物的避难所。对银露梅(Potentillaglabra)[22]、露蕊乌头(Aconitumgymnandrum)[23]和西川红景天(Rhodiolaalsia)[24]等的研究揭示,在第四纪冰期时高原台面的居群并没有退缩到高原边缘避难所,而是在高原台面上的一个或几个较大的避难所中保留下来,间冰期或冰期后再由避难所向附近扩散。此外,对西藏圆柏复合群(Juniperustibeticacomplex)[25]、西藏沙棘(Hippophaetibetana)[26]、菊叶红景天(Rhodiolachrysanthemifolia)[27]等的研究发现,这些物种第四纪冰期时在高原台面存在多个微型避难所,这些微型避难所在物种的分布范围内呈均匀分布。然而,具有不同分布范围、生境和生活史特征的植物可能具有不同的第四纪冰期进化历史。对青藏高原地区更多的植物进行谱系地理学研究,不但可为高山植物演化历史与第四纪冰期的关系提供依据,同时对青藏高原生物多样性保护具有重要启示。
虎耳草属是虎耳草科(Saxifragaceae)最大的属,约有450~500种,主要分布在欧洲和亚洲的高山地区,某些物种环北极分布,另有一些物种扩散到南美洲的落基山脉、安第斯山脉和火地岛[10-11]。中国产虎耳草属植物约220种,主要分布在青藏高原-喜马拉雅地区[28]。虎耳草属植物被广泛地用于谱系地理学和系统发育学研究,来揭示北极和高山地区植物的分化式样[10-11, 29-32]。然而,青藏高原作为虎耳草属物种多样性中心之一,该地区虎耳草属植物种内分化与第四纪冰期的关系尚不明确。唐古特虎耳草(S.tanguticaEngl.)为多年生草本植物,生于海拔2 900~4 600 m的高山草甸、灌丛、石隙,分布于青海、甘肃、四川、西藏等地,是青藏高原地区高山草甸的重要组成成分,对维持高寒草甸生态系统的稳定具有重要意义。本研究选取叶绿体DNA(cpDNA)非编码区trnL-trnF和rpl16片段对青藏高原地区的唐古特虎耳草进行谱系地理学研究,目的在于揭示该物种居群内与居群间的遗传分布式样,讨论唐古特虎耳草对于第四纪气候波动的响应,探讨其现有地理分布格局的历史成因。
1 材料和方法
1.1 实验材料
研究材料于2007年至2014年采自青海、西藏、四川、甘肃,共采集唐古特虎耳草18个居群209个个体(表1,图1)。每个居群随机采集2~24个个体,个体之间相隔至少10 m以上。采集生长良好植株的新鲜嫩叶后立即用硅胶干燥,带回实验室存放于-20 ℃冰箱备用。凭证标本由R. J. Gornall教授和高庆波副研究员共同鉴定,馆藏于中国科学院西北高原植物研究所青藏高原生物标本馆(HNWP)。
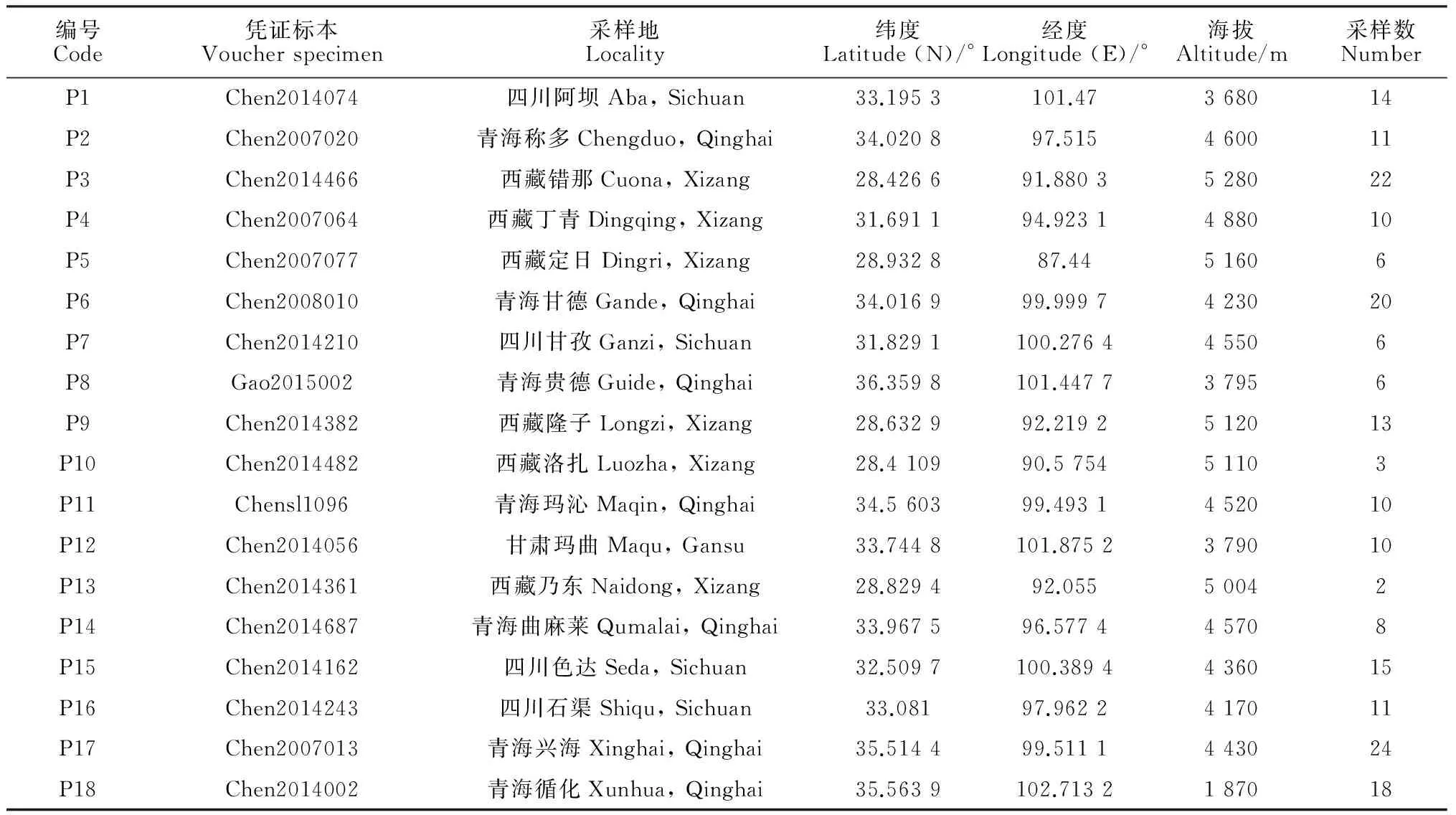
表1 唐古特虎耳草18个居群的采集信息

饼状图表示每个居群中单倍型的频率图1 唐古特虎耳草18个居群的采样图以及叶绿体单倍型分布图Pie charts show the proportions of haplotypes within each populationFig.1 Map of the 18 sampled populations of S. tangutica and the distribution of cpDNA haplotypes in the species
1.2 DNA提取与PCR扩增
本研究采用改良的CTAB法从硅胶干燥的叶片中提取总DNA[33]。采用通用引物对所有个体的cpDNAtrnL-trnF和rpl16片段进行PCR扩增[34-35]。PCR扩增反应体系为50 μL:包含5 μL的10 × PCR Buffer(含1.5 mmol/L MgCl2),2 μL的10 mmol/L dNTPs,正反引物各1.0 μL(5 pmol/L),TaqDNA聚合酶(TaKaRa,大连)0.4 μL,1 μL的DNA模板,用双蒸水补足到50 μL。PCR扩增反应程序:94 ℃预变性10 min;94 ℃变性1 min,58 ℃退火50 s,72 ℃延伸1 min,32循环;72 ℃延伸10 min。PCR产物用1%的琼脂糖凝胶电泳进行检测,然后送测序公司进行双向测序。
1.3 数据分析
利用Chromas 2.6.2软件(http://www.technelysium.com.au)对测序峰图进行肉眼核对。利用MEGA 7.0.26软件对序列进行对位排列并统计序列的碱基组成[36],发现trnL-trnF序列上存在一段约20 bp的poly A结构,为了确保碱基变异的可靠性,poly A及两侧约40 bp的碱基被删除。同样,对rpl16序列中出现的大于11 bp的poly A和poly T结构也进行了类似的删除处理。利用DnaSP 5.10软件统计cpDNA联合序列的变异位点并确定单倍型[37]。总遗传多样性(HT)、居群内平均遗传多样性(Hs)、居群间遗传分化系数GST和NST值均利用PERMUT软件进行计算[38]。其中GST仅考虑单倍型的频率,而NST兼顾了单倍型频率和单倍型之间的相似性。使用U-统计方法对GST和NST进行比较(1 000次重复的置换检验)以检测单倍型变异的地理分布模式。NST大于GST且显著,表明居群在分布范围内具有显著的谱系地理结构。由于PERMUT软件要求每个居群至少要有3个个体,所以,居群P13(2个个体)在进行PERMUT分析时未包含在内。
运用Arlequin 3.5.2计算每个居群的单倍型多样性(He)和核苷酸多样性(π)[39]。利用Arlequin软件包中的分子变异分析(AMOVA)检测居群内和居群间的遗传变异水平,并对单倍型分布的FST进行评价(1 000次置换检验)[40]。Tajima’sD、Fu & Li’sD*两种无限突变位点模型的中性检验及歧点分布分析都在DnaSP 5.10程序中完成[41-42]。
以最大简约法为原则,利用NETWORK 4.6.1.3软件构建单倍型中央连接网状图[43-44]。在运行该软件时,对于多碱基的插入、缺失或突变位点,被认为是通过一步突变形成。在单倍型Network图中,原始的单倍型多存在于网络图的内部,而近代起源的单倍型发生在网络图的边缘位置[45-46]。
2 结果与分析
2.1 唐古特虎耳草的单倍型分布
对18个居群209个个体的trnL-trnF和rpl16片段进行测序,MEGA软件对位排列后的矩阵长度为1 626 bp,通过DnaSP软件鉴定出74个单倍型,有25个简约信息位点(表2)。在检测出的74个单倍型中,有53个单倍型是居群特有单倍型,单倍型H5出现的频率最高,有46个个体拥有此单倍型。表3显示在18个居群中,16个居群拥有特有单倍型。居群的遗传多样性(He)范围为0.490 9~1.000 0,平均为0.853 6;居群的核苷酸多样性(π)范围为0.007 506~0.046 481,平均为0.022 145。
2.2 居群遗传结构特征
通过PERMUT程序计算得出唐古特虎耳草居群内平均遗传多样性HS值(0.843)、总遗传多样性HT值(0.933)、居群间遗传分化GST(0.097)和NST值(0.109)。使用U-统计方法对唐古特虎耳草单倍型变异的地理结构进行检验后发现NST大于GST,但不显著(P> 0.05),且居群间的遗传分化水平很低,表明唐古特虎耳草在现有分布区域内没有明显的谱系地理结构。分子变异分析(AMOVA)的结果表明,居群间的遗传变异仅为8.15%(FST= 0.081 45;P< 0.01),而居群内的遗传变异为91.85%(表4),进一步揭示了唐古特虎耳草遗传变异主要存在于居群内。
2.3 基于cpDNA联合序列的居群历史动态分析
对唐古特虎耳草cpDNA联合序列所有个体进行中性检验,结果表明,Tajima’sD(-2.045 07,P< 0.05)和Fu & Li’D*(-3.629 27,P< 0.05)均为显著的负值,推测其分布范围或者个体数近期经历过扩张。歧点分布分析结果显示,在唐古特虎耳草整个分布范围内观测到的歧点分布呈单峰曲线(图2),其SSD值(0.007 2)和Raggednes index值(0.007 6)均不显著(P> 0.05),说明该物种在整个分布范围内经历过近期扩张现象,这与中性检验结果一致。
2.4 单倍型之间的系统发育关系
最大简约法(maximum parsimony, MP)、最大似然法(maximum likelihood, ML)、贝叶斯法(Bayesian inference, BI)构建的单倍型系统发育树均呈现“刷子”结构,不能很好地揭示单倍型之间的系统发育关系,也表明了唐古特虎耳草单倍型的快速辐射分化(结果未显示)。NETWORK软件可更好揭示遗传分化较浅序列之间的系统发育关系。基于最大似然法构建74个单倍型的中央链接网状图,结果显示(图3),居群共享单倍型,如H1、H2、H3、H5、H16等位于网状图的中央位置,而居群特有单倍型一般位于网状图的边缘。根据溯祖理论,共享单倍型为较古老的单倍型,而居群特有单倍型可能是通过近期辐射分化衍生而来的较年轻的单倍型。

表2 唐古特虎耳草74个单倍型trnL-trnF和rpl16片段简约信息位点
续表2Continued Table 2

单倍型Hap简约信息位点ParsimonyinformativesitetrnL-trnFrpl16230355414456611792813928966988100010901124119213341352135913601414145114571465147915511562H53-------------------------H54--------------〛A----------H55---------〛C--〛A------------H56--------〛T--------〛GC-----〛TH57-----------〛T-〛A------〛A---〛TH58-------------------------H59-------------------------H60----------------〛A--------H61----------〛A--------------H62-------------------------H63---------〛C--〛A------------H64--------〛T---------------〛TH65-C-----C-〛C---------------H66----C------〛T-----------〛GTH67--------〛T--〛T-------〛AA-〛A-〛TH68-------------------------H69-C----A-------〛A----------H70--------------〛A----------H71-------------------------H72A----T------------------〛TH73----------------〛A--------H74----------〛A--------------
注:-代表与单倍型H1相同的碱基
Note: - stands for the same base ashaplotypeH1
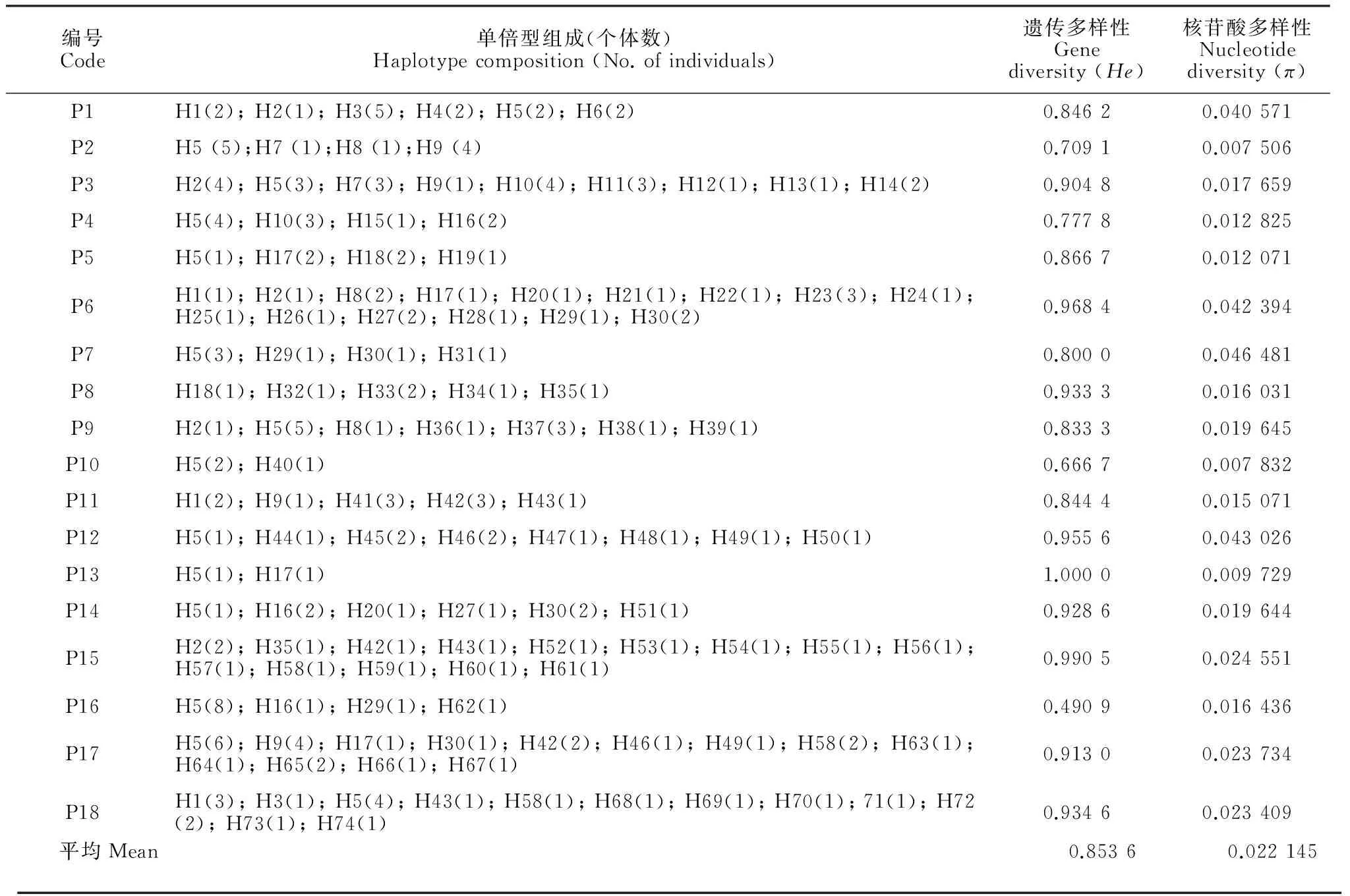
表3 唐古特虎耳草18个居群的单倍型组成、遗传多样性和核苷酸多样性

表4 唐古特虎耳草18个居群的分子遗传变异(AMOVA)分析结果

图2 唐古特虎耳草209个个体叶绿体DNA2个片段联合数据的歧点分布分析Fig.2 Mismatch distribution for 209 individuals of S. tangutica based on two cpDNA fragments
3 讨 论
3.1 唐古特虎耳草的居群遗传结构
遗传变异分析表明唐古特虎耳草cpDNA片段总遗传多样性水平较高,HT= 0.933。类似的结果也在青藏高原很多高山植物中被检测到,如偏花报春(Primulasecundiflora,HT= 0.966)[47]、西藏沙棘(Hippophaetibetana,HT= 0.956)[26]、绵参(Eriophytonwallichii,HT= 0.979)[48]、西川红景天(Rhodiolaalsia,HT= 0.950)[24]、菊叶红景天(Rhodiolachrysanthemifolia,HT= 0.937)[27]等。然而,这些物种大多数都具有低的居群内平均遗传多样性。在本研究中,唐古特虎耳草不仅具有较高的总遗传多样性,居群内的平均遗传多样性也很高(HS= 0.843),表明唐古特虎耳草较高的遗传变异主要由居群内遗传变异所致。这一结果也得到了分子变异分析(AMOVA)的证实。AMOVA分析表明,唐古特虎耳草居群内遗传变异占总变异的91.85%,而居群间遗传变异仅为8.15%,进一步揭示了唐古特虎耳草遗传变异主要存在于居群内。如此高的居群内遗传变异在青藏高原的植物中并不多见,推测造成该遗传结构的原因如下:一方面,由于检测到的74个单倍型中只有少数共享单倍型,但其个体数所占比例很高(67.46%),并且这些共享单倍型在居群中随机分布,因此降低了居群间的遗传差异;另一方面,居群特有单倍型比例高(53/74),某些居群固定多个特有单倍型,从而增加了居群内的遗传变异。特有单倍型比例较高的物种往往不能揭示出明显的谱系地理格局[22, 24, 27]。 此外,对虎耳草属山羊臭组(Saxifragasect.CiliataeHaw.)系统发育和物种形成的研究表明,唐古特虎耳草所在的唐古拉亚组(subsect.HirculoideaeEngl. & Irmsch.)具有较快的分化速率,该亚组约110个物种是在最近的2.12百万年(Ma)分化而来[10]。由此推测,唐古特虎耳草具有更短的居群分化历史。事实上,基于NETWORK构建的单倍型系统发育关系表明,大量的特有单倍型位于NETWORK图的边缘,揭示了唐古特虎耳草单倍型的近期快速分化。在第四纪冰期前,唐古特虎耳草可能广泛分布于青藏高原及其周边地区,并广泛固定某些古老单倍型(如H5)。随着第四纪冰期气候的波动,原来连续的居群片段化,形成了相互隔离的居群,并经历了异域分化,产生大量特有单倍型。在此过程中,瓶颈效应和遗传漂变等作用使得特有单倍型在居群中固定下来,抹去了居群原有的部分遗传结构。由于单独进化时间较短,特有单倍型之间的差异大多表现在单个碱基的变异。
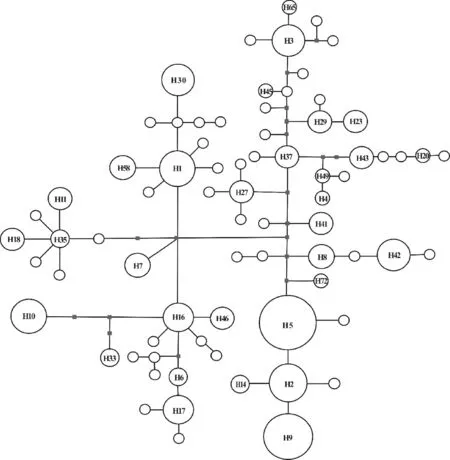
圆的大小表示单倍型频率,实心方块代表缺失单倍型图3 唐古特虎耳草cpDNA联合片段74种单倍型的中央连接网状图Circle size is proportional to haplotype frequencies, parallelograms represent missing haplotypesFig.3 Median-joining network of 74 haplotypes of S. tangutica based on two cpDNA fragments
3.2 冰期避难所及居群进化历史的推测
第四纪循环的气候波动对生物区系的分布和遗传结构产生了巨大的影响[49]。植物对第四纪冰期的反应可以根据现有的遗传结构进行追溯。于海彬等[50]综述了36种高山植物的谱系地理格局,总结出高山植物面对青藏高原复杂的地理结构、微气候和生境条件所呈现出的谱系历史。一部分高山植物在第四纪冰期时退缩到青藏高原边缘避难所,冰期后回迁到高原台面。符合这种“退缩-回迁”模式的物种,其遗传结构表现为遗传多样性较高的居群和古老单倍型往往集中在高原边缘避难所内,居群遗传多样性由高原边缘向高原台面递减。此外,一些耐寒植物,在冰期时没有完全退缩到低海拔的边缘地区,而是在高原台面形成了一个或几个相互隔离的避难所,冰期后发生局部扩张。符合这种“高原台面避难所-局部扩张”模式的物种,其遗传结构表现为特有单倍型或者遗传多样性较高的居群分布在高原台面上的一个或几个相互隔离的区域。另外,某些物种第四纪冰期时在高原台面形成多个微型避难所,间冰期或冰期后发生小范围的扩张,其遗传多样性较高的居群和特有单倍型在物种的分布范围内均匀分布。
本研究没有揭示出唐古特虎耳草遗传多样性较高的某一地理区域,也没有发现遗传多样性由高原边缘向高原台面递减。相反,本研究所涉及的唐古特虎耳草居群普遍具有较高的遗传多样性,特有单倍型在分布范围内均匀分布。我们推测,由于青藏高原地形的高度异质性以及复杂的局部微气候和生境条件,在第四纪冰期时,唐古特虎耳草在高原台面上未被冰川覆盖的多个微型避难所中存活下来。唐古特虎耳草居群对第四纪冰期气候波动的反应可能更多地表现为垂直海拔高度的迁移,而非大规模的水平迁移,这与银露梅(Potentillaglabra)[22]和西川红景天(Rhodiolachrysanthemifolia)[27]的进化历史相似。居群在垂直迁移的过程中发生了瓶颈效应和奠基者效应,居群随机固定少量古老单倍型。随后的异域分化和遗传漂变使得较年轻的衍生特有单倍型在居群中固定下来,形成了唐古特虎耳草现有的居群遗传结构。居群历史动态检验结果表明,Tajima’sD和Fu & Li’D*均为显著的负值,歧点分布分析呈单峰曲线,说明唐古特虎耳草经历过近期分布范围或者个体数的扩张[51-53]。物种经历分布范围扩张事件,其原分布中心应具有较高的遗传多样性和特有单倍型,并且原分布中心的居群包含扩散区域居群所具有的单倍型。但在本研究中,大量的特有单倍型在唐古特虎耳草的整个分布范围内均匀分布,遗传多样性和核苷酸多样性较高的居群也呈现均匀分布,并未检测到分布范围扩张的信号。由此推测,该物种可能经历过个体数快速扩张事件,从而导致了中性检验的负值和错配分布的单峰曲线。
综上所述,唐古特虎耳草具有较高的遗传变异,且主要由居群内遗传变异所致,大量特有单倍型是近期辐射分化的结果。唐古特虎耳草的进化历史与西藏圆柏复合群(Juniperustibeticacomplex)[25]、西藏沙棘(Hippophaetibetana)[26]、菊叶红景天(Rhodiolachrysanthemifolia)[27]相似,第四纪冰期时在高原台面存在多个微型避难所。第四纪冰期气候波动使得原来连续的居群片段化,形成了相互隔离的居群,并发生异域快速分化,形成大量特有单倍型。唐古特虎耳草居群对第四纪冰期气候波动的反应可能更多地表现为垂直海拔高度的迁移,而非大规模的水平迁移。此研究为青藏高原地区谱系地理学研究提供了一个新的案例,为了解第四纪冰期高山植物的避难所和现有分布格局的成因提供了参考依据。
参考文献:
[1]MYERS N, MITTERMEIER R A, MITTERMEIER C G,etal. Biodiversity hotspots for conservation priorities [J].Nature, 2000,403(6 772): 853-858.
[2]HOORN C, MOSBRUGGER V, MULCH A,etal. Biodiversity from mountain building [J].NatureGeoscience, 2013,6(3): 154.
[3]HUGHES C E. The tropical Andean plant diversity powerhouse [J].NewPhytologist, 2016,210(4): 1 152-1 154.
[5]WEN J, ZHANG J-Q, NIE Z-L,etal. Evolutionary diversifications of plants on the Qinghai-Tibetan Plateau [J].FrontiersinGenetics, 2014,5: 4.
[6]XING Y, REE R H. Uplift-driven diversification in the Hengduan Mountains, a temperate biodiversity hotspot [J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2017,114(17): E3444-E3451.
[7]EATON D A R, FENSTER C B, HEREFORD J,etal. Floral diversity and community structure inPedicularis(Orobanchaceae) [J].Ecology, 2012,93(8): S182-S194.
[8]ZHANG J Q, MENG S Y, WEN J,etal. Phylogenetic relationships and character evolution ofRhodiola(Crassulaceae) based on nuclear ribosomal ITS and plastidtrnL-FandpsbA-trnHsequences [J].SystematicBotany, 2014,39(2): 441-451.
[9]WANG Y J, SUSANNA A, VON RAAB-STRAUBE E,etal. Island-like radiation ofSaussurea(Asteraceae: Cardueae) trigged by uplifts of the Qinghai-Tibetan Plateau [J].BotanicalJournaloftheLinneanSociety, 2009,97(4): 893-903.
[10]GAO Q B, LI Y H, GORNALL R J,etal. Phylogeny and speciation inSaxifragasect.Ciliatae(Saxifragaceae): evidence frompsbA-trnH,trnL-Fand ITS sequences [J].Taxon, 2015,64(4): 703-713.
[11]EBERSBACH J, MUELLNER-RIEHL A N, MICHALAK I,etal. In and out of the Qinghai-Tibet Plateau: divergence time estimation and historical biogeography of the large arctic-alpine genusSaxifragaL [J].JournalofBiogeography, 2017,44(4): 900-910.
[12]LIU J Q, WANG Y J, WANG A L,etal. Radiation and diversification within theLigularia-Cremanthodium-Paraseneciocomplex (Asteraceae) trigged by uplift of the Qinghai-Tibetan Plateau [J].MolecularPhylogeneticsandEvolution, 2006,38(1): 31-49.
[13]ZHENG B, XU Q, SHEN Y. The relationship between climate change and Quaternary glacial cycles on the Qinghai-Tibetan Plateau: review and speculation [J].QuaternaryInternational, 2002,97-98(1): 93-101.
[14]OWEN L A, BENN D I. Equilibrium-line altitudes of the Last Glacial Maximum for the Himalaya and Tibet: an assessment and evaluation of results [J].QuaternaryInternational, 2005,138-139(3): 55-78.
[15]LEHMKUHL F, OWEN L A. Late Quaternary glaciation of Tibet and the bordering mountains: a review [J].Boreas, 2005,34(2): 87-100.
[16]HEWITT G M. Post-glacial re-colonization of European biota [J].BiologicalJournaloftheLinneanSociety, 1999,68(1/2): 87-112.
[17]HEWITT G M. Speciation, hybrid zones and phylogeography-or seeing genes in space and time [J].MolecularEcology, 2001,10(3): 537-549.
[18]ZHANG D, FENGQUAN L, JIANMIN B. Eco-environmental effects of the Qinghai-Tibet Plateau uplift during the Quaternary in China [J].EnvironmentalGeology, 2000,39(12): 1 352-1 358.
[19]HEWITT G M. Genetic consequences of climatic oscillations in the Quaternary [J].PhilosophicalTransactionsoftheRoyalSocietyofLondonB:BiologicalSciences, 2004,359(1 442): 183-195.
[20]ZHANG Q, CHIANG T Y, GEORGE M,etal. Phylogeography of the Qinghai-Tibetan Plateau endemicJuniperusprzewalskii(Cupressaceae) inferred from chloroplast DNA sequence variation [J].MolecularEcology, 2005,14(11): 3 513-3 524.
[21]MENG L, YANG R, ABBOTT R J,etal. Mitochondrial and chloroplast phylogeography ofPiceacrassifoliaKom. (Pinaceae) in the Qinghai-Tibetan Plateau and adjacent highlands [J].MolecularEcology, 2007,16(19): 4 128-4 137.
[22]WANG L Y, IKEDA H, LIU T L,etal. Repeated range expansion and glacial endurance ofPotentillaglabra(Rosaceae) in the Qinghai-Tibetan Plateau [J].JournalofIntegrativePlantBiology, 2009,51(7): 698-706.
[23]WANG L, ABBOTT R J, ZHENG W,etal. History and evolution of alpine plants endemic to the Qinghai-Tibetan Plateau:Aconitumgymnandrum(Ranunculaceae) [J].MolecularEcology, 2009,18(4): 709-721.
[24]GAO Q, ZHANG D, DUAN Y,etal. Intraspecific divergences ofRhodiolaalsia(Crassulaceae) based on plastid DNA and internal transcribed spacer fragments [J].BotanicalJournaloftheLinneanSociety, 2012,168(2): 204-215.
[25]OPGENOORTH L, VENDRAMIN G G, MAO K,etal. Tree endurance on the Tibetan Plateau marks the world’s highest known tree line of the Last Glacial Maximum [J].NewPhytologist, 2010,185(1): 332-342.
[26]WANG H, QIONG L, SUN K,etal. Phylogeographic structure ofHippophaetibetana(Elaeagnaceae) highlights the highest microrefugia and the rapid uplift of the Qinghai-Tibetan Plateau [J].MolecularEcology, 2010,19(14): 2 964-2 979.
[27]GAO Q B, ZHANG F Q, XING R,etal. Phylogeographic study revealed microrefugia for an endemic species on the Qinghai-Tibetan Plateau:Rhodiolachrysanthemifolia(Crassulaceae) [J].PlantSystematicsandEvolution, 2016,302(9):1 179-1 193.
[28]WU C Y, RAVEN P H. Flora of China [M]. Beijing: Science Press; St. Louis: Missouri Botanical Garden Press, 2001,8: 208-344.
[29]ABBOTT R J, COMES H P. Evolution in the Arctic: a phylogeographic analysis of the circumarctic plant,Saxifragaoppositifolia(Purple saxifrage) [J].NewPhytologist, 2003,161(1): 211-224.
[30]DECHAINE E G, ANDERSON S A, MCNEW J M,etal. On the evolutionary and biogeographic history ofSaxifragasect.Trachyphyllum(Gaud.) Koch (Saxifragaceae Juss.) [J].PLoSOne, 2013,8(7): e69814.
[31]EBERSBACH J, SCHNITZLER J, FAVRE A,etal. Evolutionary radiations in the species-rich mountain genusSaxifragaL [J].BMCEvolutionaryBiology, 2017,17(1): 119.
[32]GAO Q B, LI Y, GENGJI Z M,etal. Population genetic differentiation and taxonomy of three closely related species ofSaxifraga(Saxifragaceae) from southern Tibet and the Hengduan Mountains [J].FrontiersinPlantScience, 2017,8: 1 325.
[33]DOYLE J J, DOYLE J L. A rapid DNA isolation procedure for small quantities of fresh leaf material [J].PhytochemicalBulletin,BotanicalSocietyofAmerica, 1987,19(1): 11-15.
[34]TABERLET P, GIELLY L, PAUTOU G,etal. Universal primers for amplification of three non-coding regions of chloroplast [J].PlantMolecularBiology, 1991,17(5): 1 105-1 109.
[35]SCARCELLI N, BARNAUD A, EISERHARDT W,etal. A set of 100 chloroplast DNA primer pairs to study population genetics and phylogeny in Monocotyledons [J].PLoSOne, 2011,6(5): e19954.
[36]KUMAR S, STECHER G, TAMURA K. MEGA 7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets [J].MolecularBiologyandEvolution, 2016,33(7): 1 870-1 874.
[37]LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data [J].Bioinformatics, 2009,25(11): 1 451-1 452.
[38]PONS O, PETIT R J. Measuring and testing genetic differentiation with ordered versus unordered alleles [J].Genetics, 1996,144(3): 1 237-1 245.
[39]EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows [J].MolecularEcologyResources, 2010,10(3):564-567.
[40]WEIR B S, COCKERHAM C C. Estimating F-Statistics for the analysis of population structure [J].Evolution, 1984,38(6): 1 358-1 370.
[41]TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism [J].Genetics, 1989,123(3): 585-595.
[42]FU Y X, LI W H. Statistical tests of neutrality of mutations [J].Genetics, 1993,133(3): 693-709.
[43]BANDELT H J, FORSTER P, RÖHL A. Median-joining networks for inferring intraspecific phylogenies [J].MolecularBiologyandEvolution, 1999,16(1): 37-48.
[44]POLZIN T, DANESHMAND S V. On Steiner trees and minimum spanning trees in hypergrahs [J].OperationsResearchLetters, 2003,31(1): 12-20.
[45]GOLDING G B. The detection of deleterious selection using ancestors inferred from a phylogenetic history [J].GeneticsResearch, 1987,49(1): 71-82.
[46]CRANDALL K A, TEMPLETON A R. Empirical tests of some predictions from coalescent theory with applications to intraspecific phylogeny reconstruction [J].Genetics, 1993,134(3): 959-969.
[47]WANG F Y, GONG X, HU C M,etal. Phylogeography of an alpine speciesPrimulasecundiflorainferred from the chloroplast DNA sequence variation [J].JournalofSystematicsandEvolution, 2008,46(1): 13-22.
[48]王晓雄,乐霁培,孙航,等.青藏高原高山流石滩特有植物绵参的谱系地理学研究 [J]. 植物分类与资源学报, 2011,33(6): 605-614.
WANG X X, YUE J P, SUN H,etal. Phylogeographical study onEriophytonwallichii(Labiatae) from Alpine scree of Qinghai-Tibetan Plateau [J].PlantDiversity&Resources, 2011,33(6): 605-614.
[49]DYNESIUS M, JANSSON R. Evolutionary consequences of changes in species' geographical distributions driven by Milankovitch climate oscillations [J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2000,97(16): 9 115-9 120.
[50]于海彬, 张镱锂. 青藏高原及其周边地区高山植物谱系地理学研究进展 [J]. 西北植物学报, 2013,33(6): 1 268-1 278.
YU H B, ZHANG Y L. Advances in phylogeography of alpine plants in the Tibetan Plateau and adjacent regions [J].ActaBotanicaBoreali-OccidentaliaSinica, 2013,33(6): 1 268-1 278.
[51]RAY N, CURRAT M, EXCOFFIER L. Intra-deme molecular diversity in spatially expanding populations [J].MolecularBiologyandEvolution, 2003,20(1): 76-86.
[52]EXCOFFIER L. Patterns of DNA sequence diversity and genetic structure after a range expansion: lessons from the infinite-island model [J].MolecularEcology, 2004,13(4): 853-864.
[53]HARPENDING H C, BATZER M A, GURVEN M,etal. Genetic traces of ancient demography [J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 1998,95(4): 1 961-1 967.

