皮肤黑色素瘤中神经母细胞瘤大鼠肉瘤基因突变分析
林 琳 倪 楠 赵海鹰 姜晓峰*
黑色素瘤作为皮肤肿瘤中侵袭性最高的恶性肿瘤之一,占皮肤恶性肿瘤的7%~20%[1]。研究表明,很多基因突变与黑色素瘤的发生、转移的相关性越来越密切[2-3]。黑色素瘤恶性度高、侵袭能力强,而且对化疗极不敏感,近年来,相关基因抑制剂在治疗晚期黑色素瘤上,取得了显著的疗效[4]。由于黑色素瘤发生相关基因较多,发现更多有效的治疗靶点成为研究的方向之一。
神经母细胞瘤大鼠肉瘤(neuroblastoma rat sarcoma,NRAS)基因全长85 kb,位于1号染色体,编码含495个氨基酸的蛋白质[5]。研究显示,在黑素瘤中,NRAS基因突变作为Ras家族中最常见的突变类型,突变率为20%[6-7]。NRAS是黑素瘤中第2个最常见的突变基因[8]。NRAS基因突变主要以Q6、G12、G13为主[9]。Uhara等[10],通过对127例黑素瘤患者检测,发现NRAS基因突变率7.1%且主要突变发生在Q61位点上,总比例为77.8%,对NRAS基因进行突变研究,对个体化的靶向治疗有着重要的意义。本研究通过癌症基因组图谱集(the cancer genome atlas,TCGA)[11]公共数据,研究NRAS基因突变在黑色素瘤中的发生情况,为靶向治疗提供有价值的线索。
1 资料与方法
1.1 一般资料
选取1978-2013年TCGA数据集中收集的黑色素瘤病471例,数据的使用符合TCGA的出版要求。按NRAS基因突变情况,将471例病例资料分为两组,未突变组373例,年龄15~90岁,中位年龄59岁;突变组98例,年龄30~86岁,中位年龄56岁;性别、肿瘤位置、肿瘤分期、发病人种等临床资料见表1。
1.2 统计学方法
应用cBioPortal和SPSS 17.0统计分析软件,分析TCGA 数据集中NRAS基因突变情况,基因共表达、基因网络调控及生存分析。生存分析采用Kaplan-Meier和log-rank检验法,以P<0.05为差异有统计学意义。
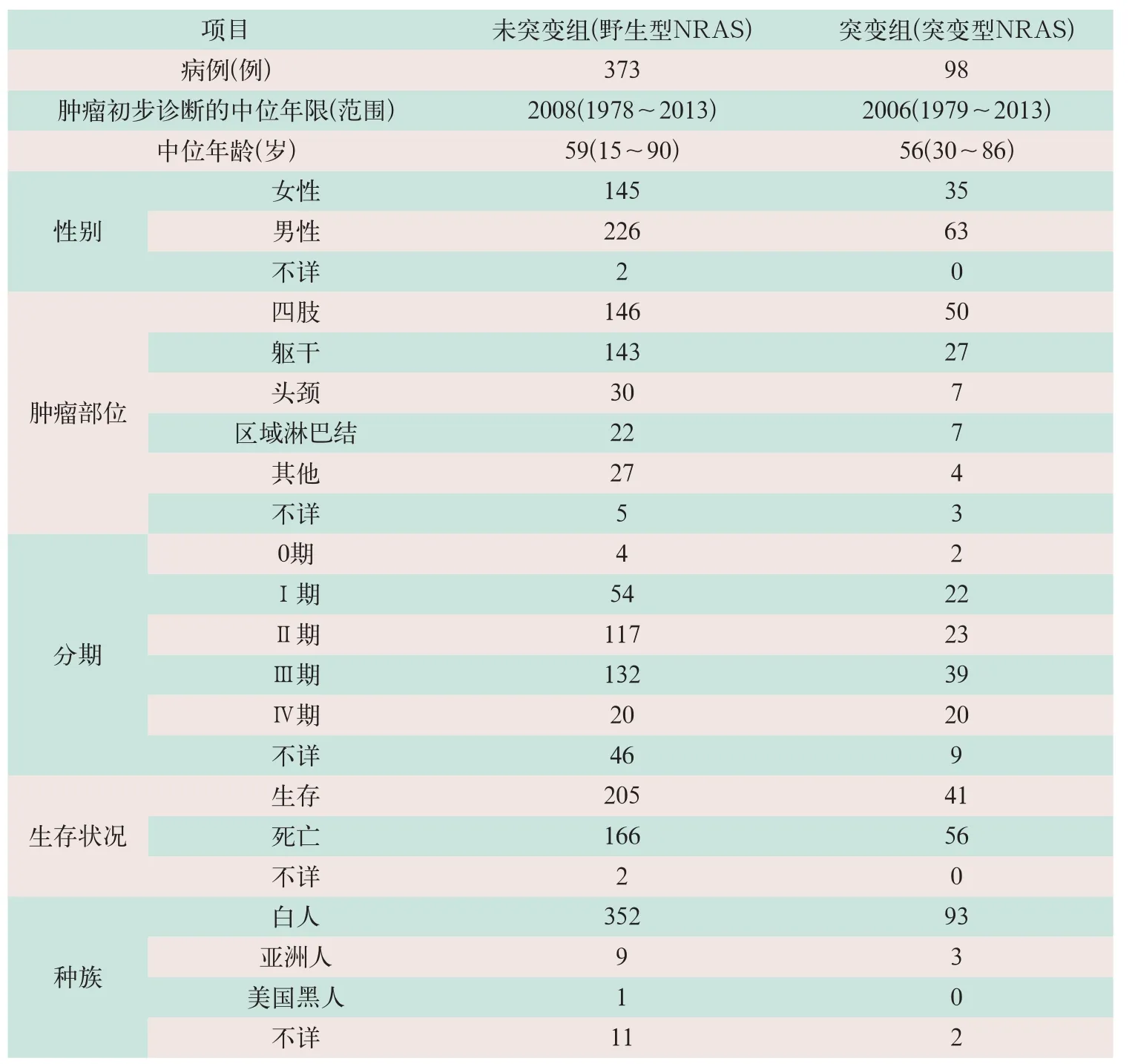
表1 471例黑色素瘤患者临床特征[例(%)]
2 结果
2.1 NRAS基因突变
在471个病例中,NRAS基因突变98例,占比20.80%(98/471);在98例NRAS基因突变病例中,突变位点以Q61R为主,共41例,占41.84%(41/98),Q61K突变31例,占31.63%(41/98),Q61L突变10例,占10.20%(10/98),Q61H突变4例,G12A突变1例,G12D突变1例,G12R突变2例,G13D突变1例,G13R突变2例,其他突变5例(见表2)。
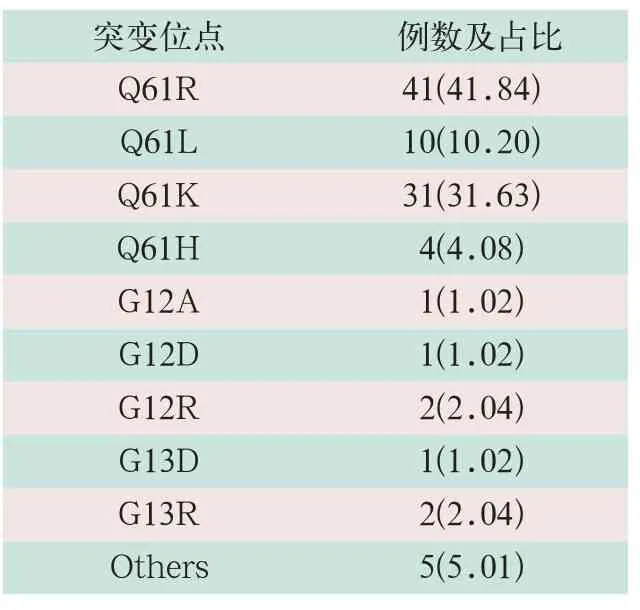
表2 98例NRAS基因突变 [例(%)]
2.2 基因共表达分析
应用cBioPortal分析黑色素瘤TCGA数据集,进行基因共表达研究,结果显示BCAS2、CSDE1、GNAT3、SIKE1基因mRNA与NRAS基因表达呈现一定程度的正相关(如图1所示)。
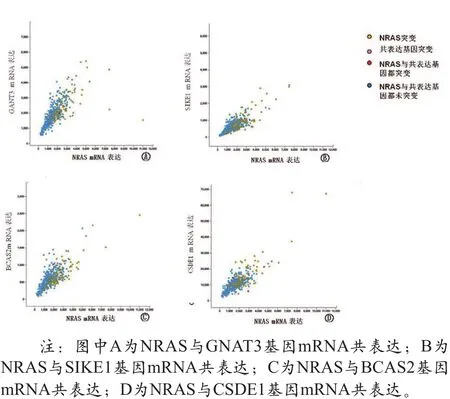
图1 NRAS 基因共表达分析示图
2.3 生存期分析
应用cBioPortal分析黑色素瘤TCGA数据集,采用Kaplan-Meier Plotter在线数据进行生存分析,结果显示,NRAS基因突变组与未突变组生存期无统计学差异,如图2所示。
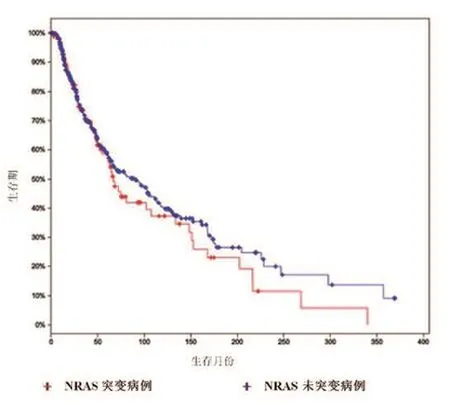
图2 黑色素瘤患者生存期分析曲线图
2.4 调控网络分析
cBioPortal整合来自人类蛋白参考数据库(human reference protein database,HPRD)、反应组学(Reactome)、国立癌症研究所自然版(National Cancer Institute,NCI)-Nature和斯隆-凯特琳癌症中心(The Memorial Sloan-Kettering Cancer Center(MSKCC) Cancer Cell Map)的数据,并对此进行分析,此调控网络中包含51节点,包含NRAS基因和其50个相关基因,分析结果如图3所示。
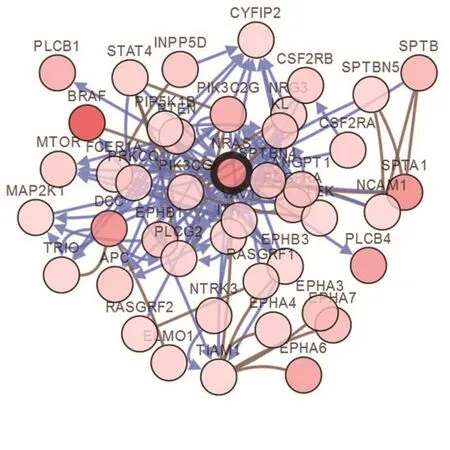
图3 调控网络分析示图
3 讨论
Ras基因家族包括NRAS基因、KRAS基因和HRAS基因,能调节细胞生长、增殖和分化。多项研究表明,NRAS基因突变与结直肠癌、黑色素瘤、急性髓细胞白血病等肿瘤的进程有着密切的联系[11-13]。本研究结果显示,在471个皮肤黑色素瘤病例中,NRAS基因突变表明,NRAS(R132)突变与黑色素瘤的发生、进展有较高程度的相关性。本研究还发现BCAS2、CSDE1、GNAT3、SIKE1等基因mRNA与NRAS基因表达呈现一定程度的正相关,其中CSDE1基因已有研究报道与黑色素瘤的侵袭、转移相关[14]。BCAS2、GNAT3、SIKE1基因与黑色素瘤的相关性还需要进一步的研究。调控网络分析结果显示,调控网络中共含51节点,包含NRAS基因和其50个相关基因,这些基因表明黑色素瘤的发生、发展与MAPK/ERK信号通路相关,此结果与Tonks等[15]研究报道一致。
目前,黑色素瘤的治疗方法以手术切除为主,同时根据其他因素结合放疗、化疗、免疫治疗等措施,而这些治疗都具有相应的局限性,探索更多的治疗方法尤为重要。有研究表明,针对一些基因突变的病例,如BRAFV600E、KIT基因突变,一些基因靶向的药物已经进入临床Ⅰ期、临床Ⅱ2期实验,并证实其有效性[16-17]。
这些研究为进一步探讨NRAS基因突变与黑色素瘤的发生奠定了一定的基础,并提示NRAS的Q61基因突变有可能成为黑色素瘤新的靶向治疗位点,为靶向治疗研究提供新的思路。
[1]乌新春,段晰所.恶性黑色素瘤的研究进展[J].承德医学院学报,2008,25(3):309-312.
[2]孔燕,梁龙,斯璐,等.43例中国人眼部恶性黑色素瘤cKit和BRAF基因突变分析[J].基础医学与临床,2012,32(2):154-157.
[3]许三雄,许建波,张晋煜.进展期恶性黑色素瘤侵袭和转移相关基因研究进展[J].现代肿瘤医学,2012,20(3):651-654.
[4]Sosman JA,Kim KB,Schuchter L,et al.Survival in BRAF V600-mutant advanced melanoma treated with vemurafenib[J].New England J Med,2012,366(366):707-714.
[5]郭芳,康晓静.BRAF、NRAS癌基因与恶性黑素瘤的研究[J].国际皮肤性病学杂志,2012,38(1):48-50.
[6]Fedorenko IV,Gibney GT,Smalley KS.NRAS mutant melanoma:biological behavior and future strategies for therapeutic management[J].Oncogene,2013,32(25):3009-3018.
[7]Singhal R,Kandel ES.The response to PAK1 inhibitor IPA3 distinguishes between cancer cells with mutations in BRAF and Ras oncogenes[J].Oncotarget,2012,3(7):700-708.
[8]Takashima A,English B,Chen Z,et al.Protein kinase Cδis a therapeutic target in malignant melanoma with NRAS mutation[J].ACS Chem Biol,2014,9(4):1003-1014.
[9]Kelleher FC,Mcarthur GA.Targeting NRAS in melanoma[J].Cancer J,2012,18(2):132.
[10]Uhara H,Ashida A,Koga H,et al.NRAS mutations in primary and metastatic melanomas of Japanese patients[J].Inter J Clin Oncol,2014,19(3):544-548.
[11]刘影,郑细闰,朱亚珍,等.252例结直肠癌组织中KRAS、NRAS、BRAF、PIK3CA的基因突变分析[J].临床与实验病理学杂志,2016,32(8):851-855.
[12]杨建刚,孙雪梅,顾香芳,等.急性髓系白血病中Nras基因点突变的检测以及信号传导通路蛋白的表达[J].现代医学,2009,37(4):273-277.
[13]刘广勇,康晓静.黑素瘤与NRAS基因研究进展[J].新疆医科大学学报,2015(2):247-249.
[14]Wurth L,Papasaikas P,Olmeda D,et al.UNR/CSDE1 drives a post-transcriptional program to promote melanoma invasion and metastasis cancer cell[J].2016,30(5):694-707.
[15]Tonks ID,Mukhopadhyay P,Schroder WA,et al.Melanocyte transformation requires complete loss of all pocket protein function via a mechanism that mitigates the need for MAPK pathway activation[J].Oncogene,2017,36(26):3789-3795.
[16]CSCO黑色素瘤专家委员会.中国黑色素瘤诊治指南(2011版)[J].临床肿瘤学杂志,2012,17(2):159-171.
[17]Montagut C,Settleman J.Targeting the RAFMEK-ERK pathway in cancer therapy[J].Cancer Lett,2009,283(2):125.

