基于转录组数据库的高羊茅HD-Zip I转录因子的鉴定及表达模式解析
庄黎丽,王剑,杨志民
(南京农业大学草业学院,江苏 南京 210095)
在干旱、高温、低温或高盐等这些逆境非生物胁迫条件下,植物中很多胁迫响应基因的转录水平发生了改变。一些转录因子在非生物胁迫响应过程中起重要作用,是称之为调节子的转录调控网络的主要组分,调控植物下游靶标基因的表达[1-2]。这些转录因子是对植物品种性状进行改良,提高其逆境胁迫下抗性的潜在工具[3-4]。例如,研究表明,过量表达脱水响应元件结合蛋白(dehydration-responsive element binding,DREBs),脱落酸响应元件结合蛋白(ABA responsive element binding factors, AREBs)和NAM, ATAF1/2, CUC2 (NACs)等转录因子能够产生抗逆性增强的转基因株系,且不影响产量[1-2]。然而,目前大部分转录因子参与非生物胁迫的功能研究仍然缺乏。
I类同源异型-亮氨酸拉链蛋白(class I homeodomain-leucine zipper, HD-Zip I)属于高等植物所特有的HD-Zip家族的一个亚类,其序列包括一个高度保守的同源异型结构域(homeodomian, HD)及一个位于HD下游的亮氨酸拉链(leucine zipper, LZ)结构域[5]。在模式植物拟南芥(Arabidopsisthaliana)和水稻(Oryzasativa)中分别有17和14个成员[5-8]。根据系统进化树,可划分为8个进化分枝[9-10]。HD-Zip I主要参与植物对生物和非生物等逆境胁迫因子的应答反应、激素和光信号应答以及器官发育调控等过程[11]。例如,拟南芥HD-Zip I因子HB6为蛋白磷酸酶ABA-insensitive(ABI)的靶标,调控脱落酸(ABA)信号响应[12],而HB7和HB12响应脱落酸信号,调控水分缺失条件下的植物生长[13]。月季(Rosachinensis)RhHB1参与ABA和乙烯对赤霉素氧化酶基因(RhGA20ox1)的抑制[14]。水稻中,HD-Zip I因子OsHOX12抑制赤霉素合成,调控水稻节间的伸长[15];OsHOX4则通过影响赤霉素的信号转导途径来调控植株高矮及分蘖数[16-17],而OsHOX22和OsHOX24调控水稻的耐旱性及耐盐性[18-19]。玉米(Zeamays)中HD-ZipI基因家族的成员Grassytillers1(GT1)抑制分蘖芽的伸长[20]。大麦(Hordeumvulgare)中Six-rowedspike1 (VRS1)则调控花序的分枝发育[21]。拟南芥中3个GT1同源基因(HB21,HB40及HB53)促进脱落酸的合成,从而抑制遮阴条件下腋芽的伸长[22]。然而,即使在模式植物水稻、拟南芥中,目前对于HD-Zip I的功能研究主要集中在δ和γ进化枝,其他分枝的较少涉及,而对于非模式植物中HD-Zip I的功能,研究更少。
高羊茅(Festucaarundinacea)为禾本科羊茅属多年生草本植物,是优良的冷季型草种,具有生长迅速、再生性强、耐湿耐热、耐瘠薄、抗病、适应性广等特点,在气候过渡地带城市绿化及人工草地建植中广泛应用[23-26]。然而,在高羊茅的整个生命周期中,经常遭遇极端高温、寒冷、干旱等逆境胁迫,制约高羊茅的生长发育,造成绿地景观效果变差或草地生产力下降。因此,培育抗旱耐极端温度的品种,有助于提高高羊茅的抗逆能力。随着分子生物学的飞速发展,转基因育种因其准确、直接、高效的优势,将成为重要的生物育种手段[27-28]。优异的基因资源则为转基因育种提供分子基础。然而长期以来,对于高羊茅等草类植物的研究多集中在种质资源收集及生理评价,对分子机理的研究比较缺乏。这主要是由于高羊茅缺乏全基因组参考序列导致的。高通量转录组测序技术的出现,为高羊茅等无参考序列物种的分子生物学研究提供了丰富的转录组数据库[29-31]。目前已有多个物种通过分析转录组数据库得到了较为理想的转录因子家族序列的信息,如菊花(Dendranthemamorifolium)和茶树(Camelliasinensis)中SBP-like、DOF、Trihelix和NAC转录因子家族的分析及鉴定[32-35]。这说明非模式植物中基于转录组数据库分析和鉴定重要的转录因子家族成为可能。基于此,本研究拟在高羊茅转录组数据库的基础上,分析和鉴定高羊茅HD-Zip I转录因子家族,并对其结构特征及干旱胁迫下的表达模式进行分析,以期为研究高羊茅中转录因子家族的功能提供一种新的思路和前期基础。
1 材料与方法
1.1 植物材料与生长条件
本研究选择经典的高羊茅品种‘Kentucky-31’(K-31)为研究材料。2017年1月,将K-31种子放置在铺两层滤纸的培养皿中,加入适量自来水,放置于4 ℃冰箱春化3 d,随后将培养皿置于光照培养箱萌发。将萌发10 d的小苗转移到人工气候室进行水培培养,一共培养9桶材料。培养条件为:白天、夜晚各12 h,白天温度25 ℃,夜晚温度20 ℃,湿度70%,光照强度为500 μmol·m-2·s-1。将小苗根颈部位以海绵包裹,种植于打孔的黑色泡沫板上,根系浸入1/2霍格兰营养液[36],并且用增氧泵通气。每隔3 d监测一次营养液pH值,每周换一次营养液。
1.2 植物处理
2017年2月,对1/2霍格兰营养液中水培培养的K-31植株进行模拟干旱处理,具体方法为:将聚乙二醇6000(polyethylene glycol, PEG6000)加入营养液中使其终浓度达到20%。短期干旱处理持续24 h,在20% PEG6000处理0, 3, 6, 12, 24 h时进行取样。长期干旱在20% PEG6000处理7及14 d时取样。取样时,采取根颈在液氮中速冻,随后保存在-70 ℃冰箱中。每个时间点都选取处理植株及相应的对照植株。对照组、短期干旱处理及长期干旱处理分别处理3桶植株,取样时均有3个生物学重复。
1.3 全长高羊茅HD-Zip I (FaHD-Zip I) cDNA的数据库搜索、克隆与测序
拟南芥HD-Zip I序列从http://www.arabidopsis.org/下载获得。水稻HD-Zip I序列根据Agalou等[37]提供的登录号,到NCBI(https://www.ncbi.nlm.nih.gov/)获得。将3个已发表[29-31]及本实验室两个未发表的高羊茅转录组数据库配置到BioEdit软件的本地数据库中。以14个水稻HD-Zip I蛋白序列作为鉴定FaHD-Zip I的请求序列,采用‘tblastn’程序,选取E-10作为期望值,搜索本地高羊茅数据库。所得序列到NCBI进一步进行blastX搜索比对,并且进行拼接组装,获得候选FaHD-Zip I序列。根据预测cDNA的5′和3′端序列设计第1轮引物,根据开放阅读框(open reading frame, ORF)设计第2轮引物。采用巢式PCR技术,通过Takara LA taq扩增FaHD-Zip I的候选目的条带。将扩增条带利用AxyPrep DNA凝胶回收试剂盒(Axygen)纯化后,连接到pMD19-T,转化大肠杆菌DH5α,经菌落PCR验证阳性克隆后送上海生工测序。
1.4 系统进化树的构建及序列分析
采用MEGA 6.0软件,用邻接法构建系统进化树。采用Compute pI/Mw在线工具预测高羊茅HD-Zip I蛋白的等电点(isoelectric point, pI)及分子量(molecular weight, Mw),采用PSORT预测亚细胞定位,采用MEME program v4.10.2预测保守结构域。MEME鉴定的所有结构域均在SMART和Pfam数据库中进行搜索。
1.5 荧光定量PCR (qRT-PCR)
采用植物RNA提取试剂盒提取样品总RNA。取1 μg RNA进行第一链cDNA的合成,所用试剂盒为PrimerScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time),反转录之前去除基因组DNA。所用定量PCR试剂为SYBR Green I Master reaction system。定量PCR的条件设置为:95 ℃预变性10 min,随后进行40个循环的PCR扩增(95 ℃变性15 s,60 ℃退火15 s,72 ℃延伸20 s)。每个循环都设定在65 ℃采集数据。内参基因为FaTublin[38],每个生物学样品都进行3次技术重复,采用2-ΔΔCT法计算基因的相对表达水平。
1.6 数据分析
采用SPSS 19.0 (SPSS Inc, Chicago, IL, USA)软件进行方差分析并用SigmaPlot 12.5 (Systat Software, Inc, San Jose, CA, USA)绘图,平均值采用最小显著差异法(LSD)进行多重比较,显著水平为P<0.05。
2 结果与分析
2.1 高羊茅HD-Zip I转录因子的鉴定及系统进化分析
采用14个水稻HD-Zip I蛋白序列(表1)作为搜索请求,在高羊茅本地转录组数据库进行搜索,经分析、组装和拼接,巢式PCR扩增(引物序列见表2),单克隆测序鉴定,得到13个高羊茅HD-Zip I全长ORF序列(表1)。基于水稻的名称(OsHOX)来命名高羊茅HD-Zip I序列(FaHOXs),并且将其登录到NCBI (GenBank登录号: MG189709~MG189721)。结果表明,13个FaHOX候选蛋白序列长度从221(FaHOX24)到345(FaHOX16)个氨基酸,等电点为4.66(FaHOX16)到6.32(FaHOX14),分子质量从24604.12(FaHOX24)到37611.29(FaHOX16)。亚细胞定位预测结果表明,这13个FaHOX基因均定位在细胞核中,与其转录因子功能相吻合。

表1 FaHD-Zip I候选基因及其水稻同源序列概况Table 1 Summary of FaHD-Zip I sequence and the identity of likely O. sativa homologs
为了评价高羊茅、水稻及拟南芥中HD-Zip I蛋白的系统进化关系,对所得高羊茅的氨基酸序列与水稻及拟南芥中蛋白进行对比分析。采用邻接法及靴值分析(1000次重复)构建系统进化树(图1)。结果表明,植物HD-Zip I家族蛋白表现出极大的多样性,可以分为8个进化分枝,13个FaHOX蛋白及14个OsHOX蛋白均分布在α、β1、δ、γ、ζ这5个进化枝,而双子叶植物拟南芥除了没有蛋白在ζ进化枝分布,其他7个进化枝均有分布。其中,β2、ε及φ1为双子叶植物拟南芥特有。此外,从进化树可以看出,所有13个FaHOX蛋白均与相应的水稻蛋白聚类在一起,表明其亲缘关系非常接近。此外,高羊茅中未找到OsHOX23的同源蛋白(表1,图1)。
2.2 FaHD-Zip I蛋白的保守结构域分析
采用MEME软件预测高羊茅及水稻HOX蛋白的保守结构域,一共鉴定到5个保守的结构域[结构域宽度设置为6~50个氨基酸残基,E值(expectation value,期望值)小于1.8×10-45],详细信息见表3。结果表明,基于保守的氨基酸结构域,可将高羊茅HOX基因划分为5个亚类。结构域1, 2, 3和4在FaHOX13, FaHOX8, FaHOX20和FaHOX4中同时出现,且排布方式一致。结构域1, 2, 3在FaHOX6, FaHOX24和FaHOX22中排布方式一致。FaHOX12和FaHOX14均只有1和2两个结构域。FaHOX21与其他蛋白明显不同,含有最多数量的结构域,单独成为一个亚族。其他HOX蛋白,如FaHOX25, FaHOX5以及FaHOX16,以结构域5开头,不同于其他亚族蛋白均是结构域1在N端。此外,结果还表明,除了β1亚族的FaHOX5/OsHOX5,高羊茅中比水稻中多一个结构域4,大部分高羊茅HOX蛋白与水稻HOX蛋白在结构域的排布方式及数量上有很好的一致性(图2)。
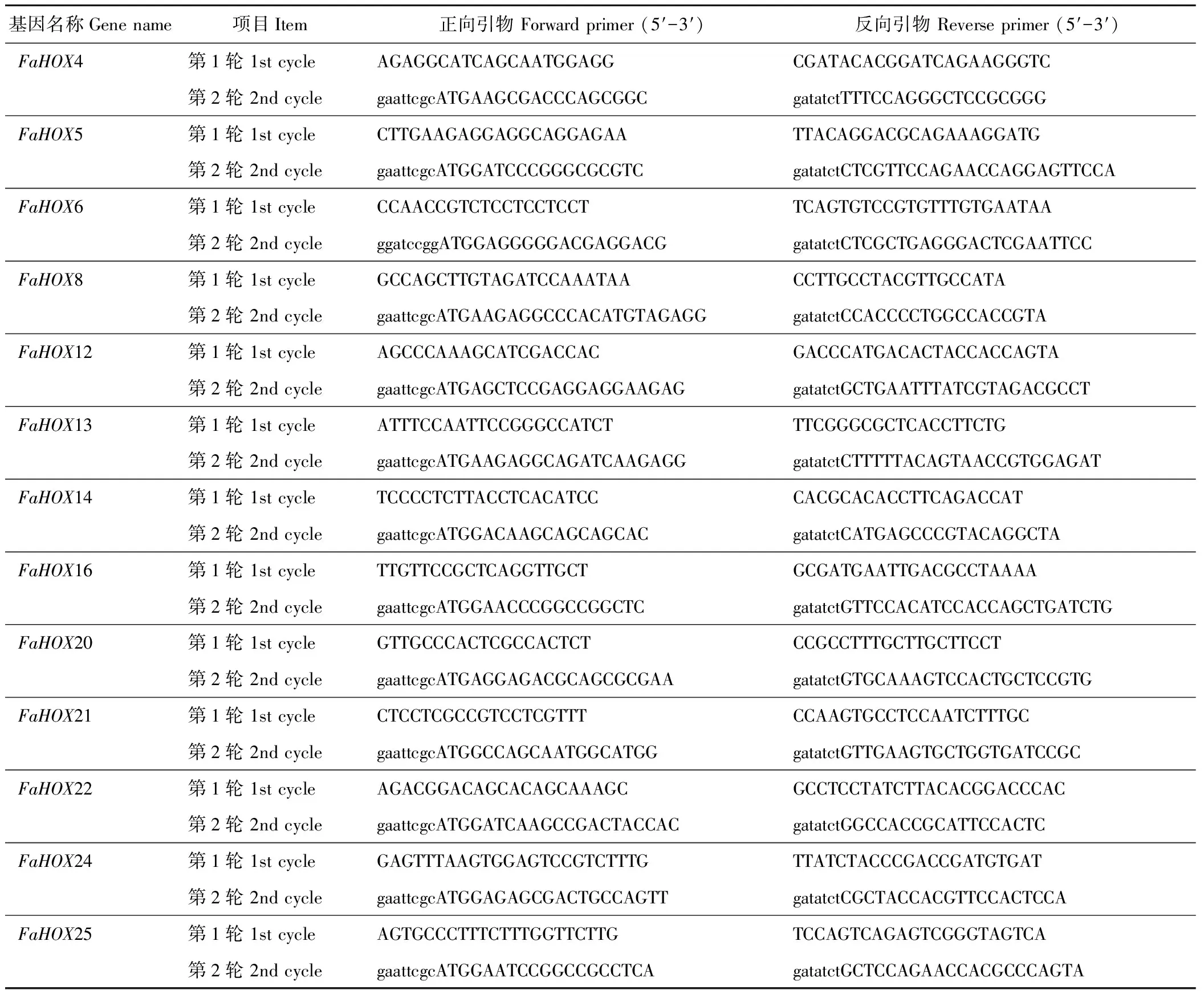
表2 FaHD-Zip I基因克隆用引物Table 2 Primers used for FaHD-Zip I cloning
2.3 短期干旱胁迫下FaHD-Zip I基因的表达
干旱胁迫是目前影响植物生长最大的逆境条件之一,研究植物中耐旱因子有着重要的意义。采用qRT-PCR技术对20% PEG6000模拟干旱处理下高羊茅根颈中HD-ZipI基因的表达进行分析。首先设计定量PCR引物(表4),产物长度108~197 bp。PCR扩增目的条带,进行琼脂糖凝胶电泳检测,FaHOX20及FaHOX21均未扩出预期条带。对其余11个基因进行qRT-PCR检测,结果表明,FaHOX4和FaHOX6的mRNA表达水平在短期24 h内均不受干旱胁迫影响。FaHOX5,FaHOX8,FaHOX14在处理24 h略微上调表达, 而FaHOX25在干旱处理24 h时下调表达。FaHOX12,FaHOX13,FaHOX16,FaHOX22,FaHOX24对干旱胁迫的响应比较快,处理3 h其表达水平均发生显著的上调或者下调。其中,FaHOX22的表达水平受到干旱胁迫的影响最大,干旱处理6 h时,其表达水平上升了约14倍(图3)。此外,从图3可以看到,几乎所有FaHOX基因在24 h内其本身的表达也发生变化,这说明其表达量可能受到昼夜节律的影响。

表3 高羊茅HD-Zip I家族蛋白主要MEME结构域Table 3 Major MEME motif sequences in tall fescue HD-Zip I protein
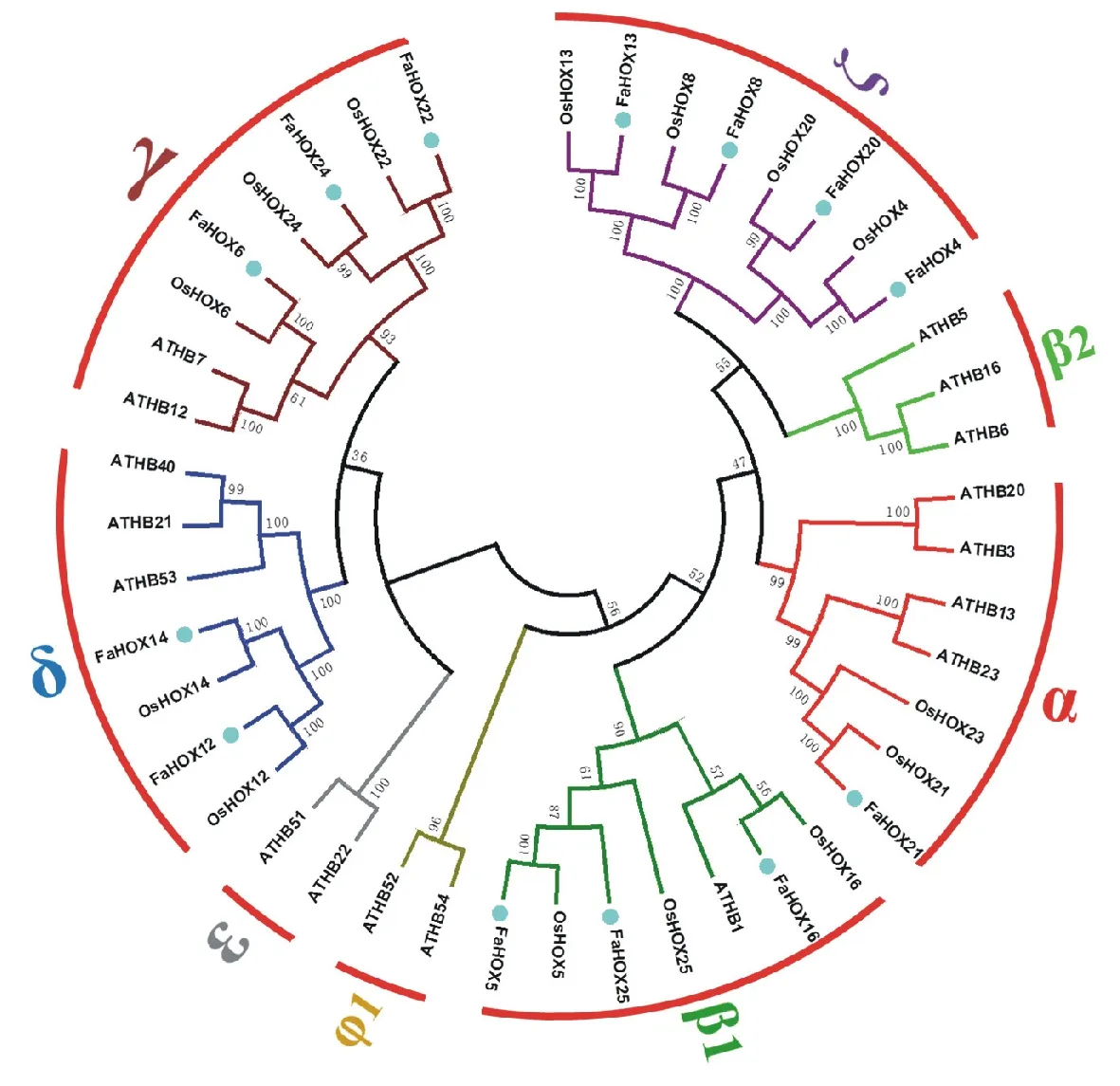
图1 高羊茅、水稻及拟南芥HD-Zip I全长氨基酸序列的系统进化树分析Fig.1 Phylogenetic tree analysis of full length amino acids of HD-Zip I in tall fescue, rice and arabidopsis
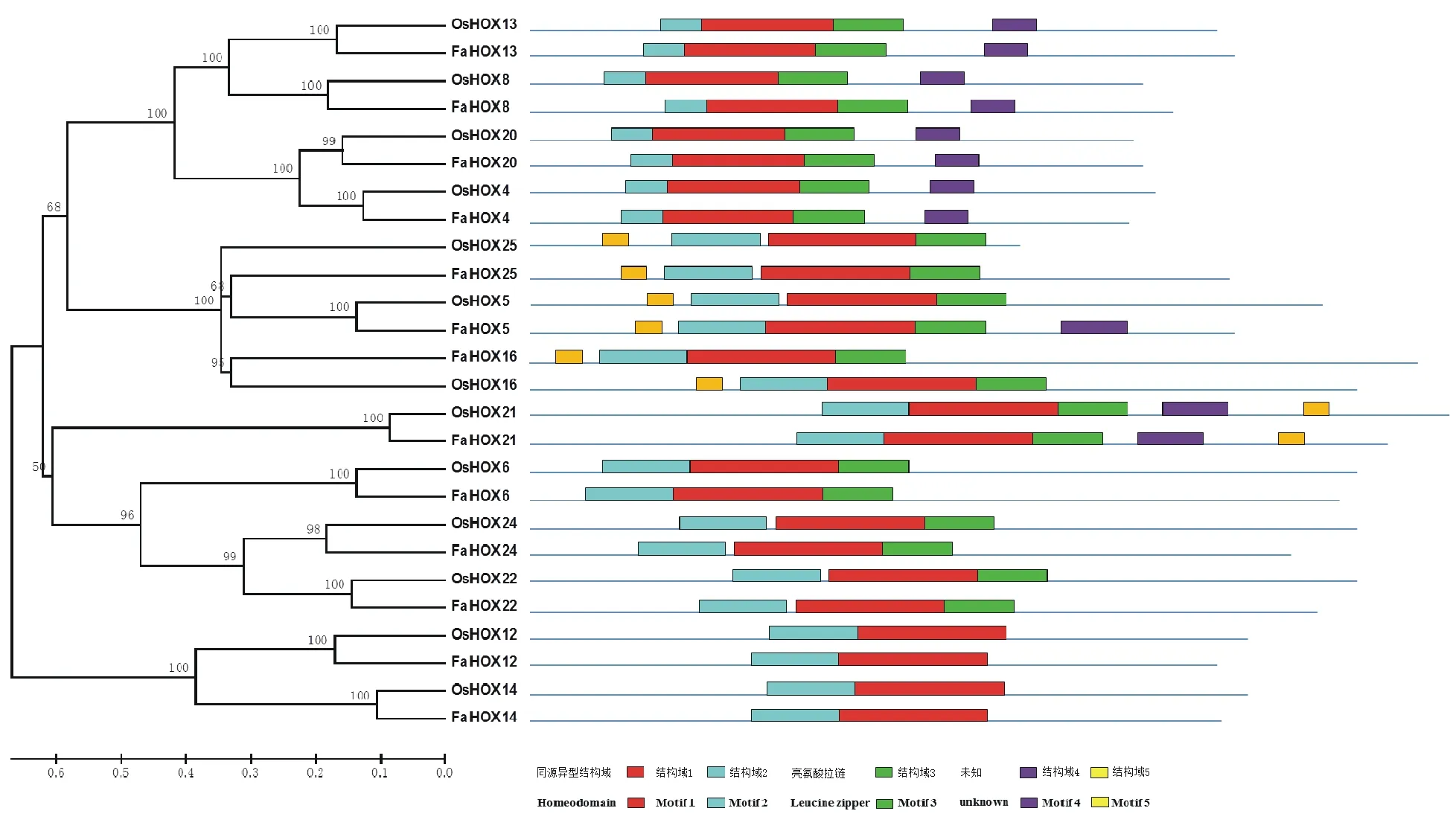
图2 高羊茅与水稻中HD-Zip I蛋白的保守结构域分析Fig.2 Distribution of conserved motifs for the tall fescue and rice HD-Zip I proteins

基因名称Genename正向引物Forwardprimer(5′-3′)反向引物Reverseprimer(5′-3′)产物长度Productlength(bp)FaHOX4AAGCTGGAGGAGGACGAGAGTGTCTCTACCAGGCCTCAT130FaHOX5CGAAGGGTGCAGTTTCCACAGGACGCAGAAAGGATGGAG184FaHOX6CAGAGTACCACCACCACCAGAAGCTTTGCAGATTGTGGCA195FaHOX8TCTTTGTACCACGGATCGCAGGATGCATGGTGGACAACAA193FaHOX12GCTACTTGATGGACTTGGCGCCCATGACACTACCACCAGT185FaHOX13AGCACGTCTCCAACCTCAATACTGTAGTACGCCCTGTCG126FaHOX14CGATGACCTGATGATGTGCGTTGAACACGCACACCTTCAG195FaHOX16GGTGGATGTGGAACTAGGCTGCGATCGATCCCAACATGAC137FaHOX20GTGTTCTTCCACGGGCATCTGCTACCTCAGTTCAGTGCA169FaHOX21CCTCCTCCGTCCCAAAGATGCAAGCCGCGACCATACATG197FaHOX22CTATCGAGTGGAATGCGGTGCGCCTCCTATCTTACACGGA161FaHOX24GTGGTAGCGTGAATCAGCGCGATGTGATCTCCGGAGTCA108FaHOX25GTGGTTCTGGAGCTGATCGACCAGTCAGAGTCGGGTAGTC111
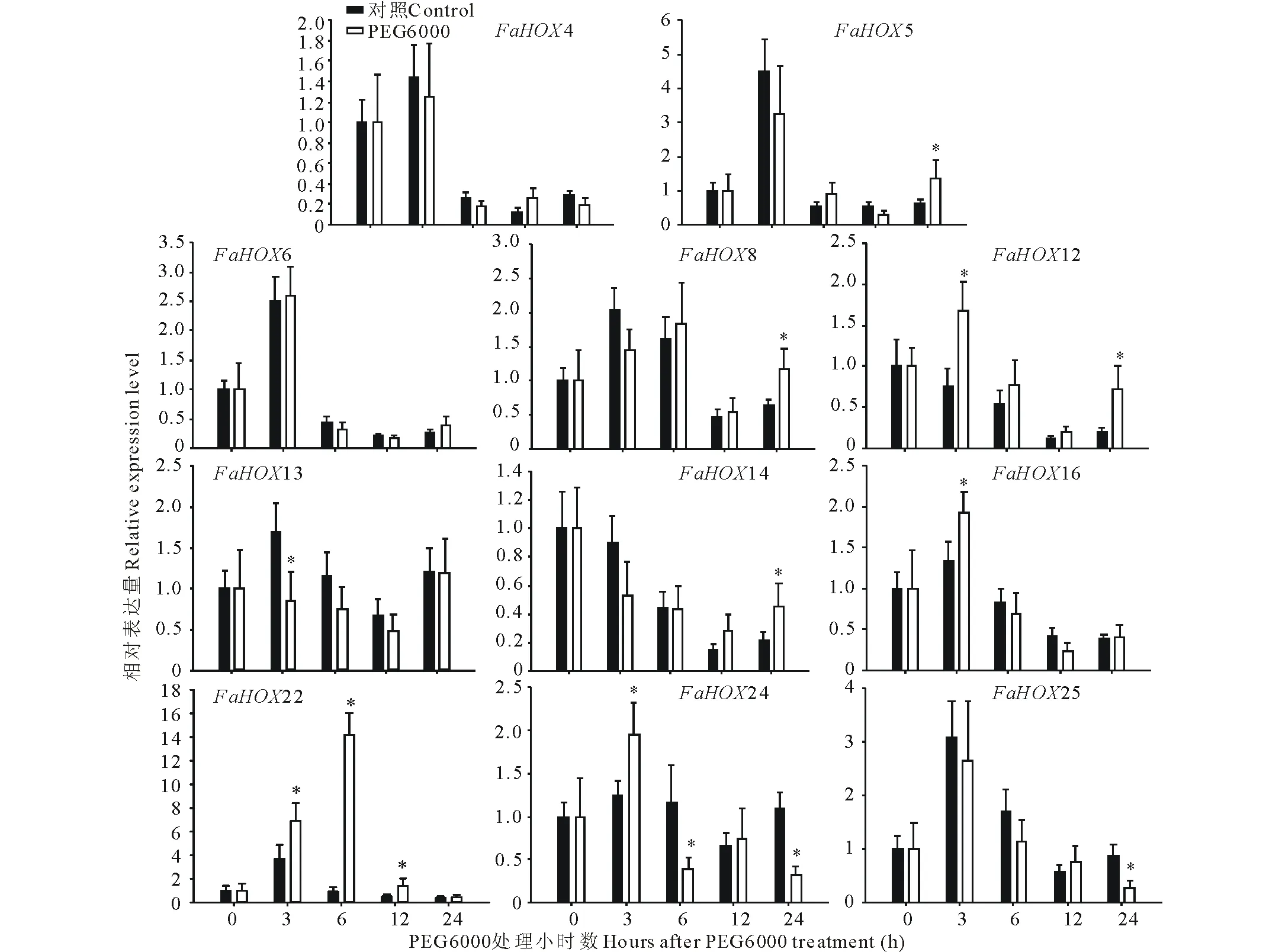
图3 短期干旱胁迫下FaHD-Zip I的qPCR分析Fig.3 PCR analysis of relative expression level of FaHD-Zip I under 24 h PEG6000 treatment 所有基因的相对表达量都以0 h对照植株根颈中基因表达量为1。柱子上部*表示相应时间点PEG6000处理与对照植株中基因表达量差异显著(P<0.05),下同。Gene expression level of control plants at 0 h was designated as 1. Stars (*) on the top of the bars indicated significant difference between the control and PEG6000-treated plants at a certain time point (P<0.05). The same below.
2.4 长期干旱胁迫下FaHD-Zip I基因的表达
进一步通过qPCR技术检测长期干旱胁迫下FaHD-ZipI的表达水平。结果表明,干旱处理7 d时,FaHOX4和FaHOX5的表达水平均显著低于对照,FaHOX8,FaHOX12,FaHOX13,FaHOX22及FaHOX25均显著上调,其他几个基因(FaHOX6,FaHOX14,FaHOX16及FaHOX24)的表达水平则不受影响。干旱处理14 d时,FaHOX6,FaHOX8,FaHOX14,FaHOX16,FaHOX22,FaHOX24及FaHOX25的表达水平均显著高于对照植株,而FaHOX4,FaHOX5,FaHOX12及FaHOX13的表达水平则与对照无显著差异(图4)。其中,干旱处理14 d时,FaHOX22的表达水平比对照上调76.3倍,比其他基因的响应更为明显。
3 讨论
3.1 转录组数据库可成为非模式植物进行分子生物学研究的有力工具
长期以来,参考基因组序列的缺乏极大地限制了非模式植物中进行分子生物学研究的步伐。高通量测序技术的出现,极大地丰富了非模式植物的转录谱信息。这不仅能够从组学水平上对特定发育时期特定器官和组织中转录发生变化的基因水平进行一个评估,同时也为分子生物学研究提供了大量的序列信息。目前已有多个基于转录组数据库对非模式植物某一类基因进行克隆与分析的研究[32-35]。本研究通过搜集他人已发表的高羊茅转录组数据库[29-31],整合自己的转录组数据库信息,结合序列搜索、比对分析、拼接组装及后期PCR克隆等步骤,较为快速地获得13个高羊茅HD-Zip I转录因子全长序列。这说明在物种转录组信息丰富的情况下,采取这种方式能够为非模式植物中基因的克隆提供很好的平台,为后续进行这些基因的功能研究打下了基础。
3.2 高羊茅中HD-Zip I转录因子的分析
目前已在不同物种中克隆了多个HD-Zip I因子并且对其功能进行了研究。本研究中,共获得13个高羊茅HD-Zip I因子,比水稻中少1个。这一方面可能是由于其物种特异性,另外一方面也可能是由于转录组数据库还没有足够丰富造成的。其他物种,如番茄(Solanumlycopersicum)中有22个HD-Zip I蛋白[9],柑橘(Citrussinensis)中16个[39],桃(Prunuspersica)中14个[40],大豆(Glycinemax)中30个[10]。由于高羊茅为异源六倍体,会存在同一个基因多个拷贝的情况,因此其实际的HD-Zip I蛋白数目必然远超过13个。这13个HD-Zip I蛋白均包括保守的同源异形盒及亮氨酸拉链结构域,且生物信息学预测均定位于细胞核,这与其转录因子的特性也是吻合的。系统进化树表明13个高羊茅HD-Zip I蛋白,14个水稻HD-Zip I蛋白及17个拟南芥HD-Zip I蛋白可以分为8个进化枝,高羊茅与水稻中对应的HD-Zip I蛋白序列更为接近,且与水稻的相应蛋白共同分布在5个进化枝,与拟南芥的有较大差异,这说明HD-Zip I蛋白在单双子叶植物中有明显的差异。
HD-Zip转录因子家族分为I~IV亚类,其中,I类的结构最简单[5]。本研究通过MEME软件分析13个高羊茅HD-Zip I蛋白的保守结构域,共发现5个结构域。高羊茅和水稻中对应蛋白在结构域数目及排布顺序方面几乎一致,且与系统进化树结果也比较吻合,也划分为5个亚类。这些结果说明高羊茅HD-Zip I蛋白的功能可能也与水稻的同源蛋白比较一致。同时,FaHOX5比OsHOX5多一个结构域,说明两个物种的HD-Zip I蛋白也有不同。这些信息为后期研究高羊茅中蛋白功能提供了借鉴意义。
总的来说,位于同一个进化枝内的同源蛋白,其功能也类似。例如拟南芥HB7和HB12对脱落酸信号作出响应,调控干旱胁迫条件下的植物生长[41],同样,其在水稻中同源蛋白OsHOX22和OsHOX24通过调控ABA合成来调节水稻的耐旱性[18-19,42]。然而,也有同一亚类内蛋白功能不同的报道。如δ亚族内单双子叶蛋白的功能明显不同,且同样单子叶植物中的同源蛋白都可能不一致。例如,目前报道OsHOX12抑制赤霉素合成,调控水稻节间的伸长[15],而玉米中OsHOX12同源基因GT1则抑制分蘖芽的伸长[20]。拟南芥中与OsHOX12同源的HB21,HB40及HB53这3个蛋白同时缺失抑制了遮阴胁迫下腋芽的伸长[22]。由此,在进化过程中,HD-Zip I蛋白的功能发生了很大歧化。另外,就目前的研究来说,其他进化枝的蛋白的分子功能如何,也值得以后的工作进一步来鉴定。
3.3 干旱胁迫下高羊茅HD-Zip I基因的表达
研究表明,HD-Zip I蛋白参与植物对生物及非生物胁迫的应答,HD-Zip I可能为激素介导植物响应逆境与自身发育的关键节点。多个研究表明HD-ZipI基因的表达水平受到逆境胁迫的显著影响[6-8,14,18,41-42]。考虑到某些基因的表达本身还有昼夜节律性,本研究中每个取样时间点均做了相应的对照。结果表明,在24 h内,大部分基因的表达都表现出节律性。在短期PEG6000模拟干旱处理下,大部分基因的表达或多或少在某个时间点发生上调或者下调表达,然而除FaHOX22以外,其余基因的表达变化倍数均不大。这与其他文献中报道的结果有差异,是由本研究设定的对照导致,且本研究的结果更为准确和有参考意义。在干旱处理7及14 d时,有多个基因表达水平比对照发生了较大程度的上调(如FaHOX6,FaHOX8,FaHOX14,FaHOX16,FaHOX22,FaHOX24及FaHOX25)。目前仅有FaHOX22和FaHOX24在拟南芥及水稻中的同源基因有较深入的研究,其余均未见报道。由此,该结果为高羊茅中这些因子的功能研究提供了参考。此外,本研究没有涉及高温、高盐等其他逆境胁迫和植物激素处理下FaHD-ZipI的表达分析,这些也需要在今后的工作完成。
4 结论
基于高羊茅本地转录组数据库,通过生物信息学分析及分子克隆技术,获得13个FaHD-ZipI全长开放阅读框,预测均定位于细胞核。系统进化树分析表明其分布在5个进化枝,与水稻的HD-ZipI基因有很高的相似性。MEME结构域分析揭示高羊茅与水稻FaHD-Zip I在保守结构域的数目及排布方式基本一致,且与系统进化树结果吻合。qRT-PCR分析FaHD-ZipI基因在短期及长期PEG6000模拟的干旱胁迫下高羊茅根颈中的表达水平,结果表明11个FaHD-ZipI均能响应干旱胁迫,但是响应快慢和响应的程度有很大区别。本研究结果为后续进行高羊茅HD-ZipI基因的功能研究奠定了工作基础。
References:
[1] Todaka D, Shinozaki K, Yamaguchi-Shinozaki K. Recent advances in the dissection of drought-stress regulatory networks and strategies for development of drought-tolerant transgenic rice plants. Frontiers in Plant Science, 2015, 6: 84.
[2] Nakashima K, Yamaguchi-Shinozaki K. Transcriptional regulatory networks in response to abiotic stresses inArabidopsisand grasses. Plant Physiology, 2009, 149(1): 88-95.
[3] Golldack D, Lüking I, Yang O. Plant tolerance to drought and salinity: stress regulating transcription factors and their functional significance in the cellular transcriptional network. Plant Cell Reports, 2011, 30(8): 1383-1391.
[4] Wang H Y, Wang H L, Shao H B,etal. Recent advances in utilizing transcription factors to improve plant abiotic stress tolerance by transgenic technology. Frontiers in Plant Science, 2016, 7: 67.
[5] Ariel F D, Manavella P A, Dezar C A,etal. The true story of the HD-Zip family. Trends in Plant Science, 2007, 12(9): 419-426.
[6] Meijer A H, de Kam R J, D’Erfurth I,etal. HD-Zip proteins of families I and II from rice: interactions and functional properties. Molecular & General Genetics, 2000, 263(1): 12-21.
[7] Jain M, Tyagi A K, Khurana J P. Genome-wide identification, classification, evolutionary expansion and expression analyses of homeobox genes in rice. The FEBS Journal, 2008, 275(11): 2845-2861.
[8] Henriksson E, Olsson A S B, Johannesson H,etal. Homeodomain leucine zipper class I genes inArabidopsis. Expression patterns and phylogenetic relationships. Plant Physiology, 2005, 139(1): 509-518.
[9] Zhang Z Z, Chen X L, Guan X,etal. A genome-wide survey of homeodomain-leucine zipper genes and analysis of cold-responsive HD-Zip I members’ expression in tomato. Bioscience, Biotechnology, and Biochemistry, 2014, 78(8): 1337-1349.
[10] Chen X, Chen Z, Zhao H L,etal. Genome-wide analysis of soybean HD-Zip gene family and expression profiling under salinity and drought treatments. PLoS One, 2014, 9(2): e87156.
[11] Brandt R, Cabedo M, Xie Y K,etal. Homeodomain leucine-zipper proteins and their role in synchronizing growth and development with the environment. Journal of Integrative Plant Biology, 2014, 56(6): 518-526.
[12] Himmelbach A, Hoffmann T, Leube M,etal. Homeodomain protein ATHB6 is a target of the protein phosphatase ABI1 and regulates hormone responses inArabidopsis. The EMBO Journal, 2002, 21(12): 3029-3038.
[13] Olsson A S, Engström P, Söderman E. The homeobox genesATHB12 andATHB7 encode potential regulators of growth in response to water deficit inArabidopsis. Plant Molecular Biology, 2004, 55(5): 663-677.
[14] Lü P T, Zhang C Q, Liu J T,etal. RhHB1 mediates the antagonism of gibberellins to ABA and ethylene during rose (Rosahybrida) petal senescence. Plant Journal, 2014, 78(4): 578-590.
[15] Gao S P, Fang J, Xu F,etal. Rice HOX12 regulates panicle exsertion by directly modulating the expression ofELONGATEDUPPERMOSTINTERNODE1. The Plant Cell, 2016, 28(3): 680-695.
[16] Zhou W, Malabanan P B, Abrigo E.OsHox4 regulates GA signaling by interacting with DELLA-like genes and GA oxidase genes in rice. Euphytica, 2015, 201(1): 97-107.
[17] Dai M Q, Hu Y F, Ma Q,etal. Functional analysis of riceHOMEOBOX4 (Oshox4) gene reveals a negative function in gibberellin responses. Plant Molecular Biology, 2008, 66(3): 289-301.
[18] Zhang S X, Haider I, Kohlen W,etal. Function of the HD-Zip I geneOshox22 in ABA-mediated drought and salt tolerances in rice. Plant Molecular Biology, 2012, 80(6): 571-585.
[19] Bhattacharjee A, Khurana J P, Jain M. Characterization of rice homeobox genes,OsHOX22 andOsHOX24, and over-expression ofOsHOX24 in transgenicArabidopsissuggest their role in abiotic stress response. Frontiers in Plant Science, 2016, 7: 627.
[20] Whipple C J, Kebrom T H, Weber A L,etal.Grassytillers1 promotes apical dominance in maize and responds to shade signals in the grasses. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(33): 506-512.
[21] Komatsuda T, Pourkheirandish M, He C,etal. Six-rowed barley originated from a mutation in a homeodomain-leucine zipper I-class homeobox gene. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(4): 1424-1429.
[22] González-Grandío E, Pajoro A, Franco-Zorrilla J M,etal. Abscisic acid signaling is controlled by aBRANCHED1/HD-ZIPIcascade inArabidopsisaxillary buds. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(2): 245-254.
[23] Sun M, Chen X F, Su X D. Introduction and application of cool season type tall fescue turfgrass cultivars in east China. Acta Agriculturae Shanghai, 1999, (1): 44-46.
孙明, 陈雪芳, 苏旭东. 冷季型草坪高羊茅草种在中国华东地区的引进和应用. 上海农业学报, 1999, (1): 44-46.
[24] Wang Z Y, Hopkins A, Mian R. Forage and turf grass biotechnology. Critical Reviews in Plant Sciences, 2001, 20(6): 573-619.
[25] Barnes R F. Biotechnology in tall fescue improvement//Kasperbauer M J. Importance and problems of tall fescue. Boca Raton, FL: CRC Press, 1990: 1-12.
[26] Yu G J, Lin Q T, Ke Q M,etal. Progress and prospects of tall fescue turfgrass. Pratacultural Science, 2005, 22(7): 77-82.
余高镜, 林奇田, 柯庆明, 等. 草坪型高羊茅的研究进展与展望. 草业科学, 2005, 22(7): 77-82.
[27] Yao N, Han L B, Zeng H M,etal. Application and development of turfgrass breeding transgenic technology. Biotechnology Bulletin, 2008, 3: 66-70.
姚娜, 韩烈保, 曾会明, 等. 草坪草转基因育种技术的应用和发展. 生物技术通报, 2008, 3: 66-70.
[28] Wang Y, Li J L, Pan Y N,etal. Research progress in transgenic breeding of excellent warm-season turfgrass. Grassland and Turf, 2007, (4): 13-17.
王艳, 李建龙, 潘永年, 等. 草坪草转基因育种研究进展. 草原与草坪, 2007, (4): 13-17.
[29] Li H Y, Hu T, Amombo E,etal. Transcriptome profilings of two tall fescue (Festucaarundinacea) cultivars in response to lead (Pb) stress. BMC Genomics, 2017, 18(1): 145.
[30] Hu T, Sun X Y, Zhang X Z,etal. An RNA sequencing transcriptome analysis of the high-temperature stressed tall fescue reveals novel insights into plant thermotolerance. BMC Genomics, 2014, 15(1): 1147.
[31] Wang Y, Dai Y, Tao X,etal. Heat shock factor genes of tall fescue and perennial ryegrass in response to temperature stress by RNA-seq analysis. Frontiers in Plant Science, 2015, 6: 1226.
[32] Song A P, Wu D, Fan Q Q,etal. Transcriptome-wide identification and expression profiling analysis ofChrysanthemumtrihelix transcription factors. International Journal of Molecular Sciences, 2016, 17(2): 198.
[33] Song A P, Gao T W, Wu D,etal. Transcriptome-wide identification and expression analysis of chrysanthemum SBP-like transcription factors. Plant Physiology and Biochemistry, 2016, 102: 10-16.
[34] Song A, Gao T, Li P,etal. Transcriptome-wide identification and expression profiling of the DOF transcription factor gene family inChrysanthemummorifolium. Frontiers in Plant Science, 2016, 7: 199.
[35] Wang Y X, Liu Z W, Wu Z J,etal. Transcriptome-wide identification and expression analysis of the NAC gene family in tea plant [Camelliasinensis(L.) O. Kuntze]. Plos One, 2016, 11: e0166727.
[36] Hoagland D R, Arnon D I. The water-culture method for growing plants without soil. California Agricultural Experiment Station Circular, 1950, 347(5406): 357-359.
[37] Agalou A, Purwantomo S, Overnas E,etal. A genome-wide survey of HD-Zip genes in rice and analysis of drought-responsive family members. Plant Molecular Biology, 2008, 66(1/2): 87-103.
[38] Zhuang L L, Wang J, Huang B R. Drought inhibition of tillering inFestucaarundinaceaassociated with axillary bud development and strigolactone signaling. Environmental and Experimental Botany, 2017, 142: 15-23.
[39] Ge X X, Liu Z, Wu X M,etal. Genome-wide identification, classification and analysis of HD-ZIP gene family in citrus, and its potential roles in somatic embryogenesis regulation. Gene, 2015, 574(1): 61-68.
[40] Zhang C H, Ma R J, Shen Z J,etal. Genome-wide analysis of the homeodomain-leucine zipper (HD-ZIP) gene family in peach (Prunuspersica). Genetics and Molecular Research, 2014, 13(2): 2654-2668.
[41] Valdés A E, Overnäs E, Johansson H,etal. The homeodomain-leucine zipper (HD-Zip) class I transcription factors ATHB7 and ATHB12 modulate abscisic acid signalling by regulating protein phosphatase 2C and abscisic acid receptor gene activities. Plant Molecular Biology, 2012, 80(4/5): 405-418.
[42] Bhattacharjee A, Sharma R, Jain M. Over-expression ofOsHOX24 confers enhanced susceptibility to abiotic stresses in transgenic rice via modulating stress-responsive gene expression. Frontiers in Plant Science, 2017, 8: 628.

