长链非编码RNA在肝细胞肝癌中的研究进展
马 杰, 张 莉, 向作林
1. 复旦大学附属中山医院放疗科,上海 200032 2. 同济大学附属东方医院放疗科,上海 200120
肝细胞肝癌(HCC)是肝癌中最常见的病理类型。在世界范围内,肝癌患者死亡率在所有癌症总死亡率中排第2位。特别是在东亚、东南亚、非洲及欧洲南部,肝癌的发病率、死亡率仍呈上升趋势[1]。中国每年肝癌发病和死亡患者约占全球的一半[2]。传统手术治疗对于部分HCC患者的病情改善有明显疗效,但相当数量的患者对治疗不耐受,远期复发进展。因此,作为未来精准治疗的关键,HCC相关调控因子成为目前研究的重点。
现代基因学研究[3]发现,人类基因组中只有不到2%的基因序列编译蛋白质,超过90%的序列可转录为非编码RNA。早期观点认为非编码RNA是翻译的“噪声”,不行使功能。近年来,随着高分辨率芯片及高通量测序技术的发展,人们发现长链非编码RNA(long noncoding RNA, lncRNA)不仅起转录干扰作用,而且是基因调控中不可或缺的部分。越来越多的研究表明,非编码RNA在疾病发展过程中起着重要作用。在HCC中,lncRNA的调控作用越发引起关注。
LncRNA是一类长度超过200nt的内源性RNA,在表观遗传水平、转录过程中发挥了重要的调控作用。根据与邻近编码RNA的位置关系,lncRNA可分为5类:正义lncRNA、反义lncRNA、双向lncRNA、内含子源性lncRNA、基因间lncRNA。LncRNA空间和组织形式上的多样性使其有多种功能[4]。在细胞核内,lncRNA可以参与染色质的相互作用、转录调控与RNA加工过程;在细胞质内,lncRNA在转录产物的修饰、翻译过程的调节与信号通路的调控中扮演重要角色。由于lncRNA长度大于200 nt,可以折叠为复杂的高级结构,其作用模式在多种情况下脱离了作为核苷酸序列的一级结构基础(与同源RNA序列直接结合),而偏向了二级或更高级结构[5]。目前,已有多篇文献报道,HCC相关的lncRNA以多种形式参与HCC进展调控,但由于lncRNA高级结构和作用形式的复杂性和未知性,其在HCC中的调控作用尚需深入探讨。本文就lncRNA在HCC中的研究进展进行综述。
1 LncRNA在HCC发生中的作用
HCC的发生涉及病毒感染、基因调控失调、遗传变异等多种因素。乙型肝炎病毒x蛋白(HBx)是乙型肝炎病毒(HBV)致癌效应的重要载体[6]。有研究[7-8]证实,lncRNA HULC在HBX诱导下表达上升;作为HBV的下游靶点,其在肝硬化、HCC发生过程中发挥了调控作用。但也有研究[9]显示,在HBV阴性患者的癌组织中,HULC依然高表达。该研究还发现,HULC可通过细胞外调节蛋白激酶(ERK)提高YB-1蛋白的磷酸化水平,使YB-1从YB-1/mRNAs复合物中释放,进而激活Cyclin D1、Cyclin E1、MMP3等致癌相关mRNA的表达,诱导HCC的发生。正常细胞的昼夜节律异常是肿瘤发生的重要诱因,CLOCK复合体参与管控细胞昼夜节律。研究[10]显示,HULC可以改变HCC细胞内CLOCK的表达模式、延长其表达周期,干扰细胞节律,促进HCC发生。以上研究说明,HULC在HCC发生过程中调控有多样性。
LncRNA HOTAIR与多个类型肿瘤的发生发展及临床预后密切相关。在HCC细胞中,FOXC1可通过结合HOTAIR上游区域而激活其表达,激活的HOTAIR继而通过占据miR-1结合位点来负性调节miR-1对下游的调控,发挥其促癌效应;miR-1还能通过直接结合HOTAIR序列来负性调控HOTAIR,构成一个负性反馈环路[11]。Bmi-1是miR-218的下游靶点,作为原癌基因在肿瘤形成与肿瘤干细胞特性的维持中发挥重要作用[12-14]。Fu等[15]研究发现,HOTAIR通过招募EZH2于miR-218启动子位点来沉默其表达,增强下游Bmi-1编译,从而抑制P14ARF和P16Ink4a 信号通路,促进HCC形成。HOTAIR还可抑制CREB、P300、RNA polⅡ募集于SETD2的启动子位点,降低SETD2的表达和磷酸化水平,诱导HCC干细胞的分化[16]。HOTAIR在肝肿瘤干细胞特性的维持和分化方面具有重要意义。
单核苷酸多态性(SNP)是人类可遗传变异中最常见的一种,lncRNA相关SNP参与HCC的发生。在中国人群中,rs920778C > T多态性与HOTAIR的高表达相关,影响HCC的易感性和增殖能力[17];在中国汉族人群中,rs145204276多态性与HCC易感性相关,rs145204276多态性可能通过干预lncRNA GAS5启动子区甲基化水平而影响其对HCC细胞凋亡的抑制作用,进而促进HCC的发生[18]。
2 LncRNA在HCC发展中的调节
HCC的发展主要包括肿瘤细胞的增殖和转移。在HCC发展过程中,肿瘤血管生成是维持其高代谢水平的基础,鞘氨醇激酶1(Sphk1)参与这一过程。HULC通过竞争性隔离miRNA-107的作用,上调E2F1表达水平,促进SPHK1的表达,进而调控肿瘤血管形成[19]。脂质代谢相关因子已被证实与肿瘤的转移起始有很强的相关性[20],而HULC可以通过HULC/miR-9/PPARA/ACSL1/cholesterol/RXRA正反馈环路扰乱HCC细胞的正常脂质代谢[21]。因此,HULC可能通过脂质代谢相关途径参与HCC的转移。在缺氧状态下,HCC细胞中组蛋白H3、H4的乙酰化水平会下降,lncRNA LET的表达也随之受到抑制;同时,下调的lncRNA LET会通过干扰低氧诱导因子-1α(HIF-1α)mRNA的翻译来影响HIF-1α的表达量和稳定性,从而诱发缺氧相关的癌细胞转移[22]。
肿瘤干细胞自我更新行为在肿瘤的转移、复发以及异质性形成过程中具有重要意义。LncTCF7是一种在HCC和肝肿瘤干细胞中高表达的lncRNA,目前发现其在肝肿瘤干细胞的自我更新和肿瘤扩散过程中有调控作用。LncTCF7通过募集SWI-SNF复合体于TCF7的启动子上,激活Wnt通路来促进HCC的发展[23]。LncBRM同样在HCC细胞及肿瘤干细胞中高表达,可以与BRM蛋白交联以启动BRG1/BRM交换,从而通过BRG1嵌入式BAF复合体来激活YAP1通路,使HCC干细胞维持自我更新和HCC发生[24]。除此之外,lncRNA DANCR与miRNA竞争性结合CTNNB1 的mRNA序列,通过调控CTNNB1表达来调控影响HCC干细胞特性[25]。
表观遗传学改变在HCC发展中同样具有重要作用。据报道,在亚砷酸盐诱导形成的HCC细胞内,lncRNA MALAT1与HIF-2α显著高表达并能增强肿瘤细胞的转移侵袭能力。其中,MALAT1可以通过泛素-蛋白酶体途径增强HIF-2α的表达;反之,HIF-2α对MALAT1具有转录调节作用,两者构成一个反馈环路在HCC发展中发挥作用[26]。LncRNA GIHCG通过募集EZH2及DNMT1来上调miR-200b/a/429启动子区域组蛋白H3K27的三甲基化和DNA甲基化水平,进而沉默miR-200b/a/429表达,促进HCC细胞的增殖和转移[27]。LncRNA PCAT-14通过诱导miR-372启动子甲基化而沉默miR-372的表达,从而调控ATAD2和Hedgehog通路表达水平,促进HCC细胞的增殖和侵袭[28]。
大多数基因组拷贝数变异(CNVs)是基因组不稳定造成的,因此一部分CNVs具有致癌效应。癌细胞中多种CNVs位于非编码区域,与lncRNA异常表达相关。在HCC中,lncRNA PRAL存在高频的拷贝数缺失,而其通过结合HSP90蛋白促进HSP90与p53的结合,从而提高p53核内表达,促进肿瘤细胞的凋亡,发挥抑癌作用[29]。同样,lncRNA TSLNC8在HCC中存在拷贝数缺失,而TSLNC8可通过与TKT和STAT3竞争,并诱导IL-6/STAT3通路失活,发挥抑癌作用[30]。LncRNA PRAL和TSLNC8拷贝数变异参与HCC的发生与发展。
研究[31]发现,一部分lncRNA是其他类型非编码RNA的来源,与miRNA关系密切。LncRNA H19是miR-675的前体,而miR-675在HCC细胞的多种生物行为中扮演重要角色,比如通过Rb蛋白影响HCC细胞增殖,通过Twist1调节间充质-上皮细胞转化(MET)过程等,干预HCC发展[32]。除此之外,H19在HCC细胞中的促增殖作用还可以通过抑制PHB1表达来实现,PHB1通过与CTCF的作用可以反向调控H19的表达。H19与PHB1构成的作用环路在HCC中发挥作用[33]。
以往认为恶性肿瘤转移是肿瘤发展的晚期阶段,但越来越多的证据表明,在肿瘤发展的早期,甚至原发灶肿瘤未形成时,肿瘤细胞可能已经发生了转移[34]。上皮细胞-间充质转化(EMT)是上皮细胞通过特定程序转化为具有间质表型细胞的过程,与肿瘤的转移侵袭行为密切相关。目前发现部分lncRNAs参与这一过程,详见表1。
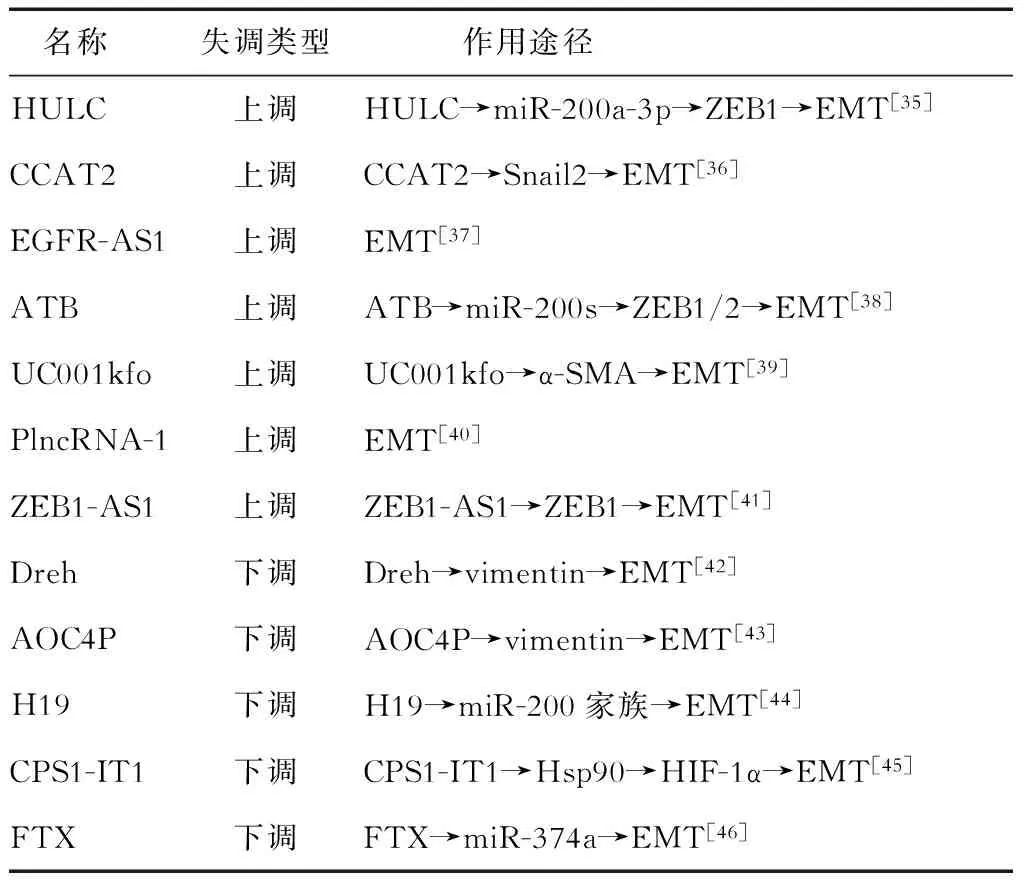
表1 通过EMT增强HCC细胞侵袭转移能力的lncRNA
3 LncRNA在HCC预后中的价值
HCC的预后相关指标与lncRNA的表达关系密切。Yuan等[47]在215例HCC患者中发现,癌组织中的MVIH高表达与无复发生存率(RFS,P<0.001) 和总存活率(OS,P=0.007)较低明显相关,而且组织中MVIH高表达是患者低RFS的独立危险因素。研究[48]通过分析110例接受肝移植术后HCC患者的临床数据发现,HCC组织中HOTAIR高表达可作为HCC复发的独立预后因素[风险比(HR)=3.564,P=0.001];尤其是在米兰标准所定义的适合行肝移植术指标范围以外的患者中,HOTAIR高表达组RFS患者更低。Yang等[49]在240例HCC患者中发现,HULC异常高表达与OS(HR=0.885,P=0.023)和无病生存率[DFS, HR=0.913,P=0.045)]相关;H19过表达是HCC患者DFS的危险因素(HR=1.071,P=0.022),在HBV阳性的HCC患者中,H19呈现高表达趋势(OR=1.14,P=0.037)。MEG3高表达同样与HCC的预后相关。研究[50]入组的72例HCC病例中,MEG3高表达组DFS、OS优于低表达组(P<0.05),其表达量可以作为HCC患者的独立预后因素(P=0.038)。研究[41]通过比较102例HCC组织和21例正常肝组织,发现ZEB1-AS1高表达者OS(P=0.017)和DFS(P=0.013)降低,说明ZEB1-AS1异常表达是患者生存率的独立预测因素。
4 LncRNA在HCC治疗中的前景
肿瘤细胞的自噬现象与肿瘤耐药性的关系是目前研究的热点。前期已有学者发现肿瘤细胞的自噬与化疗敏感性形成有关[51]。转录因子SP对HULC具有调控作用,HULC可通过稳定Sirt1来诱导自噬行为,削弱HCC的化疗敏感性[52]。因此,二甲双胍这类可降低SP及其管控基因表达的药物具有增强化疗效果的潜力[53]。HOTAIR在HCC中可以通过上调ATG3和ATG7的水平诱导自噬的发生[54]。另外,研究[55]还发现,部分基于胞外囊泡进行转运的lncRNA与HCC化疗敏感性的改变关系密切,如lincRNA-ROR。lncARSR通过胞外囊泡向周围组织传递肾癌耐药性改变的信号[56]。LncRNA对HCC细胞化疗敏感性的影响也可能通过胞外囊泡形式传递给周围癌细胞,HCC细胞中经胞外囊泡转运的lncRNA如TUC339,同样可以改变肿瘤表型[57]。
以外源lncRNA对相关调控通路进行干扰也是有潜力的治疗手段。研究[58]利用MS2病毒样颗粒(VLPs)与GE11多肽交联,成功构建了MEG3运载体,其经网格蛋白介导的内吞作用,靶向作用于EGFR阳性的HCC细胞。外源导入的MEG3在胞内通过增强p53及下游GDF15的表达,降低MDM2表达,进而抑制HCC细胞的增殖。研究结果显示了lncRNA与生物工程结合应用于HCC治疗的潜在价值。
近年来,免疫治疗成为肿瘤学治疗领域的热点,是癌症治疗探索过程中的里程碑。LncRNA在免疫调控方面的重要作用显示其在免疫治疗中的应用价值[59],如lncEGFR特异性结合EGFR,通过阻断其与c-CBL的相互作用及随后的泛素化来稳定EGFR的表达,引起调节性T细胞的分化、细胞毒性T细胞的抑制以及HCC的发展[60]。通过靶向调节lncRNA来协助免疫治疗具有较高的潜力。
5 总结和展望
在中国,HCC的发病率和死亡率非常高,故针对HCC相关的研究显得尤为迫切。前期的研究成果和临床新技术已在提高HCC疗效方面取得了较为满意的效果,但不同生理状况和病理类型等个体化因素对疗效和预后影响很大,故目前对于HCC的治疗正向个体化、精准化的方向发展。LncRNA作为在肿瘤发展过程中起广泛调控作用的因子,是个体化精准治疗的重要一环。虽然目前对lncRNA在HCC中的作用还未充分了解,但最新发现的lncRNA TERRA与端粒酶之间的密切联系和对端粒长度的监控作用显示出lncRNA在肿瘤领域作用的深度[61- 62]。因此,lncRNA在HCC组织中的调控作用是今后研究的重点,也是临床检测、诊断和治疗的潜在靶点。
[ 1 ] BERTUCCIO P, TURATI F, CARIOLI G, et al. Global trends and predictions in hepatocellular carcinoma mortality[J]. J Hepatol, 2017,67(2): 302-309.
[ 2 ] TORRE L A, BRAY F, SIEGEL R L, et al. Global cancer statistics, 2012[J]. CACancer J Clin, 2015,65(2):87-108.
[ 3 ] ESTELLER M. Non-coding RNAs in human disease[J]. Nat Rev Genet, 2011,12(12):861-874.
[ 4 ] MA L, BAJIC V B, ZHANG Z. On the classification of long non-coding RNAs[J]. RNA Biol, 2014,10(6):924-933.
[ 5 ] GUTTMAN M, RINN J L. Modular regulatory principles of large non-coding RNAs[J]. Nature, 2012,482(7385):339-346.
[ 6 ] ZHANG X D, WANG Y, YE L H. Hepatitis B virus X protein accelerates the development of hepatoma[J]. Cancer Biol Med, 2014,11(3):182-190.
[ 7 ] ZHAO J, FAN Y, WANG K, et al. LncRNA HULC affects the differentiation of Treg in HBV-related liver cirrhosis[J]. Int Immunopharmacol, 2015,28(2):901-905.
[ 8 ] Du Y, KONG G, YOU X, et al. Elevation of highly up-regulated in liver cancer (HULC) by hepatitis B virus X protein promotes hepatoma cell proliferation via down-regulating p18[J]. J Biol Chem, 2012,287(31):26302-26311.
[ 9 ] LI D, LIU X, ZHOU J, et al. Long noncoding RNA HULC modulates the phosphorylation of YB-1 through serving as a scaffold of extracellular signal-regulated kinase and YB-1 to enhance hepatocarcinogenesis[J]. Hepatology, 2017,65(5):1612-1627.
[10] CUI M, ZHENG M, SUN B, et al. A long noncoding RNA perturbs the circadian rhythm of hepatoma cells to facilitate hepatocarcinogenesis[J]. Neoplasia, 2015,17(1):79-88.
[11] SU D N, WU S P, CHEN H T, et al. HOTAIR, a long non-coding RNA driver of malignancy whose expression is activated by FOXC1, negatively regulates miRNA-1 in hepatocellular carcinoma[J]. Oncol Lett, 2016,12(5):4061-4067.
[12] JIANG L, LI J, SONG L. Bmi-1, stem cells and cancer[J]. Acta Biochim Biophys Sin (Shanghai), 2009,41(7):527-534.
[13] XU C R, LEE S, HO C, et al. Bmi1 functions as an oncogene independent of Ink4A/Arf repression in hepatic carcinogenesis[J]. Mol Cancer Res, 2009,7(12):1937-1945.
[14] CHIBA T, MIYAGI S, SARAYA A, et al. The polycomb gene product BMI1 contributes to the maintenance of tumor-initiating side population cells in hepatocellular carcinoma[J]. Cancer Res, 2008,68(19):7742-7749.
[15] FU W M, ZHU X, WANG W M, et al. Hotair mediates hepatocarcinogenesis through suppressing miRNA-218 expression and activating P14 and P16 signaling[J]. J Hepatol, 2015,63(4):886-895.
[16] LI H, AN J, WU M, et al. LncRNA HOTAIR promotes human liver cancer stem cell malignant growth through downregulation of SETD2[J]. Oncotarget, 2015,6(29):27847-27864.
[17] LI H, TANG X M, LIU Y, et al. Association of functional genetic variants of HOTAIR with hepatocellular carcinoma (HCC) susceptibility in a Chinese population[J]. Cell Physiol Biochem, 2017,44(2):447-454.
[18] TAO R, HU S, WANG S, et al. Association between indel polymorphism in the promoter region of lncRNA GAS5 and the risk of hepatocellular carcinoma[J]. Carcinogenesis, 2015,36(10):1136-1143.
[19] LU Z, XIAO Z, LIU F, et al. Long non-coding RNA HULC promotes tumor angiogenesis in liver cancer by up-regulating sphingosine kinase 1 (SPHK1)[J]. Oncotarget, 2016,7(1):241-254.
[20] PASCUAL G, AVGUSTINOVA A, MEJETTA S, et al. Targeting metastasis-initiating cells through the fatty acid receptor CD36[J]. Nature, 2017,541(7635):41-45.
[21] CUI M, XIAO Z, WANG Y, et al. Long noncoding RNA HULC modulates abnormal lipid metabolism in hepatoma cells through an miR-9-mediated RXRA signaling pathway[J]. Cancer Res, 2015,75(5):846-857.
[22] YANG F, HUO X S, YUAN S X, et al. Repression of the long noncoding RNA-LET by histone deacetylase 3 contributes to hypoxia-mediated metastasis[J]. Mol Cell, 2013,49(6):1083-1096.
[23] WANG Y, HE L, Du Y, et al. The long noncoding RNA lncTCF7 promotes self-renewal of human liver cancer stem cells through activation of Wnt signaling[J]. Cell Stem Cell, 2015,16(4):413-425.
[24] ZHU P, WANG Y, WU J, et al. LncBRM initiates YAP1 signalling activation to drive self-renewal of liver cancer stem cells[J]. Nat Commun, 2016,7:13608.
[25] YUAN S X, WANG J, YANG F, et al. Long noncoding RNA DANCR increases stemness features of hepatocellular carcinoma by derepression of CTNNB1[J]. Hepatology, 2016,63(2):499-511.
[26] LUO F, SUN B, LI H, et al. A MALAT1/HIF-2alpha feedback loop contributes to arsenite carcinogenesis[J]. Oncotarget, 2016,7(5):5769-5787.
[27] SUI C J, ZHOU Y M, SHEN W F, et al. Long noncoding RNA GIHCG promotes hepatocellular carcinoma progression through epigenetically regulating miR-200b/a/429[J]. J Mol Med (Berl), 2016,94(11):1281-1296.
[28] WANG Y, HU Y, WU G, et al. Long noncoding RNA PCAT-14 induces proliferation and invasion by hepatocellular carcinoma cells by inducing methylation of miR-372[J]. Oncotarget, 2017,8(21):34429-34441.
[29] ZHOU C C, YANG F, YUAN S X, et al. Systemic genome screening identifies the outcome associated focal loss of long noncoding RNA PRAL in hepatocellular carcinoma[J]. Hepatology, 2016,63(3):850-863.
[30] ZHANG J, LI Z, LIU L, et al. Long noncoding RNA TSLNC8 is a tumor suppressor that inactivates the interleukin-6/STAT3 signaling pathway[J]. Hepatology, 2018,67(1):171-187.
[31] HUANG M S, ZHU T, LI L, et al. LncRNAs and circRNAs from the same gene: masterpieces of RNA splicing[J]. Cancer Lett, 2017,415:49-57.
[32] HERNANDEZ J M, ELAHI A, CLARK C W, et al. MiR-675 mediates downregulation of Twist1 and Rb in AFP-secreting hepatocellular carcinoma[J]. Ann Surg Oncol, 2013,20Suppl 3:S625-S635.
[33] RAMANI K, MAVILA N, KO K S, et al. Prohibitin 1 regulates the H19-Igf2 axis and proliferation in hepatocytes[J]. J Biol Chem, 2016,291(46):24148-24159.
[34] HUSEMANN Y, GEIGL J B, SCHUBERT F, et al. Systemic spread is an early step in breast cancer[J]. Cancer Cell, 2008,13(1):58-68.
[35] LI S P, XU H X, YU Y, et al. LncRNA HULC enhances epithelial-mesenchymal transition to promote tumorigenesis and metastasis of hepatocellular carcinomaviathe miR-200a-3p/ZEB1 signaling pathway[J]. Oncotarget, 2016,7(27):42431-42446.
[36] XU Y, WANG B, ZHANG F, et al. Long non-coding RNA CCAT2 is associated with poor prognosis in hepatocellular carcinoma and promotes tumor metastasis by regulating snail2-mediated epithelial-mesenchymal transition[J]. Onco Targets Ther, 2017,10:1191-1198.
[37] QI H L, LI C S, QIAN C W, et al. The long noncoding RNA, EGFR-AS1, a target of GHR, increases the expression of EGFR in hepatocellular carcinoma[J]. Tumour Biol, 2016,37(1):1079-1089.
[38] YUAN J H, YANG F, WANG F, et al. A long noncoding RNA activated by TGF-beta promotes the invasion-metastasis cascade in hepatocellular carcinoma[J]. Cancer Cell, 2014,25(5):666-681.
[39] PAN Y, QIN T, YIN S, et al. Long non-coding RNA UC001kfo promotes hepatocellular carcinoma proliferation and metastasis by targeting alpha-SMA[J]. Biomed Pharmacother, 2017,87:669-677.
[40] DONG L, NI J, HU W, et al. Upregulation of long non-coding RNA plncRNA-1 promotes metastasis and induces epithelial-mesenchymal transition in hepatocellular carcinoma[J]. Cell Physiol Biochem, 2016,38(2):836-846.
[41] LI T, XIE J, SHEN C, et al. Upregulation of long noncoding RNA ZEB1-AS1 promotes tumor metastasis and predicts poor prognosis in hepatocellular carcinoma[J]. Oncogene, 2016,35(12):1575-1584.
[42] HUANG J F, GUO Y J, ZHAO C X, et al. Hepatitis B virus X protein (HBx)-related long noncoding RNA (lncRNA) down-regulated expression by HBx (Dreh) inhibits hepatocellular carcinoma metastasis by targeting the intermediate filament protein vimentin[J]. Hepatology, 2013,57(5):1882-1892.
[43] WANG T H, LIN Y S, CHEN Y, et al. Long non-coding RNA AOC4P suppresses hepatocellular carcinoma metastasis by enhancing vimentin degradation and inhibiting epithelial-mesenchymal transition[J]. Oncotarget, 2015,6(27):23342-23357.
[44] ZHANG L, YANG F, YUAN J H, et al. Epigenetic activation of the miR-200 family contributes to H19-mediated metastasis suppression in hepatocellular carcinoma[J]. Carcinogenesis, 2013,34(3):577-586.
[45] WANG T H, YU C C, LIN Y S, et al. Long noncoding RNA CPS1-IT1 suppresses the metastasis of hepatocellular carcinoma by regulating HIF-1alpha activity and inhibiting epithelial-mesenchymal transition[J]. Oncotarget, 2016,7(28):43588-43603.
[46] LIU F, YUAN J H, HUANG J F, et al. Long noncoding RNA FTX inhibits hepatocellular carcinoma proliferation and metastasis by binding MCM2 and miR-374a[J]. Oncogene, 2016,35(41):5422-5434.
[47] YUAN S X, YANG F, YANG Y, et al. Long noncoding RNA associated with microvascular invasion in hepatocellular carcinoma promotes angiogenesis and serves as a predictor for hepatocellular carcinoma patients’ poor recurrence-free survival after hepatectomy[J]. Hepatology, 2012,56(6):2231-2241.
[48] YANG Z, ZHOU L, WU L M, et al. Overexpression of long non-coding RNA HOTAIR predicts tumor recurrence in hepatocellular carcinoma patients following liver transplantation[J]. Ann Surg Oncol, 2011,18(5):1243-1250.
[49] YANG Z, LU Y, XU Q, et al. HULC and H19 played different roles in overall and disease-free survival from hepatocellular carcinoma after curative hepatectomy: a preliminary analysis from gene expression omnibus[J]. Dis Markers, 2015,2015:191029.
[50] ZHUO H, TANG J, LIN Z, et al. The aberrant expression of MEG3 regulated by UHRF1 predicts the prognosis of hepatocellular carcinoma[J]. Mol Carcinog, 2016,55(2):209-219.
[51] AMARAVADI R K, LIPPINCOTT-SCHWARTZ J, YIN X M, et al. Principles and current strategies for targeting autophagy for cancer treatment[J]. Clin Cancer Res, 2011,17(4):654-666.
[52] XIONG H, NI Z, HE J, et al. LncRNA HULC triggers autophagy via stabilizing Sirt1 and attenuates the chemosensitivity of HCC cells[J]. Oncogene, 2017,36(25):3528-3540.
[53] GANDHY S U, IMANIRAD P, JIN U H, et al. Specificity protein (Sp) transcription factors and metformin regulate expression of the long non-coding RNA HULC[J]. Oncotarget, 2015,6(28):26359-26372.
[54] YANG L, ZHANG X, LI H, et al. The long noncoding RNA HOTAIR activates autophagy by upregulating ATG3 and ATG7 in hepatocellular carcinoma[J]. Mol Biosyst, 2016,12(8):2605-2612.
[55] TAKAHASHI K, YAN I K, KOGURE T, et al. Extracellular vesicle-mediated transfer of long non-coding RNA ROR modulates chemosensitivity in human hepatocellular cancer[J]. FEBS Open Bio, 2014,4:458-467.
[56] QU L, DING J, CHEN C, et al. Exosome-transmitted lncARSR promotes sunitinib resistance in renal cancer by acting as a competing endogenous RNA[J]. Cancer Cell, 2016,29(5):653-668.
[57] KOGURE T, YAN I K, LIN W L, et al. Extracellular vesicle-mediated transfer of a novel long noncoding RNA TUC339: a mechanism of intercellular signaling in human hepatocellular cancer[J]. Genes Cancer, 2013,4(7-8):261-272.
[58] CHANG L, WANG G, JIA T, et al. Armored long non-coding RNA MEG3 targeting EGFR based on recombinant MS2 bacteriophage virus-like particles against hepatocellular carcinoma[J]. Oncotarget, 2016,7(17):23988-24004.
[59] ATIANAND M K, CAFFREY D R, FITZGERALD K A. Immunobiology of long noncoding RNAs[J]. Annu Rev Immunol, 2017,35:177-198.
[60] JIANG R, TANG J, CHEN Y, et al. The long noncoding RNA lnc-EGFR stimulates T-regulatory cells differentiation thus promoting hepatocellular carcinoma immune evasion[J]. Nat Commun, 2017,8:15129.
[61] RIPPE K, LUKE B. TERRA and the state of the telomere[J]. Nat Struct Mol Biol, 2015,22(11):853-858.
[62] ROAKE C M, ARTANDI S E. Approaching TERRA firma: genomic functions of telomeric noncoding RNA[J]. Cell, 2017,170(1):8-9.

