中国荷斯坦牛CSN3基因多态性及其与泌乳性状的关联性
胡 言,桂林生,余横伟,Haider Abbas Raza,昝林森,2(西北农林科技大学 动物科技学院,陕西 杨凌7200;2现代牛业生物技术与应用国家地方联合工程研究中心,陕西 杨凌7200)
泌乳性状的选育是奶牛育种的重要内容之一,因为其直接决定着奶牛养殖的经济效益。乳成分包括乳脂、乳蛋白以及乳糖等,而乳蛋白分为酪蛋白(Casein)和乳清蛋白两大类[1],其中酪蛋白含有大量的钙和磷,属于特有的一种磷蛋白,按照生化性质分为αs1酪蛋白(CSN1S1)、αs2酪蛋白(CSN1S2)、β酪蛋白(CSN2)、κ酪蛋白(CSN3)和γ酪蛋白(γ Casein)5种亚型[2]。牛CSN3基因位于第6号染色体上,基因全长2 554 bp,含1个外显子,其中编码序列(Coding sequence,CDS)长483 bp。有研究证实,CSN1S1、CSN1S2、CSN2和CSN3基因在牛6号染色体q31~33位置上紧密连锁[3],显著影响牛奶中的乳蛋白率、乳脂率和产奶量等性状[4]。
在山羊上的研究发现,CSN3基因多态性与产奶量、乳品质、繁殖力等有一定的相关性[5]。在小鼠模型上,敲除CSN3基因后发现,尽管其它酪蛋白表达量未发生变化,但母鼠却丧失了泌乳能力,说明CSN3基因在泌乳过程中发挥着重要的调控作用[6]。此外,κ酪蛋白是大部分哺乳动物中唯一的糖基化酪蛋白,水解后产生的配糖巨肽可以预防胎儿对外源性蛋白过敏[7],还能为其提供氨基酸和碳水化合物[8]。
牛CSN3基因位于第6号染色体上,基因全长2 554 bp,含1个外显子。基于CSN3基因在泌乳过程中的重要调控功能,本研究采用DNA测序法,对565头中国荷斯坦牛CSN3基因进行群体变异检测分析,将检测到的单核苷酸多态性(Single nucleotide polymorphism,SNP)位点与中国荷斯坦牛的泌乳性状进行关联性分析,同时分析不同双倍型对中国荷斯坦牛泌乳性状的影响,以期为中国荷斯坦牛分子育种提供新的理论方法和技术途径。
1 材料与方法
1.1 血液采集及泌乳性状指标的获取
供试血样采自陕西省华阴市的西安草滩牧业有限公司华阴奶牛一场。采样时,随机选择健康的中国荷斯坦牛母牛565头,每头牛尾静脉采血10 mL,样品采集完毕后置于-80 ℃保存。荷斯坦牛胎次信息及DHI数据均来自采样牛场资料库,本试验选用DHI指标包括乳脂率、乳蛋白率及乳糖率。
1.2 中国荷斯坦牛CSN3基因的PCR扩增
外显子区域的SNP可能会引起氨基酸结构发生改变,进而影响基因的翻译;3′UTR区域SNP可能改变MicroRNA结合,导致基因的转录水平发生改变。故本试验针对CSN3基因外显子和3′UTR区域设计引物。根据GenBank公布的牛CSN3基因序列(GenBank登录号:NM_174294.2),利用Primer 5.0软件设计引物(表1),交由上海生工生物工程股份有限公司合成。由于CSN3基因3′UTR区域全长2 071 bp,无法一次进行扩增,因此设计2对引物进行分段(3′UTR1和3′UTR2)扩增。

表1 CSN3基因引物序列信息Table 1 Sequence information of primers for CSN3 gene
使用TIANamp Blood DNA kit试剂盒(天根,北京)提取DNA。DNA质量和浓度分别采用0.5%琼脂糖凝胶电泳和紫外分光光度计法进行检测。CSN3基因PCR反应体系为30.0 μL:含有核酸染料的dNTPs、TaqDNA聚合酶、10×Buffer的Mix共计15.0 μL,ddH2O 11.8 μL,上、下游引物各(10 pmol/μL)0.6 μL,模板DNA(50 ng/μL)2.0 μL。PCR反应条件为:95 ℃预变性5 min;94 ℃ 30 s,退火30 s(退火温度见表1),72 ℃ 30 s,35个循环;72 ℃延伸10 min,4 ℃保存。PCR扩增产物用1.0%琼脂糖凝胶进行电泳检测。
1.3 SNP检测
将扩增好的PCR产物送至上海生工生物工程股份有限公司进行纯化并测序,测序结果使用DNAMAN软件进行对比分析,筛选突变位点。本试验中的CSN3基因3′ UTR区域所扩增的2个片段分别独立分析。
1.4 数据统计与分析
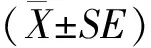
2 结果与分析
2.1 CSN3基因外显子和3′ UTR的PCR产物检测结果
图1、2为CSN3基因外显子、3′ UTR1和3′ UTR2的PCR扩增结果,其片段长度分别为375,905和511 bp。结果显示,扩增产物电泳条带清晰,特异性较好,可以用于后续测序分析。
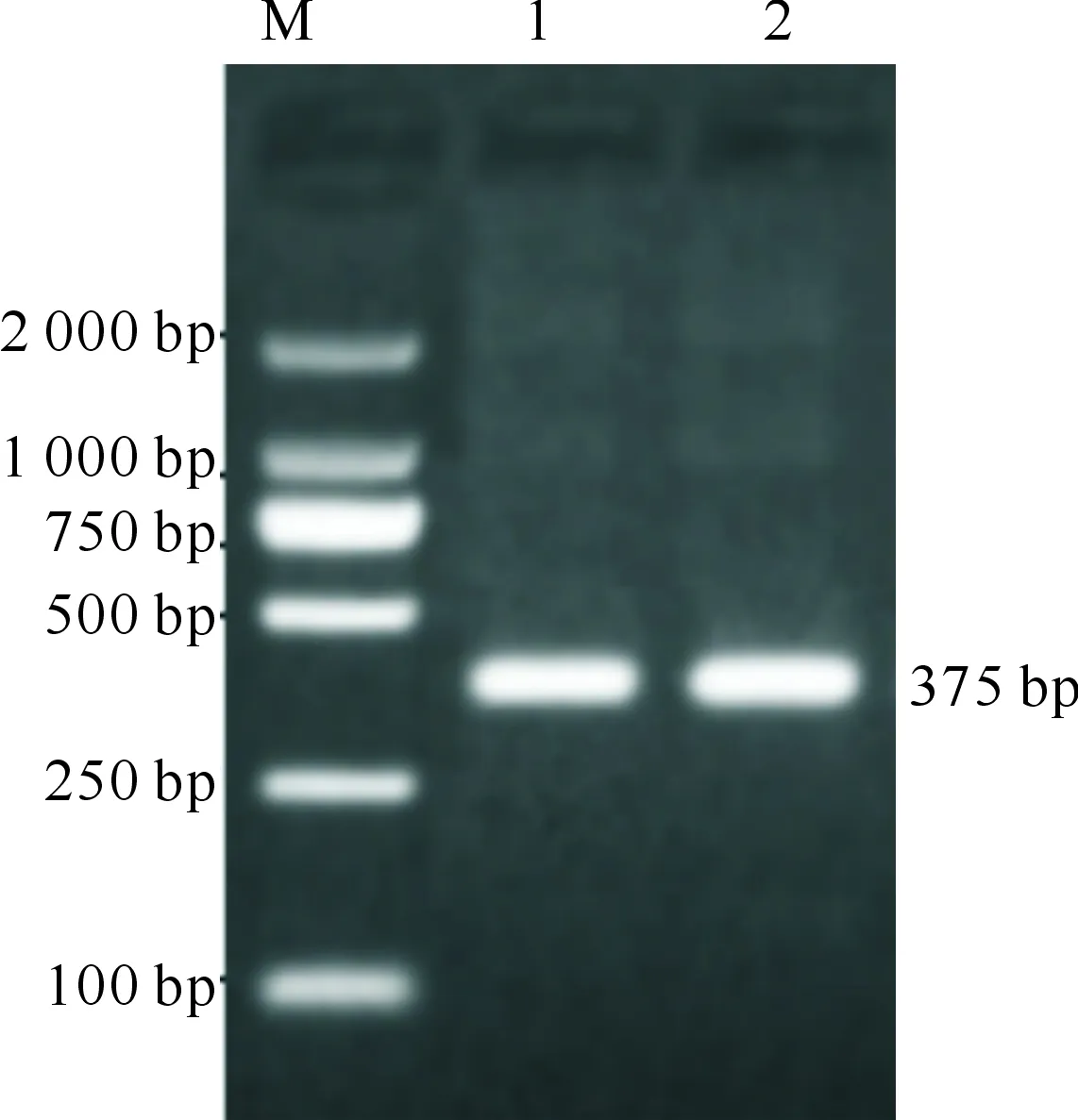
M.DNA Marker;1,2.外显子扩增产物M.DNA Marker;1,2.PCR product of exon图1 中国荷斯坦牛CSN3基因外显子PCR产物的电泳检测Fig.1 Electrophorogram of PCR product of exon1 in CSN3 gene of Chinese Holstein Cattle

M.DNA Marker;1,2.3′ UTR1;3,4.3′ UTR2图2 中国荷斯坦牛CSN3基因3′ UTR分段PCR扩增产物的电泳检测Fig.2 Electrophorogram of PCR product of 3′ UTR in CSN3 gene of Chinese Holstein Cattle
2.2 CSN3基因多态位点的遗传学分析
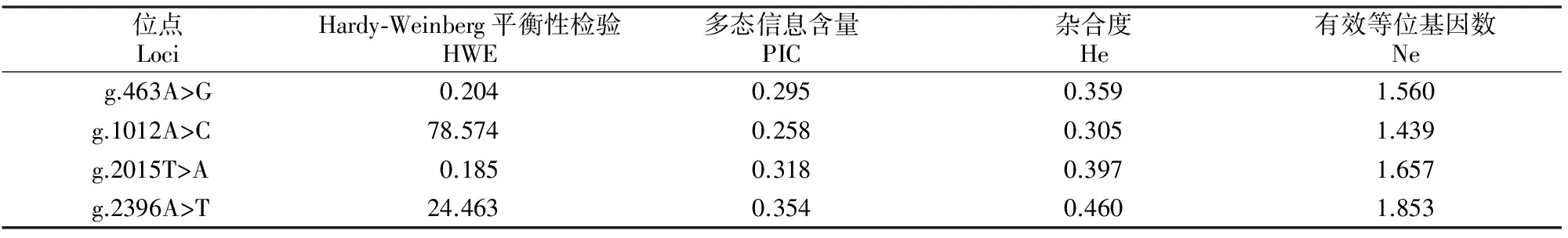
通过测序分析发现,CSN3基因外显子上存在1个SNP位点,为g.463A>G(图3-A);在3′UTR区域存在3个SNPs,分别是g.1012A>C(图3-B)、g.2015T>A(图3-C)和g.2396A>T(图3-D),尽管这3个SNP位点均不能改变氨基酸结构(同义突变),但可能改变MicroRNA结合,导致基因转录活性发生变化,因此具有一定的研究价值。
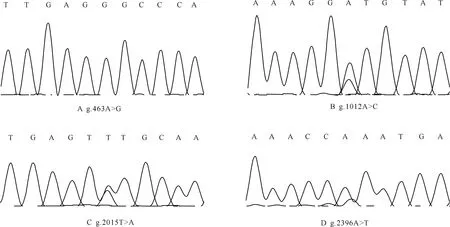
图3 中国荷斯坦牛CSN3基因突变区域基因型突变序列比对Fig.3 Sequencing comparison between different genotypes of mutational regions of CSN3 gene in Chinese Holstein cattle
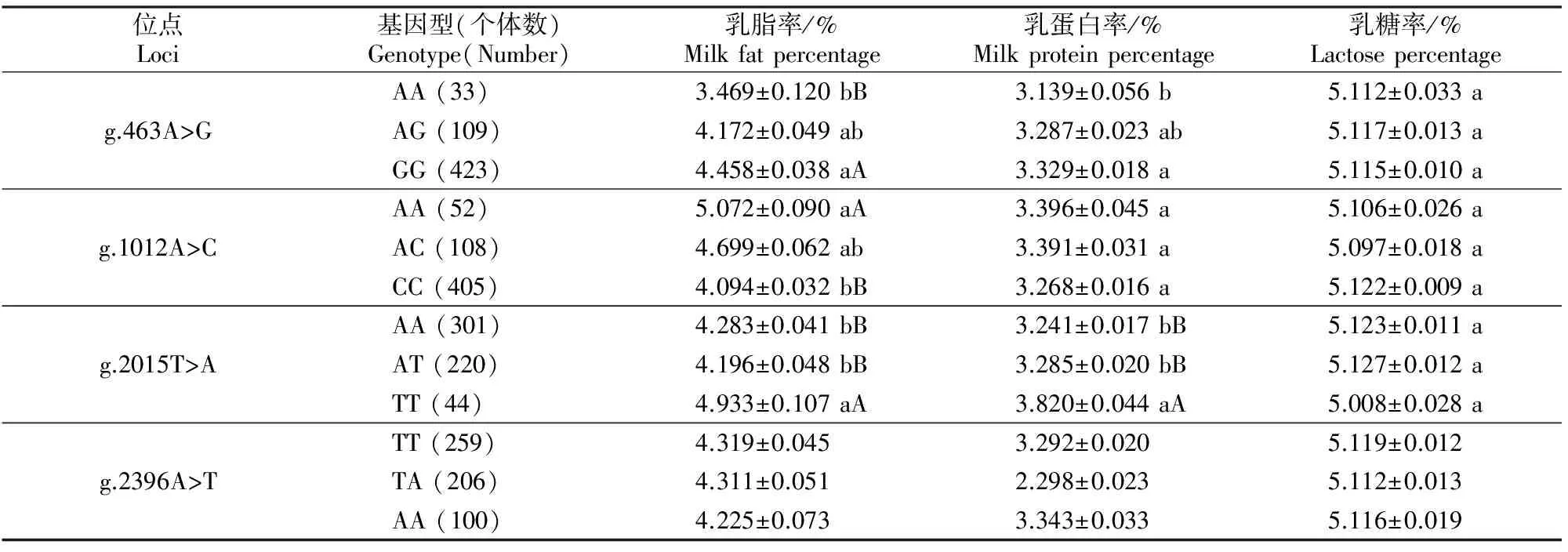
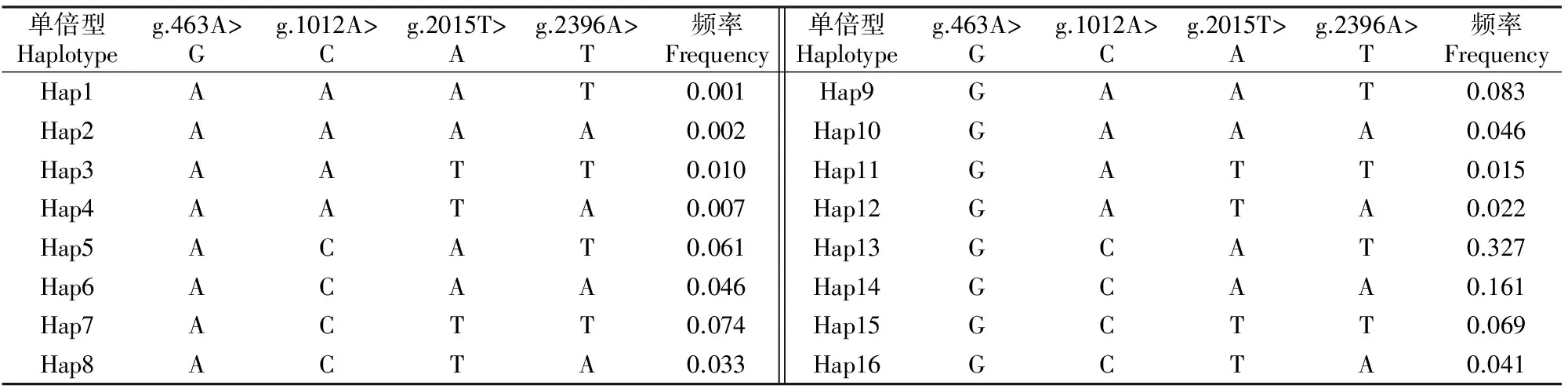
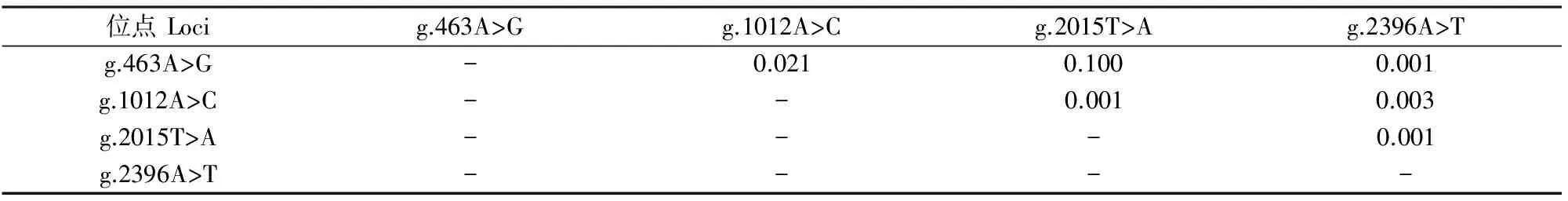
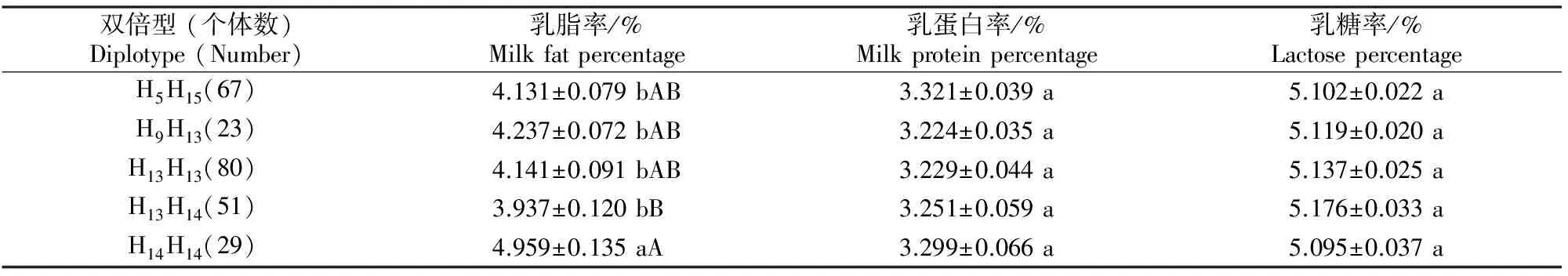
本研究分析了这4个SNPs位点的基因型频率、等位基因频率、杂合度、有效等位基因数和多态信息含量等遗传指标,并进行卡方检验检测Hardy-Weinberg平衡状态,结果见表2和表3。表2显示,CSN3基因g.463A>G处,GG为优势基因型;g.1012A>C处,CC是优势基因型;g.2015T>A处,AA为优势基因型;g.2396A>T处,TT为优势基因型。表3显示,g.463A>G和g.2015T>A位点处于Hardy-Weinberg平衡状态(HWE<5.991);4个SNPs位点均处于中度多态(0.25 表2 中国荷斯坦牛CSN3基因4个SNP位点的基因型频率和等位基因频率Table 2 Genotypes and allele frequencies among four SNP loci in CSN3 gene of Chinese Holstein cattle 表3 中国荷斯坦牛CSN3基因4个SNP位点的遗传参数Table 3 Genetic parameters among four SNP loci in CSN3 gene of Chinese Holstein cattle 将565头荷斯坦母牛的3个泌乳性状分别与CSN3基因突变位点的基因型进行关联分析,结果(表4)显示,g.463A>G位点上的GG基因型泌乳性状表现最优,乳脂率极显著高于AA基因型(P<0.01),乳蛋白率显著高于AA基因型(P<0.05);g.1012A>C位点上的AA基因型为优良基因型,其乳脂率极显著高于CC基因型(P<0.01);g.2015T>A位点不同基因型在乳脂率和乳蛋白率方面差异显著(P<0.01),TT基因型极显著高于AT和AA基因型。g.2396A>T位点3种基因型各泌乳性状指标无显著差异。 表4 中国荷斯坦牛CSN3基因的基因型与泌乳性状的关联性分析Table 4 Association of CSN3 gene polymorphisms with milk production traits of Chinese Holstein cattle 注:同一位点同列数据后标不同小写字母表示差异显著(P<0.05),标不同大写字母表示差异极显著(P<0.01)。 Note:Different lowercase letters in each column indicate significant difference (P<0.05) and different uppercase letters mean extremely significant difference (P<0.01). 对CSN3基因上4个SNPs位点进行单倍型分析,总共检测到16种单倍型,分别为Hap1~Hap16(表5),其中单倍型Hap13的发生频率最高,达到0.327,其次为单倍型Hap14和单倍型Hap9,发生频率分别为0.161和0.083。由于发生频率低于0.050的单倍型在统计学分析中没有意义,故只保留Hap5、Hap7、Hap9、Hap13、Hap14和Hap15等6种用于双倍型的关联性分析。 连锁不平衡分析结果(表6)表明,4个SNPs位点两两间的r2值均小于0.330。r2值大于0.330,表明2个位点间具有较强的连锁不平衡,因此本研究发现的4个SNPs位点间不存在强连锁性。 表5 中国荷斯坦牛CSN3基因4个SNPs位点的单倍型频率分析Table 5 Haplotypic frequencies among four SNPs loci in CSN3 gene of Chinese Holstein cattle 表6 中国荷斯坦牛CSN3基因4个SNPs位点的连锁不平衡分析Table 6 Estimated LD values of four SNPs loci in CSN3 gene of Chinese Holstein cattle 经分析得出,CSN3基因4个SNPs位点共有15种双倍型,将其中发生频率高于5.00%的双倍型与中国荷斯坦牛泌乳性状进行关联性分析,结果如表7所示。由表7可以看出,双倍型H14H14个体的乳脂率极显著高于双倍型H13H14(P<0.01),并且显著高于双倍型H5H15、H9H13和H13H13(P<0.05)。因此,在本试验群体中,双倍型H14H14是最优秀的双倍型。 表7 中国荷斯坦牛CSN3基因双倍型与泌乳性状的关联性分析Table 7 Diplotypes of CSN3 gene associated with milk production traits of Chinese Holstein cattle 注:同列数据后标不同小写字母表示差异显著(P<0.05),标不同大写字母表示差异极显著(P<0.01)。 Note:Different lowercase letters in each column indicate significant difference (P<0.05) and different uppercase letters mean extremely significant difference (P<0.01). CSN3基因编码的κ酪蛋白是哺乳动物乳蛋白的主要成分之一,其除起到营养作用外,还有一个重要的功能就是稳定蛋白质,防止牛奶蛋白质凝聚和沉淀[9],在自然状态下,使牛奶保持稳定的乳浊液状态[10]。酪蛋白参与调控酪蛋白微团的稳定性,影响乳微粒的大小,进而对乳品质具有重要的调控作用[11]。酪蛋白含有动物体几乎全部的必需氨基酸,是营养价值很高的蛋白质,并且能提高幼仔对钙、磷等的吸收能力[12]。基于此,CSN3基因可被视为对哺乳动物泌乳性能具有重要作用的候选基因。李玲等[13]发现,在3组泌乳性能不同的西农萨能奶山羊中,CSN3基因在乳腺中的表达量对乳蛋白和产奶量具有显著影响。赵春江等[14]通过酶切分型法分析了187头中国荷斯坦牛CSN3基因的多态性,发现CSN3的突变位点与乳蛋白率显著相关。Alim等[15]通过飞行质谱法,在荷斯坦牛CSN3基因中检测到6个SNP位点,这6个SNP位点均对产奶量、乳脂率和乳蛋白率有影响。本研究发现了4个SNP位点,这些位点与Alim所检测到的SNP位点不一致,导致这一结果的原因可能是样本选择以及样本量不相同造成的。Alim采集样本的地点在山东,样本数量为398头,而本试验的采样地点在陕西,样本数量为565头。分析表明,本试验检测出的CSN3基因的SNP位点与中国荷斯坦牛泌乳性状紧密关联。Caravaca等[16]就CSN3基因的多态性与产奶量、乳脂率、乳蛋白率、乳糖率的关联性进行了研究,证实CSN3基因BB型具有较高的产奶量和乳蛋白率。在捷克弗莱维赫牛中,Matějicek等[17]通过组合CSN3和LGB基因的SNP位点发现,组合基因型ABAA与泌乳性能紧密关联。Moioli等[18]在绵羊和山羊奶品质的研究中对CSN3的多态性进行检测,发现CSN3基因对乳蛋白含量有显著影响。Alipanah等[19]在俄罗斯牛种中发现,BB型个体比AA型和AB型有更高的乳蛋白率。有人通过筛选候选基因的方法,对CSN3基因外显子启动子区进行研究,发现了多个与产奶性状显著相关的SNP位点[20-21]。奶牛的乳脂率一般为3.5%左右,而本研究的乳脂率都在4%以上,平均值为4.345%。这是由于牛奶中乳脂率受到诸多因素影响,包括奶牛品种、营养摄入、管理水平及泌乳阶段等。本试验采样过程中,所涉及到的奶牛基本处于产后的第4~8周,这一阶段由于能量供给不足导致奶牛处于能量负平衡状态,奶牛会动用脂肪组织来供给能量,从而导致乳脂率较高。本试验在中国荷斯坦牛CSN3基因上检测到4个SNP位点,除g.2396A>T位点外,其余3个SNP位点与泌乳性状紧密关联,具体而言,g.463A>G和g.2015T>A均可以显著影响乳脂率和乳蛋白率,g.1012A>C可以显著影响乳脂率。双倍型关联性分析发现,H14H14乳脂率显著高于其余双倍型。综上,CSN3基因可以作为候选基因应用于中国荷斯坦牛泌乳性状的选育工作中。 目前,越来越多的研究指出,3′UTR区域上的突变位点可以通过改变mRNA的稳定性,进而影响到蛋白的表达和表型[22]。在中国荷斯坦牛上,FADS2基因3′UTR区域上的c.1571A>G和c.2776A>G能够显著影响牛乳脂肪酸组成[23];在秦川牛上,SIRT2基因的3′UTR区域的g.19676G>A突变与背膘厚显著关联[24];在南阳牛上,LHX4基因3′UTR区域的g.463A>G位点与牛的生长发育密切关联[25]。本试验在中国荷斯坦牛CSN3基因外显子和3′UTR区域共检测到4个SNP位点(g.463A>G,g.1012A>C,g.2015T>A和g.2396A>T),除g.463A>G外,其余3个SNP位点均定位于3′UTR区域,且不改变氨基酸结构,但关联性分析发现,g.1012A>C和g.2015T>A与中国荷斯坦牛泌乳性状密切关联。本试验中,g.1012A>C和g.2015T>A 2个SNP位点影响中国荷斯坦牛泌乳性状的具体作用机制还有待进一步研究。 本研究发现,CSN3基因在中国荷斯坦牛中存在多态性,其中g.463A>G、g.1012A>C和g.2015T>A与泌乳性状紧密关联,优势双倍型为H14H14,其个体的乳脂率显著或极显著高于其他双倍型,可以尝试将CSN3基因作为影响泌乳性状的候选基因用于标记辅助选择。 [1] Chen H,Lan X Y,Li R B,et al.The effect ofCSN1S2,CSN3 andβ-lg genes on milk performance in Xinong Saanen Dairy Goat [J].Acta Genetica Sinica,2005,32(8):804-810. [2] 杨永强,龚 俞,焦仁刚,等.牛αs2酪蛋白、κ-酪蛋白基因启动子区多态性研究 [J].畜牧与兽医, 2013,45(4):132-136. Yang Y Q,Gong Y,Jiao R G,et al.Study on the polymorphism ofCSN3 andCSN1S2 genes promoter region in cattle [J].Animal Husbandry & Veterinary Medicine,2013,45(4):132-136. [3] Bovenhuis H,Welled J I.Mapping and analysis of dairy cattle quantitative trait loci by maximum likelihood methodology using milk protein genes as genetic markers [J].The Genetics Society of America,1994,137(1):267-280. [4] Mehar S K,Peter C T,Imke T,et al.Quantitative trait loci mapping in dairy cattle: review and meta-analysis [J].EDP Sciences,2004,36:163-190. [5] 田 青.CSN3基因在动物生产中的研究进展 [J].饲料博览,2013,10(1):9-12 Tian Q.Research progress ofCSN3 gene in animal production [J].Feed Expo,2013,10(1):9-12 [6] Shekar P C,Goel S,Rani S D S,et al.kappa-Casein-deficient mice fail to lactate [J].Proceedings of the National Academy of Sciences of the United States of America,2006,23:8000-8005. [7] Melanie R G,Murray R G.Comparative aspects of milk caseins [J].Comparative Biochemistry and Physiology Part B,1999,124:133-145. [8] Swaisgood H.Primary sequence of Kappa-Casein [J].Journal of Dairy,1975,58(4):583-592. [9] Thorn D C,Meehan S,Sunde M,et al.Amyloid fibril formation by bovine milk kappa-casein and its inhibition by the molecular chaperones alphaS- and beta-casein [J].Biochemistry,2005,44(51):17027-17036. [10] 鞠志花,杨桂文.κ-酪蛋白基因及其功能研究进展 [J].中国畜牧兽医,2008,35(10):58-60. Ju Z H,Yang G W.Advances in the study of kappa casein gene and its function [J].Chinese Animal Husbandry and Veterinary,2008,35(10):58-60. [11] 祁 宏,刘丽仙,袁 峰,等.水牛κ-酪蛋白基因(CSN3)外显子4序列多态性研究 [J].家畜生态学报,2012,33(3):128-133. Qi H,Liu L X,Yuan F,et al.Study on the polymorphism of exon 4 of buffaloCSN3 [J].Journal of Animal Ecology,2012,33(3):128-133. [12] Martin P,Szymanowska M,Zwierzchowski L,et al.The impac t of genetic polymorphisms on the protein composit ion of ruminant m ilks [J].Reproduction Nutrition Development,2002,42(5):433-459 . [13] 李 玲,李 广,安小鹏,等.CSN1S2和CSN3基因表达量与西农萨能奶山羊产奶性状的关系 [J].西北农林科技大学学报(自然科学版),2010,38(1):5-10. Li L,Li G,An X P,et al.The expression ofCSN1S2 andCSN3 the relationship between the amount of Saanen milk production traits in dairy goats [J].Journal of Northwest Agriculture and Forestry University (Natural Science Edition),2010,38(1):5-10. [14] 赵春江,张 沅,李 宁.中国荷斯坦牛乳蛋白分子遗传多态性和产奶性状相关性研究 [J].黄牛杂志,1999,25(1):13-16. Zhao C J,Zhang Y,Li N.Study on relationship between genetic polymorphism of milk protein and milk performance in Chinese Holstein [J].Journa l of Yellow Cattle Science,1999,25(1):13-16. [15] Alim M A,Dong T,Xie Y,et al.Effect of polymorphisms in theCSN3 (κ-casein) gene on milk production traits in Chinese Holstein Cattle [J].Mol Biol Rep,2014,41:7585-7593. [16] Caravaca F,Carrizosa J,Urrutia B,et al.Effect of alpha(S1)-casein(CSN1S1) and kappa-casein(CSN3) genotypes on milk composition in Murciano-Granadina goats [J].Journal Dairy Science,2009,92(6):2960-2964 . [17] Matějícek A,Matějícková J,Němcová E,et al.Joint effects of CSN3 and LGB genotypes and their relation to breeding values of milk production parameters in Czech Fleckvieh [J].Czech J Anim Sci,2007,52(4):83-87. [18] Moioli B,Pilla F,Tripaldi C.Detection of milk protein genetic polymorphisms in order to improve dairy traits in sheep and goats:a review [J].Small Ruminant Research,1998,27(8):185-195 . [19] Alipanah M,Alexandrovna K L,Veladimirovich R G.Kappa-casein andPRL-RSAⅠgenotypic frequencies in two Russian cattle breeds [J].Archivos de Zootecnia,2008,57(218):131-138. [20] 林福玉,李 宁,陈永福.中国荷斯坦牛κ-酪蛋白基因多态性与产奶量的相关分 [J].中国畜牧杂志,1999,35(1):8-9. Lin F Y,Li N,ChenY F.Analysis of relationship between genotype ofbovine k-casein and milk yield [J].Chinese Journal of Animal Science,1999,35(1):8-9. [21] 鞠志花,李秋玲,王洪梅,等.中国荷斯坦奶牛κ-酪蛋白基因第四外显子的多态性与产奶性状的关联分析 [J].遗传,2008,30(10):1312-1318. Ju Z H,Li Q L,Wang H M,et al.Genetic polymorphism of κ-casein gene exon4 and its correlation with milk production traits in Chinese Holsteins [J].Hereditas,2008,30(10):1312-1318. [22] Yie S M,Li L H,Xiao R,et al.A single base-pair mutation in the 30-untranslated region of HLA-G mRNA is associated with preeclampsia [J].MHR:Basic Science of Reproductive Medicine,2008,14(6):49-53. [23] 徐晨希,王梦琦,朱小瑞,等.中国荷斯坦牛FADS2基因3′端SNP突变对乳中脂肪酸组成的影响 [J].中国农业科学,2016,49(11):2194-2202. Xu C X,Wang M Q,Zhu X R,et al.FADS2 gene 3′ end effects of SNP on milk fatty acids composition in Chinese Holstein [J].Chinese Agricultural Science,2016,49(11):2194-2202. [24] Gui L S,Hao R J,Zhang Y R,et al.Haplotype distribution in the class 1 sirtuin genes and their associations with ultrasound carcass traits in Qinchuan cattle (Bostaurus) [J].Molecular and Cellular Probes,2015,29(3):167-171. [25] Ren G,Chen H,Zhang L Z,et al.Acoding SNP ofLHX4 gene is associated with body weight and body length in bovine [J].Mol Biol Rep,2010,37(4):17-22.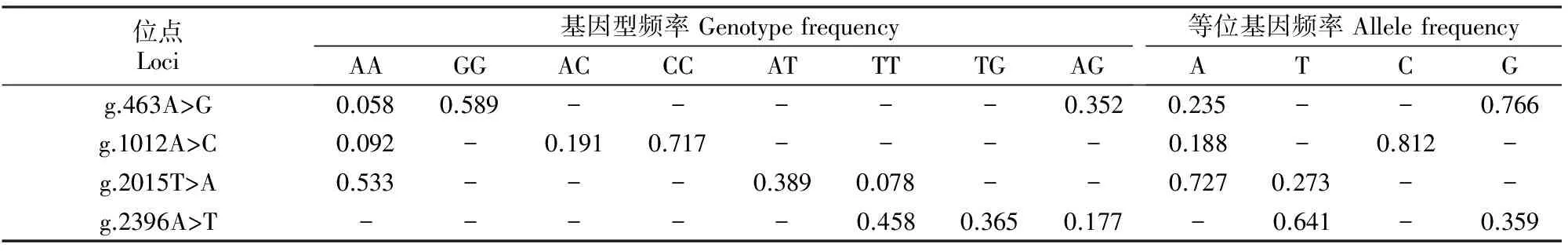
2.3 CSN3基因SNP位点的不同基因型对中国荷斯坦牛泌乳性状的影响
2.4 CSN3基因SNP位点的单倍型和连锁不平衡分析
2.5 CSN3基因SNP位点双倍型对中国荷斯坦牛泌乳性状的影响
3 讨 论
4 结 论

