胃癌组织中HBO1的表达变化及其意义
王燕,靳钦,刘颖,田薇
(南通大学附属医院,江苏南通226001)
胃癌是最常见的恶性肿瘤之一,病死率居恶性肿瘤的第五位[1]。2016年,中国国家癌症中心发布的数据显示我国胃癌的发病率及病死率率高居恶性肿瘤第二位[2]。但胃癌早期临床症状不典型,多数患者就诊时己处于中晚期。目前临床尚无有效治疗方法。研究胃癌的发病机制,寻找影响预后的肿瘤分子标志物,是目前的研究热点。
组蛋白乙酰转移酶结合至起源识别复合物1(HBO1)是组蛋白乙酰转移酶MYST家族中的重要一员,含611个氨基酸,定位于17号染色体上(17q21.3),是组蛋白特异的乙酰转移酶,在结合至起源识别复合物1(ORC1)上首次被发现[3]。HBO1具有典型的激酶催化活性区,在HBO复合体中起催化作用[4]。HBO1可与雄激素受体(AR)、孕激素受体(PR)、细胞周期蛋白依赖性激酶11B(CDK11B)、微染色体维持复合物2(MCM2)、ORC1等蛋白相互作用,参与细胞复制、转录的执行与调控,进而调控细胞增殖、脂肪形成,甚至导致淋巴瘤等肿瘤的发生[4,5]。同时HBO1还能与肿瘤抑制因子p53结合并抑制其功能[6]。目前关于HBO1在胃癌组织中的表达报道较少。我们观察了胃癌组织中HBO1的表达变化,并分析其与临床病理参数、患者预后的关系。现将结果报告如下。
1 资料与方法
1.1临床资料选择南通大学附属医院2009年 1 月~2011年12月收治的胃癌患者125例,男89例、女36例,年龄21~75( 55.24 ± 17.16) 岁;均行胃癌切除手术,术中留取胃癌组织、癌旁组织及正常胃组织标本。患者术前均未接受过放疗或化疗。所有标本经液氮冷冻,-80 ℃冰箱保存备用。所有患者均临床资料完整及电话随访记随访率100%,随访截至2016年6月。本研究经本院伦理委员会批准。
1.2主要试剂鼠抗人HBO1购自Abcam公司,HRP标记山羊抗鼠IgG购自Sigma公司、TRIzol试剂盒、qRT-PCR试剂盒购自美国Invitrogen公司,DAB显色试剂盒购自北京中杉金桥生物技术有限公司。
1.3胃癌组织、癌旁组织及正常胃组织HBO1蛋白检测采用免疫组化法。取胃癌组织、癌旁组织及正常胃组织,免疫组织化学染色后,将切片置于高倍显微镜下观察胃癌组织、癌旁组织及正常胃组织表达HBO1的情况。HBO1定位于细胞核中,测算HBO1的阳性表达率。
1.4 胃癌组织、癌旁组织及正常胃组织HBO1 mRNA检测采用qRT-PCR法。将胃癌、癌旁组织及正常胃组织用玻璃研磨器研磨,TRIzol法提取总RNA,测其浓度和纯度,用反转录试剂盒进行逆转录合成cDNA,以β-actin为内参,用PCR扩增试剂盒扩增,特异性引物序列:HBO1上游引物5′-TTTTGGCCGCTATGAACTG -3′,下游引物5′-GGAGGATTGTCTGGCTCTTC -3′,产物大小为755 bp。β-actin上游引物5′-AAGGCCAACCGTGAAAAGAT-3′,下游引物5′-GTGGTACGACCAGAGGCATAC-3′,产物大小为336 bp。PCR反应条件:95 ℃ 3 min、95 ℃ 40 s、55 ℃ 40 s、72 ℃ 40 s,40次循环,72 ℃ 10 min。采用2-ΔΔCt表示HBO1 mRNA 相对表达量。实验重复3 次,结果取平均值。
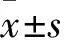
2 结果
2.2HBO1表达与胃癌患者临床病理参数的关系HBO1表达与胃癌肿瘤分化程度、TNM分期、浸润程度、淋巴结转移及术前血CEA水平有关(P均<0.05),而与患者性别、年龄及组织类型无关(P均>0.05)。见表1。
2.3HBO1表达与胃癌患者预后的关系Kaplan-Meier统计分析显示,HBO1高表达患者的总生存率降低(P<0.05)。单因素生存分析显示, HBO1表达、肿瘤分化程度、TNM分期、肿瘤浸润深度、淋巴结转移及术前血CEA值与胃癌患者的预后有关(P均<0.05,而与年龄、性别及组织类型等无关(P均>0.05),多因素分析显示HBO1表达、肿瘤分化程度、TNM分期及淋巴结转移为胃癌患者预后的独立危险因素(P均<0.05)。
3 讨论
HBO1是MYST家族的一员,为核定位蛋白,在细胞核中有不同的亚结构,它最初被确定为起源识别复合物(ORC)的伴侣,因其含有保守的MYST结构域,可乙酰化组蛋白和调控染色体结构,在小鼠胚胎干细胞中,Myst2/Kat7/HBO1三个蛋白相互作用参与H3和H4组蛋白的乙酰[7],在酵母中,Sas2作为HBO1的同源蛋白参与基因的沉默。另外,HBO1也与细胞周期有关[8],有可能参与了细胞周期的调控。HBO1不仅对组蛋白H3和H4具有组蛋白乙酰转移酶活性[7,9~12],对一些非组蛋白如ORC2、MCM2、CDC6等也有一定的作用[13]。在胚胎细胞中,HBO1的缺失可引起E10.5位点的H3K14广泛乙酰化,从而使胚胎的致死率降低[11]。
注:a: 含1例腺鳞癌;b:含4例黏液腺癌。
因HBO1具有广谱的乙酰转移酶活性,在DNA复制和转录激活中也发挥了多功能蛋白的作用。在DNA 复制方面,HBO1可以通过两种方式来调节:①乙酰化H4K5/8/12来促进复制前复合物(Pre-RC)的形成[11];②乙酰化H3K14来促进CDC45的装载以激活S期中的DNA复制[12]。在转录激活方面,首先,HBO1可结合到复制开始位点和基因编码区,与ING4 /5、hEaf6 和JADE1/2/3形成复合物,在染色质基因编码区通过复合物中多个锌指结构与组蛋白H3尾相互作用,通过ING4/5和JADE1S调控转录的起始和周期的延长[14];其次,研究显示,HBO1可增强转录活性,在猴CV1细胞中,孕酮的受体在的 MYST结构域作用下转录活性增强,其同源基因chameau (Chm) 可调控Hox基因沉默的突变进而起到转录的作用,同时Chm能在HEK293细胞中协同 DFos和DJun 促进基因的转录,在这个过程中 H4K16 乙酰化的水平也会增加[15],另外HBO1可与BRPF3协同调节复制起点的激活和组蛋白H3K14的乙酰化[12]。
HBO1的过表达可引起细胞过度复制活化、促进细胞增殖,进而引起各种肿瘤,如睾丸的生殖细胞肿瘤,乳腺癌和卵巢浆液性癌等[16]。组蛋白乙酰转移酶(HAT)HBO1/KAT7/MYST2通过泛素化促进ERα的蛋白酶体依赖性降解,并参与组蛋白广泛乙酰化、DNA复制、转录和细胞增殖,同时HBO1是泛素化ERα的另一种ERα共激活因子[17],这些跟乳腺癌的发病相关; HBO1与AR间存在相互作用关系,说明HBO1也与前列腺癌的发展有关;HBO1存在于肿瘤抑制物 ING4和ING5中,其乙酰化转移酶活性含有增强 p53的转录活性,ING4 能阻止体外培养的细胞失去接触抑制,在多种人肿瘤细胞株中ING4 处于失活状态,在小鼠模型中,ING4是脑胶质瘤和血管生成的有效抑制物[18],这可能与HBO1有关。HBO1编码区域已经被确定为一个共同的逆转录病毒整合位点,可与转录相关病毒结合,导致肿瘤发生,在鼠中能够导致骨髓白血病和淋巴瘤[19];另外HBO1可与不同的受体结合,对肿瘤性疾病的发生及进展产生不同的促进或抑制作用[20]。
鲁列斯三角形可以紧紧地嵌在一个正方形内部,即正方形的各边上都有鲁列斯三角形边缘上的点,如图2.设鲁列斯三角形的顶点A、B、C间距离为1cm,则正方形边长也是1cm.鲁列斯三角形三个顶点沿着正方形边界行进一周的过程中,正三角形ABC的外心T也在运动.
综上所述, 胃癌组织中HBO1高表达,其可能与胃癌的发生、发展有关,HBO1是影响胃癌患者预后的独立危险因素。
参考文献:
[1] Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015,65(2):87-108.
[2] Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016,66(2):115-132.
[3] Iizuka M, Stillman B. Histone acetyltransferase HBO1 interacts with the ORC1 subunit of the human initiator protein[J]. J Biol Chem, 1999,274(33):23027-23034.
[4] Foy RL, Song IY, Chitalia VC, et al. Role of Jade-1 in the histone acetyltransferase (HAT) HBO1 complex[J]. J Biol Chem, 2008,283(43):28817-28826.
[5] Johmura Y, Osada S, Nishizuka M, et al. FAD24 acts in concert with histone acetyltransferase HBO1 to promote adipogenesis by controlling DNA replication[J]. J Biol Chem, 2008,283(4):2265-2274.
[6] Iizuka M, Sarmento OF, Sekiya T, et al. Hbo1 Links p53-dependent stress signaling to DNA replication licensing[J]. Mol Cell Biol, 2008,28(1):140-153.
[7] Pardo M, Yu L, Shen S, et al. Myst2/Kat7 histone acetyltransferase interaction proteomics reveals tumour-suppressor Niam as a novel binding partner in embryonic stem cells[J]. Sci Rep, 2017,7(1):8157.
[8] Zong H, Li Z, Liu L, et al. Cyclin-dependent kinase 11(p58) interacts with HBO1 and enhances its histone acetyltransferase activity[J]. FEBS Lett ,2005,579(17):3579-3588.
[9] Avvakumov N, Lalonde ME, Saksouk N, et al. Conserved molecular interactions within the HBO1 acetyltransferase complexes regulate cell proliferation[J]. Mol Cell Biol, 2012,32(3):689-703.
[10] Lalonde ME, Avvakumov N, Glass KC, et al. Exchange of associated factors directs a switch in HBO1 acetyltransferase histone tail specificity[J]. Genes Dev, 2013,27(18):2009-2024.
[11] Kueh AJ, Dixon MP, Voss AK, et al. HBO1 is required for H3K14 acetylation and normal transcriptional activity during embryonic development[J]. Mol Cell Biol, 2011,31(4):845-860.
[12] Feng Y, Vlassis A, Roques C, et al. BRPF3-HBO1 regulates replication origin activation and histone H3K14 acetylation[J]. EMBO J, 2016,35(2):176-192.
[13] Iizuka M, Matsui T, Takisawa H, et al. Regulation of replication licensing by acetyltransferase Hbo1[J]. Mol Cell Biol, 2006,26(3):1098-1108.
[14] Saksouk N, Avvakumov N, Champagne KS, et al. HBO1 HAT complexes target chromatin throughout gene coding regions via multiple PHD finger interactions with histone H3 tail[J]. Mol Cell, 2009,33(2):257-265.
[15] Miotto B, Sagnier T, Berenger H, et al. Chameau HAT and DRpd3 HDAC function as antagonistic cofactors of JNK/AP-1-dependent transcription during Drosophila metamorphosis[J]. Genes Dev, 2006,20(1):101-112.
[16] Iizuka M, Takahashi Y, Mizzen CA, et al. Histone acetyltransferase Hbo1: catalytic activity, cellular abundance, and links to primary cancers[J]. Gene, 2009,436(1-2):108-114.
[17] Iizuka M, Susa T, Tamamori-Adachi M, et al. Intrinsic ubiquitin E3 ligase activity of histone acetyltransferase Hbo1 for estrogen receptor alpha[J]. Proc Jpn Acad Ser B Phys Biol Sci, 2017,93(7):498-510.
[18] Kim S, Chin K, Gray JW, et al. A screen for genes that suppress loss of contact inhibition: identification of ING4 as a candidate tumor suppressor gene in human cancer[J]. Proc Natl Acad Sci U S A, 2004,101(46):16251-16256.
[19] Suzuki T, Shen H, Akagi K, et al. New genes involved in cancer identified by retroviral tagging[J]. Nat Genet, 2002,32(1):166-174.
[20] Yagi T, Inoue N, Yanai A, et al. Prognostic significance of geminin expression levels in Ki67-high subset of estrogen receptor-positive and HER2-negative breast cancers[J]. Breast Cancer, 2016,23(2):224-230.

