利用siRNA高通量测序技术检测平菇病毒*
丛倩倩,兰玉菲,崔晓,安秀荣
(泰安市农业科学研究院,山东泰安271000)
平菇(Pleurotus ostreatus),又名糙皮侧耳,隶属于真菌门担子菌纲(Basidiomycetes)伞菌目(A-garicales)侧耳科(Pleurotaceae)侧耳属(Pleurotus),是我国栽培面积最广泛的食用菌之一[1]。平菇营养物质丰富,适应性强,栽培成本低,周期短,且栽培原料能就地取材,可为栽培者带来良好的经济效益。但是,在平菇菌种生产种植过程中可能会受到一些病毒的侵染,导致子实体畸形,品质下降,产量降低甚至绝收[2]。
病毒的检测和诊断是预防和控制病毒病害的关键技术环节,对生产实践具有重要的指导意义。相对于其他作物病毒病害的研究,食用菌病毒病害的研究范围较窄,研究基础较为薄弱。目前,对食用菌病毒的检测和鉴定通常采用电镜法[3-4]、血清学方法[5-6]、dsRNA技术[7-10]、PCR[11-12]等。上述方法仅在对病原物特征有预先了解的情况下使用,而在对病原物了解缺乏的情况下,这些检测方法的使用就受到了极大的限制。
应用siRNA高通量测序技术鉴定病毒是近几年快速发展起来的一项技术。在病毒与寄主的互作中,病毒复制产生的双链RNA(dsRNA)可以被寄主体内Dicer酶切割成大量针对入侵病毒的小干扰RNA(siRNA)[13-14]。siRNA分子量较小,长度为20 nt~25 nt,很容易与细胞中的其他RNA区分,因此可以利用高通量测序技术对病毒产生的siRNA序列进行测序,并利用软件对测序结果进行组装,组装结果与数据库进行比对来鉴定和发现新病毒[15-18]。
本研究利用siRNA高通量测序技术,检测疑似被病毒侵染的平菇样品中是否携带病毒,旨在为其他种类食用菌的病毒检测鉴定提供借鉴和参考。
1 材料与方法
1.1 供试菌株
平菇样品采集于山东省聊城市郑家镇,样品表现症状为子实体球形变小,菌盖黄褐色,见图1。
1.2 方法
1.2.1 RNA的提取与质量检测

图1 平菇样品症状Fig.1 Sample symptoms of Pleurotus ostreatus
将5个平菇样品混合后,采用Trizol法提取总RNA。总RNA用干冰保存,送往北京诺禾致源生物信息科技有限公司进行测序。测序前需对RNA的质量进行检测,包括:分析RNA是否降解及其是否有污染,检测RNA的纯度和完整性,精确定量RNA的浓度。
1.2.2 文库的构建
样品检测合格后,使用Small RNA Sample Pre Kit构建cDNA文库。
1.2.3 测序数据过滤
构建好的文库利用Illumina HiSeqTM 2000进行测序。为了保证分析质量,必须对测序数据进行过滤,去除接头序列和低质量的读段(reads),得到干净的读段(clean reads),最后选取18 nt~26 nt的reads来进行后续分析。
1.2.4 siRNA的序列拼接和候选病毒筛选
所得的序列去除3’接头,然后分别利用PFOR(参数为-x4)和Velvet(参数为k-mer 17)软件对筛选所得siRNA进行序列拼接,获得较长的重叠群(contigs)。首先将拼接所得的contigs与寄主基因组数据库(Host Genome)进行比对,除去寄主的基因组序列,然后将剩余的contigs用BLASTN与核酸数据库(NCBI Nt)进行比对,寻找与这些contigs高度同源的序列,再然后用BLASTX将没有找到高度同源序列的contigs与蛋白数据库(NCBI Nr)进行比对,寻找与contigs部分同源的序列,最后将与contigs高度同源序列和部分同源序列分别与病毒核酸数据库(Virus RefSeq Nucleotide)和病毒蛋白数据库(Virus RefSeq Protein)进行比对,筛选出和病毒序列同源的contigs,从而得到候选病毒。比对采用参数限制e期望值(e-value)(1e-6 for BLASTN,1e-4 for BLASTX)。
1.2.5 PCR验证
根据所得候选病毒,设计特异性引物分别进行扩增以进一步验证测序结果的准确性。
2 结果与分析
2.1 平菇样品总RNA质量分析
平菇样品总RNA质量分析结果见表1,检测结果合格,符合建立cDNA文库的要求。
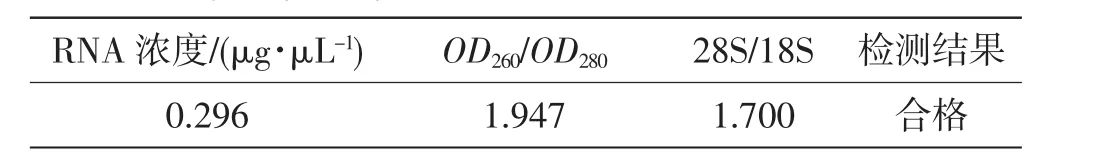
表1 平菇样品总RNA质量分析Tab.1 Quality analysis on total RNA of Pleurotus ostreatus
2.2 测序数据质量情况
对siRNA测序结果进行分析,去除接头序列和低质量的读段,测序得到的数据统计结果见表2。
对数据库的测序质量进行评估,其中干净的读段占总读段的百分比达到了95%以上,表明测序质量较高,达到信息分析的要求。
2.3 siRNA的拼接与注释
5个数据库所注释的contigs维恩图和结果统计见图2和表3。
从clean reads中筛选长度18 nt~26 nt的siRNA来进行后续分析,经长度筛选后所得的总reads数为2 749 087。使用PFOR软件和velvet软件对筛选的siRNA进行序列拼接,得到总的重叠群数为479,将拼接所得的contigs分别与Host Genome、NCBI Nr、NCBI Nt、Virus RefSeq Nucletide和Virus RefSeq Protein五个数据库进行比对,发现被Host Genome注释所得的contigs数为85,未被Host Genome而只被NCBI Nr和NCBI Nt注释的contigs数分别为5和17,被Virus RefSeq Nucleotide和Virus RefSeqProtein注释所得的contigs数分别为2和7。
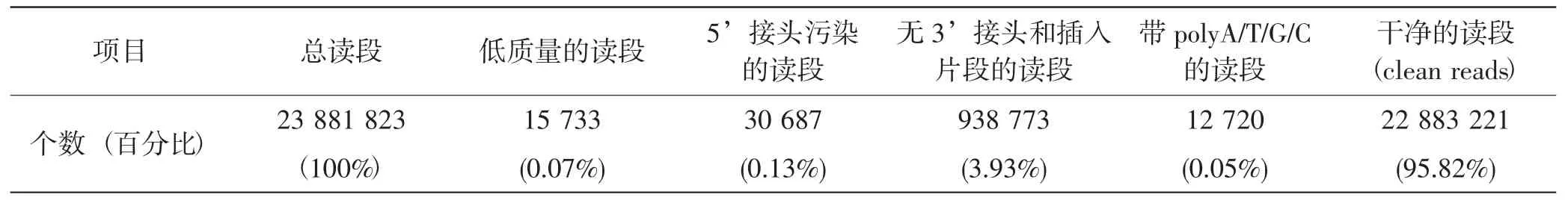
表2 测序数据质量统计Tab.2 Quality statistics of sequencing data
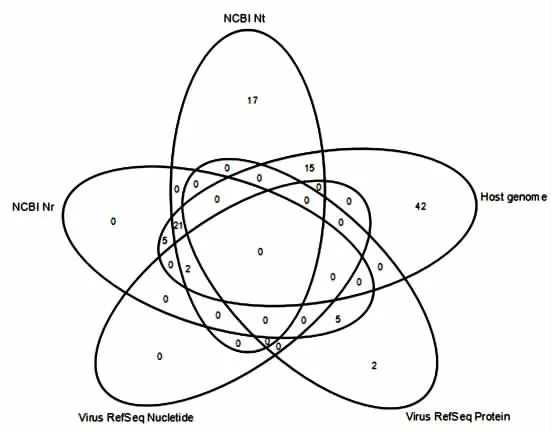
图2 5个数据库所注释的contigs维恩图Fig.2 Contigs Venn diagrams annotated by 5 databases
2.4 候选病毒检测结果
在以上分类注释结果中分离与病毒相关的con tigs,其病毒检测结果见表4。
从结果可以看出,文库中检测到8种候选病毒。其中,与Burdock mottle virus同源的contigs数目最高为3,其次检测到了与Choristoneura occidentalisgranulovirus和Beet soil-borne mosaic virus同源的contigs数都为2,样本中还检测到与Culex originated Tymoviridae-like virus、Fig fleck-associated virus、Beet necrotic yellow vein virus、Blackberry virus E、Grapevine fleck virus同源的contigs数都为1。由于检测到的与8种病毒同源的contigs数目均较少,而且能比对上的contigs总长度都较短,因此测序结果需要进一步的试验验证才能明确所采集的样品里是否含有这几种病毒。
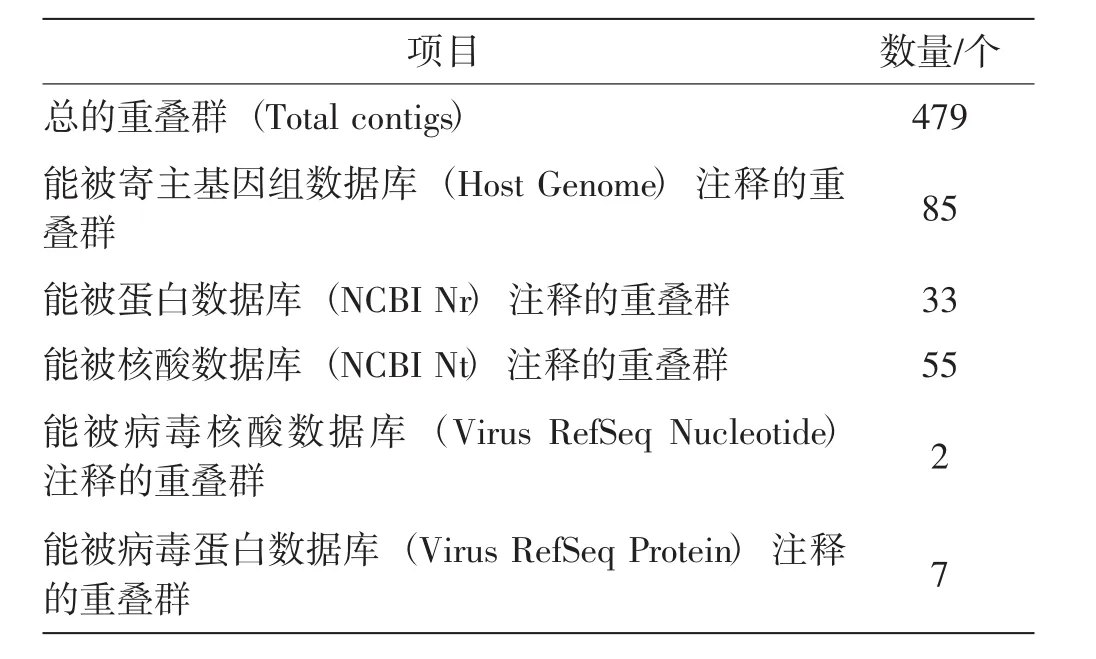
表3 5个数据库所注释的contigs结果统计Tab.3 Contigs statistics annotated by 5 databases
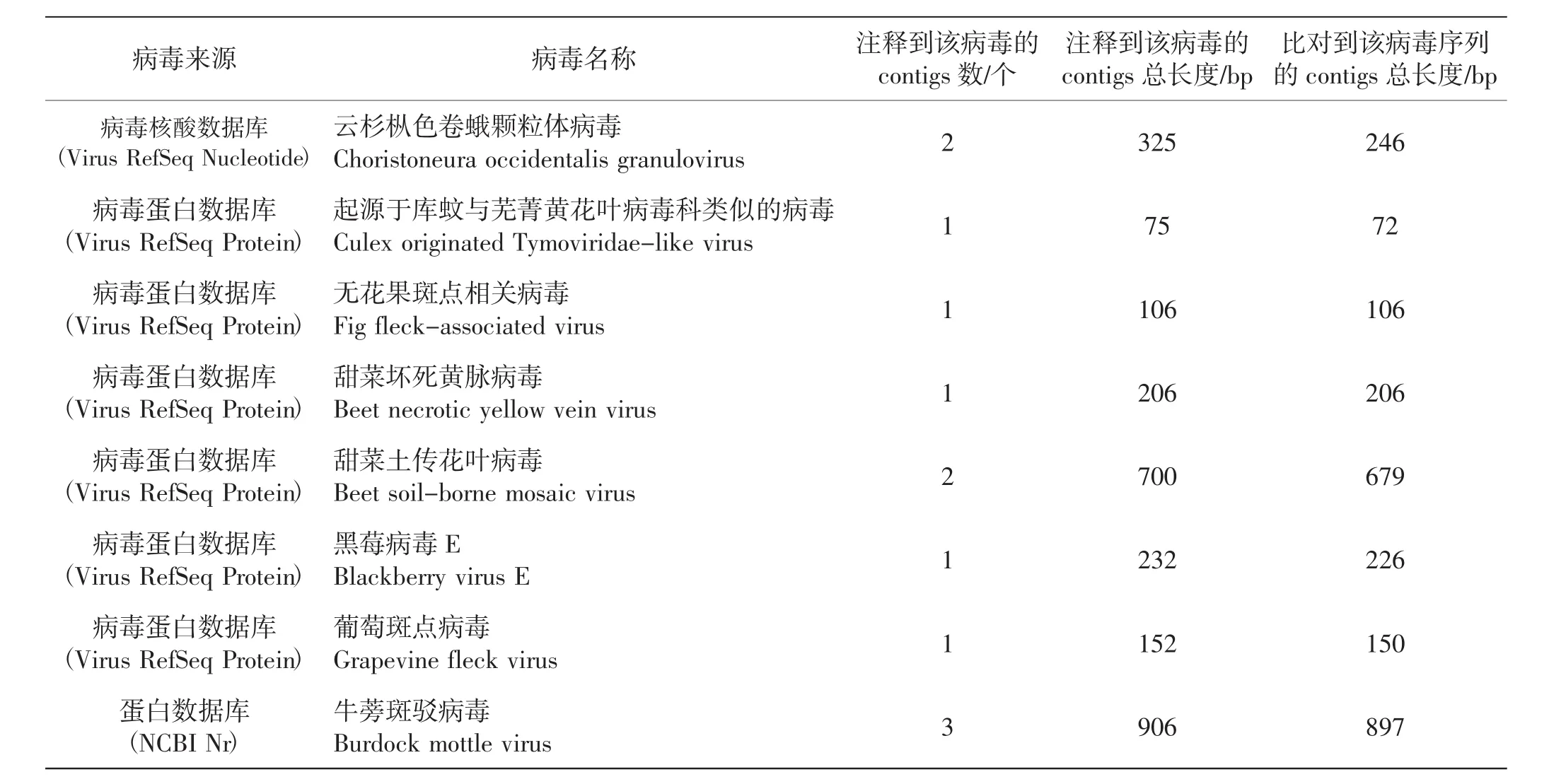
表4 平菇siRNA文库病毒检测结果Tab.4 Detection results of virus in Pleurotus ostreatus siRNA library
2.5 RT-PCR验证
根据序列拼接结果,设计针对这八种候选病毒的特异性引物,利用RT-PCR方法进行扩增验证,RT-PCR结果却未检测到目的条带,说明采集的平菇样品中不存在这八种病毒,推测平菇球形症状的产生可能不是由于病毒侵染所导致的。
3 讨论
常规的检测方法在病毒检测和新病毒发现中存在一定缺陷,适用于研究比较清楚的病毒,其针对性强,但对于新病毒的发现较困难。与其相比,应用深度测序技术可在病毒序列、特征等信息未知情况下,只需要提取样品总RNA,通过筛选,建立小RNA文库,然后对小RNA进行测序,并通过组装来检测和发现病毒,灵敏度高,适用性强,获得的信息丰富。近年来,利用siRNA深度测序技术已在鉴定许多动植物的未知病原物中发挥重要作用[15-18]。
目前,对食用菌病毒研究相对较少,可以利用的序列信息也较少,利用常规检测方法难以对食用菌病毒做到全面检测和鉴定,特别是尚未报道的病毒[19]。本研究利用siRNA高通量测序技术直接对球形平菇样品中提取的siRNA进行测序和生物信息学分析,鉴定其是否受病毒侵染,结果证明本研究采集的球形平菇样品中不存在病毒,表明这种症状的产生并不是由病毒侵染所导致的。平菇畸形症状产生的原因有很多,包括栽培环境不适宜、管理不当、菌种退化或变异等,我们所采集的样品具体由哪种原因所导致还有待于进一步的试验证明。
[1]陈玉梅.平菇高产高效栽培[J].农业知识:瓜果菜,2013(23):46-47.
[2]李彦鹏,梁振普,张小霞,等.食用菌病毒研究进展[J].中国农学通报,2006,22(8):408-413.
[3]Hollings M.Viruses associated with die-back disease of cultivated mushroom[J].Nature,1962,196(4858):962-965.
[4]刘宏迪,梁平彦.感染病毒的侧耳的超微结构[J].微生物学报,1986,26(3):221-225.
[5]Schwartz EF,Stollar BD.Antibodies to polyadenylatepolyuridylate copolymers as reagents for double strand RNA and DNA-RNA hybrid complexes[J].Biochem and Biophys Res Commun,1969,35(1):115-120.
[6]张鹤龄,马志亮.双链核糖核酸免疫化学研究[J].真菌学报,1984,3(4):233-238.
[7]Morris TJ,Dodds JA.Isolation and analysis of double-stranded RNA from virus infected plant and fungal tissue[J].Phytopathology,1979,69(8):854-858.
[8]周雪平,李德葆.双链RNA技术在植物病毒中的应用[J].生物技术,1995,5(1):1-4.
[9]方详,叶华智,曹燕.四川省稻瘟病菌中dsRNA因子的检测[J].四川农业大学学报,2000,18(4):315-318.
[10]Shigeru K,Tsutomu F,Shen RL,et al.Double-stranded RNA virus in human pathogenmic fungus blastomyces dermatitidis[J].Journal of Virology,1994,68(11):7554-7558.[11]刘莉,陈集双.利用TaqDNA聚合酶直接从双链RNA模板中扩增靶序列[J].微生物学通报,2007,34(1):57-60.
[12]章松柏,吴祖建,段永平,等.一种实用的双链RNA病毒基因克隆方法[J].长江大学学报:自然版,2005,25(1):71-73.
[13]Hamilton AJ,Baulcombe DC.A species of small antisense RNA in posttranscriptional gene silencing in plants[J].Science,1999,286(5441):950-952.
[14]Blevins T,Rajeswaran R,Shibaprasad PV,et al.Four plant dicers mediate viral small RNA biogenesis and DNA virus induced silencing[J].Nucleic Acids Res,2006,34(21):6233-6246.
[15]Kreuze JF,Perez A,Untiveros M,et al.Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs:a generic method for diagnosis,discovery and sequencing of viruses[J].Virology,2009,388:1-7.
[16]Hagem C,Frizzi A,Kao J,et al.Using small RNA sequences to diagnose,sequence,and investigate the infectivity characteristics of vegetable-infecting viruses[J].Arch Virol,2011,156(7):1209-1216.
[17]Wu QF,Luo YJ,Lu R,et al.Virus discovery by deep sequencing and assembly of virus-derived small silencing RNAs[J].Proc Natl Acad Sci USA,2010,107(6):1606-1611.
[18]Giampetruzzi A,Roumi V,Roberto R,et al.A new grapevine virus discovered by deep sequencing of virus-and viroid-derived small RNAs in Cv pinot gris[J].Virus Res,2012,163(1):262-268.
[19]徐章逸,边银丙.食用菌病毒病害研究进展[J].食用菌学报,2008,15(3):80-84.

