拟穴青蟹miR-34靶基因预测及其生物信息学分析
梁萌萌,张子平,贾锡伟,王艺磊*
(1.集美大学水产学院,福建 厦门 361021;2.福建农林大学动物科学学院,福建 福州 350002)
拟穴青蟹miR-34靶基因预测及其生物信息学分析
梁萌萌1,张子平2,贾锡伟1,王艺磊1*
(1.集美大学水产学院,福建 厦门 361021;2.福建农林大学动物科学学院,福建 福州 350002)
MiR-34是一类在进化过程中高度保守的miRNA家族,参与机体的许多重要生命活动。本研究首先比较本实验室构建的拟穴青蟹miRNA数据库中的miR-34(spa-miR-34)和通过miRBase获得的多个物种的miR-34的序列特征,然后应用RNAhybrid和Segal Lab两种在线工具预测spa-miR-34的靶基因,用blast2go软件对这些靶基因进行功能注释和信号通路富集分析。结果表明,spa-miR-34可能的靶基因有66个,这些基因的功能主要包括氧化还原、磷酸化、钙离子跨膜运输、转录等生物学过程;信号通路主要富集于精氨酸和脯氨酸代谢、抗生素生物合成、苯丙素生物合成、糖酵解和糖异生等合成代谢通路中。spa-miR-34预测的靶基因所涉及的基因家族和通路亦包括CHH家族、ERK通路、细胞周期和UPP通路、SUMO通路。此外,spa-miR-34预测的靶基因还有蜕皮激素受体,推测spa-miR-34在生殖、蜕皮及氨基酸等物质代谢方面具有重要作用。本研究为进一步了解spa-miR-34在拟穴青蟹中的作用提供了重要参考资料。
miR-34;靶基因;生物信息学;拟穴青蟹
MicroRNA(简称miRNA)是一类内源性的非编码单链小分子RNA,长度约22~25个核苷酸,在动物中其通过与靶基因mRNA的3′非翻译区(3′untranslated region,3′UTR)的不完全性互补配对,引起转录抑制或者下调,进而参与调控机体内多种重要的生命活动过程,如生长发育、生殖和免疫等[1-2]。
MiR-34是一类在进化过程中高度保守的miRNA家族,且广泛存在于后生动物中[3]。越来越多的研究表明miRNA-34在机体生命活动中发挥着重要的作用[4],如参与人类癌症的发生[5-6]及衰老过程[7-8],但在拟穴青蟹中,目前对其miRNA-34的研究未见报道。根据拟穴青蟹miR-34(spa-miRNA-34)靶基因的预测结果,通过数据整合和应用生物信息学分析方法,进一步挖掘理论上存在的可能结果,并对结果进行系统分析整理,可为spa-miR-34靶基因的实验鉴定及其生物学功能研究提供数据支持和理论指导。
1 材料与方法
1.1 材料
spa-miR-34:拟穴青蟹购自厦门集美农贸市场,分别提取不同性腺发育阶段的三个眼柄的RNA,等量混合RNA分别形成雌雄眼柄RNA两个样品池,送至杭州联川生物技术有限公司,采用illumina二代测序仪进行高通量测序并通过生物信息学分析获得拟穴青蟹眼柄miRNA数据库,从该数据库中获得了表达量较高的spa-miR-34。
不同物种的miR-34:搜索miRBase(http://www.mirbase.org/)可获得不同动物的miR-34的成熟序列。
拟穴青蟹基因的3′UTR:通过NCBI数据库(https://www.ncbi.nlm.nih.gov/)获取拟穴青蟹所有已知基因的3′UTR,用于进行靶基因的预测。
1.2 方法
通过MEGA7软件对本实验室高通量测序获得的spa-miRNA-34和miRBase获得的miR-34进行比对,分析不同物种miR-34的保守性。
利用RNAhybrid(http://bibiserv.techfak.uni-bielefeld.de/rnahybrid?id=rnahybrid_view_submission)和Segal Lab(https://genie.weizmann.ac.il/pubs/mir07/prediction.html)两种通用的靶基因预测软件对spa-miRNA-34进行靶基因预测,取预测结果的交集。根据miRNA预测靶基因的原理,RNAhybrid在线预测软件设置的参数为:种子区域第二位至第八位互补,允许G∶U配对,最小结合自由能(MFE)设置为-20 kcal/mol,结合自由能越低表示miR-34与靶基因的3′UTR区结合越紧密;Segal Lab在线预测软件设置的参数为:种子区域第二位至第八位互补,允许G∶U配对,允许单个错配。由于拟穴青蟹属非模式生物,数据库中的基因信息不足,使用RNAhybrid和Segal Lab在线软件更加方便进行靶基因预测。
利用blast2go(https://www.blast2go.com/)中的 GO注释和KEGG通路分析对spa-miR-34的所有靶基因进行功能注释和信号通路的富集分析。利用blast2go软件进行blast时设置的参数为:E值为1.0E-3。进行功能注释时的参数设置为E值为1.0E-6,其他采用默认值。
2 结果与分析
2.1 miR-34的保守性
通过搜索miRBase发现miR-34是研究比较广泛且深入的一种miRNA,分别检索人、鼠、果蝇等16个物种及实验室获得的spa-miR-34成熟序列,利用MEGA7对miRBase数据库获得的不同物种的miR-34进行ClustalW分析,比对结果发现17个物种的miR-34序列一致性极高(表1),表明miR-34在不同物种间具有高度的保守性。

表1 不同物种的miR-34的成熟序列
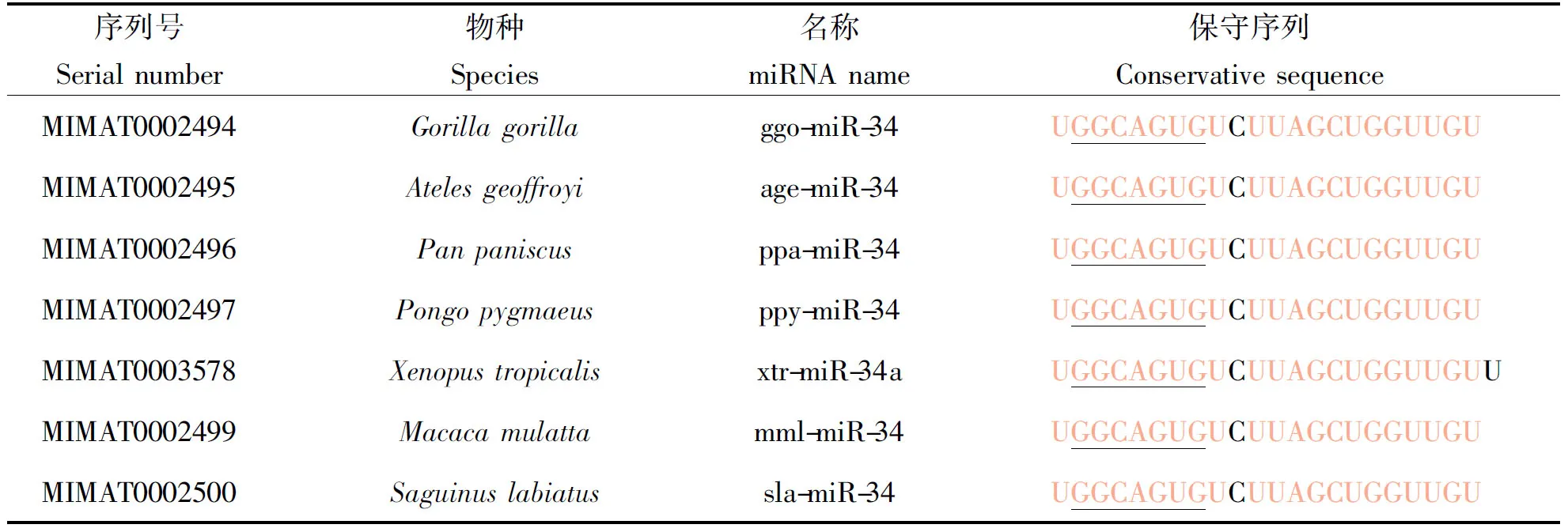
续表1
注:红色表示完全相同的碱基,下划线表示种子序列。
Note:The same nucleotidesare in red,seed sequences are underlined.
2.2 spa-miR-34靶基因的预测
用RNAhybrid和Segal Lab2在线工具预测spa-miR-34的靶基因,选取预测结果的交集,得到66个spa-miR-34的靶基因(表2)。

表2 2种在线工具预测miR-34靶基因的交集

续表2

续表2
2.3 spa-miR-34靶基因的GO分析
为进一步认识spa-miR-34的靶基因,对集合中的66个基因进行GO分类富集分析,GO注释得到56个基因的GO生物学过程注释信息,将这56个基因投到GO的生物学过程上,结果发现spa-miR-34的靶基因主要富集于氧化还原过程、磷酸化过程、钙离子跨膜运输、转录等生物学过程(表3)。
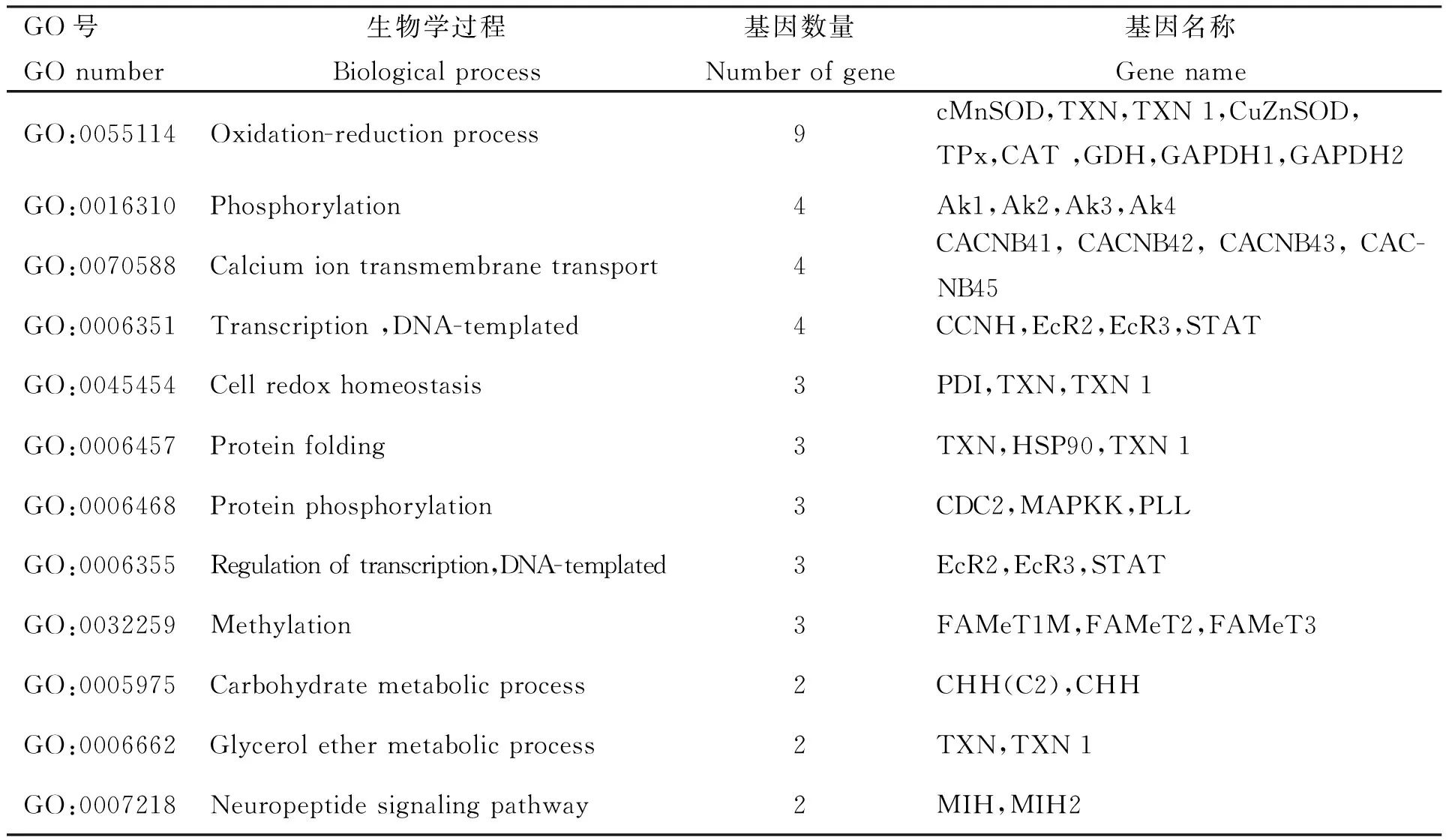
表3 spa-miR-34预测靶基因GO生物学过程分析

续表3
2.4 miR-34靶基因的KEGG分析
在GO注释分类的基础上,利用已有生物通路数据,对基因集合中的66个基因进行生物通路富集分析。结果显示:在经典通路数据库KEGG中Spa-miR-34预测靶基因集合显著富集于精氨酸和脯氨酸代谢通路、生物合成抗生素通路、苯丙素生物合成通路、糖酵解和糖异生等通路(表4)。
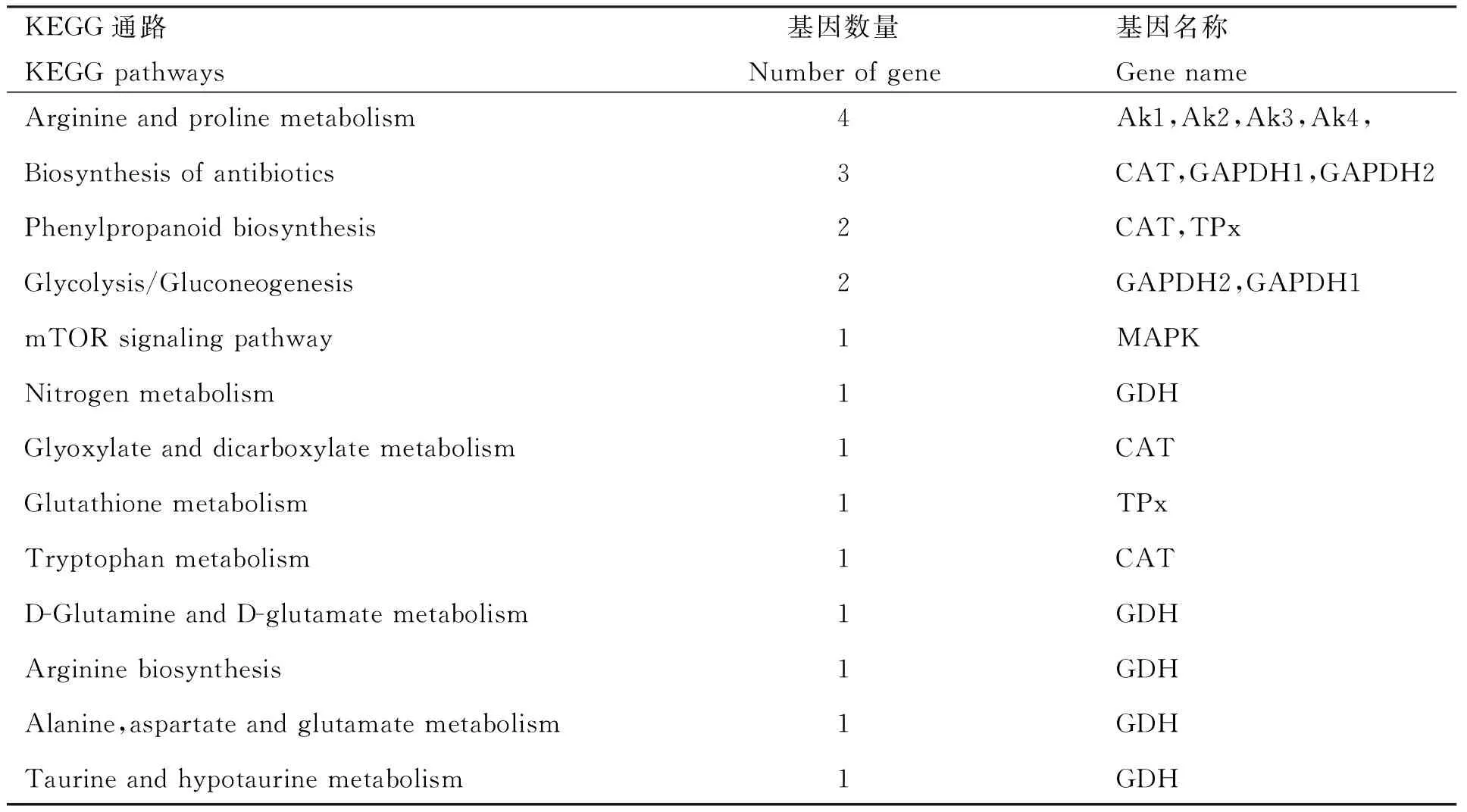
表4 spa-miR-34预测靶基因的通路分析
3 讨论
靶基因mRNA的3’端UTR区和miRNA5’端第2~8位碱基完全互补,这7个碱基序列称作“miRNA种子区”[3]。MiR-34作为进化上较为保守的转录后调控因子,一定程度地表现在与靶基因进行识别的“种子序列”中[9]。“种子序列”的保守性较高可能反映出miR-34在物种演化过程中始终扮演着重要的角色。本实验室前期在性腺miRNA组的测序中获得了spa-miR-34,关于miR-34靶基因的研究及验证,多集中在高等动物中,在水产无脊椎动物中尚未涉及。在人类中miR-34的靶基因有E2F2[10]NumbL[11]、Notch1[11]、OCT4[12]、DLL1[13]、IGFBP-3等[14]。在蜜蜂中的靶基因有eve、ftz-f1和act5C[15]。
通过生物信息学方法预测spa-miR-34靶基因,对spa-miR-34靶基因进行功能富集分析,发现其靶基因的功能主要富集于氧化还原、磷酸化、钙离子跨膜运输、转录等生物学过程中。其中富集最多的是抗氧化系统中的抗氧化酶基因,如cMnSOD、CuZnSOD、CAT等,说明spa-miR-34可能参与拟穴青蟹的抗氧化防御过程。对spa-miR-34靶基因进行生物通路的分析,发现信号通路主要富集于精氨酸和脯氨酸代谢、抗生素生物合成、苯丙素生物合成、糖酵解和糖异生等合成代谢通路中,说明spa-miR-34可能参与氨基酸等物质代谢过程。
spa-miR-34预测的靶基因所涉及的基因家族和通路亦包括CHH家族(CHH,MIH)[16]、ERK通路(MAPK,MAPKK,STAT,14-3-3)[17-19]、细胞周期和UPP通路(cyclinA,cyclinH,CDC2,UBE2I)[20-21]、SUMO通路(SUMO-2)[22-23]。CHH家族神经肽是调节甲壳动物生长发育、生殖、蜕皮等生理过程的重要激素,例如CHH可以刺激卵黄发生,促进生殖[24],MIH不仅调控蜕皮的发生,也能促进性腺的发育[24-25]。ERK通路在卵母细胞减数分裂、受精、胚胎发育、细胞凋亡、突触可塑性和记忆形成等时期均起作用[26]。ERK通路在甲壳动物性腺发育中的作用已逐渐被认识[17,27]。细胞周期和UPP通路涉及机体的各种基本生理过程,在精子发生、成熟,受精,卵母细胞减数分裂,卵子发生,早期胚胎发育,性腺发育等阶段均有作用[28]。例如,CDC2和cyclin B参与了拟穴青蟹性腺发育[21],UBE2I在小鼠卵母细胞基因表达中起SUMO化的作用[29]。SUMO通路在卵母细胞减数分裂[30]、转录活性调控、核质转运、细胞周期调控、DNA的复制与修复以及信号的转导[31]等方面发挥重要作用。戴燕彬[32]研究发现拟穴青蟹SUMO通路在卵巢的早期发育、配子发生过程亦起重要作用。预测的靶基因涉及以上通路,说明spa-miR-34在生殖方面可能起重要作用。
此外,spa-miR-34预测的靶基因还有蜕皮激素受体(EcR),它是蜕皮激素的作用靶标,因此,spa-miR-34可能也参与拟穴青蟹的蜕皮过程。
由于拟穴青蟹属非模式生物,数据库中的基因信息不足,无法进行更深入的挖掘,相信随着拟穴青蟹基因在数据库中的丰富,spa-miR-34的作用会获得更深入的阐释。通过对miR-34靶基因进行生物通路的分析,可以从调控网络层面挖掘miR-34的调控机制,看到基因层面看不到的信息,从而在调控层面更全面地了解miR-34的生物学功能,对了解miR-34对拟穴青蟹的调控机制奠定基础。但所预测的miR-34靶基因在拟穴青蟹中是否为miR-34的直接靶基因或间接靶基因需要进一步实验验证,目前本实验室正在进行相关的工作。
[1]Hwang H W,Mendell J T.MicroRNAs in cell proliferation,cell death,and tumorigenesis[J].British Journal of Cancer,2006,94(6):776-780.
[2]王天宇,董园园,李海燕,等.MicroRNAs的分子进化与调控机制[J].遗传,2010,32(9):277-279.
[3]高佳莉,罗玉萍,李思光.miR-34基因家族的分子进化[J].动物学研究,2007,28(3):271-278.
[4]Engkvist M E,Stratford E W,Lorenz S,et al.Analysis of the miR-34 family functions in breast cancer reveals annotation error of miR-34b[J].Scientific Reports,2017,7(1):9655.
[5]Hermeking H.The miR-34 family in cancer and apoptosis[J].Cell Death and Differentiation,2010,17(2):193-199.
[6]Slabáková E,Culig Z,Remšík J,et al.Alternative mechanisms of miR-34a regulation in cancer[J].Cell Death and Disease,2017,8(10):e3100.
[7]Liu N,Landreh M,Cao K,et al.The microRNA miR-34 modulates ageing and neurodegeneration in Drosophila[J].Nature,2012,482(7386):519-523.
[8]Aw S,Cohen S M.Time is of the essence:microRNAs and age-associated neurodegeneration[J].Cell Research,2012,22(8):1218-1220.
[9]Grimson A,Farh K K,Johnston W K,et al.MicroRNA targeting specificity in mammals:determinants beyond seed pairing[J].Molecular Cell,2007,27(1):91-105.
[10]Pulikkan J A,Peramangalam P S,Dengler V,et al.C/EBPα regulated microRNA-34a targest E2F3 during granulopoiesis and is down-regulated in AML with CEBPA mutations[J].Blood,2010,116(25):5638-5649.
[11]Mollinari C,Racaniello M,Berry A,et al.MiR-34a regulates cell proliferation,morphology and function of newborn neurons resulting in improved behaviouraloutcomes[J].Cell Death and Disease,2015,6(1):e1622.
[12]Ng W L,Chen G,Wang M,et al.OCT4 as a target of miR-34a stimulates p63 but inhibits p53 to promote human cell transformation[J].Cell Death and Disease,2014,5(1):e1024.
[13]Pu Y,Zhao F,Wang H,et al.MiR-34a-5p promotes multi-chemoresistance of osteosarcoma through down-regulation of the DLL1 gene[J].Scientific Reports,2017,7:44218.
[14]Yin H,Zhang S,Sun Y,et al.MicroRNA-34/449 targets IGFBP-3 and attenuatesairway remodeling by suppressing Nur77-mediated autophagy[J].Cell Death and Disease,2017,8(8):e2998.
[15]Freitas F C,Pires C V,Claudianos C,et al.MicroRNA-34 directly targets pair-rule genes and cytoskeleton component in the honey bee[J].Scientific Reports,2017,7:40884.
[16]王晓伟,张子平,王艺磊,等.虾蟹类卵巢发育调节机制研究进展[J].生物技术通报,2013,(7):29-35.
[17]Ma A N,Wang Y L,Zou Z H,et al.Erk2 in ovarian development of green mud crabScyllaparamamosain[J].DNA and Cell Biology,2012,31(7):1233-1244.
[18]马阿妮.ERK信号转导通路相关基因在拟穴青蟹卵巢发育中的作用研究[D].厦门:集美大学,2011.
[19]王晓伟.调控拟穴青蟹卵巢发育的眼柄转录组学分析[D].厦门:集美大学,2013.
[20]Zou Z H,Zhang Z P,Wang Y L,et al.EST analysis on the gonad development related organs and microarray screen for differentially expressed genes in mature ovary and testis ofScyllaparamamosain[J].Comparative Biochemistry and Physiology Part D:Genomics and Proteomics,2011,6(2):150-157.
[21]Han K H,Wang Y L,Zhang Z P,et al.Molecular characterization and expression profiles of cdc2 and cyclin B during oogenesis and spermatogenesis in green mud crab(Scyllaparamamosain)[J].Comparative Biochemistry and Physiology Part B:Biochemistry & Molecular Biology,2012,163(3-4):292-302.
[22]Dai Y B,Wang Y L,Zhang Z P,et al.SUMO-1 of mud crab(Scyllaparamamosain)in gametogenesis[J].Gene,2012,503(2):260-268.
[23]Gao J,Wang Y L,Zhang Z P,et al.Transcriptome analysis of the differences in gene expression between testis and ovary in green mud crab(Scyllaparamamosain)[J].BMC Genomics,2014,15(1):585.
[24]Zmora N,Trant J,Zohar Y,et al.Molt-inhibiting hormone stimulates vitellogenesis at advanced ovarian developmental stages in the female blue crab,Callinectessapidus1:an ovarian stage dependent involvement[J].Saline Systems,2009,5(1):7.
[25]Tiu S H K,Chan S M.The use of recombinant protein and RNA interference approaches to study the reproductive functions of a gonad-stimulating hormone from the shrimpMetapenaeusensis[J].The FEBS Journal,2007,274(17):4385-4395.
[26]马阿妮,王艺磊,张子平,等.MAPK 信号途径及其在水生无脊椎动物的研究进展[J].生命科学,2010,22(10):979-984.
[27]Devaraj H,Saravanakumar M,Thiyagu M.Induction of ovarian maturation inPenaeusmonodonby molecular signal interventional approach[J].Journal of Experimental Zoology Part B:Molecular and Developmental Evolution,2012,318(7):572-585.
[28]韩坤煌,张子平,王艺磊,等.Cyclin-CDK-CKI 及UPP 参与生殖调控及在甲壳动物性腺发育中的研究进展[J].生物技术通报,2010,(7):49-54.
[29]Ihara M,Stein P,Schultz R M.UBE2I( UBC9),a SUMO-conjugating enzyme,localizes to nuclear speckles and stimulates transcription in mouse oocytes[J].Biology of Reproduction,2008,79(5):906-913.
[30]袁一峰.SUMO-1在小鼠卵母细胞减数分裂中的作用及其分子机制研究[D].武汉:华中农业大学,2014.
[31]陈玲.Sumo信号通路对DAPK蛋白表达的调控[D].福州:福建师范大学,2016.
[32]戴燕彬.SUMO化信号通路相关基因在拟穴青蟹性腺发育过程中的作用研究[D].厦门:集美大学,2012.
PredictionandbioinformaticanalysisofmiR-34targetgenesinScyllaparamamosain
LIANG Mengmeng1,ZHANG Ziping2,JIA Xiwei1,WANG Yilei1*
(1.College of Fisheries,Jimei University,Xiamen 361021,China;2.College of Animal Science,Fujian Agriculture and Forestry University,Fuzhou 350002,China)
MiR-34 is a family of microRNAs that is highly conserved in evolution and is involved in many important life activities.In this study,the sequences characteristics between spa-miR-34 ofScyllaparamamosainfrom the database constructed by our laboratory and miR-34 of other animals from the miRBase were compared,and then the spa-miR-34 target genes were predicted by two online tools:RNAhybrid and Segal Lab.The functional annotation and signal pathway enrichment of these target genes were analyzed by using blast2go software.The results showed that the number of the predicted pa-miR-34 target gene was 66,and the functions of these genes are mainly distributed in the biological processes of oxidation-reduction,phosphorylation,calcium ion transmembrane transport,transcription,etc.Signal pathways related to these genes are mainly concentrated in the metabolism of arginine and proline metabolism,antibiotic biosynthesis,phenylpropanoid biosynthesis,glycolysis and glycogendysis,and other synthetic metabolic pathways.The predicted gene family and pathways of the spa-miR-34 target genes also include CHH family,ERK pathway,cell cycle and UPP pathway,and SUMO pathway.In addition,ecdysteroid receptor gene was also one of the predicted target genes.It is speculated that spa-miR-34 plays an important role in the reproduction,molt and the metabolism of amino acids and other substances.This study provides an important reference for further understand the role of spa-miR-34 inS.paramamosain.
miR-34;target genes;bioinformatics;Scyllaparamamosain
2017-11-15
国家自然科学基金(41676161,31672681,31472266).
梁萌萌(1988-),女,研究生,从事水产动物生殖生物学研究.E-mail:1638785935@qq.com
王艺磊(1963-),女,教授,从事水产动物生殖生物学研究.E-mail:ylwang@jmu.edu.cn
梁萌萌,张子平,贾锡伟,等.拟穴青蟹miR-34靶基因预测及其生物信息学分析[J].渔业研究,2017,39(6):419-428.
Q811.4
A
1006-5601(2017)06-0419-10

