青藏高原盐湖细菌群落与超盐环境因素的相关性
朱德锐 ,韩 睿 ,石 晴 ,沈国平 ,龙启福 ,双 杰 (.青海大学医学院,基础医学研究中心,青海 西宁8006;2.青海大学农林科学院,青海省蔬菜遗传与生理重点实验室,青海 西宁 8006)
基于不同离子的水质化学特征,盐环境可分为海盐环境和盐碱环境两种基本类型[1].海盐环境源自海水的蒸发,水体中离子组成与海水相似,pH近中性,主导盐分为NaCl,大多数已知的嗜盐微生物可适应性的生存其中.盐碱环境的离子成分与地质演化起源异于海盐环境(总溶盐>3.5%),主要类型是盐/碱湖泊,富含有Na+、高浓度的二价阳离子(Mg2+与 Ca2+)和碳酸盐,通常能反映当地的地质化学性质[2],可分为低盐盐湖(35~50g/L)、中盐盐湖(50~250g/L)和超盐盐湖(>250g/L)[3].通常,海水水体的平均盐度为 35.0g/L,NaCl成分约占 86.0%[4],而超盐盐湖的盐度极高,如死海(盐度 347.0g/L)、美国大盐湖(盐度333.0g/L)和非洲马加迪湖(盐度315.0g/L)等[5-7].
超盐环境是一类极其特殊的极端环境[8],作为一种陆生生态模型系统,由不同的极端条件组合可形成湿地超盐系统、干旱超盐系统、寒冷超盐系统和酸性超盐系统等[9].湿地超盐系统研究主要涉及盐井、盐场、盐池和盐湖水体等,多以Na+、K+、Ca2+、Mg2+、SO42-、Br-、Cl-、和 CO32-等离子的组合盐分为主要特征.许多嗜盐微生物,如古生菌、细菌、真核生物和嗜盐病毒均能在超盐环境中适应性生存[4,10].在总盐度、离子组成、温度、压力和营养状态等方面,不同的极端盐湖存在显著差异,致使嗜盐微生物的多样性、群落结构组成和分布亦存在明显不同[11].超盐环境微生物具有丰富的物种和遗传多样性,而这些微生物在极端盐湖生态系统的物质与能量循环以及生态演替中发挥重要的作用.
青藏高原极端盐湖资源丰富,分布着地球上面积最大(2×106km2)、海拔最高(平均 4500m 以上)、数量最多的超盐盐湖群[12].国内学者曾以青海湖、纳木错湖、普莫雍错湖和阿翁错盐湖等为研究对象[12-17],开展了湖泊微生物种群结构、多样性调查、放线菌/氨氧细菌资源收集以及极端微生物胞内次生代谢物(嗜盐菌素、抗生素、相容溶质和抗肿瘤活性物质)等不同方面的相关研究,然而针对极端盐湖细菌的群落结构差异与超盐环境因子的相关性研究,尚未深入开展.基于此,本研究以青藏高原 4个极端盐湖为研究对象,采用16S rRNA基因(V3~V4区)高通量测序方法分析盐境栖息细菌的群落结构差异和超盐环境因子的制约相关性,从而为后续的微生物资源利用和极端盐环境胁迫适应性研究提供重要的参考依据.
1 材料与方法
1.1 样品采集与处理
2015年 7月中旬采集察尔汉盐湖(95°18′55.72″E,36°50′7.02″N, 标 记 Q1/Q2/Q3/Q4)、茶卡盐湖(99°4′41.67″E,36°45′0.05″N,标记C1/C2/C3/C4) 、 柯 柯 盐 湖 (98°15′3.4″E,36°58′19.97″N,标记 K1/K2/K3/K4)和小柴旦盐湖(95°32′58.38″E,37°28′0.65″N,标记 X1/X2/X3/X4)的水样,附带盐湖底泥和盐结晶物,浑浊状态,水样采集深度 10~25cm,采样温度 20~27.7℃,各盐湖样点间距大于100m,共计16个样品.样本储存于 4℃车载冰箱,运回实验室.各样点理化性质(Na+、K+、Mg2+、Ca2+、Li+、Cl-、SO42-、CO32-和NO3-等离子)测定由上海微谱化工技术服务公司完成,离子检测标准参照离子色谱分析方法通则( JY/T 020-1996).
1.2 提取基因组DNA及16S rRNA基因PCR扩增
水样于 0.22μm 细菌滤膜上进行真空抽滤,将滤膜剪碎放入 DNA试剂盒(QI Aamp Fast DNA Stool Mini Kit,德国),参照试剂盒步骤提取基因组DNA,纯度检测分析采用Nanodrop 2000(美国Thermo公司).细菌采用16S rRNA基因通用引物[18]338F(5'-ACTCCTACGGGAGGCAGCA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')进行 V3~V4区目的基因扩增(美国 ABI Gene Amp 9700).PCR采用 TransStart Fastpfu DNA Polymerase,反 应 条 件 为 (20μL 体 系 ):95℃ ( 3min),94 ℃( 30s), 55℃ (30s),72℃ (45s),27个循环,最后在 72℃下延伸 10min.采用 Axyprep DNA Gel Extraction Kit(美国 Axygen scientific公司)进行PCR产物纯化,电泳检测.
1.3 测序分析与优化
采用Quanti Fluor™-ST蓝色荧光定量系统(Promega公司)进行DNA精准定量分析.检测合格的PCR纯化产物进行16S rRNA基因高通量测序(Illumina MiSeq 300PE),由上海美吉生物科技有限公司完成,每个样本获得双端序列数据为3~5万个Reads.根据测序Reads之间Overlap关系进行拼接(Merge)和质控过滤;根据序列首尾两端的 Barcode和引物序列区分,优化有效序列.使用 Usearch 7.1 软件(http://drive5.com/uparse/)对原始数据进行处理,在相似度 97%水平上聚类出可操作分类单元OTU picking.
1.4 群落结构分析
基 于 Mothur V.1.30.1软 件(http://www.mothur.org/wiki/Schloss)进行alpha稀释曲线、微生物群落的丰度和多样性分析,并计算菌群丰度Chao1与ACE指数、多样性Shannon-Wiener与Simpson指数以及测序深度Coverage指数.采用Qiime 平 台 (http://qiime.org/scripts/assign_taxonomy.html)和 RDP classifier贝叶斯算法(Version 2.2, http://sourceforge.net/projects/rdpclassifier/)进行OTU序列分类学分析,参照Silva数据库(Release 119, http://www.arb-silva.de),并在门Phylum、纲Class与属Genus分类水平统计每个样品的群落组成,观测样本在不同分类水平上的群落结构.
1.5 相似性聚类与环境因子相关分析
根据每个样本与 Silva或 RDP数据库(Release 11.3, http://rdp.cme.msu.edu/)的分类学比对结果,选出优势物种.采用R语言软件Vegan包,Vegdist和 Hclust进行距离计算和聚类分析,构建Heatmap图,距离算法为Bray-Curtis,聚类方法为 Complete.基于水质离子特征测定数据和OUT注释数据(Species和Sample数据,97%相似度),采用多元直接梯度回归分析菌群与环境因子之间关系.采用R软件语言Vegan包中RDA或者 CCA分析和作图,Bioenv函数判断环境因子与样本群落分布差异的最大 Pearson相关系数,Permutest分析来判断CCA分析的显著性.
1.6 数据提交
本研究所得细菌序列均已提交 NCBI数据库(http://www.ncbi.nlm.nih.gov/)中,BioProject 登录号为 PRJNA307208,BioSample登录号为SAMN04376877-SAMN04376892.
2 结果与分析
2.1 超盐环境样本水质特征
采用离子色谱法分析极端盐湖样本的水质离子特征(表1),表明环境样本 Q1/2/3/4的总盐度(TS)为 310.01~332.82g/L,样 本 K1/2/3 为279.97~327.26g/L,样 本 C2/3/4 为 315.81~325.17g/L,均属于超盐极端盐湖类型(>250g/L)[3],盐晶盐华富集,地区气候干燥、寒冷,具有综合性超盐系统的特征,离子组成类型亦与盐碱环境特征相一致[1].对照组样本 K4、C1和 X1/2/3/4的总盐度分别为 110.12,210.12,110.25~113.84g/L,属于中盐极端盐湖类型(50~250g/L)[3].对比分析,表明超盐环境样本组的总盐度明显高于对照组 X1/2/3/4、C1和 K4样本, 其中样本 C1和 K4因采样位点汇集淡水的补给,致使样本的TS、TOC、TN与盐湖饱和区样本存在显著差异.此外,水体的pH值和各种离子的浓度(Na+、K+、Mg2+、Ca2+、Cl-、SO42-与 CO32-),因样本的来源不同而有所差异.

表1 不同极端盐湖样本的离子特征参数Table 1 Ion characteristic parameters of salt lake in different samples
2.2 高通量测序数据统计
提取环境样本基因组 DNA,采用细菌引物338F和806R进行16S rRNA基因V3~V4区PCR扩增,构建Miseq文库进行Miseq 300PE平台测序,检测获得目的基因为 447bp.采用Trimmomatic软件完成数据优化与统计,获得细菌的总Sequences为532,615条,441~460bp长度序列分布比例为90.59%;在相似度97%水平上聚类可操作分类单元OUT,并进行OTU picking.利用Mothur做Rarefaction分析,制作稀释曲线(图1),随着测序序列数目的增加,测序样本的曲线基本趋向平坦,测序覆盖度为 99.41%~99.94%,表明测序数据渐进合理.
统计分析察尔汗盐湖、柯柯盐湖和茶卡盐湖超盐环境样本的 OTUs数目分别为 98~111、94~132 和 86~170,多样性 Shannon 指数为 2.26~2.56(表2).通常,超盐环境中的总盐度和水质化学特征对细菌的群落结构组成、多样性丰度和分布规律等具有重要的制约影响,盐度趋高则致使细菌的种类和多样性指数降低[19-20].Zhong等[13]采用高通量研究青藏高原7个湖泊的细菌多样性,其中获得超盐盐湖嘎斯库勒(盐度 317~ 344g/L)OUT为 347个,Shannon指数 3.45;托素盐湖(24~35g/L)OUT 为 1036 个,Shannon 指 数7.33.Simachew等[21]利用焦磷酸高通量测序分析Abijata Lake盐碱池,表明饱和盐晶区的OUT为19~42 个,Shannon 指数为 0.11~0.91.Tazi等[22]采用16S rRNA基因文库测序分析美国大盐湖(盐度 30%)的细菌多样性,获得 OUT为 42个,Shannon指数为 3.97.由此表明,超盐环境中细菌的群落结构复杂度和多样性显著低于非超盐环境,此与相关报道一致,即饱和盐度显著制约细菌的丰度比例.

图1 高通量测序稀释曲线Fig.1 Rarafaction curve of high-throughput sequencing

表2 超盐环境盐湖微生物的可观察物种和多样性分析Table 2 Statistical analysis of observed species and microbial diversity in the hypersaline lakes

续表2
2.3 超盐环境中细菌的群落结构与组成分析
通过在RDP和Silva数据库进行相似性比对和 OTUs注释,获得超盐环境中分类地位明确的细菌17门28纲121属,以及大量未确定分类地位的细菌3门6纲41属,并在门层次下对各样品的类群丰度和聚类层次进行分析(图2).盐湖超盐环境(总盐度>250g/L)中主要的细菌优势类群是Firmicutes (74.04%~81.04%),再次为Proteobacteria (15.51%~20.06%)、Bacteroidetes(2.68%~4.84%)和 Actinobacteria (0.71%~2.45%).相对于盐环境而言,超盐环境的细菌群落结构和微生物生态多样性趋于稳定,细菌类群的优势度明显,且样本类群组成和相似性亦趋于一致.随着总盐度的降低(如小柴旦盐湖X1/X2/X3/X4、C1和K4样点),Firmicutes的丰度比例急剧下降,而 Proteobacteria、Bacteroidetes、Cyanobacteria和 Actinobacteria类群的丰度比例则显著升高.

图2 超盐环境样本的群落结构组成及相似性特征Fig.2 Community compositions and similarity features of hypersaline environmental samples
超盐环境饱和卤水(>250g/L)中的主要栖息者是嗜盐古菌,然而亦发现大量的嗜盐或耐盐细菌亦生存其中[23].Abdallah等[24]采用 16S rRNA基因 PCR-DGGE分析吉利特盐湖(Chott El Jerid,346g/L)的细菌群落结构,表明优势类群依次是 Proteobacteria (α-、γ-和 δ-纲)、Firmicutes、Deinococcus-Thermus门和未分类细菌(Unclassified).Çınar 等[25]利 用 荧 光 原 位 杂 交(FISH)和 454焦磷酸测序分析土耳其安纳托利亚不同盐田(盐度 25%~27%)的细菌多样性,结果显示主要的细菌类群依次是 Bacteroidetes、Proteobacteria、Cyanobacteria、Actinobacteria、Firmicutes和Planctomycetes,而不同的盐境中,优势类群和丰度显著差异.Tazi等[22]采用 16S rRNA基因文库测序分析美国大盐湖(盐度为30%)的细菌多样性,聚类分析表明优势类群依次是 Proteobacteria、Firmicutes、Bacteroidetes-Chlorobi、Cyanobacteria、Deinococcus-Thermus、Actinobacteria、Aquificae、Spirochaetes 以及Chlamydiae.本研究与上述报道相比较而言,优势细菌类群存在显著差异,多以 Firmicutes为主,存在优势类群完全不一致[22,24-25],可能与盐湖的水化学特征有关.
厚壁菌门Firmicutes细菌,形态和生理特征高度多样化,栖息环境亦多元化,尤其是适应生存于极端超盐环境,主要代表类群是芽胞杆菌纲Bacilli的芽孢杆菌科Bacillaceae、动性球菌科 Planococcaceae和 葡 萄 球菌 科Staphylococcaceae,以及梭菌纲Clostridia的盐厌氧菌科 Halanaerobiaceae和盐拟杆菌科Halobacteroidaceae[4].Mesbah 等[26]采 用 16S rRNA基因文库测序分析埃及超盐盐湖 Wadi An Natrun(总盐 393.9g/L),表明生长优势类群Firmicutes在盐泥沉积层中的OTU丰度比例约为 34%~39%,其次 为 α-Proteobacteria(22%~27%).然而,青藏高原超盐盐湖(总盐度>300g/L)所有样本中,优势类群 Firmicutes的比例高达74.04%~81.04%,与上述超盐盐湖相比,具有显著差异.
2.4 超盐环境中细菌的优势种群分析
基于属层次分类统计分析超盐环境样本(前100种优势属群)的丰度比例和系统发育,并构建热图(图3).分析表明:超盐环境样本中细菌的优势属群进化相似性聚类明显,分为两个热度区域.高丰度区域主要类聚包括有 Bacilli纲中的Bacillus(50.63%~58.35%)、Lactococcus (9.28%~11.81%)、海洋芽孢杆菌属 Oceanobacillus(8.41%~10.52%)和环丝菌属Brochothrix (1.57%~2.17%);γ-Proteobacteria纲中的假单胞菌属Pseudomonas(9.15%~11.37%)和寡养单胞菌属Stenotrophomonas(3.17%~4.12%)、嗜冷杆菌属Psychrobacter(2.07%~2.90%);Acidobacteria纲中的节杆菌属 Arthrobacter(0.87%~2.36%)以及Flavobacteria纲中的香味菌属 Myroides(1.85%~2.60%).次丰度区域(≤1%)主要类聚有Lysinibacillus、Janthinobacterium、Solibacillus、Serratia、 Carnobacterium、 Hafnia、Chryseobacterium、Leuconostoc、Flavobacterium、Rahnella、Acinetobacter、Streptococcus 和Enhydrobacter.
青藏高原超盐环境中,优势种群 Bacillus主要包括有大量未确定属分类的潜在新种(Bacillus unclassified,49.97%~58.05%),其 次 是Bacillus oceanisediminis(0.21%~0.34%) 和Bacillus badius(<0.03%),与其他超盐环境盐湖的优势属群相比较,存在显著差异.通常情况下,Bacillus具有严格需氧或兼性厌氧的生理特性,具有荚膜,能够产生具有特殊抵抗力的芽孢,能良好生存于碱性或高盐极端环境中,常见于盐碱土壤或盐碱深层淤泥之中[4].
乳球菌属Lactococcus是第二大属群,属于青藏高原超盐环境中的特殊类群,涉及超盐环境盐湖的相关报道极少,主要的代表种群是Lactococcus piscium(6.19%~7.76%)和未确定属分类的潜在新种(3.15%~4.04%).Lactococcus细菌属于革兰氏阳性细菌,已有报道存在于鲜牛奶、活性污泥泡沫、动物(鳟鱼肠粘液)或植物(卷心菜)以及发酵食品之中[27].双鱼乳球杆菌 L.piscium属于嗜冷乳酸细菌,首次从红鳟鱼中分离发现,后陆续从各种食品中分离获得(如冷冻肉类和海产品)[28-29].Smith等[30]研究乳酸乳球杆菌L. lactis的耐盐生长能力,表明在3%~5%的盐浓度下,菌株生长良好,且能积聚甜菜碱抵抗盐渗透压的刺激变化.由此表明,Lactococcus细菌具备在极端寒冷和盐环境的生存能力,亦反映了Lactococcus细菌的存在与青藏高原超盐环境的相关性.
青藏高原超盐环境中第三大属群是海洋芽孢杆菌属 Oceanobacillus,主要代表有Oceanobacillus profundus(9.8%)和少量未确定分类的新种(<0.5%).Oceanobacillus细菌广泛存在于海洋、活性污泥、土壤、发酵食物、盐湖或超盐盐湖等各类环境之中[31],截止到目前共计有24个种分类(http://www.bacterio.net/oceanobacillus.html).Kim 等[32]曾从深海岩芯中分离获得Oceanobacillus profundus菌株 CL-MP28T,该细菌具有耐盐嗜碱的特性,好氧生长.本研究超盐环境中,Oceanobacillus profundus多有存在且丰度较高,相比较其他超盐盐湖的细菌多样性研究,则鲜有报道.

图3 超盐环境样本的优势属群Heatmap聚类分析(前100属)Fig.3 Heatmap cluster analysis of dominant genera in hypersaline samples (the top 100genera)
2.5 超盐环境样本的进化相似性及差异性分析
基于样本两两之间的比较分析获得Unifrac距离矩阵(图4A),评估微生物群落差异的显著性以及进行样品群落组成 Unifrac PCoa差异分析(图4B).相对于盐环境样本X1/2/3/4、C1和K4而言,超盐环境样本Q1/2/3/4、K1/2/3和C2/3/4的矩阵距离较小,进化类聚趋同效应明显.PCoa分析表明:在OTU的丰度水平上,超盐环境样本间的细菌群落结构差异较小.

图4 超盐环境样本间的Unifrac距离矩阵与差异性分析Fig.4 Unifrac distance matrix and difference analysis of different hypersaline samples
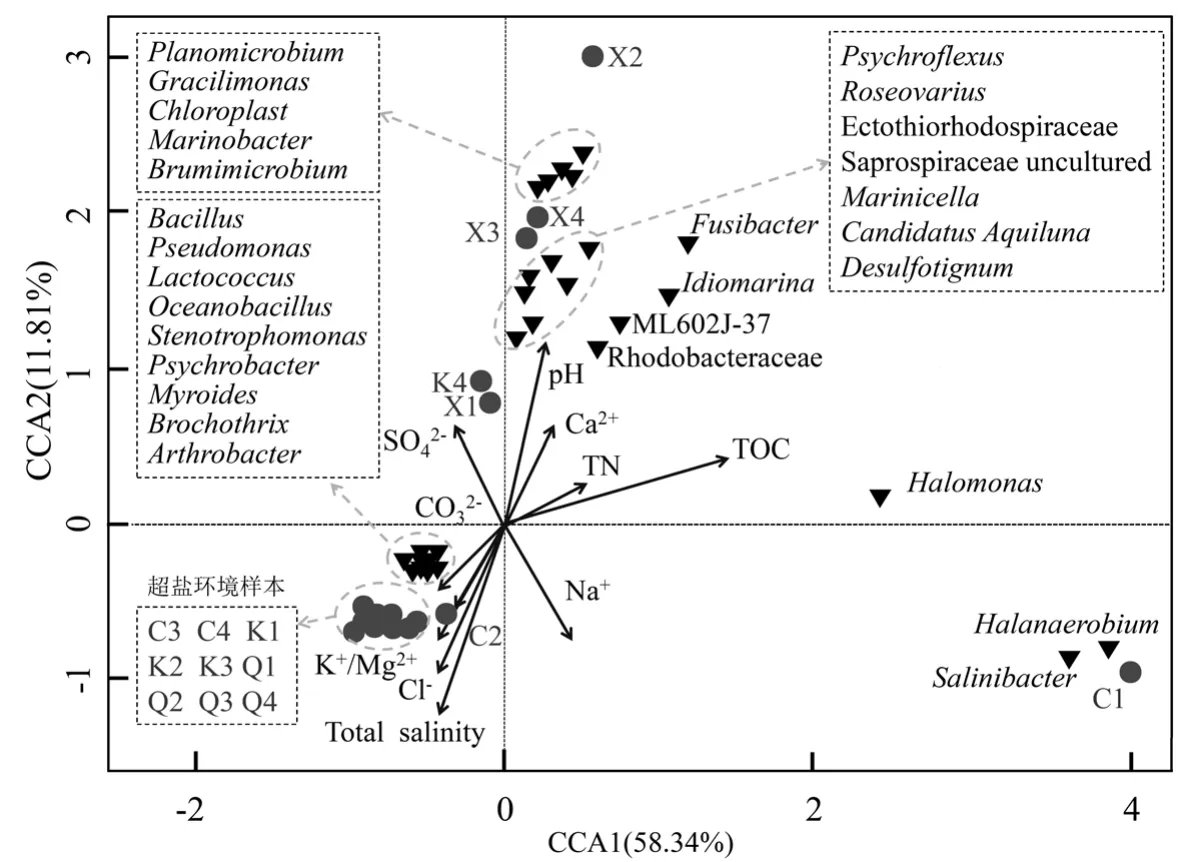
图5 超盐环境因子与细菌群落结构相关性CCA分析Fig.5 Canonical Correspondence analysis of bacterial community structure and hypersaline environment factors
2.6 超盐环境因子相关性分析
基于水质离子特征测定数据和 Speciessample丰度数据,采用多元直接梯度回归分析样本、菌群与环境因子之间关系,并构建典范对应CCA分析(图5).超盐环境样本Q1/2/3/4、K1/2/3和 C2/3/4聚集在第三象限,样本的属性值相似,与环境因子 TS、Cl-、K+、Mg2+和 CO32-的垂线投影点相近,表明环境因子的制约影响作用最强.通过分析超盐环境中优势微生物的类群分布,结果显示 Bacillus、Pseudomonas、Lactococcus、Oceanobacillus、 Stenotrophomonas、Psychrobacter、Myroides、Brochothrix 和Arthrobacter聚集,与环境因子 TS、Cl-、Mg2+、K+和 CO32-呈锐角态势毗邻,正相关作用显著,尤其是受到 TS、Cl-和 Mg2+浓度因素的制约影响(TS>Cl->Mg2+>K+>CO32-).
诸多研究表明,许多嗜盐微生物能在宽泛的盐度浓度范围内适应生存,如大多数盐单胞菌科Halomonadaceae细菌具备低盐环境生存能力,同时亦可在饱和盐度中生存.盐境中嗜盐细菌的多样性和分布特征与水体的离子组成有关,其中NaCl是其主要因素,某些情况下还受制于二价阳离子(Mg2+和 Ca2+)的影响[4].然而就相对总盐度而言,高原超盐样本与嘎斯库勒盐湖(盐度317~344g/L)研究[13]相一致,但其他离子浓度因素的制约影响又具有独特性.
非超盐样本X1/K4受到pH值、SO42-和Ca2+环境因子的影响制约最大,而样本C1和X2/3/4受到环境因子的制约影响作用较小,且优势菌属多聚集于第一象限,与 pH 值、Ca2+、TN、SO42-和TOC等环境因子呈正相关.Yuan等[33]研究沙漠盐湖细菌群落的垂直分布差异时,发现菌群分布随取样的深度不同,环境制约的关联因子亦不同,其中Marinobacter和Halomonas与NO3-浓度呈正相关,以及 Thioalkalivibrio与溶解氧呈负相关而与SO42-浓度呈正相关.Zhong等[13]研究青藏高原 7个中低度盐湖的多样性时,发现 TS和离子浓度(K+、Cl-、Na+、SO42-和 Ca2+)直接或间接的制约细菌的群落结构组成、多样性分布以及个体的进化分支.因此,在不同的盐境中,细菌优势类群组成和特征分布的主要驱动因素各有所差异,可能与优势类群的代谢方式和群体适应生存机制有关.
3 结论
3.1 青藏高原超盐盐湖(总盐度>300g/L)的细菌群落结构组成和优势属群相对稳定,与其它超盐环境相比,存在显著差异,而且群落结构复杂度和多样性显著低于非超盐环境.
3.2 青藏高原超盐环境盐湖中主要的细菌优势类群依次是 Firmicutes(74.04%~81.04%),Proteobacteria(15.51%~20.06%)、Bacteroidetes (2.68%~4.84%)和Actinobacteria(0.71%~2.45%).
3.3 高原超盐环境样本中细菌优势属群的同源聚类明显,分为两个热度区域,其中高丰度区域为高原特色优势属群,依次是 Bacillus(50.63%~58.35%)、Lactococcus(9.28%~11.81%)和 Oceanobacillus(8.41%~10.52%).
3.4 高原超盐环境样本的属性值基本一致,同源类聚明显,进化趋同保守,各样品间的微生物群落结构无明显差异.优势属群依次是 Bacillus、Pseudomonas、Lactococcus、Oceanobacillus、Stenotrophomonas、Psychrobacter、Myroides、Brochothrix和Arthrobacter,其群落组成与聚集分布与超盐环境因子(TS、Cl-、Mg2+和K+浓度)呈正相关关系,即超盐环境因子积极影响优势微生物的类群分布和适应生存.
[1] Oren A. Life at High Salt Concentrations [J]. Prokaryotes,2006,4(5):421—440.
[2] Maheshwari D K, Saraf M. Halophiles [M]. Switzerland: Springer International Publishing, 2015:355—357.
[3] Sorokin D Y, Berben T, Melton E D, et al. Microbial diversity and biogeochemical cycling in soda lakes [J]. Extremophiles Life Under Extreme Conditions, 2014,18(5):791—809.
[4] Horikoshi K, Antranikian G, Bull A T, et al. Extremophiles Handbook [M]. Japan: Springer International Publishing, 2011:256—400.
[5] Boutaiba S, Hacene H, Bidle K A, et al. Microbial diversity of the hypersaline Sidi Ameur and Himalatt Salt Lakes of the Algerian Sahara [J]. Journal of Arid Environments, 2011,75(10):909—916.
[6] Kjeldsen K U, Loy A, Jakobsen T F, et al. Diversity of sulfate-reducing bacteria from an extreme hypersaline sediment,Great Salt Lake (Utah) [J]. FEMS Microbiology Ecology, 2007,60(2):287—298.
[7] Oren A, Baxter B K, Weimer B C. Microbial communities in salt lakes: phylogenetic diversity, metabolic diversity, and in situ activities [J]. Natural Resources & Environmental Issues, 2009,15(1):1—7.
[8] Ventosa A, Oren A, Ma Y. Halophiles and Hypersaline Environments [M]. Berlin: Springer International Publishing,2011:48—49.
[9] Oren A. Halophilic archaea on Earth and in space: growth and survival under extreme conditions [J]. Philosophical Transactions of the Royal Society, 2014,372(2030):1—12.
[10] Ghai R, Pašić L, Fernández AB, et al. New abundant microbialgroups in aquatic hypersaline environments [J]. Scientific Reports,2011,1(9):135—145.
[11] Ventosa A, Fernández A B, León M J, et al. The Santa Pola saltern as a model for studying the microbiota of hypersaline environments [J]. Extremophiles, 2014,18(5):811—824.
[12] Liu Y, Priscu J C, Yao T, et al. A comparison of pelagic, littoral,and riverine bacterial assemblages in Lake Bangongco, Tibetan Plateau [J]. FEMS Microbiology Ecology, 2014,89(2):1—11.
[13] Zhong Z P, Liu Y, Miao L L, et al. Prokaryotic community structure driven by salinity and ionic concentrations in plateau lakes of the Tibetan Plateau [J]. Applied & Environmental Microbiology, 2016,82(6):1846—1858.
[14] Yun J, Ju Y, Deng Y, et al. Bacterial community structure in two permafrost wetlands on the Tibetan Plateau and Sanjiang Plain,China [J]. Microbial Ecology, 2014,68(2):360—369.
[15] Xiong J, Liu Y, Lin X, et al. Geographic distance and pH drive bacterial distribution in alkaline lake sediments across Tibetan Plateau [J]. Environmental Microbiology, 2012,14(9):2457—2466.
[16] Jiang H, Huang Q, Deng S, et al. Planktonic actinobacterial diversity along a salinity gradient of a river and five lakes on the Tibetan Plateau [J]. Extremophiles, 2010,14(4):367—376.
[17] Hu A, Yao T, Jiao N, et al. Community structures of ammoniaoxidising archaea and bacteria in high-altitude lakes on the Tibetan Plateau [J]. Freshwater Biology, 2010,55(11):2375—2390.
[18] Yu Y, Lee C, Kim J, et al. Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction [J]. Biotechnology & Bioengineering,2005,89(6):670—679.
[19] Demergasso C, Escudero L, Casamayor EO, et al. Novelty and spatio-temporal heterogeneity in the bacterial diversity of hypersaline Lake Tebenquiche (Salar de Atacama) [J].Extremophiles, 2008,12(4):491—504.
[20] Oren A. Molecular ecology of extremely halophilic Archaea and Bacteria [J]. FEMS Microbiology Ecology, 2002,39(1):1—7.
[21] Simachew A, Lanzén A, Gessesse A, et al. Prokaryotic community diversity along an increasing salt gradient in a Soda Ash concentration pond [J]. Microbial Ecology, 2016,71(2):326—338.
[22] Tazi L, Breakwell D P, Harker A R, et al. Life in extreme environments: microbial diversity in Great Salt Lake, Utah [J].Extremophiles, 2014,18(3):525—535.
[23] Ali I, Prasongsuk S, Akbar A, et al. Hypersaline habitats and halophilic microorganisms [J]. Maejo International Journal of Science and Technology, 2016,10(3):330-345.
[24] Abdallah M B, Karray F, Mhiri N, et al. Prokaryotic diversity in a Tunisian hypersaline lake, Chott El Jerid [J]. Extremophiles,2016,20(2):125—138.
[25] Çınar S, Mutlu M B. Comparative analysis of prokaryotic diversity in solar salterns in eastern Anatolia (Turkey) [J].Extremophiles, 2016,20(5):589—601.
[26] Mesbah N M, Abou-El-Ela S H, Wiegel J. Novel and unexpected prokaryotic diversity in water and sediments of the alkaline,hypersaline lakes of the Wadi An Natrun, Egypt [J]. Microbial Ecology, 2007,54(4):598—617.
[27] Odamaki T, Yonezawa S, Kitahara M, et al. Novel multiplex polymerase chain reaction primer set for identification of Lactococcus species [J]. Letters in Applied Microbiology, 2011,52(5):491—496.
[28] Marché L, Saraoui T, Remenant B, et al. Complete genome sequence of Lactococcus piscium CNCM I-4031, a bioprotective strain for seafood products [J]. Genome Announcements, 2017,5(4):1—2.
[29] Saraoui T, Leroi F, Björkroth J, et al. Lactococcus piscium: a psychrotrophic lactic acid bacterium with bioprotective or spoilage activity in food-a review [J]. Journal of Applied Microbiology, 2016,121(4):907—918.
[30] Smith W M, Dykes G A, Soomro A H, et al. Current Research,Technology and Education Topics in Applied Microbiology and Microbial Biotechnology [M], Spain: Formatex Research Center Publisher, 2010:1106—1118.
[31] Amoozegar M A, Bagheri M, Makhdoumi A, et al.Oceanobacillus halophilus sp. nov. a novel moderately halophilic bacterium from a hypersaline lake [J]. International Journal of Systematic & Evolutionary Microbiology, 2016,66(3):1317—1322.
[32] Kim Y G, Choi D H, Hyun S, et al. Oceanobacillus profundus sp.nov. isolated from a deep-sea sediment core [J]. International Journal of Systematic & Evolutionary Microbiology, 2007,57:409—413.
[33] Yuan W, Su X, Cui G, et al. Microbial community structure in hypolentic zones of a brine lake in a desert plateau, China [J].Environmental Earth Sciences, 2016,75(15):1—14.

