甜樱桃栽培种指纹图谱构建及遗传多样性分析
王丹丹,付化瑞,张彦文(. 辽东学院 农学院,辽宁丹东 8003;.北票市林业局,辽宁北票 00)
甜樱桃栽培种指纹图谱构建及遗传多样性分析
王丹丹1,付化瑞2,张彦文1
(1. 辽东学院 农学院,辽宁丹东 118003;2.北票市林业局,辽宁北票 122100)
为了对甜樱桃品种进行分子鉴别及分析不同品种间的亲缘关系,以叶片直接进行PCR扩增,采用EST-SSR分子标记技术构建生产上常用的40个甜樱桃栽培品种的数字指纹图谱和模式指纹图谱数据库(Cluster Project)并进行遗传多样性分析。采用24对甜樱桃核心引物对所有品种基因组进行扩增,共检测到129个等位位点,包括118个多态性位点,多态性比率为91.2%。每对引物扩增出的等位位点数3~10个,每对引物平均扩增5.38个基因型,扩增条带大小介于100~500 bp,多态信息含量(PIC)为0.654,基因流(Nm)为1.402,平均杂合率为0.495。8对引物,即PA17、PA32、PA51、PA54、PA73、PA99、PA101和PA202可作为指纹图谱构建的首选引物,利用其中的PA17、PA51、PA99、PA101和PA202以引物组合方式构建指纹图谱可区分40个品种;聚类分析表明在遗传距离0.71处40个品种被分为5类,聚类结果与品种的特性及成熟期具有较高的一致性。
甜樱桃;EST-SSR标记;指纹图谱;遗传多样性;聚类分析
欧洲甜樱桃(PrunusaviunLinn.)又名大樱桃、甜樱桃,隶属于蔷薇科(Rosaceae)李亚科(Prunoideae)李属(Prunus),果粒大、颜色鲜艳、营养丰富、酸甜可口,经济价值高[1];原产于西亚及欧洲东南部,19世纪末、20世纪初引入中国,由于甜樱桃冬季休眠期需7.2 ℃以下低温处理900~1 400 h,而中国长江以南绝大部分地区不能满足这一条件,所以甜樱桃以江北栽培为主,西南高地有少量发展,但品质远不如西北黄土高原产区,总体上,中国甜樱桃在布局上呈现低纬度高海拔、高纬度低海拔分布态势,初步形成山东、辽宁、陕西三大产区,安徽、江西、河北、甘肃、新疆、山西、北京、四川、重庆、云南、贵州、青海等多省(市)局部有所发展的产业布局,经过多年不断繁衍扩展,目前全国种植总面积超过10万hm2,甜樱桃年产量达60余万t,已成为中国水果的重要组成成分,甜樱桃的种植成为典型的高效产业之一,在中国方兴未艾,具有蓬勃发展之势[2-8]。20世纪80、90年代后,由于甜樱桃生产价值较高,各地纷纷引种,呈现适宜区全面开花式发展,种植范围、生产规模持续扩大,进入快速发展阶段,但大都忽略种质资源的保护和优良品种的选育,且个别种植户采收母本的同时人为掺杂父本或其他品种,导致种质真实性及纯度受到严重影响;此外,异物同名、同物异名、变种以及遗传倍性等问题,使甜樱桃市场处于混乱状态,给育种业和种植业造成损失。甜樱桃杂交种的真实性和纯度是其制种面临的关键问题,也是难关[9]。中国甜樱桃种类繁多,每年还会有新品种发布,然而,目前还没有一套适合鉴定其种质真实性和纯度的可靠方法。因此,利用分子标记技术构建甜樱桃DNA指纹图谱对其种质的快速鉴定、新品种选育以及种内亲缘关系的研判等具有重要意义,对规范种植业市场、维护种植者的权益也具有指导性意义。
品种真实性和纯度鉴定的方法主要有形态学鉴定法、生物化学法及分子标记鉴定法等。因受材料本身与环境条件的影响,前两者已很少作为单一的评判标准。由于EST-SSR(Expressed Sequence Tag-Simple Sequence Repeats)分子标记为共显性、扩增谱带少、易识别、统计方便,以及EST来自DNA编码区,其序列保守性相对较高等优点,已成为品种真实性和纯度鉴定的有效方法[10]。中国已建立多种植物的指纹图谱以鉴定种质的真实性和纯度,如《水稻品种鉴定DNA指纹法》NY/T1433-2007、《玉米品种鉴定DNA指纹法》NY/T1432-2007等。利用EST-SSR分子标记构建甜樱桃DNA指纹谱图的研究尚未见报道。本研究中,采用EST-SSR分子标记技术建立甜樱桃DNA指纹谱图,为其种质资源的真实性和纯度鉴定奠定基础。同时,在研究过程中以叶片直接进行PCR扩增,简化试验程序、优化PCR体系。
1 材料与方法
1.1 试验材料
采用常规种植的40个具有不同产地的甜樱桃品种为材料(表1),由大连旅顺、烟台、北京等甜樱桃研究机构或甜樱桃品种引进机构提供。春季或夏季采集生长旺盛的健康、幼嫩叶片于硅胶中干燥保存,以备提取基因组DNA[11]。试验分析工作在辽东学院农学院进行。
1.2 EST-SSR引物的开发
从美国国家生物技术信息中心(National Center for Biotechnology Infoemation,NCBI)EST数据库下载甜樱桃的所有EST序列,分别利用Blastclust、est_timmer、misa.pl和primer 3批量设计EST-SSR引物;用Blast对所开发的引物进行比对验证[12-13],最后引物由生工生物工程(上海)股份有限公司合成。
1.3 叶片的碱处理
将叶片剪碎,大小不宜超过2 mm2,否则不利于后续的PCR扩增。将叶片放入含有40 μL 0.25 mol·L-1NaOH的Eppendorf管中,沸水煮30 s;加入40 μL 0.25 mol·L-1HCl和20 μL 0.5 mol·L-1Tris-HCl(pH 8.0,含有φ=0.25% Nonidet-P-40),再次置沸水中2 min,随后取出叶片,直接进行PCR扩增[12-16]。
1.4 EST-SSR PCR扩增
在50 μL PCR反应体系加入1片经碱处理的叶片。反应液组成为:dNTPs (2.5 mmol·L-1) 4 μL,引物(10 μmol·L-1)各2 μL,TaqDNA聚合酶(5 U·μL-1)0.5 μL,MgCl2(1.5 mmol·L-1) 3 μL,10×缓冲液5.0 μL,加灭菌ddH2O至总体积50 μL。PCR反应程序:95 ℃预变性5 min;95 ℃变性50 s,59 ℃或60 ℃退火50 s,72 ℃延伸50 s,35个循环;72 ℃延伸10 min,产物于4 ℃保存[17-21]。扩增产物采用6%非变性聚丙烯酰胺凝胶电泳分离、银染和图谱分析[22]。所用的24对引物是从笔者已开发的300余对EST-SSR引物中筛选得到的,具有多态性高、稳定性好等特点(表2)。引物由生工生物工程(上海)股份有限公司合成。
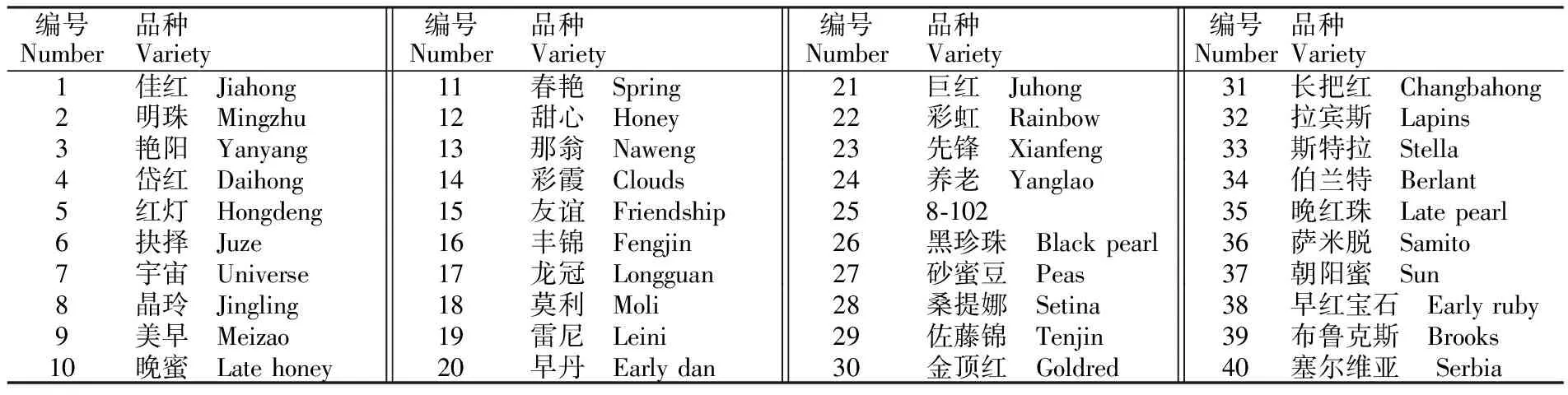
表1 40种甜樱桃栽培品种Table 1 The 40 varieties of sweet cherry
1.5 数据处理
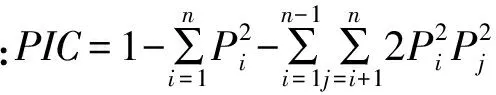
2 结果与分析
2.1 EST-SSR引物扩增
利用24对引物对40份甜樱桃进行PCR扩增,部分引物扩增图见图1,共获得129个等位位点,包括118个多态性位点,多态性比率为91.2%。每对引物扩增出的等位位点数在3~10个,每对引物平均扩增5.38个基因型,扩增的条带大小介于100~500 bp。多态信息含量(PIC)是表示微卫星DNA变异程度高低的一个指标,反映微卫星DNA多态性高低,其取决于检测的等位基因的数目和该基因的基因频率分布,一般来说,PIC>0.5,多态性较高;0.25≤PIC≤0.5多态性适中;PIC<0.25,多态性较低。而本研究24对EST-SSR引物的平均PIC值为0.654(0.500~0.807,见表2),表现出较高的多态性;基因流(Nm)平均值为1.402(0.496~3.128,见表2),FST平均值为0.180(0.074~0.335,P<0.05,见表2)。
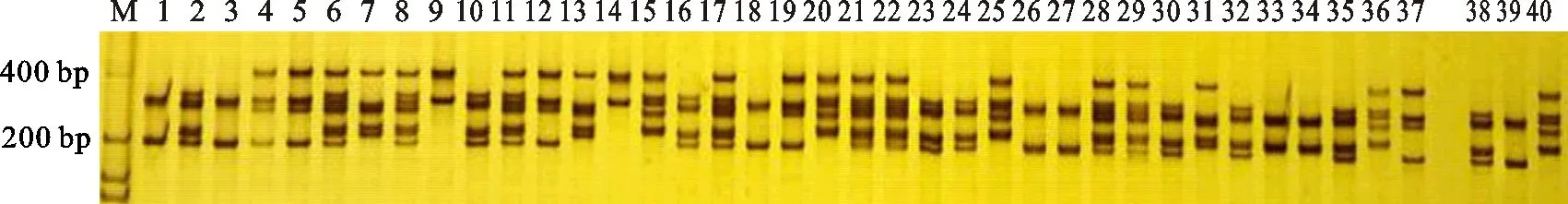
M.DM 2 000 DNA marker;1~40.品种编号 The numbers of the 40 varieties
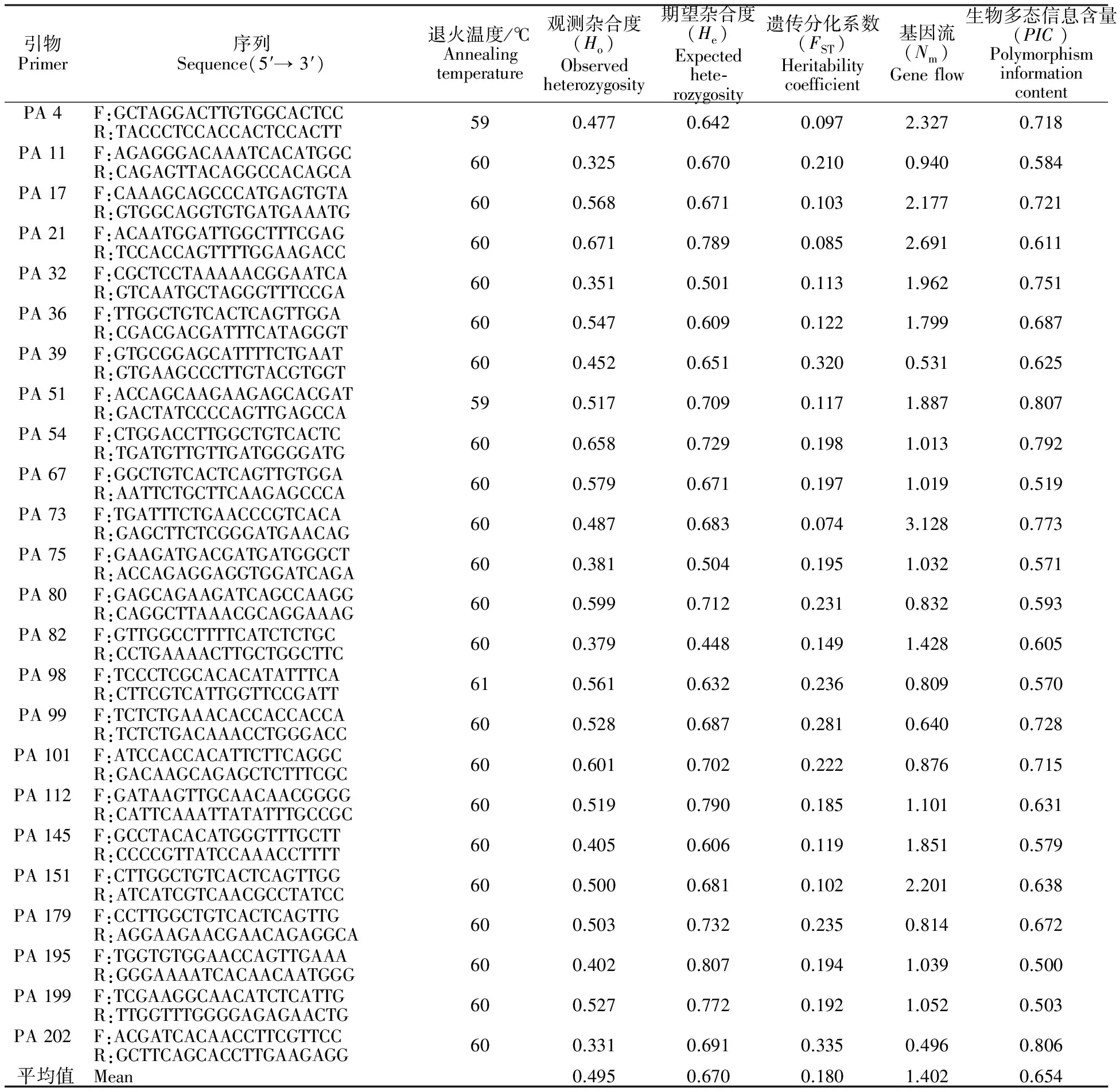
表2 EST-SSR引物在40份供试材料中的扩增情况Table 2 The amplification of EST-SSR primers in 40 varieties
2.2 EST-SSR指纹图谱的构建
24对引物中有8对引物具有区别不同品种的能力,即PA17、PA32、PA51、PA54、PA73、PA99、PA101和PA202(表3)。PA17区别品种最多为1、6、9、11、14、16、17、20、21、26、28、30、34、37和40,其中2、5、8、18、23、31、33、38和39带型一致;3、7、10、12、13、24、27、29和36带型一致;15、19、22和25带型一致;32和35带型一致。
根据8对引物的多态性,采用特殊引物法可鉴定40份甜樱桃品种,其余5种不能鉴定,即5、10、18、25和32。因此,采用引物组合法只需5对EST-SSR引物(即PA17、PA51、PA99、PA101和PA202)便可完全区分40份甜樱桃品种(指纹图谱见表4)。采用ClusterProject软件对构建的指纹图谱数据进行分析,形成指纹模式图谱(图2),由此,以2种EST-SSR指纹分析方式共同建立甜樱桃的指纹图谱数据库。
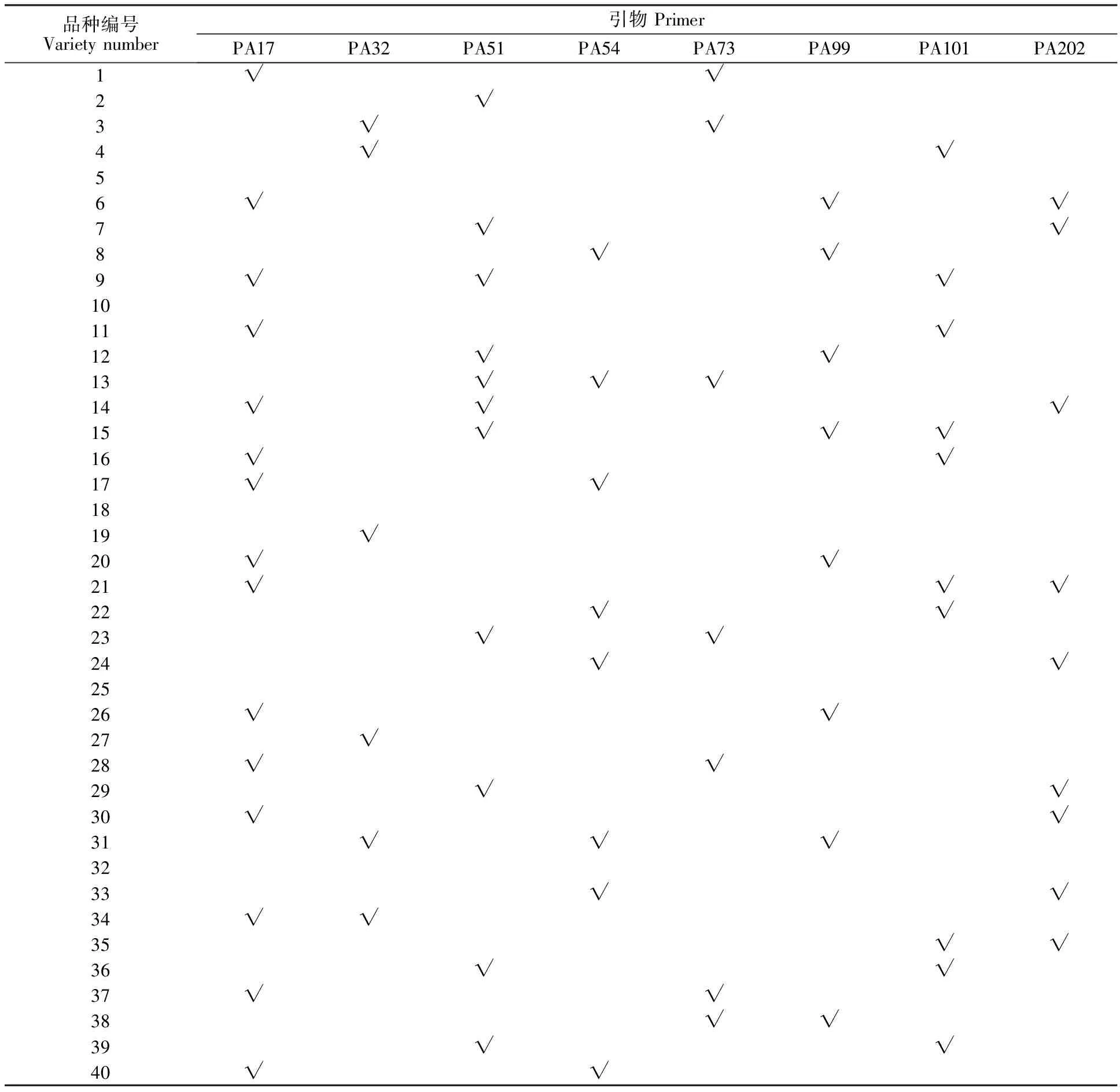
表3 基于8对EST-SSR引物区分40份甜樱桃品种的能力Table 3 The ability to distinguish 40 sweet cherry varieties based on 8 pairs of EST-SSR primers
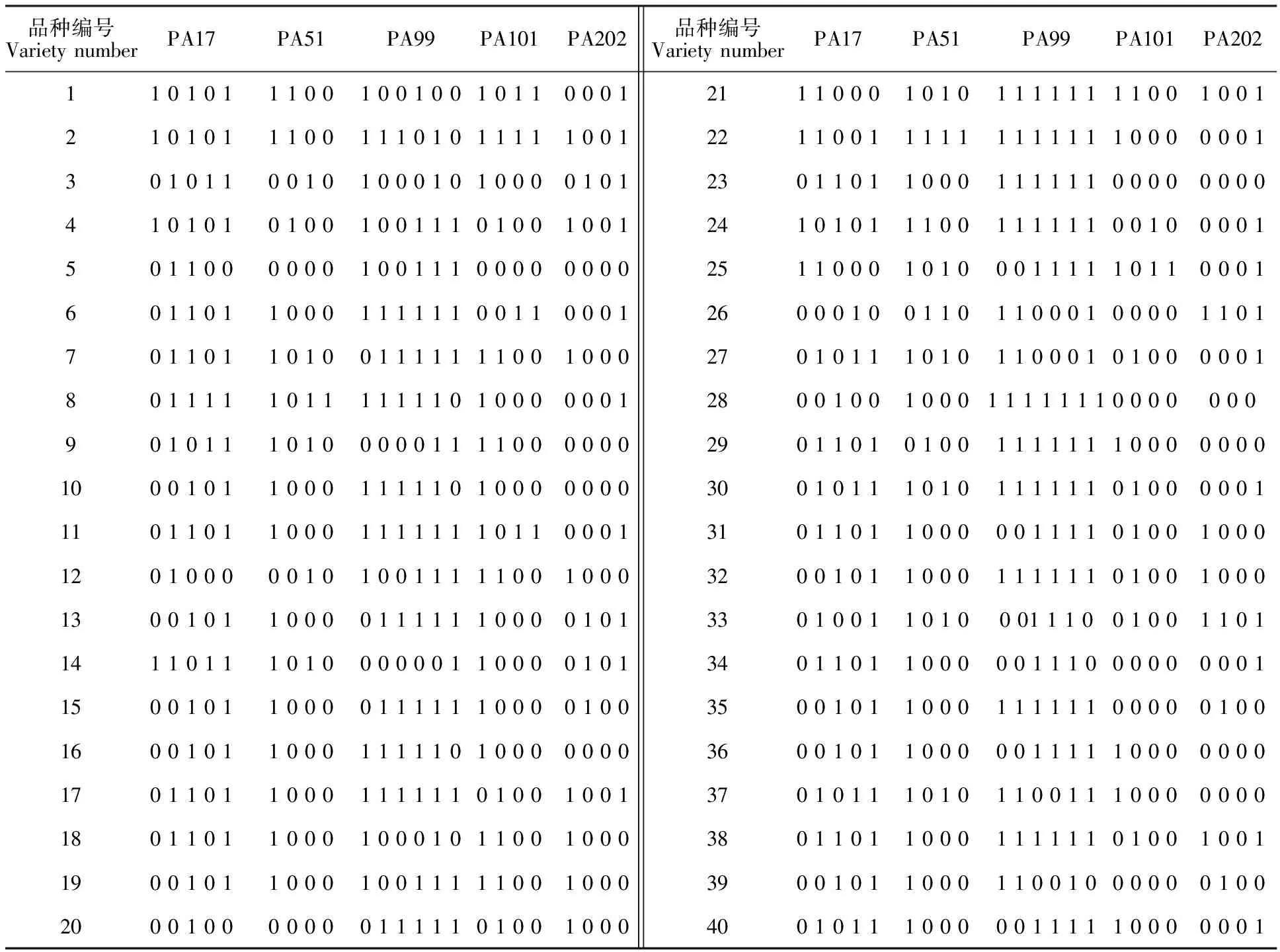
表4 甜樱桃品种的DNA数字指纹图谱Table 4 The digital fingerprinting of sweet cherry varieties

1~40.品种编号 The numbers of the 40 varieties
2.3 聚类分析
采用UPGMA进行聚类分析,构建40份甜樱桃品种的遗传关系树状图(图3)。40份甜樱桃品种被划分为5个类群,聚类结果与品种的形态特征及来源具有较高的一致性,即形态特性相似和来源于同一地区的品种聚在一个类群种,第Ⅰ类群有7个品种,包括‘友谊’‘金顶红’‘养老’’黑珍珠’‘伯兰特’‘桑提娜’‘塞尔维亚’,该类群品种果型均为心脏型,果皮深红色,且均晚熟;第Ⅱ类群有8个品种,包括‘莫利’‘8-102’‘艳阳’‘长把红’‘砂蜜豆’‘朝阳蜜’‘抉择’‘晚红珠’,大部分品种果型鸡心型或肾型,果皮洋红色,早熟;第Ⅲ类群有7个品种,即‘先锋’‘萨米脱’‘美早’‘宇宙’‘龙冠’‘岱红’‘拉宾斯’,大部分果型为近圆形,中早熟品种,果皮多为浓红色至紫红色,果肉红色或紫红色;第Ⅳ类群有‘晶玲’‘甜心’‘红灯’‘彩霞’‘早红宝石’‘布鲁克斯’‘早丹’7个品种聚在一起,果型圆形、多为早熟品种,果皮紫红色,果肉红色至紫红色;第Ⅴ类群包括11个品种,有‘佳红’‘明珠’‘晚蜜’‘春艳’‘那翁’‘丰锦’‘佐锦藤’‘斯特拉’‘雷尼’‘巨红’‘彩虹’,果型宽心脏型,果皮黄色或黄色带红晕,果肉黄色。
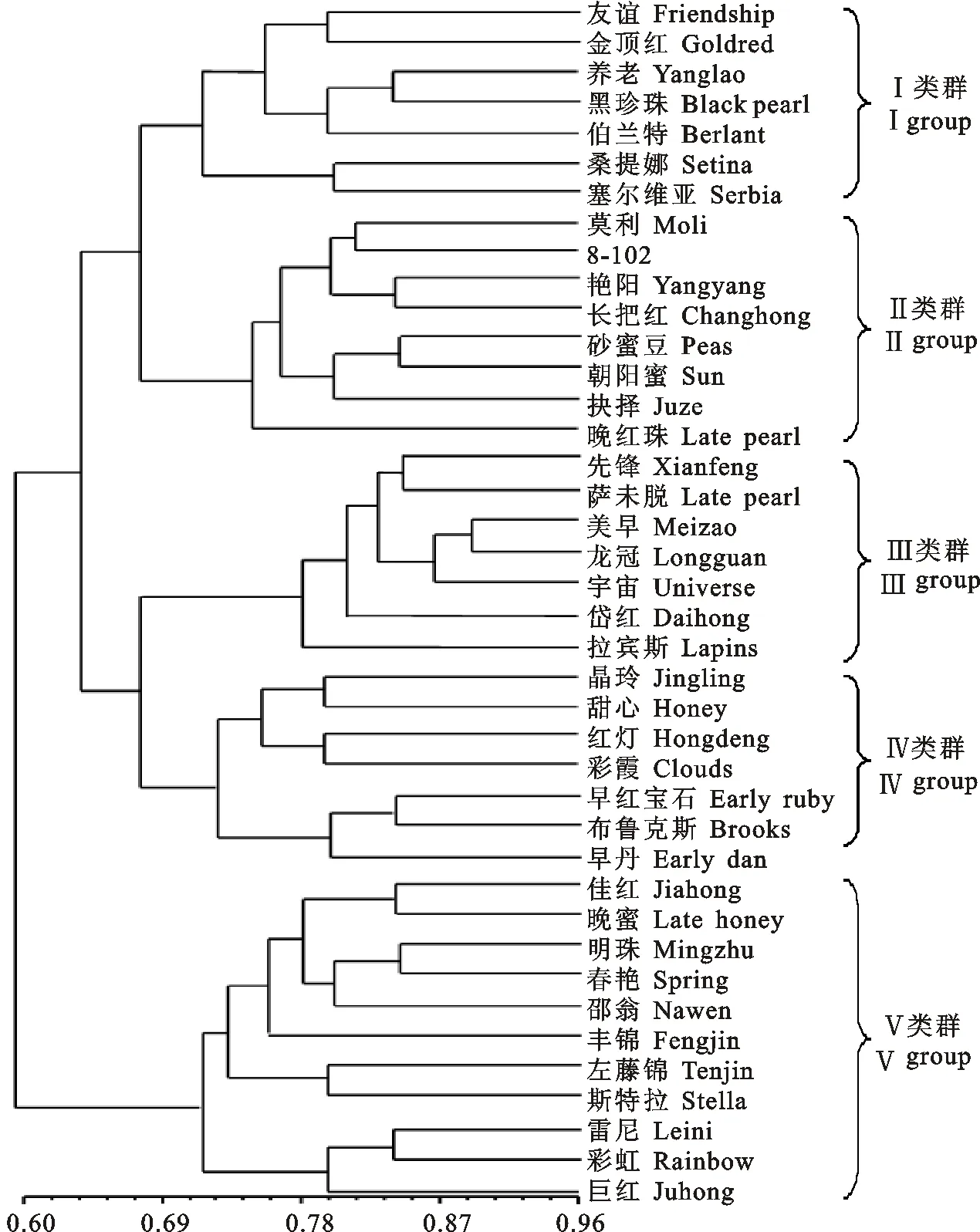
图3 40份甜樱桃品种聚类图Fig.3 The dendrogram of 40 sweet cherry cultivars
3 讨 论
本研究的40个甜樱桃品种在遗传距离0.71处被划分为5类,大部分以品种形态特征和成熟季节进行的类别划分。此外,同一产地的品种聚在一起的较为普遍,比如由大连农科院选育的新品种‘佳红’‘巨红’和‘明珠’3个品种聚为一类,说明目前生产上同一地区反复使用同一授粉系或者不育系杂交配种的现象较普遍,导致群体内部基因流较大(Nm=1.402)。观测杂合度(Ho=0.495)都略低于期望杂合度(He=0.670),性状相似现象亦较为普遍,造成同一地区遗传基础较差。此外,有些国产品种和国外品种聚在一起,亲缘关系较近,说明国际合作育种工作已有成效。为了避免中国甜樱桃品种的遗传基础出现狭窄现象,未来有必要加强国内外合作育种或杂交配育更加优良的甜樱桃品种。
目前,很多国家审定新品种都需要DUS证明新品种的特性与现有品种不同,而部分品种仅是育种者提供品种,经过区域试验后,对于达到要求的品种即可申请为新品种,而对于新品种的描述也只限于形态学的特征,造成品种同物异名和异物同名等现象。利用EST-SSR为基础的分子标记技术具有明显的优势,如不含内含子,在各组织、各发育时期均可检测到,且不受季节、环境等因素的限制;数量之多可覆盖整个基因组;多态性高,成共显性,能够鉴别出纯合、杂合基因型等,即使品种间形态学差异较小,只要在遗传基础上仍有差异,EST-SSR就可发挥作用。
本研究对供试的40个甜樱桃品种,采用叶片直接进行PCR扩增,简化PCR程序、筛选出适合鉴定真实性和纯度的5对核心引物,构建现有甜樱桃种质的DNA指纹数字图谱和EST-SSR指纹模式图谱数据库,指纹图谱数据库的构建对未来解决甜樱桃出现的品种纠纷提供服务。随着甜樱桃新品种的不断出现,指纹图谱构建工作需不断加强,要大量筛选新的甜樱桃EST-SSR核心引物,为新品种做指纹图谱,以保证中国甜樱桃产业健康有序地繁荣发展。
Reference:
[1] 艾呈祥,张力思,魏海蓉,等.甜樱桃品种SSR指纹图谱数据库的建立[J].农业生物技术学报,2007,23(5):55-58.
AI CH X,ZHANG L S,WEI H R,etal.Construction of molecular fingerprinting database for sweet cherry using SSR markers [J].JournalofAgriculturalBiotechnology,2007,23(5):55-58(in Chinese with English abstract).
[2] SEANNA H,BENJAMIN K,RAMYYA H,etal.Evaluation of multiple approaches to identify genome-wide polymorphisms in closely related genotypea of sweet cherry(PrunusserotinaL.) [J].ComputationalandStructuralBiotechnologyJournal,2017,15:290-298.
[3] 蔡宇良,李 珊,曹东伟,等.利用DNA扩增片段序列对樱桃种质资源的遗传分析[J].园艺学报,2006,33(2):249-254.
CAI Y L,LI SH,CAO D W,etal.Use of amplified DNA sequence for the genetic analysis of the cherry germplasm [J].ActaHorticulturaeSinica,2006,33(2):249-254(in Chinese with English abstract).
[4] 艾呈祥,刘庆忠,李国田,等.甜樱桃品种SSR指纹图谱的构建[J].山东农业科学,2010(4):8-10.
AI CH X,LIU Q ZH,LI G T,etal.Establishment of fingerprint for sweet cherry cultivars using SSR markers [J].ShandongAgriculturalSciences,2010(4):8-10(in Chinese with English abstract).
[5] DOYLE J.A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J].PhytochemicalBulletin,1987,19(5):11-15.
[6] CHAT J,JAUREGUI B,PETIT R J,etal.Reticulate evolution in kiwifruit(Actinidia,Actinidiaceae) identified by comparing their maternal and paternal phylogenies[J].AmericanJournalofBotany,2004,91(6):736-747.
[7] 陈晓流,陈学森,束怀瑞,等.15个樱桃品种的RAPD分析[J].果树学报,2004,21(6):556-559.
CHEN X L,CHEN X S,SHU H R,etal.RAPD analysis of 15 cherry cultivars [J].JournalofFruitScience,2004,21(6):556-559(in Chinese with English abstract).
[8] ELLIS J R,PASHLEY C H,BURKE J M,etal.High genetic diversity in a rare and endangered sunflower as compared to a common congener[J].MolecularEcology,2006,15(2):2345-2355.
[9] DOWNEY S L,LEZZONI A F.Polymorphic DNA markers in black cherry(PrunusserotinaL.) are identified using sequences from sweet cherry,peach,and sour cherry [J].JournaloftheAmericanSocietyHorticculturalScience,2000,125(1):76-80.
[10] ZHANG L,YUAN M,HE X,etal.Development and universality evaluation of EST-SSR makers from GenBank in jute [J].ActaAgronomicaSinica,2014,19(5):11-15.
[11] STRUSS D,BORITZKI M,GLOSSER K,etal.Detection of genetic diversity among populations of sweet cherry(PrunusserotinaL.) by AFLP [J].JournaloftheHorticulturalScience&Biotechnology,2001,76(3):362-367.
[12] STANYS V,BANIULIS S,MORKUNAITE J B,etal.Characterizing the genetic diversity of Lithuanian sweet cherry(PrunusserotinaL.) cultivars using SSR markers [J].ScientiaHorticulturae,2009,142(13):136-142.
[13] DIRLEWANGER E,COSSON P,TAVAUD M,etal.Develpoment of microsatellite markers in peach [Prunusserotina(L.) Batsch] and their use in genetic diversity analysis in peach and sweet cherry(PrunusserotinaL.) [J].TheoreticalandAplliedGenetics,2002,105(1):127-138.
[14] PROHENS J,MUNOZ-FLACON J E,RODRIGUEZ-BU RRUEZO A,etal.H15 an Almzgro-type pickling eggplant with high yield and reduced prickliness [J].Hortscience,2009,44:2017-2019.
[15] HAY M J,SHARP P J.Targeted development of informative microsatellite(SSR) markers [J].NucleicAcidsResearch,2001,29(8):44-47.
[16] INCE A G,KARACA M,ONUS A N.Polymorphic microsatellite markers transferable across species [J].PlantMolecularBiologyReporter,2010,28(2):285-291.
[17] LATOCHLA P.The comparison of some biological features ofActinidiaargutacultivars fruit [J].AnnalsofWarsawUniversityofLifeSciences-SGGWHorticultureandLandscapeArchitecture,2007,28(3):105-109.
[18] 段艳凤,刘 杰,卞春松,等.中国88个马铃薯审定品种SSR指纹图谱构建与遗传多样性分析[J].作物学报,2009,35(8):1451-1457.
DUAN Y F,LIU J,BIAN CH S,etal.Construction of fingerprinting and analysis of genetic diversity with SSR markers for eighty-eight approved potato cultivar(SolanumtuberosumL.)[J].ActaAgronomicaSinica,2009,35(8):1451-1457(in Chinese with English abstract).
[19] 王彩虹,田义柯,赵 静,等.樱桃品种资源间遗传差异的RAPD分析[J].西北植物学报,2005,25(12):2431-2435.
WANG C H,TIAN Y K,ZHAO J,etal.RAPD analysis of the genetic differences among several cherry varieties [J].ActaBotanicaBoreali-OccidentaliaSinica,2005,25(12):2431-2435(in Chinese with English abstract).
[20] MAZZUCATO A,FICCADENTI N,CAIONI M.Genetic diversity and distinctiveness in tomato(SolanumlycopersicumL.) landraces:the Italian case of a pera Abruzzese [J].ScientiaHorticulturae,2010,125(1):55-62.
[21] CASTRO I,MARTIN J P,ORTIZ J M,etal.Varietal discrimination and genetic relationships ofVitisviniferaL.cultivars from two major controlled appellation(DOC) regions in Portugal [J].ScientiaHorticulturae,2011,127(3):507-514.
[22] PETIT R J,DUMINIL J,FINESCCHI S,etal.Comparative organization of chloroplast,mitochondrial and nuclear diversity in plant populations [J].MolecularEcology,2005,14(1):689-701.
[23] RAIGON M D,RODRIGUEZ-BURRUEZO A,PROHE NS J.effects of organic and conventional cultivation methods on composition of eggplant fruits [J].JournalofAgriculturalandFoodChemistry,2010,58(1):6833-6840.
[24] ZHANG L W,CAI R R,YUAN M H,etal.Genetic diversity and DNA fingerprinting in jute(Corchorusspp.) based on SSR markers [J].TheCropJournal,2015,3(5):416-422.
[25] GUNARS L,ISAAK R,SILVIJA,etal.Assessment of genetic diversity of Latvian and Swedish sweet cherry(PrunusserotinaL.) genetic resources collections by using SSR markers[J].ScientiaHorticulturae,2009,121(4):451-457.
[26] NAVASCUES M,EMERSON B C.Chloroplast microsatellites:measures of genetic diversity and the effect of homoplasy [J].MolecularEcology,2005,14(5):1333-1441.
[27] ZONG X J,WANG W W,WEI H R,etal.A multiplex RT-PCR assay for simultaneous detection of four viruses from sweet cherry [J].ScientiaHorticulturae,2014,180:118-122.
[28] 王红意,翟 红,王玉萍,等.30个中国甘薯主栽品种的RAPD指纹图谱构建及遗传变异分析[J].分子种植育种,2009,7(5):879-884.
WANG H Y,ZHAI H,WANG Y Petal.RAPD fingerprints and genetic variations of the 30 main sweetpotato varieties in China [J].MolecularPlantBreeding,2009,7(5):879-884(in Chinese with English abstract).
[29] WU G,XU X,XU J,etal.Singh,Multi-locus DNA fingerprinting and genetic diversity in jute(Corchorusspp.) based on sequence-related amplified polymorphism [J].JournalofPlantBiochemistryBiotechnology,2012,22(1):1-8.
EstablishmentofDNAFingerprintingandAnalysisofGeneticDiversityamongPrunusaviunCultivars
WANG Dandan1,FU Huarui2and ZHANG Yanwen1
(1.College of Agriculture, Eastern Liaoning University, Dandong Liaoning 118003, China;2.Forestry Burean of Beipiao,Beipiao Liaoning 122100,China)
In order to identify the cultivars ofPrunusaviunand analyze the genetic relationship among different cultivars at a molecular level, we constructed the digital fingerprint and model fingerprint(ClusterProject)for 40 commonPrunusaviuncultivars using EST-SSR(Expressed Sequence Tag-Simple Sequence Repeats) molecular markers. The 24 pairs of primers amplified a total of 129 alleles including 118 polymorphic alleles with a polymorphism rate of 91.2%. Each pairs of primers can amplify 3-10 alleles, with an average of 5.38 alleles per locus. The length of amplified products was 100-500 bp, with the mean value ofPIC(Polymorphism information content) being 0.654,Nm(Gene flow) being 1.402, and the mean value ofHo(Heterozygosity) being 0.495. Among the 24 primers, 8 primers can be used as a preferred choice for fingerprinting, namely PA17, PA32, PA51, PA54,PA73, PA99,PA101 and PA202, in which 5 primers(PA17, PA51, PA99, PA101 and PA202) can be employed in combination to identify the 40 cultivars. Cluster analysis showed that the 40 cultivars can be divided into five groups at a genetic distance of 0.71; the genetic relationship as indicated by the cluster completely reflected the characteristics and the maturation stage of the cultivars.
Prunusaviun; EST-SSR markers; Fingerprinting; Genetic diversity; Cluster analysis
2017-06-05
2017-10-30
The National Natural Science Fund(No.31370400); Dandong Project of Science and Techenology in 2016 (No.2016KJ001); Eastern Liaoning University Foundation Youth Fund Project(No.2015QN006).
WANG Dandan,female,lecturer.Research area:biochemistry and molecular biology.E-mail:wangdandansq@sina.com
郭柏寿ResponsibleeditorGUOBaishou)
日期:2017-12-21
网络出版地址:http://kns.cnki.net/kcms/detail/61.1220.S.20171221.1650.012.html
2017-06-05
2017-10-30
国家自然科学基金(31370400);丹东市2016科技攻关(2016KJ001);辽东学院青年基金(2015QN006)。
王丹丹,女,硕士,讲师,从事分子生物学研究。E-mail:wangdandansq@sina.com
Q663.4
A
1004-1389(2017)12-1813-08

