土壤甲烷氧化菌群落结构研究进展
曹淑贞, 沈媛媛, 王风芹, 宋安东, 桑玉强
(1. 河南农业大学 生命科学学院, 郑州 450002; 2. 河南农业大学 林学院, 郑州 450002)
土壤甲烷氧化菌群落结构研究进展
曹淑贞1, 沈媛媛2, 王风芹1, 宋安东1, 桑玉强2
(1. 河南农业大学 生命科学学院, 郑州 450002; 2. 河南农业大学 林学院, 郑州 450002)
CH4气体具有极强的红外线吸收能力,单分子CH4造成的增温效应可以达到单分子CO2的15~30倍,更是远远高于其他温室气体。地表甲烷氧化和释放对整个生态系统的物质循环和动态平衡意义重大,近年来科学家围绕土壤甲烷氧化细菌做了大量研究工作,取得进展的同时也存在诸多问题。以甲烷氧化菌为出发点,总结目前国内外关于土壤甲烷氧化的研究进展,重点介绍了甲烷氧化菌群落结构研究的各种分子生物学方法,旨在推进对土壤氧化甲烷菌作用机制的研究,完善引起温室效应的生物学因素,为进一步对地表土壤甲烷通量的分析和研究提供理论依据。
甲烷氧化细菌;菌落结构;土壤
CH4作为重要的温室气体,对全球变暖效应的贡献率为20%,仅比第一大温室气体CO2低了35%[1]。工业革命使得煤、石油等化石能源的消耗剧增,与此同时大气中的CH4浓度也在日益升高。据调查显示,大气甲烷浓度已在过去200年内增加了约1.48倍,目前仍以较高的速度逐年增加[2]。甲烷氧化研究已成为研究全球变暖效应不可或缺的重要环节。
陆地土壤对CH4的氧化和释放影响着大气中的甲烷含量。对于单一系统,甲烷吸收大于甲烷氧化,称为甲烷吸收汇,反之则为甲烷排放源。近年来,大气中甲烷浓度急剧升高就是吸收汇减少和排放源增加的结果。土壤中含有大量的甲烷氧化细菌,研究表明不饱和土壤在大气甲烷排放源与吸收汇的平衡中起着重要作用,CH4的年平均摄取量可达到36 Tg±23 Tg[3]。自然因素和人类活动都密切影响着土壤中甲烷氧化菌对甲烷的释放和吸收,研究不同条件地表土壤中甲烷氧化菌的生态多样性对分析甲烷氧化机制和净甲烷排放核算都具有深远的意义。
1 森林甲烷氧化菌
1.1 甲烷氧化菌分类
甲烷氧化细菌(Methanotrophic bacteria)属于革兰氏阴性菌,广泛存在于土壤、水体等各种生态环境中,其最大的特点是能够以甲烷作为唯一的碳源和能量维持生存[4]。甲烷氧化菌分为好氧甲烷氧化菌(Aerobic Methanotrophs)和厌氧甲烷氧化菌(Anaerobic Methanotrophs)两种,其中厌氧甲烷氧化菌能够在缺氧或无氧环境中氧化甲烷,多为古生菌,大多在极地、热泉等极端条件下被发现,本文的主要探讨对象为好氧型甲烷氧化细菌。
好氧型甲烷氧化菌于19世纪初被荷兰微生物学家Sohngen分离出来[5],直到1970年才开始被科学家进行分类,奠定了现代甲烷氧化菌分类学的基础[6]。甲烷氧化菌分为3个菌门,分别为变形菌门(Proteobacteria)、疣微菌门(Verrucomicrobia)[7]和NC10门[8]。疣微菌门只在极端嗜酸嗜热环境下发现过,NC10门细菌可以在厌氧条件下进行甲烷和亚硝酸盐耦合反应,又叫反硝化厌氧甲烷氧化菌,传统的甲烷氧化菌都归类为变形菌门。传统好氧甲烷氧化细菌氧化甲烷的代谢途径也存在差异,其中TypeⅠ型菌利用单磷酸核酮糖途径氧化代谢甲烷,Type II型菌则利用丝氨酸途径。Ⅰ型甲烷氧化菌被划分为γ-变形菌纲,又可以细分为Ⅰa型和Ⅰb型,其中Ⅰb型菌又名X型甲烷氧化菌[4]。X型甲烷氧化菌代谢甲烷的途径与TypeⅠ型菌相同,但其和I型甲烷氧化菌还是存在一定差异:X型菌含有少量的丝氨酸代谢途径酶,最适生长温度高于其他类型的甲烷氧化细菌,并且具有大多数Ⅰ型菌缺乏的固氮能力[9]。Ⅱ型甲烷氧化菌属于α-变形菌纲,包括Methylocystaceae和Beijerinckiaceae两个科,前者有Methylocystis和Methylosinus属,后者包括Methylocapsa、Methylocella和Methyloferula属。
不同类型的甲烷氧化细菌具有不同的环境耐受能力。TypeⅠ甲烷氧化菌偏向于高氧浓度低含量甲烷以及营养物质丰富的环境,而Type II型菌则完全相反。关于Ⅰ型和Ⅱ型的具体分类参见表1。

表1 甲烷氧化菌分类
1.2 甲烷氧化机理
土壤中的甲烷氧化菌首先利用细胞内的关键酶甲烷单加氧酶(MMO)将大气中的CH4氧化为甲醇。不同类型的甲烷氧化菌所利用的甲烷单加氧酶也存在一定的差别:Ⅰ型菌只能利用可溶性甲烷单加氧酶(sMMO),而TypeⅡ甲烷氧化菌既可同时利用可溶性甲烷单加氧酶(sMMO)和颗粒性甲烷单加氧酶(pMMO)。两种酶的作用机理都是破坏甲烷分子C-H键,最终生成甲醇(CH3OH)和水。后续反应中,产生的甲醇被苹果酸脱氢酶(MDH)氧化成甲醛,甲醛的代谢过程是TypeⅠ菌和TypeⅡ菌的最重要区别,前者利用磷酸核酮糖途径氧化甲醛,后者则利用的是丝氨酸途径。该过程反应见图1。
2 甲烷氧化菌标记基因
传统的检测甲烷氧化菌群落结构的方法都是依靠培养法实现,操作复杂、工作量大、不能一次性实现大批样品的快速检测等使得该方法存在许多缺点[10]。1995年,Amann等研究人员通过相关实验证实了传统的培养方法可能会错失99%以上的微生物种类,这使得人们开始开拓创立更为前沿的生物多样性研究技术[11]。
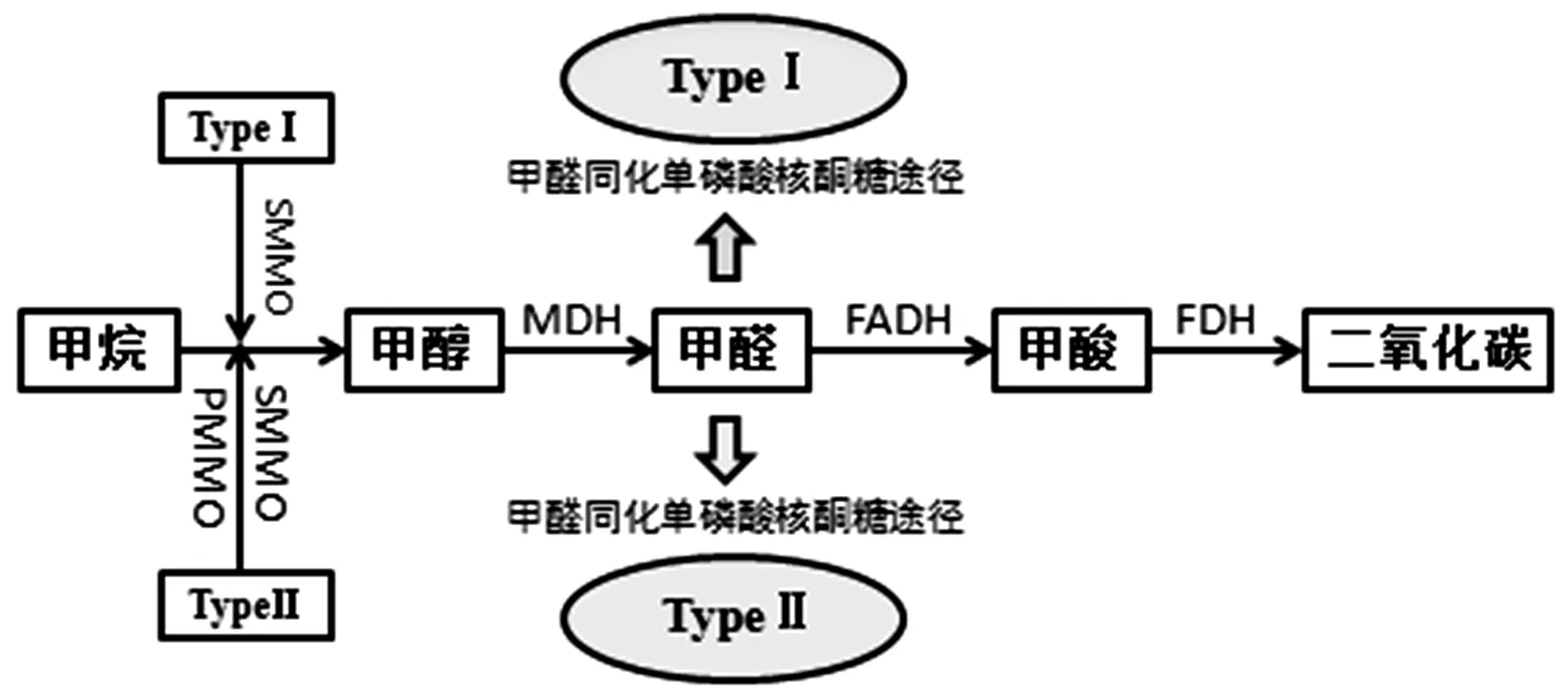
图1 甲烷氧化途径[4]
随着分子生态学的发展,分子检测法逐渐取代了传统的甲烷氧化细菌检测方法,采用标记基因法测定微生物群落结构的方法已得到广泛应用。标记基因是细菌内能够起特异性标记作用的一种基因,通常具有已知的功能和序列。16s rRNA基因作为最常用的标记基因,负责编码细菌内特定的DNA序列,是微生物多样性研究的重要工具[12]。特殊情况下,需要对具有特定功能的微生物进行研究,此时需要更为专一负责编码关键酶的功能基因作为研究对象[13]。
pmoA是甲烷氧化细菌的功能基因,在关键酶甲烷单加氧酶(MMO)的编码中发挥着不可替代的作用。因此,环境中甲烷氧化菌的多样性研究常选pmoA作为标记基因[14]。继寡核苷酸引物A189f/A682r之后[15],人们又设计出更多pmoA的特异性引物来实现该功能基因的PCR扩增。除了pmoA,其他能编码甲烷单加氧酶的功能基因也相继被发现,共同组成甲烷氧化菌生态学研究的强有力工具[16]。
3 甲烷氧化菌群落结构的分子生物学测定方法
近年来分子生物学分析群落结构的技术被广泛应用,包括限制性片段长度多态分析(Terminal restriction fragment length polymorphism, T-RFLP)、实时荧光定量PCR (Real-time PCR, RT-PCR)、高通量基因芯片(GeoChip)、磷酸脂肪酸分析(Phospholipid fatty acid, PLFA)、荧光原位杂交(Fluorescence in situ hybridization, FISH)、变性梯度凝胶电泳(Denaturing gradient gel electrophoresis, DGGE)以及高通量测序技术(High throughput sequencing, HTS)。这些技术的应用拓展了土壤甲烷氧化菌等微生物多样性的研究手段,以下对目前已应用的土壤甲烷氧化菌群落结构的分子生物学研究方法做相应总结。
3.1 限制性片段多态性分析
限制性片段多态性分析(T-RFLP)是研究微生物多样性的一种高效便捷的手段。该技术以分子系统学为基础,克服了传统微生物培养方法造成的物种缺失弊端,不但应用快速,而且具有较高的灵敏度,如今已被广泛应用到微生物种群多样性的研究分析领域[17]。该技术测定甲烷氧化菌菌落结构的原理是用荧光标记土壤样品中的甲烷氧化菌功能基因片段,通过DNA测序仪进行电泳和荧光检测,分析带有荧光标记基因片段的长度和含量,从而揭示土壤氧化菌的丰度和菌落结构等特征。Singh等[18]以T-RFLP技术研究了新西兰的原始山毛榉林,发现该林区土壤中typeⅡ型甲烷氧化菌是最主要的活跃种群;Lüke等[19]运用该技术研究了意大利某地多种水稻根际甲烷氧化菌群落结构,结果证明typeⅡ和typeⅠb甲烷氧化菌比其他甲烷氧化菌在水稻根际土壤中更占优势;Ho等[20]采用T-RFLP与qPCR结合的技术研究了反复性的干旱和洪涝干扰对水稻田土壤中甲烷氧化菌活性、丰度以及菌落结构的影响,结果证明甲烷氧化菌对抗外界干扰有一定的自我恢复能力,但不同类型的甲烷氧化菌的抗受能力不同,typeⅠ型菌在不利干扰条件下相比typeⅡ型菌有更强的自我恢复能力,这同时表明不同的甲烷氧化菌采用不同的策略来应对外界干扰作用。
3.2 实时荧光定量PCR
实时荧光定量PCR作为一种高灵敏度、强特异性、高自动化的新兴分子生物学研究技术[10],可以结合16s rRNA 基因或功能基因作为标记来检测环境样品中微生物多样性[21]。该技术的原理是将过量的具有荧光作用的染料(如SYBR Green I)加入到传统的PCR反应体系,荧光染料可以特异性结合DNA双链从而对其进行标记,PCR的整个反应进程就可以通过荧光累积实时进行监测控制,最后通过标准曲线定量计算分析。杨铭德等[10]利用实时荧光定量PCR 技术,研究探讨了不同盐碱程度下土壤甲烷氧化菌活性与甲烷氧化速率的关系;刘雅慈等[21]在传统荧光定量PCR 技术的基础上,结合甲烷氧化细菌功能基因pmoA,探索建立了快速、灵敏和特异性的油气田土壤甲烷氧化菌定量 PCR 检测技术;Yun等[22]运用该技术研究了青藏高原若尔盖湿地3种典型植被条件下甲烷氧化菌丰度、活性以及群落结构的差异,其中甲烷氧化菌的16s rRNA以及pomA基因拷贝量均显示出湿地环境中typeⅠ型菌在数量上占有优势,而mRNA/DNA的结果又表明typeⅡ型菌在该地区虽然丰度较低,但在所有样品中活性最高。
3.3 高通量基因芯片
高通量基因芯片技术(GeoChip)研究物种多样性的原理是将高密度的DNA 探针通过原位合成或微印刷的方法固化于载体的表面,探针进一步与样品DNA进行杂交,杂交点信号可用激光扫描仪进行采集,最后通过软件分析处理对生物样品进行快速有效地检测和医疗诊断。Martineau等[23]通过基因芯片与克隆文库结合的方法研究了加拿大高纬度北极圈土壤甲烷氧化情况,结果发现了在极地土壤中有许多未被研究报道的甲烷氧化细菌。Wang等[24]为探讨低水位及氮沉降对青藏高原高寒湿地的影响,运用基因芯片技术(Geochip 5.0)对土壤中温室气体相关微生物(甲烷氧化菌、产甲烷菌等)进行研究,结果表明温室气体的排放影响着土壤中的相关微生物结构,在探求温室气体释放对环境影响的同时不能忽视对微生物机制的研究。如今,微阵列芯片技术已作为甲烷氧化菌多样性研究的一个重要手段被广泛应用[19]。
3.4 基因组高通量测序
基因组高通量测序(HTS),也称下一代测序技术(Next generation sequencing, NGS),其原理是将待测样品基因长链打断成短片段并在两端加上接头,然后使这些单个的小片段DNA分子结合在固相表面实现单分子的独立扩增,最后通过高分辨率的成像系统检测分析荧光信号,实现基因组测序的目的[25]。科研人员以16s rRNA为标记基因研究了新西兰部分草地和森林土壤,分析比较高通量测序和 DGGE 技术两种方法在土壤微生物丰度、菌落结构以及多样性检测方面的差异,结果发现前者能够更全面和准确地反映土壤微生物群落结构及多样性,DGGE仅能够反应有限的微生物类群,在极大程度上会低估物种组成结构[26]。Duan等[27]用基因组高通量测序法研究了畜牧业表层土壤中甲烷氧化菌多样性,表明该环境中Ⅰ型甲烷氧化细菌在物种多样性和丰度方面更具优势。
3.5 磷酸脂肪酸分析技术
磷酸脂肪酸分析(PLFA)技术可以通过检测微生物中独特的磷酸脂肪酸类型和含量来评估不同种类微生物活细胞生物量。不同类型的甲烷氧化菌内磷酸脂肪酸组成比例也有差异,Ⅰ型和X 型甲烷氧化菌活细胞内占优势的为16-C脂肪酸,而18-C脂肪酸在Ⅱ型菌中含量更为丰富,C18:1ω8c和C18:1ω7c常作为标志性脂肪酸来检测环境中TypeⅡ甲烷氧化菌的特异性存在[28]。实际应用中,PLFA常与同位素13C标记相结合,13C-PLFA技术已成为目前研究土壤甲烷氧化菌的有力工具[29]。Boschker等[30]在对淡水湖泊底层沉积物甲烷氧化菌多样性的研究中首次使用了13C-PLFA 技术,研究表明在该环境下氧化CH4的主导菌群为Ⅰ型甲烷氧化菌。Bodelier等[31]利用该技术标记研究22种TypeⅡ甲烷氧化菌并构建其磷酸脂肪酸数据库,结合多变量方差分析的方法,极大地提高了甲烷氧化菌系统发育分析的分辨率。但目前甲烷氧化菌的特征性脂肪酸数据信息不是很全面,并且该分析所用仪器较为昂贵,使得13C-PLFA技术的研究还存在一定的困难和挑战[25]。
3.6 荧光原位杂交
荧光原位杂交技术(FISH)由Pardue和John两个工作小组于1969年合作研究发明,该技术以细胞遗传学为基础,用荧光替代传统的同位素标记,可以使样品DNA进行原位杂交,不仅可以鉴定环境中特定种类的微生物,还能对其进行种群多样性分析和动态追踪监测[32]。目前,FISH 技术在甲烷氧化菌种群鉴定和种群密度定量描述等方面已取得重大进展,精确程度甚至可以达到物种水平[33]。Dekus等[34]利用该技术对海底甲烷渗漏区沉积物中的厌氧甲烷氧化菌与硫酸盐还原菌展开研究,证实了两者之间的互养共栖关系。Elizabeth研究课题组将FISH技术与标签测序和细胞捕获技术结合起来,研究某甲烷渗漏区沉淀物中甲烷氧化菌与其他微生物之间的关系,推测出甲烷氧化菌的氧化作用与沉积物中的其他微生物功能间存在着复杂的内部联系[35]。然而,由于荧光标记的探针与细胞 rRNA 的拷贝数直接相关,同时也影响着细胞生长速率,而环境中细胞生长率过低会导致对靶细胞的检出率降低,再加上强荧光背景的环境对特定细胞的检测的干扰作用等[36],这些因素都会影响到FISH 技术的准确性。
3.7 变性梯度凝胶电泳
变性梯度凝胶电泳技术(DGGE)创立于19世纪80年代,最初的目的是用以分析基因突变,随后Myuzer等在1993年利用该技术进行了微生物菌落结构的研究并取得良好的效果,开启了DGGE技术研究微生物生态多样性的先河。DGGE技术可以将微生物基因组的长链DNA打断成小片段进行研究,该过程可以通过加入了变性剂(甲酰胺和尿素)的凝胶电泳来实现。最近研究中,相关学者采用PCR-DGGE技术对某地污泥干化芦苇床中的甲烷氧化菌进行了研究,过程中以pmoA作为分子标记,分析比较了不同生长时期以及不同位置存积淤泥中甲烷氧化菌的群落结构和丰度变化[37]。此外,Chi等[38]利用PCR-DGGE和荧光定量PCR技术分析了中国南方地区垃圾填埋表层土壤甲烷氧化菌丰度和群落结构,更进一步拓宽了PCR-DGGE技术在甲烷氧化细菌生态多样性研究领域的应用。但DGGE检测技术也存在很多缺点:一方面,DGGE只能分析较短的(﹤500 bp)基因片段,这就造成了得到的系统发育信息不完善;另一方面,对一些进化关系相近的微生物类群来说,其基因PCR产物的分离不彻底也是造成分析微生物群落结构组成偏差的重要原因[36]。
4 展望
土壤甲烷氧化菌影响着大气甲烷的氧化与吸收,在全球的温室气体平衡中起重要作用,并且甲烷氧化菌能够降解卤化烃类化合物,具有潜在的商业价值。目前为止,对甲烷氧化菌的研究还处于不断探索的阶段,土壤甲烷氧化菌多样性是研究的重要环节,其中包括不同地域、不同植被类型的土壤等。分子生物学方法的兴起为土壤甲烷氧化菌群落结构的研究提供了强有力的工具,为揭示甲烷氧化菌的生态功能及生物多样性提供了大量的信息。鉴于这些方法有各自的优缺点,在实际应用中,通常是多种技术相互补充印证,并且与传统的分离培养方法相结合,以期得到的信息更加全面准确地反映出甲烷氧化细菌的群落结构等生物学特征。
[1]RODHE H. A Comparison of the contribution of various gases to the greenhouse Effect[J]. Science, 1990, 248(4960): 1217-1219.
[2]王明星, 张仁健, 郑循华. 温室气体的源与汇[J]. 气候与环境研究, 2000, 5(1):75-79.
[3]BORKEN W, BRUMME R. Methane uptake by temperate forest soils[J]. Functioning and Management of European Beech Ecosystems, 2009, 208: 369-385.
[4]HANSON R S, HANSON T E. Methanotrophic bacteria[J]. Microbiological Reviews, 1996, 60(2): 439-471.
[5]SOHNGEN N L. Uber Bakterien, welche methan ab kohlenstoffnahrung and energiequelle gebrauchen[J]. Parasitenkd Infectionsker Abt, 1906, 15: 513-517.
[6]WHITTENBURY R, PHILLIPS K C, WILKINSON J F. Enrichment, isolation and some properties of methane-utilizing bacteria[J]. J Gen Microbiol, 1970, 61(2): 205-218.
[7]DUNFIELD P F, YURYEV A, SENIN P, et al. Methane oxidation by an extremely acidophilic bacterium of the phylumVerrucomicrobia[J]. Nature, 2007, 450( 7171): 879-882.
[8]ETTWIG K F, BUTLER M K, LE P D, et al. Nitrite-driven anaerobic methane oxidation by oxygenic bacteria[J]. Nature, 2010, 464(7288): 543-548.
[9]MURRELL J C, DALTON H. Nitrogen fixation in obligate methanotrophs[J]. Microbiology, 1983, 129(11): 3481-3486.
[10]杨铭德, 焦 燕, 李 新, 等. 基于实时荧光定量PCR技术对不同盐碱程度土壤甲烷氧化菌比活性的研究[J]. 生态环境学报, 2015(5):797-803.
[11]AMANN R I, LUDWING W, SCHLEIFER K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143-169.
[12]HEAD I M, SAUNDERS J R, PICKUP R W. Microbial evolution, diversity, and ecology: a decade of ribosomal RNA analysis of uncultivated microorganisms[J]. Microbial Ecology, 1998, 35(1): 1-21.
[13]邓永翠,车荣晓,吴伊波,等.好氧甲烷氧化菌生理生态特征及其在自然湿地中的群落多样性研究进展[J].生态学报,2015, 35(14): 4579-4591.
[14]WEN X, YANG S, LIEBNER S. Evaluation and update of cutoff values for methanotrophic pmoA gene sequences[J]. Archives of Microbiology, 2016, 198(7):629-636.
[15]HOLMES A J, COSTELLO A, LIDSTROM M E, et al. Evidence that particulate methane monooxygenase and ammonia monooxygenase may be evolutionarily related.[J]. FEMS Microbiology Letters, 1995, 132(3):203-208.
[16]BODROSSY L, STRALIS-PAVESE N, MURRELL J C, et al. Development and validation of a diagnostic microbial microarray for methanotrophs [J]. Environ Microbiol, 2003, 5(7): 566-582.
[17]王洪媛,江晓路,管华诗,等. 微生物生态学一种新研究方法-T-RFLP 技术[J].微生物学通报,2004, 31(6): 90-94.
[18]SINGH B K, TATE K. Biochemical and molecular characterization of methanotrophs in soil from a pristine New Zealand beech forest[J]. FEMS Microbiology Letters, 2007, 275(1): 89-97.
[19]LÜKE C, BODROSSY L, LUPOTTO E, et al. Methanotrophic bacteria associated to rice roots: the cultivar effect assessed by T-RFLP and microarray analysis[J]. Environmental Microbiology Reports, 2011, 3(5):518-525.
[20]HO A, VAN D B E, REIMA, et al. Recurrence and frequency of disturbance have cumulative effect on methanotrophic activity, Abundance, and community structure[J]. Frontiers in Microbiology, 2015, 6:1493.
[21]刘雅慈,何 泽,张 胜,等.油气田土壤甲烷氧化菌实时荧光定量PCR检测技术的建立与应用[J].微生物学通报,2014, 41(6):1071-1081.
[22]YUN J L, ZHUANG G Q, MA A Z, et al. Community structure, abundance, and activity of methanotrophs in the zoige wetland of the Tibetan Plateau[J]. Microbial Ecology, 2012, 63(4): 835-843.
[23]MARTINEAU C, PAN Y, BODROSSY L, et al. Atmospheric methane oxidizers are present and active in Canadian high Arctic soils[J]. FEMS Microbiology Ecology, 2014, 89(2):257-69.
[24]WANG H, YU L, ZHANG Z, et al. Molecular mechanisms of water table lowering and nitrogen deposition in affecting greenhouse gas emissions from a Tibetan alpine wetland[J]. Global Change Biology, 2017, 23(2):815-829.
[25]汤华阳, 杜卫东, 张学军. 焦磷酸测序技术及应用[J]. 医学分子生物学杂志, 2007, 4(3):272-275.
[26]夏围围, 贾仲君. 高通量测序和DGGE分析土壤微生物群落的技术评价[J]. 微生物学报, 2014, 54(12):1489-1499.
[27]DUAN Y F, AL-SOUD W A, BREJNROD A. Methanotrophs, methanogens and microbial community structure in livestock slurry surface crusts[J]. Journal of Applied Microbiology, 2014, 117(4): 1066-1078.
[28]BOWMAN J P, SKERRATT J H, NICHOLS P D, et al. Phospholipid fatty acid and lipopolysaccharide fatty acid signature lipids in methane-utilizing bacteria[J]. FEMS Microbiology Ecology, 1991, 85(1): 15-21.
[29]李增强, 赵炳梓, 张佳宝.13C 标记磷脂脂肪酸分析在土壤微生物生态研究中的应用[J]. 中国生态农业学报, 2016, 24(4):470-477.
[30]BOSCHKER H T S, NOLD S C, WELLSBURY P, et al. Direct linking of microbial populations to specific biogeochemical processes by13C-labelling of biomarkers[J]. Nature, 1998, 392(6678): 801-805.
[31]BODELIER P L, GILISEN M J, HORDIJK K, et al. A reanalysis of phospholipid fatty acids as ecological biomarkers for methanotrophic bacteria[J]. International Society for Microbial Ecology, 2009, 3(5): 606-617.
[32]佘晨兴, 仝 川. 自然湿地土壤产甲烷菌和甲烷氧化菌多样性的分子检测[J]. 生态学报, 2011, 31(14):4126-4135.
[33]DEDYSH S N, DERAKSHANI M, LIESACK W. Detection and enumeration of methanotrophs in acidicSphagnumpeat by 16S rRNA fluorescence in situ hybridization, including the use of newly developed oligonucleotide probe forMethylocellapalustris[J]. Applied and Environmental Microbiology, 2001, 67( 10) : 4850-4857.
[34]DEKAS A E, CONNON S A, CHADWICK G L, et al. Activity and interactions of methane seep microorganisms assessed by parallel transcription and FISH-NanoSIMS analyses[J]. Isme Journal, 2015, 10(3):678-692.
[35]TREMBATH-REICHERT E, CASE D H, ORPHAN V J. Characterization of microbial associations with methanotrophic archaea and sulfate-reducing bacteria through statistical comparison of nested Magneto-FISH enrichments[J]. Peerj, 2016, 4: e1913.
[36]梁战备, 史 奕, 岳 进. 甲烷氧化菌研究进展[J]. 生态学杂志, 2004, 23(5):198-205.
[37]王世全. 活性污泥中甲烷菌的研究[D]. 大连:大连工业大学, 2014.
[38]CHI Z F, LU W J, WANG H T. Spatial patterns of methane oxidation and methanotrophic diversity in landfill cover soils of Southern China[J]. Journal of Microbiology & Biotechnology, 2015, 25(4): 423-430.
Researchadvanceinsoilmethanotrophscommunitystructure
CAO Shu-zhen1, SHEN Yuan-yuan2, WANG Feng-qin1, SONG An-dong1, SANG Yu-qiang2
(1. College of Life Science, Henan Agricultural University, Zhengzhou 450002; 2. College of Forestry, Henan Agricultural University, Zhengzhou 450002, China)
Methane, one of the greenhouse gases, has a strong ability in absorbing infrared radiation and its warming potential of single molecule is 15-30 times higher than CO2. Soil methane absorption plays the significant role in the carbon and nitrogen cycle in ecosystem and carbon balance. In recent years, scientists have done a lot of researches on soil methanotrophic bacterias, and there are also many problems at the same time. This article starts with the methanotrophic microorganisms and summarizes the research advance in soil methane oxidation domestically and internationally. The various molecular biology methods used in methane oxidative bacteria community structure were introduced. It aims to synthesize the research advance in soil methane oxidation and the influence mechanism at home and abroad, which provides theoretical reasons to recognize the role of soil methanotrophic bacterias in global climate change and a theoretical guidance for carrying out the research of surficial methane flux.
methanotrophic bacteria; community structure; soil
2016-10-28;
2016-12-16
国家科技支撑计划子课题(2015BAD07B050601);林业公益性行业科研专项(201404206-03);河南省教育厅重点研究项目(16A220003)
曹淑贞,硕士,专业方向为微生物生态学,E-mail: 13154665225@163.com
桑玉强,副教授,从事林业生态学研究,E-mail: syuqiang@163.com
10.3969/j.issn.2095-1736.2017.06.078
Q938.1+3
A
2095-1736(2017)06-0078-05

