代表性鱼类物种全基因组测序研究进展
陈泓宇, 陈艳艳, 李 蓉, 肖 蘅, 陈善元
(云南大学 生命科学学院, 昆明 650091)
代表性鱼类物种全基因组测序研究进展
陈泓宇, 陈艳艳, 李 蓉, 肖 蘅, 陈善元
(云南大学 生命科学学院, 昆明 650091)
鱼类不仅是脊椎动物中种类最多的一个类群,还是研究脊椎动物演化过程的重要环节之一。基于已完成全基因组测序的鱼类代表性物种信息,从鱼类的基因组倍增、重要基因的演化、免疫系统的演化等3个主要方面进行了概述,以期了解脊椎动物从原始无颌类到软骨鱼类再到硬骨鱼类演化过程中发生的基因组或基因变化,为今后鱼类进化基因组学研究提供一定的参考。
鱼类;全基因组测序;脊椎动物演化
基因组学(Genomics)在1986年首次被提出后,Venter等于1995年率先报道了第一个微生物(Haemophilusinfluenza)基因组序列,为生命科学研究打开了新的大门。相比传统的单基因研究,基因组学催生了生命科学的大数据时代。基因组学极大的数据量使得测序的核苷酸序列剧增,提供了丰富全面的DNA信息,对生物系统发育和抗病力等方面研究的发展产生了极大地推动作用[1]。随着测序技术日渐成熟,在测序费用越来越低,二三代测序技术精度越来越高地推动下[2],各生物的全基因组测序也越来越普遍。全基因测序使得生物学在分子层面的研究更加深入,为探讨物种间和种内群体间的遗传多样性、自然选择及物种形成等方面都带来了便捷[3]。
鱼类是脊椎动物中最为繁盛的一个类群,截至2016年就有超过36 000个物种被描述,主要包括软骨鱼类和硬骨鱼类。鱼类作为古老世系的生物,由于分化强烈和多样的生境而导致其多样的生殖策略和生存策略。有些特殊鱼类为适应特殊生境在形态、感官系统和免疫系统方面都产生了巨大的变异。随着基因组学的研究手段逐渐运用到鱼类研究中,对其基因组层面信息的挖掘还为鱼类生物多样性保护、资源合理利用和寻找新的分子标记等方面提供参考[4-5]。
基于已完成全基因组测序的鱼类代表性物种的信息(表1),本文将从鱼类的基因组倍增、重要基因的演化、免疫系统的演化等3个主要方面进行综述,并对今后的鱼类全基因组测序工作做了展望,以期了解脊椎动物从原始无颌类到软骨鱼类再到硬骨鱼类演化过程中发生的基因组或基因变化,为今后鱼类进化基因组学研究提供一定的参考。
1 鱼类基因组倍增
脊椎动物的进化起始于无颌类(Agnatha),之后逐渐向软骨鱼类(Chondrichthyes)、硬骨鱼类(Osteichthyes)、两栖类(Amphibian)和爬行类(Reptilia)不断地演化和发展[6],无颌脊椎动物逐渐向四足动物演化的过程中,出现过4次全基因组倍增事件并在基因组中留下了演化的痕迹,但是倍增事件的细节仍然不够清楚[7]。第1次全基因组倍增事件发生于脊椎动物祖先,被认为是脊椎动物进化的基础;第2次全基因组倍增事件出现在软骨鱼类和硬骨鱼类分化之前[8];第3次全基因组倍增事件是硬骨鱼类中的真骨鱼类(Teleostei)的一次全基因倍增(TSD)[9];第4次特殊的基因组倍增事件在八千万年前单独出现在鲑属鱼类中[10]。全基因组倍增后产生的副本拷贝基因和原拷贝基因会出现相互调节,以避免基因冗余造成表达错误。这些基因的调节是无颌类向有颌类演化过程中表型改变的主要因素,对脊椎动物进化产生了重要作用。传统的分子生物学手段无法完整地对这些基因演变进行研究,而全基因组测序为研究这些冗余重复基因的变化提供了有效的方法。
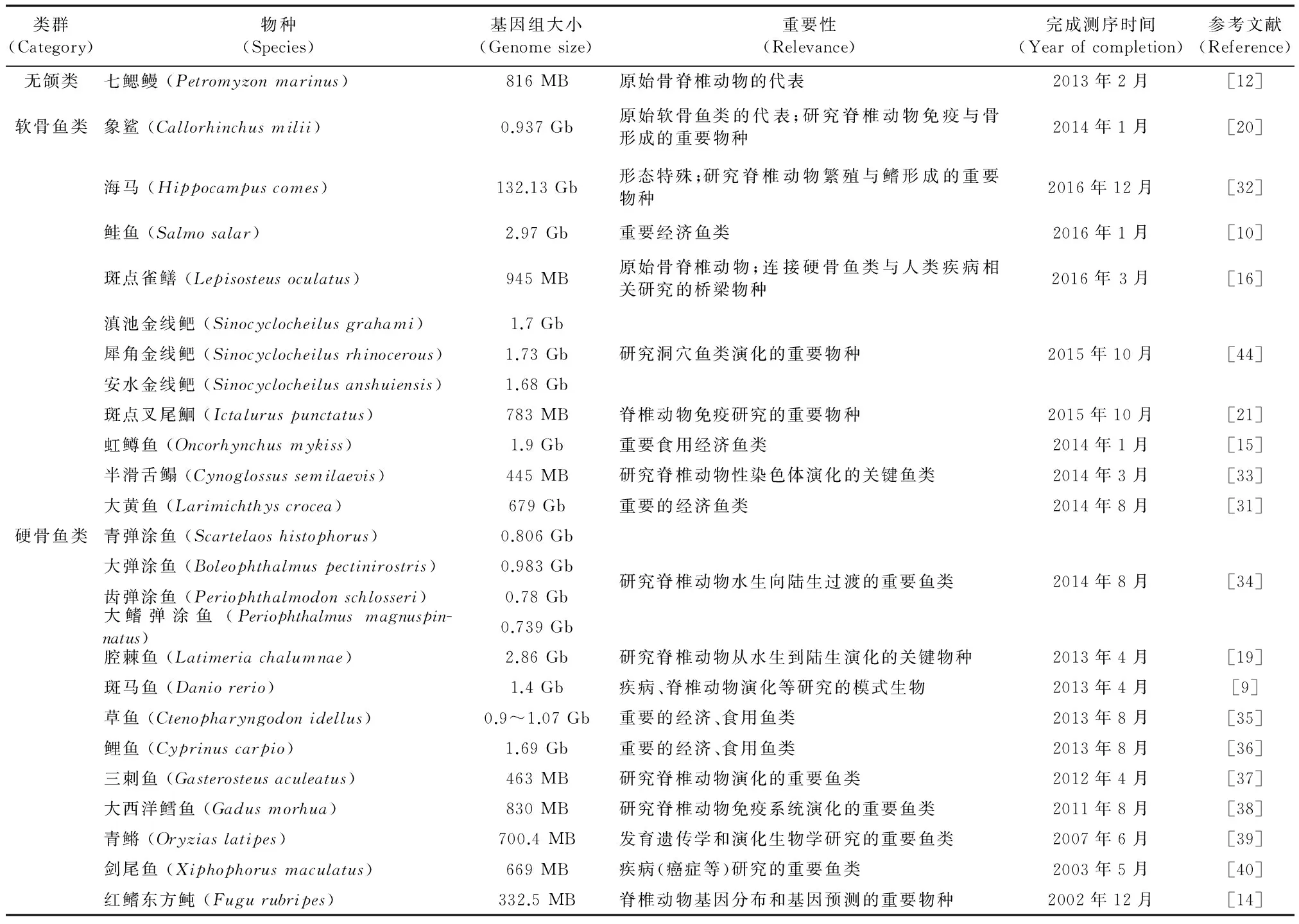
表1 近年来完成全基因组测序的代表性鱼类物种
无颌类圆口纲(Cyclostomata)七鳃鳗(Petromyzonmarinus)虽然不属于鱼类类群,但作为脊索动物门中的原始类群,经历了首次基因组倍增,是研究全基因组倍增事件的重要物种。Jermiah等对七鳃鳗进行了全基因组测序,发现七鳃鳗基因组中存在高度一致的重复元件,但大部分七鳃鳗基因并没有发现相同的两个拷贝[11],可能是由于倍增事件后同源基因会产生丢失[12]。基因组倍增会造成基因冗余,部分冗余基因会出现丢失或假基因化,有的会出现新功能化和亚功能化并协同作用确保基因的正确表达[13]。虹鳟鱼(Oncorhynchusmykiss)属于辐鳍鱼类(Actinopterygii),辐鳍鱼类基因组大规模倍增事件出现在3.8亿万年以前[14]。而特殊的鲑属(Salmo)鱼类其祖先在8千万年前又出现了一次额外的全基因组倍增事件,代表第4次全基因组倍增(Ss4R)后拥有特殊基因组的类群,由于倍增事件发生较晚,依然清晰保留倍增事件后的原本状态。对于研究基因组增倍造成的新功能化、亚功能化以及重复基因分馏提供了一个机会。Berthelot等[15]对虹鳟鱼进行了全基因组测序,利用鳟鱼基因组对基因重复后冗余的多拷贝基因进行了研究,结果表明随基因组倍增,基因重复拷贝最终只有部分保留,冗余的基因重复拷贝会由于分馏作用失活。在虹鳟鱼中选取的569对基因中发现,48%保留同源重复拷贝,其中一半的蛋白质编码基因重复拷贝丢失,而micRNA基因的副本却基本保留。前3次基因组倍增事件保留的基因副本,更有可能在Ss4R后保留,这些保留基因的功能似乎都是与进化发育有关,更多富集于胚胎发育、神经系统和转录因子有关的通路中。而虹鳟鱼中的micRNA可能是由于亚功能化,作为协同调控元件而保留。通常认为,全基因组倍增事件与物种的适应辐射和进化有关,通过全基因组倍增推动了脊椎动物的进化。斑点雀鳝(Lepisosteusoculatus)属全骨鱼类(Holostei),其分化在真骨鱼类特殊的基因组倍增事件之前,进化缓慢并保留了很多古老脊椎动物的基因,对研究基因组倍增事件之后的进化具有重要的作用。Braasch等[16]对斑点雀鳝进行了全基因组测序,利用基因组数据对斑点雀鳝和硬骨鱼类进行了进化速率分析对比,结果显示斑点雀鳝进化速率慢于真骨鱼类,证明TSD可能推动了真骨鱼类进化速率增快;通过与真骨鱼类基因对比发现,每一个斑点雀鳝基因片段在真骨鱼类基因组中都有两个对应,并且每一个TSD起源的真骨鱼类基因都表现出保守共线性;研究表明斑点雀鳝和真骨鱼类于TSD之前分化,并且基因重排事件发生于TSD之后。
2 鱼类重要基因的演化
通过全基因组测序来研究脊椎动物进化过程中的遗传变化,揭示形态改变下的遗传机制,是对鱼类进行全基因组测序的主要目的。在脊椎动物进化过程中,有颌类的主要进化事件是成对附肢的形成[12]。在对四足动物附肢形成问题的研究上发现,Shh基因参与脊椎动物发育的许多过程,与肢体、体节、神经管等发育均有关[17]。但肢的形成从何时开始,如何起源与演化还有待进一步研究。
比较无颌类与四足动物几个关键物种的全基因组中与肢发育和相关调控元件,可以进一步了解附肢演化的问题。七鳃鳗作为古老无颌类,保留了最原始脊椎动物的部分基因。将七鳃鳗全基因组测序数据与四足动物等其他脊椎动物类群比对,以搜寻基因组中与成对附肢发育有关的基因,对了解附肢的起源起到了重要帮助。结果显示Shh附肢特异性调控元件(ShARE)在真骨鱼类和四足动物中都有发现,在七鳃鳗全基因组中却未发现ShARE相关基因,推测与成对肢发育有关的Shh基因和调控元件只在有颌类中产生[12]。有颌类分化出软骨鱼纲和硬骨鱼纲两大类。其中腔棘鱼(Coelacanth)隶属于硬骨鱼纲辐鳍亚纲,不仅是进化缓慢的古老的物种,还是研究脊椎动物从水生到陆生过渡的关键物种。Amemiya等对腔棘鱼进行了全基因组测序,对腔棘鱼和四足动物HoxD基因家族进行对比。Hox基因家族主要调控细胞分裂、硬毛及附肢等的发育,在四足动物中与肢形成有关[18]。对比结果表明,在腔棘鱼中发现一个与四足动物中共有的与肢特殊表达驱动有关的调控区域Island1,推测远端末梢的生长调控是从祖先肉鳍鱼上演化而来的。并且通过系统发育研究表明,是肺鱼(Lungfish)而不是腔棘鱼与四足动物的关系更密切[19]。
软骨鱼类和硬骨鱼类的另一个明显差异是鳞和骨骼的发育。软骨鱼类内骨骼完全由软骨组成,外骨骼不发达。而硬骨鱼类具有较为发达的骨组织,也许在软骨鱼类与硬骨鱼类分化之后硬骨鱼类出现了特殊的基因表达,造成骨的硬化。象鲨(Callorhinchusmilii)是软骨鱼类中比较具有代表性的物种,进化缓慢。VenRatesh等对象鲨进行了全基因组测序,对比硬骨鱼类,寻找骨骼形成有关的基因,发现象鲨可能由于缺乏Scpp(掺锶聚磷酸钙)家族基因而影响了骨的发育。可能Scpp相关的基因(Sparc、Sparcl1)调节了脊椎动物骨的矿化[20]。斑点雀鳝发育缓慢,基因组中保留了古老的骨矿化有关的基因。通过对斑点雀鳝全基因组测序,在斑点雀鳝基因组中找到了35个Scpp有关的基因,5个与真骨鱼类中的Scpp基因同源,6个与肉鳍鱼类特有的Scpp基因同源[16]。古脊椎动物祖先可能有丰富的Scpp基因存在,但在第2次全基因组倍增过程中在软骨鱼类基因组中出现了丢失,只保留在硬骨鱼类中表达。在软骨鱼类中丢失的原因可能是基因组倍增事件造成的基因分馏或者出现了亚功能化,也可能是在基因组倍增后硬骨鱼类的冗余重复基因出现了特殊调控机制,使Scpp基因及相关的ShARE得以表达。在真骨鱼类研究中发现,Scpp基因还与鱼鳞的形成有关,为了确定鳞片形成的机制,Liu等[21]对无鳞片鲶鱼(lctaluruspunctatus)进行了全基因组测序,将鲶鱼与有鳞鱼转录组数据进行了比对,确定了13个与鳞片形成有关的基因,其中占主导作用的是载脂基因和Scpp基因,而鳞片形成的过程中,SCPP7基因的表达明显上升。
3 鱼类免疫系统的演化
鱼类虽是低等脊椎动物,但与哺乳动物一样,拥有先天性免疫应答和适应性免疫应答两种免疫应答类型[22]。通过对鱼类的全基因组测序,能够更全面地探索鱼类进化过程中免疫系统方面的变化,还可作为研究高等脊椎动物疾病的医学模型。通常认为,适应性免疫系统起源于有颌类脊椎动物,在无颌类脊椎动物中并不存在[23]。在高等脊椎动物的适应性免疫系统中,MHC(组织相容性复合体)、免疫球蛋白Ig以及T/B淋巴受体是发挥主要作用的3个核心组份[24]。Jermiah等在对七鳃鳗基因组研究中发现,其基因组拥有与有颌类相似的两类免疫细胞,存在适应性免疫相关的因子,其适应性免疫受体与有颌类不同,也许反映了祖先脊椎动物的免疫受体,但在之后的演化过程中出现了变化。
七鳃鳗为研究原始无颌类脊椎动物向有颌类脊椎动物演化中适应性免疫系统的变化提供了线索[12]。在免疫球蛋白方面,哺乳动物有5类免疫球蛋白[25]而鱼类是最早产生免疫球蛋白(Ig)的脊椎动物之一,其中IgM是鱼类最主要的免疫球蛋白。近年来新发现的IgW是一类在软骨鱼类中的原始Ig[26]。有颌鱼类中,通过对进化缓慢的腔棘鱼进行全基因组测序发现,腔棘鱼中并不存在硬骨鱼类中共有的IgM,而具有软骨鱼类特有的IgW。据此推测免疫球蛋白IgW起源于无颌类和有颌类的共同祖先,但是在硬骨鱼类和爬行动物演化的过程中被丢失了[19]。
主要组织相容性复合体(MHC),是脊椎动物中发现的编码免疫球蛋白样受体的基因群,主要分为两类——I(ClassI)和II(ClassII)。脊椎动物许多与免疫相关的重要基因都在MHC位点上[27]。通常认为,MHC两类基因在四足动物和软骨鱼类中存在紧密联系,但是与硬骨鱼类没有关联。硬骨鱼类作为初等脊椎动物重要类群,其MHC基因结构与高等脊椎动物相比存在较大差异[28]。通过对斑点雀鳝MHC相关的基因与真骨鱼类和四足动物对比显示,斑点雀鳝基因组中既有部分的MHCI类基因与真骨鱼类同源,又有部分与四足动物同源[16]。同样对模式生物斑马鱼全基因组测序后对MHC基因搜索,结果显示斑马鱼中具有与哺乳动物MHC相关的同源基因[9]。斑点雀鳝和斑马鱼成了连接真骨鱼类与四足动物的桥梁。
在先天性免疫方面,Toll样受体(TLR)家族是鱼类免疫的第一道防线,并连接先天性免疫与获得性免疫[29]。Amaia等在虹鳟鱼中鉴定了首个鱼类TLR基因[30]。人工饲养大黄鱼与野生大黄鱼相比抗病力差,容易暴发疾病,Wu等对野生大黄鱼(Larimichthyscrocea)全基因组测序,旨在揭示大黄鱼免疫系统基因层面的信息。研究结果显示,大黄鱼Toll样受体相关的一系列因子、ILs(白细胞介素)、TNFs(诱导肿瘤坏死因子)在大黄鱼基因组中都存在,TNF相关的基因在野生大黄鱼基因组中得到扩张,表明野生大黄鱼的先天性免疫系统由TNF基因的加倍得到加强。同时快速进化的和受正选择作用的基因富集于补体系统和凝血瀑布有关的基因中。依此推测大黄鱼抗病力差的原因可能是由于大黄鱼虽然有一套有效的先天性免疫系统抵抗常规疾病,但是适应性免疫系统对鱼特殊病原体的抵抗较弱[31]。
4 结语与展望
作为分子生物学重要的分析方法之一,DNA测序为生物遗传信息的揭示和基因表达调控研究等提供了重要的数据[41]。在全基因组测序之前,线粒体基因组测序手段被运用于生物学的研究中,为研究物种系统发育关系等方面提供了一定的便捷[42]。但是相比于核基因组的双亲遗传信息,线粒体基因组仅提供母系方面的遗传信息,没有全基因组测序提供的遗传信息全面,很多问题无法解决。通过对部分鱼类全基因组测序分析,使无颌类、软骨鱼类和硬骨鱼类进化过程中一系列基因变化,包括基因组倍增事件以及倍增之后冗余拷贝的状况都得到进一步了解,而进化过程中起重要作用的基因如HOX基因家族、肢和鳞形成有关的矿化基因、免疫基因等,也通过全基因组测序得到了研究。就目前开展的鱼类全基因组测序而言,如表1所示对软骨鱼类的研究较为缺乏[43],但软骨鱼类是脊椎动物演化过程中具有重要作用的一大类群,若想要系统地研究脊椎动物的演化历史,了解软骨鱼类与硬骨鱼类进化时发生的基因变化,还需要对一些具有代表性的软骨鱼类进行全基因组测序分析。
此外,对一些具有独特性状(如眼睛退化、体色透明)的鱼类物种开展全基因组测序分析,有助于了解这些独特性状形成背后的遗传基础。Yang等[44]就对我国特有的3种金线鲃属物种进行了全基因组测序,发现洞穴金线鲃中存在很多特殊的遗传变化,如视蛋白基因丢失、晶状体蛋白基因假基因化、色素相关基因发生突变,而味觉相关的转录因子数量增加。但目前此类从独特性状出发的研究依然较少,但也在逐步开展。如Lin等[32]从海马的特殊体型和“雄性育儿”行为等特征出发,基于海马全基因组及与其他物种的比较基因组学分析,首次阐明了海马特异体型进化机制,并从基因层面探讨了育儿袋形成和怀孕过程,揭开了海马雄性育儿之谜。相信随着全基因组测序成本的持续下降和数据分析手段的常规化,越来越多的鱼类全基因组序列将被测定,相关研究也会越来越全面和深入。
[1]于 黎, 张亚平. 系统发育基因组学——重建生命之树的一条迷人途径 [J]. 遗传, 2006, 28(11):1445-1450.
[2]GOODWIN S, MCPHERSON J D, MCCOMBIE W R, et al. Coming of age: ten years of next-generation sequencing technologies [J]. Nature Reviews Genetics, 2016, 17(6):333-351.
[3]王章群, 解增言, 蔡应繁, 等. 系统发育基因组学研究进展 [J]. 遗传, 2014, 36(7):669-678.
[4]陈艺燕, 章 群, 任 岗, 等. 10种石斑鱼系统发育的线粒体细胞色素b基因序列分析 [J]. 海洋科学, 2006, 30(6):12-15.
[5]MENG F, BRAASCH I, PHILLIPS J B, et al. Evolution of the eye transcriptome under constant darkness in sinocyclocheilus cavefish [J]. Molecular Biology and Evolution, 2013, 30(7):1527-1543.
[6]解 谦. 脊椎动物从水生到陆生的结构演变 [J]. 山西农业大学学报(自然科学版), 2003, 23(4):383-385.
[7]KURATANI S, KURAKU S, MURAKAMI Y, et al. Lamprey as an evo-devo model: lessons from comparative embryology and molecular phylogenetics [J]. Genesis, 2002, 34(3):175-183.
[8]MOGHADAM H K, FERGUSON M M, DANZMANN R G, et al. Comparative genomics and evolution of conserved noncoding elements (CNE) in rainbow trout [J]. BMC Genomics, 2009, 10:278.
[9]HOWE K, CLARK M D, TORROJA C F, et al. The zebrafish reference genome sequence and its relationship to the human genome [J]. Nature, 2013, 496(7446):498-503.
[10]LIEN S, KOOP B F, SANDVE S R, et al. The Atlantic salmon genome provides insights into rediploidization [J]. Nature, 2016, 533(7602):200-205.
[11]李庆伟, 冯 波, 陈立勇, 等. 雷氏七鳃鳗(Lampetrareissneri)消化系统组成及消化液主要组分的鉴定分析 [J]. 辽宁师范大学学报(自然科学版), 2012, 35(2):238-240.
[12]SMITH J J, KURAKU S, HOLT C, et al. Sequencing of the sea lamprey (Petromyzonmarinus) genome provides insights into vertebrate evolution [J]. Nature Genetics, 2013, 45(4):415-421.
[13]LAN X, PRITCHARD J K. Coregulation of tandem duplicate genes slows evolution of subfunctionalization in mammals [J]. Science, 2016, 352(6288):1009-1013.
[14]CHRISTOFFELS A, BRENNER S, VENKATESH B. Tetraodon genome analysis provides further evidence for whole-genome duplication in the ray-finned fish lineage [J]. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2006, 1(1):13-19.
[15]BERTHELOT C, BRUNET F, CHALOPIN D, et al. The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertebrates [J]. Nature Communications, 2014, 5:3657.
[16]BRAASCH I, GEHRKE A R, SMITH J J, et al. The spotted gar genome illuminates vertebrate evolution and facilitates human-teleost comparisons [J]. Nature Genetics, 2016, 48(4):427-437.
[17]张昌盛, 刘 群, 李湘涛, 等. 多鳍鱼Shh基因克隆与同源性分析 [J]. 科技导报, 2007, 25(12):23-27.
[18]耿 波, 孙效文. 鱼类Hox基因簇结构、表达和进化方面研究进展 [J]. 水产学杂志, 2012, 25(4):60-64.
[19]AMEMIYA C T, ALFÖLDI J, LEE A P, et al. The African coelacanth genome provides insights into tetrapod evolution [J]. Nature, 2013, 496(7445):311-316.
[20]VENKATESH B, LEE A P, RAVI V, et al. Elephant shark genome provides unique insights into gnathostome evolution [J]. Nature, 2014, 505(7482):174-179.
[21]LIU Z, LIU S, YAO J, et al. The channel catfish genome sequence provides insights into the evolution of scale formation in teleosts [J]. Nature Communications, 2016, 7:11757.
[22]李亚南, 陈全震, 邵健忠, 等. 鱼类免疫学研究进展 [J]. 动物学研究, 1995, 16(1):83-94.
[23]PLOUFFE D A, HANINGTON P C, WALSH J G, et al. Comparison of select innate immune mechanisms of fish and mammals [J]. Xenotransplantation, 2005, 12(4):266-277.
[24]李风铃, 李兆新, 翟毓秀, 等. 鱼类适应性免疫系统研究概述 [J]. 水产科学, 2012, 31(4):240-244.
[25]吕 翠, 安利国, 杨桂文. 硬骨鱼新型免疫球蛋白的研究进展 [J]. 中国细胞生物学学报, 2011, 33(8):905-913.
[26]吕爱军, 胡秀彩, 张艳华,等. 鱼类中新发现的免疫球蛋白 [J]. 水产科学, 2011, 30(7):425-428.
[27]史 燕, 吴孝兵, 晏 鹏, 等. 主要组织相容性复合体(MHC)基因的研究概况 [J]. 生命科学研究, 2003, 7(2):104-109.
[28]贾震虎, 夏 春. 硬骨鱼类MHCⅠ基因结构及表达研究 [J]. 中国兽医杂志, 2008, 44(11):54-55.
[29]范泽军, 邹鹏飞, 姚翠鸾. 鱼类Toll样受体及其信号传导的研究进展 [J]. 水生生物学报, 2015, 39(1):173-184.
[30]AMAIA S, MARTIN S A M, O’DEA P G, et al. Cloning and characterization of the rainbow trout (Oncorhynchusmykiss) type II interleukin-1 receptor cDNA [J]. European Journal of Biochemistry, 2000, 267(24):7031-7037.
[31]WU C, ZHANG D, KAN M, et al. The draft genome of the large yellow croaker reveals well-developed innate immunity [J]. Nature Communications, 2014, 5(5):5227.
[32]LIN Q, FAN S, ZHANG Y, et al. The seahorse genome and the evolution of its specialized morphology [J]. Nature, 2016, 540 (7633): 395-399.
[33]CHEN S, ZHANG G, SHAO C, et al. Whole-genome sequence of a flatfish provides insights into ZW sex chromosome evolution and adaptation to a benthic lifestyle [J]. Nature Genetics, 2014, 46(3):253-260.
[34]YOU X, BIAN C, ZAN Q, et al. Mudskipper genomes provide insights into the terrestrial adaptation of amphibious fishes [J]. Nature Communications, 2014, 5:5594.
[35]WANG Y, LU Y, ZHANG Y, et al. The draft genome of the grass carp (Ctenopharyngodonidellus) provides insights into its evolution and vegetarian adaptation [J]. Nature Genetics, 2015, 47(6):625-631.
[36]XU P, ZHANG X, WANG X, et al. Genome sequence and genetic diversity of the common carp,Cyprinuscarpio[J]. Nature Genetics, 2014, 46(11):1212-1229.
[37]JONES F C, GRABHEER M G, CHAN Y F, et al. The genomic basis of adaptive evolution in threespine sticklebacks [J]. Nature, 2012, 484(7392):55-61.
[38]STAR B, NEDERBRAGT A J, JENTOFT S, et al. The genome sequence of Atlantic cod reveals a unique immune system [J]. Nature, 2011, 477(7363):207-210.
[39]KASAHARA M, NARUSE K, SASAK S, et al. The medaka draft genome and insights into vertebrate genome evolution [J]. Nature, 2007, 447(7145):714-719.
[40]SCHARTL M, WALTER R B, SHEN Y, et al. The genome of the platyfish,Xiphophorusmaculatus, provides insights into evolutionary adaptation and several complex traits [J]. Nature Genetics, 2013, 45(5):567-572.
[41]王兴春, 杨致荣, 王 敏, 等. 高通量测序技术及其应用 [J]. 中国生物工程杂志, 2012, 32(1):109-114.
[42]郑 蕾. 鸟类线粒体基因组及系统发育的研究进展 [J]. 芜湖职业技术学院学报, 2015, 17(1):90-92.
[43]谢仰杰, 翁朝红, 苏永全,等. 软骨鱼类染色体研究进展 [J]. 中国水产科学, 2006, 13(5):856-866.
[44]YANG J, CHEN X, BAI J, et al. TheSinocyclocheiluscavefish genome provides insights into cave adaptation[J]. BMC Biology, 2016, 14(1):1-13.
Researchadvancesinwhole-genomesequencingofrepresentativefishspecies
CHEN Hong-yu, CHEN Yan-yan, LI Rong, XIAO Heng, CHEN Shan-yuan
(School of Life Sciences, Yunnan University, Kunming 650091, China)
Fishes are not only one of major groups with highest species diversity among vertebrates, but also one of important parts for studying vertebrate evolutionary processes. Based on available information from whole-genome sequencing of representative fish species, in order to understand changes in genomes or genes that occurred during vertebrate evolutionary processes from Agnatha to Chondrichthyes and to Osteichthyes, this paper reviews genome duplication, evolution of important genes, and evolution of immune systems in fishes, aming to provide reference for future evolutionary genomics study on fishes.
fishes; whole-genome sequencing; vertebrate evolution
2016-12-12;
2016-12-15
国家自然科学基金(31560111);云南大学引进人才科研启动资金(XT412002)
陈泓宇,硕士,研究方向为动物遗传学,E-mail:786180187@qq.com
肖 蘅,博士,教授,博士生导师,研究方向为动物遗传与进化,E-mail:xiaoheng@ynu.edu.cn;陈善元,博士,教授,硕士生导师,研究方向为动物遗传学与基因组学,E-mail:chensy@ynu.edu.cn
10.3969/j.issn.2095-1736.2017.06.073
Q953
A
2095-1736(2017)06-0073-05

