宁夏外引水稻种质资源与选育品种遗传多样性分析
黄新玲,张振海,孙建昌*,李 华
(1.宁夏农林科学院农作物研究所,宁夏 永宁 750105;2.宁夏银川市西夏区文萃路宁夏职业教育园区,宁夏 银川 750021)
宁夏外引水稻种质资源与选育品种遗传多样性分析
黄新玲1,张振海1,孙建昌1*,李 华2
(1.宁夏农林科学院农作物研究所,宁夏 永宁 750105;2.宁夏银川市西夏区文萃路宁夏职业教育园区,宁夏 银川 750021)
【目的】本文探讨了宁夏近几年引进筛选的水稻种质资源及当地选育品种的遗传多样性和遗传相似性,为拓展宁夏水稻遗传基础,丰富水稻遗传多样性提供参考依据。【方法】利用分布于12条染色体上的24对SSR引物对宁夏引进筛选的水稻种质资源和当地自育品种(系)进行了遗传多样性和遗传相似性分析。【结果】参试170份材料中,共检测到112条等位基因,平均每条SSR标记有4.67条等位基因,Nei′s基因多样性指数为0.6079,变幅为0.2271~0.7986。将材料按地理来源划分,探讨其亲缘关系,不同地理来源品种间的相似系数介于0.4053~0.8620,平均系数为0.7109。其中东北地区和日本的品种相似系数最大为0.8620,而江苏和南方(以云南为主)的品种间相似系数最小为0.4053。本次所引进的材料与宁夏本区的材料遗传相似系数均较大,尤其东北3省的最大,达到0.8344。【结论】大多数引进资源与宁夏水稻品种的亲缘关系较近,说明宁夏种质资源的遗传多样性虽然有所增加,但遗传基础较为狭窄。在今后水稻种质资源创制利用中,要充分利用引进资源的遗传差异,加大亲本遗传距离,以丰富育成品种的遗传基础。
水稻;外引品种;自育品种;遗传多样性
【研究意义】种质资源(Germplasm resources)又称遗传资源(Genetic resources) 或基因资源(Gene resources),一直被认为是育种的基础与前提,是最原始的遗传物质的载体包括了其所在物种(包含其变种)和所有的基因类型[1]。水稻种质资源是水稻育种的关键,有效的掌握和利用水稻种质资源,才有可能取得水稻的遗传改良和突破性的育种成就。与其它农作物比较,水稻是宁夏的特色优势农作物,古时曾为贡米,以高产和优质著称[2]。建国以来,宁夏水稻品种已实现了6次更新换代,前3次以外引品种为主,后 3次以自育品种为主[3]。近年来一些优良品种被大面积推广,为当地水稻产业做出了巨大的贡献。同时,也不同程度出现了品种单一化的趋势,从而造成了遗传多样性的降低,同时大量基因也随之丢失,为病、虫等自然灾害提供了温床,提高了农户的风险点[4]。为拓宽宁夏水稻亲本的遗传基础,扩大引种范围并挖掘其有利基因,同时要利用分子标记技术,明确品种间的遗传距离,可为亲本的选择提供参考依据。【前人研究进展】前人的大量研究表明,利用SSR标记,探索种质资源的遗传多样性,确定其遗传距离,可为品种选育提供依据[5]。遗传多样性的研究对于保护生物多样性具有重要的理论和实际意义[6]。赵勇[7]等利用16对SSR功能基因标记对23份来自5个国家的水稻种质资源的遗传距离进行研究,证实SSR功能标记是研究水稻种质资源分类、生态类型、地理分布和系谱分析的有效工具。束爱萍等[8]探讨了中国不同省份间粳稻选育品种的遗传相似性和遗传差异,建议在资源创新和育种中,选配亲本要尽量选择纬度相差较大、地理位置相隔较远,生长环境不同的材料,创造出具有丰富变异类型的新种质,从而符合品种选育的目标。郝伟等[9]对东北三省参加区域试验的35个水稻品种(系)利用SSR标记分析其遗传多样性和相似性,结果表明参试材料间遗传距离狭窄,为防止遗传多样性进一步丢失,建议注重收集、保存地方品种资源;为扩大育种材料的遗传基础,还要充分利用我国丰富的水稻种质资源。孙健等[10]利用42对SSR引物对80份黑龙江省主栽水稻品种进行SSR遗传多样性分析,为拓宽黑龙江省水稻种质资源的遗传基础,建议今后要充分挖掘地方品种、杂草稻等资源的有用基因,同时加大力度引进、鉴定和利用国外资源。甘晓燕等[11]对宁夏89份粳稻种质材料进行SSR分析,以探讨宁夏粳稻品种的遗传多样性水平, 研究表明,宁夏粳稻种质之间的相似性较高,遗传差异较小,遗传背景比较单一。随着育种工作的深入,育种目标的明确化,所选育的品种趋于单一化,核心亲本的反复利用,造成品种遗传多样性下降。为了提高宁夏水稻的育种效率,降低品种抗性丧失风险点,就必须进一步引进、开发和利用遗传多样性丰富的资源,如地方品种和野生稻等。近年来,宁夏也从国内外引进了很多水稻种质资源,但为了实现育种目标,适应本区生态环境,外引品种局限性较大,遗传背景较相似。【本研究切入点】本试验通过对外引资源和宁夏本区品种(系)进行遗传多样性分析,确定其遗传距离。【拟解决的关键问题】为育种者提供很好的依据,在以后水稻杂交选育中采取遗传距离较远的种质资源相互杂交,促使在杂交后代中出现丰富多样的重组类型,这有助于扩大双亲的遗传基础和丰富宁夏水稻的遗传多样性。
1 材料与方法
1.1 供试材料
本试验选取170份2015年宁夏的外引粳稻资源、当地的选育品种(品系)、本区参加区域试验的品种以及新育成的品系作为试验材料。包括日本材料71份,江苏材料1份,宁夏本地材料54份,新疆材料6份,吉林、黑龙江、辽宁的材料共17份,山西材料1份,北京、天津材料共7份,安徽1份,云南材料12份。2015年将全部材料种植于宁夏农林科学院农作物研究所试验田,按顺序排列,单株插秧,每个材料3行,行长1 m,田间管理与大田相同。
1.2 DNA提取和SSR分析
在水稻分蘖盛期,取其单株叶片,将K. Edwards等[12]的CTAB法稍做改进,进行DNA提取与纯化。将分布于水稻12条染色体上的24对多态性较高,条带相对稳定、清晰的SSR引物筛选出来,用于参试材料的遗传分析。PCR反应体系体积10 μl:10×PCR Buffer(含Mg2+)1.0 μl,20 ng/μl DNA 1.0 μl,2.5 mmol/L dNTP 0.75 μl,2 μmol/L SSR引物1.0 μl,ddH2O 6.0 μl,5 U/μlTaq酶0.25 μl。扩增程序为94 ℃预变性5 min,94 ℃变性30 s,55~60 ℃退火30 s,72 ℃延伸30 s,共35~45个循环,72 ℃延伸5 min。采用6 %的聚丙烯酰胺变性凝胶电泳及银染法检测[13]。
1.3 数据分析
等位基因数(Na)、有效等位基因数(Ne)、Nei’s基因多样性指数(He)和Shannon信息指数(I)应用POPGENE32统计软件计算。不同地区类群的Jaccard遗传相似系数矩阵用NTSYSpc2.0计算,聚类分析采用UPGMA法。
2 结果与分析
2.1 引进稻种资源与自育品种SSR标记的多态性分析
对170份水稻品种资源,利用分布于12条染色体上的24对多态性较高、条带相对清晰的SSR引物进行遗传多样性和遗传相似性分析(表1),共检测到112条等位基因,品种间不同位点的等位基因数目不等,变幅为3~8条,平均每条SSR标记有4.67条等位基因。其中等位基因数在3~5之间的引物18对,占参试引物的75 %。RM1337检测到的等位基因数较多,为8条,其次是RM336检测到7条。24对引物检测到有效等位基因数在1.29~4.97,平均2.89条,其中RM336最高,其次为RM7217(4.58)。

表1 宁夏引进水稻品种和当地选育品种的SSR遗传多样性
24对SSR引物中平均Nei’s基因多样性指数为0.6079,变幅为0.2271~0.7986,其中Nei’s基因多样性指数在0.7以上的有:RM336(0.7986)、RM7217(0.7819)、RM1337(0.7576)、RM430(0.7375)、RM8208(0.7355)、RM5473(0.7227)、RM223(0.7131),其Shannon指数在0.4431~1.7413,平均1.1472。
2.2 不同地理来源粳稻品种的亲缘关系分析
2.2.1 遗传相似性分析 将所有材料按地理来源划分为日本、宁夏、东北、华北、南方(以云南为主)、新疆、江苏,各地区品种间的相似系数介于0.4053~0.8620,平均系数为0.7109。其中东北地区和日本的品种相似系数最大为0.8620,而江苏和南方(以云南为主)的品种间相似系数最小为0.4053。宁夏稻与日本、东北3省、华北地区、云南、新疆以及江苏地区的品种间遗传相似系数均较大,变异范围为0.6014~0.8344,平均为0.7681。尤其与东北3省的遗传相似系数最大为0.8344,其次是新疆为0.8144(表2)。
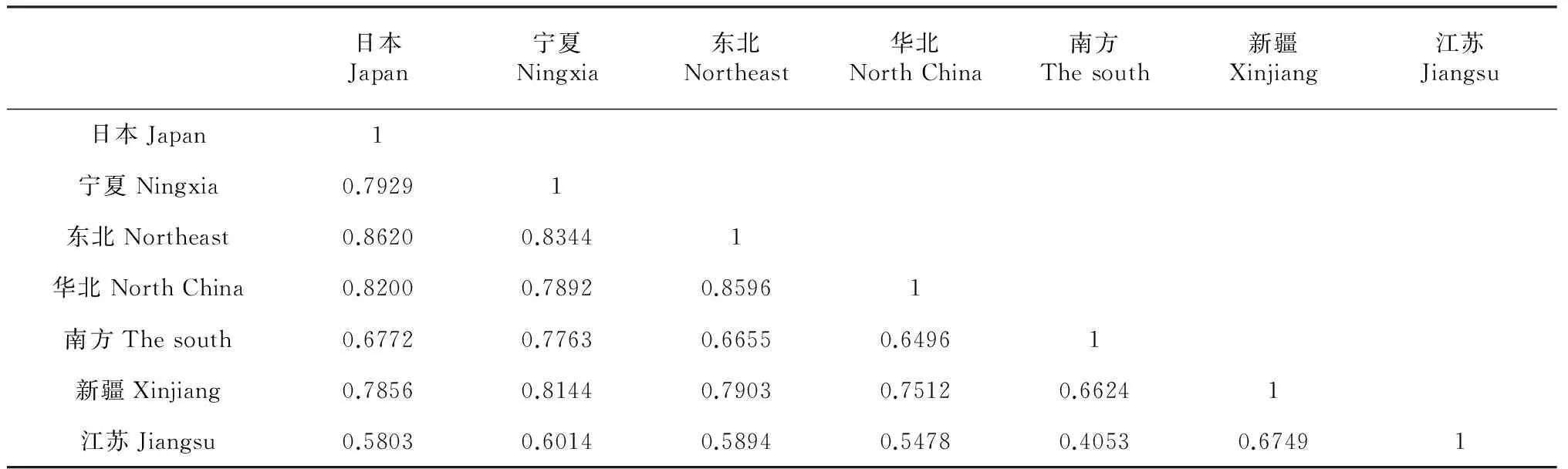
表2 以SSR标记为依据的不同地区水稻品种资源的遗传相似系数
2.2.2 聚类分析 根据试验数据的矩阵情况,按UPGMA进行聚类分析,获得了170份供试材料的聚类图(图1),共分为5大类,其中日本的2个品种Snow parl和早丰各自形成一个独立类,其他品系聚成3大类群,类群I有20个品种(系),根据遗传相似性又可分为3个亚类群。来自宁夏的XM-307、SD-8和2012X-8聚为第1个亚类群。第2个亚类群包括15个品种(系),其中除一个天津品种隆优619和一个吉林品系2012C76外,其余均为宁夏品种(系)。第3个亚类群是日本品种奇丰和黑龙江品系1335。类群Ⅱ包括9个品种(系),黑龙江的龙洋0957、云南的JD330和宁夏的14Q-100聚为一个亚类群,引自日本的梁分和关东15,宁夏自育品系JK308和2013LJ-17,引自新疆的伊粳13号和新核1号6个品种(系)聚为另一个亚类群。其余的139个品种(系)均属于类群Ⅲ,根据遗传相似性又可聚为2个亚类群,第1个亚类群包括47个品种(系),其中大部分(25份)是宁夏的品种(系),从云南引进的12份品种(系)中有11份都归于此类群,还有第2个亚类群包括92个品种(系),大部分(59份)都是日本品种(系),另有12份东北品种(系)和5份宁夏的品种(系)。
聚类结果显示,同一地区的大部分品种(系)往往聚为一类,如类群I的第2个亚类群和类群Ⅲ的第1个亚类群大部分都是宁夏品种(系),类群Ⅲ的第2个亚类群大部分都是日本品种(系)。栽培环境相似,也会导致遗传距离较近,如类群Ⅲ的第1个亚类群中,宁夏和云南的品种(系)聚为一类,这两个地方均为高海拔地区,其气候环境与维度较高的东北3省较相似,因此,黑龙江的龙洋0957、云南的JD330和宁夏的14Q-100聚为一个亚类群。这都是由于在长期的自然选择和人为选育过程中形成了具有相似特征特性的水稻品种。
3 结论与讨论
宁夏优越的自然条件,使得水稻成为宁夏具有特色的区域性优势粮食作物,而且在西北地区粮食生产中宁夏水稻的品种、品质及产量水平都具有举足轻重的地位和作用[14]。20世纪80年代中期,宁夏的水稻主栽品种实现以自育品种取代了外引品种,但由于条件的限制,宁夏地方品种的有利基因挖掘、利用不够,水稻育种的亲本材料类型单一,造成目前宁夏选育的新品种不能有突破性的进展。杨玉蓉等[15]对76份宁夏不同年代水稻品种(系)利用48对SSR引物进行遗传多样性分析,遗传多样性指数变幅0.1492~0.8945,平均为0.6032,大多数宁夏水稻品种的亲缘关系较近,2005年后宁夏水稻品种的遗传多样性趋于变小。马静等[16]选择31份宁夏近年来育成或审定的水稻品种(系),利用分布于12条染色体的36对SSR引物进行遗传距离和遗传多样性分析,共检测到了159个等位基因,Nei基因指数的变幅为0.0317~0.8444,平均为0.5088。SSR标记显示大多数品种的遗传相似系数在0.5以上,最高达到0.94,这反映了宁夏水稻的遗传基础比较狭窄。因此,在今后选育水稻新品种时,为拓宽宁夏水稻的遗传基础,不仅要考虑亲本的亲缘关系,还要继续加大力度引进和创新亲本材料。另外,还有大量研究如刘炜等[17]、甘晓燕等[11]、陈小龙等[18]都表明宁夏粳稻品种间的遗传相似性较高,遗传差异较小,亲缘关系较近,遗传基础相对狭窄。
本试验通过24对引物对170份外引水稻资源与宁夏本区的区域试验材料进行遗传多样性分析,共检测到112条等位基因,平均每条SSR标记有4.67条等位基因。Nei’s基因多样性指数为0.6079,变幅为0.2271~0.7986,高于马静等[19]的研究结果,即宁夏的59份水稻品种的平均Nei's基因多样性指数为0.4394,变幅为0.0333~0.9164,说明本次引进的水稻种质资源有效的扩宽了宁夏水稻的遗传基础。不同地理来源品种间的相似系数介于0.4053~0.8620,平均系数为0.7109。其中东北地区和日本的品种相似系数最大为0.8620,而江苏和南方(以云南为主)的品种间相似系数最小为0.4053。本次所引进的材料与宁夏本区的材料遗传相似系数均较大,尤其东北三省的最大,达到0.8344,这和孙刘臣[20]的研究结果一致,即宁夏水稻种质资源与东北水稻种质资源材料的亲缘关系较近。马静等[19]也表明可能造成宁夏水稻遗传背景单一的主要原因之一是宁夏能够直接或间接利用的资源比较匮乏,多数资源都来自于日本和东北。按UPGMA进行聚类分析,170份材料中除了2个日本材料各聚一类,其余3大类群中均分布有宁夏品种(系),也进一步证实通过近些年资源引进力度的加大,宁夏的水稻种质资源更加丰富了。另外,聚类结果也表明,不同地区的品种遗传相似性不仅和亲本的基因类型密切相关,还和选育品种时的地理位置、环境条件有关,这与束爱萍等[8]的研究结论一致。

图1 基于SSR标记的170份水稻品种资源的聚类图Fig.1 Dendrogram of 170 cultivars based on SSR markers
虽然近年来,宁夏水稻遗传多样性有增加的趋势,但宁夏选育水稻品种的遗传基础狭窄[16],究其原因主要是宁夏的资源类型比较单一,相似的品种间进行过多的杂交,或者反复利用一些优良的亲本材料,另外宁夏特定的水稻种植区的气候类型,导致选育出具备共同特征特性的品种,以此来适应宁夏特殊的生态环境[15]。为了改变这一现状,近些年也加大了对水稻种质资源的引进,但引进材料还比较单一,所引材料国外主要来自日本、韩国等,国内主要以东北、新疆为主,导致许多引进的材料和宁夏的选育材料遗传相似性较高;而且由于气候条件所致,生育期稍长的材料和籼稻在宁夏无法正常成熟,所引材料主要以生育期相对较短的粳稻为主,与籼稻的杂交较少,主要是通过室内或南繁进行,这就大大限制了宁夏水稻遗传基础的拓展。在今后宁夏水稻资源的引进中,一方面要丰富引进的材料,提高品质性状的多样性;另一方面就要通过分子生物学方法挖掘有利基因加以利用。
本试验通过对外引资源和宁夏本区品种(系)进行遗传多样性分析,确定其遗传距离,充分利用引进资源间的遗传差异,在杂交选育中采取遗传距离较远的种质资源相互杂交,促使在杂交后代中出现丰富多样的重组类型,这有助于扩大双亲的遗传基础和丰富宁夏水稻的遗传基础,从而保障水稻育种的持续发展。
[1]李振姣.宁夏外引水稻种质资源的遗传多样性研究及核心种质构建[D]. 宁夏大学农学院硕士学位论文,2016:1-5.
[2]马洪文,殷延勃. 宁夏水稻优质高产栽培技术答疑[M]. 银川:宁夏人民出版社,2009.
[3]王兴盛. 宁夏水稻[J]. 北方水稻,2007(1):19-22.
[4]Normile D. Variety spices up Chinese rice yield[J]. Science, 2000, 289: 1119-1120.
[5]罗 兵,孙海燕,杨志刚,等.基于SSR标记的太湖稻区常规粳稻DNA指纹图谱构建及遗传相似性分析 [J].南方农业学报,2015,46(1):9-14.
[6]马 静,安永平,王彩芬,等.遗传多样性研究进展[J]. 陕西农业科学,2010(1):126-130.
[7]赵 勇,杨 凯,Akbar Ali Cheema,等.利用水稻功能基因SSR标记鉴定水稻种质资源[J]. 中国农业科学,2002,35(4):349-353.
[8]束爱萍,张媛媛,曹桂兰,等. 中国不同省份粳稻选育品种的遗传相似性[J]. 中国农业科学,2009,42(10):3381-3387.
[9]郝 伟,张 旭,徐正进,等. 东北三省水稻遗传多样性和亲缘关系的SSR分析[J]. 河南农业科学,2008(4):18-24.
[10]孙 健,王敬国,刘化龙,等. 黑龙江省主栽水稻品种的遗传多样性分析[J]. 作物杂志,2011(1):63-67.
[11]甘晓燕,李 苗,关雅静,等. 宁夏89份粳稻种质遗传多样性的SSR分析[J]. 西北植物学报,2009,29(9):1772-1778.
[12]Edwards K, Johnstone C, Thompson C.A simple and rapid method for the preparation of plant genomic DNA for PCR analysis[J].Nucleic Acids Res,1991,19(6): 1349.
[13]Panaud O, Chen X, Mc Couch S R. Development of a microsatellite markers and characterization of simple sequence length polymorphism (SSLP) in rice (O.sativaL.)[J]. Mol Gen Genet,1996, 252(5):597-607.
[14]殷延勃,马洪文. 宁夏水稻遗传育种回顾与展望[J]. 宁夏农林科技,2008(3):65-67.
[15]杨玉蓉,孙建昌,王兴盛,等. 宁夏不同年代水稻品种的遗传多样性比较[J]. 植物遗传资源学报,2014,15(3):457-464.
[16]马 静,孙建昌,王兴盛,等. 宁夏水稻选育品种遗传多样性和亲缘关系分析[J]. 西北植物学报,2011,31(5):929-934.
[17]刘 炜,李子超,史延丽,等. 利用SSR标记进行粳稻品种的遗传多样性研究[J]. 西南农业学报,2005,18(5):509-513.
[18]陈小龙,马利奋,王 鹏,等. 宁夏 60 份粳稻种质资源遗传多样性分析[J]. 植物遗传资源学报,2013,14(2):226-231.
[19]马 静,孙建昌,安永平,等. 基于 SSR 标记的宁夏水稻遗传多样性分析[J]. 植物遗传资源学报,2013,14(5):826-832.
[20]孙刘臣. 宁夏和东北早稷稻种质资源遗传多样性分祈[D]. 宁夏大学农学院硕士学位论文,2016:8-26.
(责任编辑 陈 虹)
GeneticDiversityAnalysisofRiceGermplasmIntroducedfromElsewhereandLocalSelf-bredinNingxia
HUANG Xin-ling1, ZHANG Zhen-hai1, SUN Jian-chang1*, LI Hua2
(1.Institute of Crop Science, Ningxia Academy of Agriculture and Forestry Sciences, Ningxia Yongning 750105, China;2.Ningxia Vocational Education Park, Ningxia Yinchuan 750021,China)
【Objective】In this paper, the genetic diversity and similarity of rice germplasm introduced from elsewhere in recent years and self-bred in Ningxia were studied to provide some references for the abundant genetic diversity of rice and expand its genetic basis.【Method】A total of 24 SSR markers distributed in 12 chromosomes were used to analyze the genetic diversity and their genetic relationship of rice germplasm which were introduced from elsewhere and self-bred rice varieties of Ningxia.【Result】Of the 170 materials involved in trials, 112 alleles were revealed, the mean number of alleles per locus was 4.67, and total genetic diversity index of Nei varied greatly among loci from 0.2271 to 0.7986, with an average of 0.6079. To investigate the genetic relationship of different geographical rice germplasm, the similarity coefficient of different varieties of geographical source was between 0.4053 and 0.8620, with the average coefficient of 0.7109. The maximum similarity coefficient between The northeast China and Japan varieties was 0.8620, and the minimum between the Jiangsu and the south (mainly in Yunnan) varieties was 0.4053. There was a high genetic similarity among the rice germplasm of introduction and self-bred, especially in the three provinces of northeast's largest, reaching at 0.8344.【Conclusion】Most of the introduced rice variety resources and Ningxia were close relatives, indicating that the genetic diversity of germplasm resources in Ningxia although had a tendency to increase but the narrow genetic basis. In rice creating and utilizing in the future, we should make full use of the introduction of the genetic differences of resources, increase the genetic distance of the parents and enrich the genetic basis of bred varieties.
Rice; Introduced varieties; Self-bred varieties; Genetic diversity
S511
A
1001-4829(2017)11-2406-06
10.16213/j.cnki.scjas.2017.11.003
2017-03-20
宁夏农业育种专项“水稻新品种选育-资源的引进、鉴定与评价研究”(2013NYYZ0301)
黄新玲(1982-),女,宁夏中卫人,硕士,助理研究员,主要从事水稻品种资源的研究,E-mail:smallbell_512@126.com,Tel:13995193856;*为通讯作者,孙建昌(1975-),男,博士,研究员,主要从事水稻遗传育种的研究,E-mail: nxsjch@163.com。

