谷氨酸棒杆菌天冬氨酸激酶G359D突变解除赖氨酸与苏氨酸协同抑制的研究
徐德雨 郑小梅 赵晶 郑平 赵树欣
(1. 天津科技大学生物工程学院,天津 300457;2. 中国科学院系统微生物工程重点实验室,天津 300308)
谷氨酸棒杆菌天冬氨酸激酶G359D突变解除赖氨酸与苏氨酸协同抑制的研究
徐德雨1,2郑小梅2赵晶2郑平2赵树欣1
(1. 天津科技大学生物工程学院,天津 300457;2. 中国科学院系统微生物工程重点实验室,天津 300308)
天冬氨酸激酶(Aspartate Kinase,AK)是赖氨酸合成途径中关键酶,其受到代谢产物赖氨酸与苏氨酸的协同抑制,旨在发现其解除协同抑制的新突变体并解析其机理。通过对赖氨酸高产菌Corynebacterium glutamicum ZL5与野生型C. glutamicum ATCC 13032的天冬氨酸激酶氨基酸序列比对,发现在高产菌的天冬氨酸激酶中存在G359D突变。通过体外天冬氨酸激酶野生型和G359D突变体酶活检测,发现该G359D突变体在10 mmol/L赖氨酸和苏氨酸同时存在时仍保留了76.94%±1.61%的酶活,而野生酶的酶活仅残留4.38%±1.28%。这一结果在赖氨酸高产菌谷氨酸棒杆菌的回复突变中也可得到验证,其回复突变后使赖氨酸产量下降15.57%。通过同源建模和结构分析发现,与野生酶相比,G359D突变体结合赖氨酸后,其位于活性中心附近的Arg151与Glu74之间无法形成离子键,允许底物分子的结合而具有较高活性。发现天冬氨酸激酶G359D突变体可阻断赖氨酸引起的别构效应,从而有效解除赖氨酸与苏氨酸的协同抑制。
谷氨酸棒杆菌;天冬氨酸激酶;赖氨酸;苏氨酸;协同抑制
赖氨酸是人和动物体内的必需氨基酸之一[1-2],广泛应用于医药、食品与饲料工业[3-5]。目前,赖氨酸的主要生产方法是发酵法,其中谷氨酸棒杆菌(Corynebacterium glutamicum)是赖氨酸的重要生产菌株,年产量达到220万吨以上[6-7]。在赖氨酸合成途径中,天冬氨酸激酶(Aspartate kinase,AK,EC 2.7.2.4)是非常重要的限速酶,可利用ATP将天冬氨酸磷酸化成天冬氨酰-P[6,8]。谷氨酸棒杆菌的天冬氨酸激酶(CgAK)由于受到赖氨酸与苏氨酸的反馈别构调控而成为研究热点。
CgAK属于II类型天冬氨酸激酶,为α2β2型异源四聚体[9-10],即具有2个α亚基和2个β亚基,且均由lysC基因编码(图1)。CgAK催化结构域位于α亚基的N-末端,其别构调控结构域由α亚基的C-末端区域和β亚基组成,在α亚基与β亚基上分别含有两个参与别构调控结合的ACT区(AK(A),chorismate mutase(C)and prep-Henate dehydrogenase(T,for TyrA)), 即 ACT1 和 ACT2[11-12]。 一 个 αβ调节区的ACT结构域中含有两个苏氨酸结合位点和两个赖氨酸结合位点,但在CgAK中有一个赖氨酸结合位点是空置的,因此只结合两个苏氨酸和一个赖氨酸[12]。α亚基的ACT1(253-342)与β亚基的ACT2(1-13,94-160)构成苏氨酸的结合位点,β亚基的ACT1(14-93)与α亚基的ACT2(250-262,343-409)构成苏氨酸的另一个结合位点[12]。另外,β亚基的ACT1与α亚基ACT2也可构成赖氨酸的结合位点,Ile44(β)-N、Val360(α)-N、Thr361(α)-N和Thr361(α)-Oγ1分别与赖氨酸的羧基形成氢键相连,且Gly359-N和Ile42-O也有利于稳定赖氨酸的结合[12]。
在天冬氨酸激酶中,参与赖氨酸与苏氨酸结合的关键氨基酸在其协同抑制中起重要作用。其突变将打破赖氨酸与苏氨酸与天冬氨酸激酶之间的非共价结合,从而解除赖氨酸与苏氨酸对天冬氨酸激酶的协同抑制。Yoshida等[13]利用定点突变与结构分析,发现位于苏氨酸结合位点的G277A与位于调节域αβ二聚体界面的V360A、T361A、E363A的突变体可完全解除协同抑制,在10 mmol/L赖氨酸和苏氨酸的条件下突变体的残留酶活为110%、100%、100%与110%。他们同时发现将CgAK中Gly359突变为丙氨酸时却完全不能解除协同抑制。但在北京棒杆菌(Corynebacterium Pekinense)中的天冬氨酸激酶[14]中将与CgAK的Gly359对应的Gly377位点突变为苯丙氨酸时部分解除了协同抑制,在10 mmol/L赖氨酸和苏氨酸的条件下其酶活可保留24.81%。由此可见,Gly359突变为不同氨基酸时解除协同抑制的程度不同,那么突变何种氨基酸能有效解除协同抑制以及突变后解除抑制的具体机理还需深入研究。
针对上述问题,本研究首先利用野生型C. glutamicum ATCC 13032与赖氨酸高产菌C.glutamicum ZL5的AK的蛋白比对,发现高产菌株中的AK存在G359D的突变。体外酶活检测发现其高效解除赖氨酸和苏氨酸的协同抑制。后续进一步通过对赖氨酸高产菌C. glutamicum ZL5中的突变位点进行回复突变,体内验证该突变位点回复突变后对赖氨酸产量的影响。最后,以CgAK的蛋白结构3AAW和3AB4为模板,利用同源建模来重建突变体的蛋白三维结构,再通过蛋白结构与成键分析来解析突变体解除协同抑制的机理。

图 1 谷氨酸棒杆菌天冬氨酸激酶的α2β2型晶体结构图(PDB ID :3AAW)[12]
1 材料与方法
1.1 材料
1.1.1 菌株和质粒(表1)。
1.1.2 主要试剂和仪器 谷氨酸棒杆菌基因组提取试剂盒为Biomiga公司Promega基因组提取试剂盒;质粒提取试剂盒为北京全式金生物技术有限公司EasyPure Plasmid MiniPrep Kit;胶回收试剂盒为北京全式金生物技术有限公司EasyPure Quick Gel Extraction Kit;克隆载体的一步重组试剂盒为南京诺唯赞生物科技有限公司ClonExpress®II One StepCloning Kit;蛋白定量试剂盒为Pierce®BCA Protein Assay Kit。所有分子生物学工具酶均购自Thermo公司;Ni2+-树脂亲和层析柱购自GE公司;羟胺、天冬氨酸、ATP均购自Sigma公司;其余生化药品为进口或国产分析纯试剂;使用的主要仪器有凝胶成像仪、酶标仪、电转仪、PCR 基因扩增仪、蛋白电泳槽、台式高速冷冻离心机、紫外分光光度计、SBA-40D 生物传感分析仪。
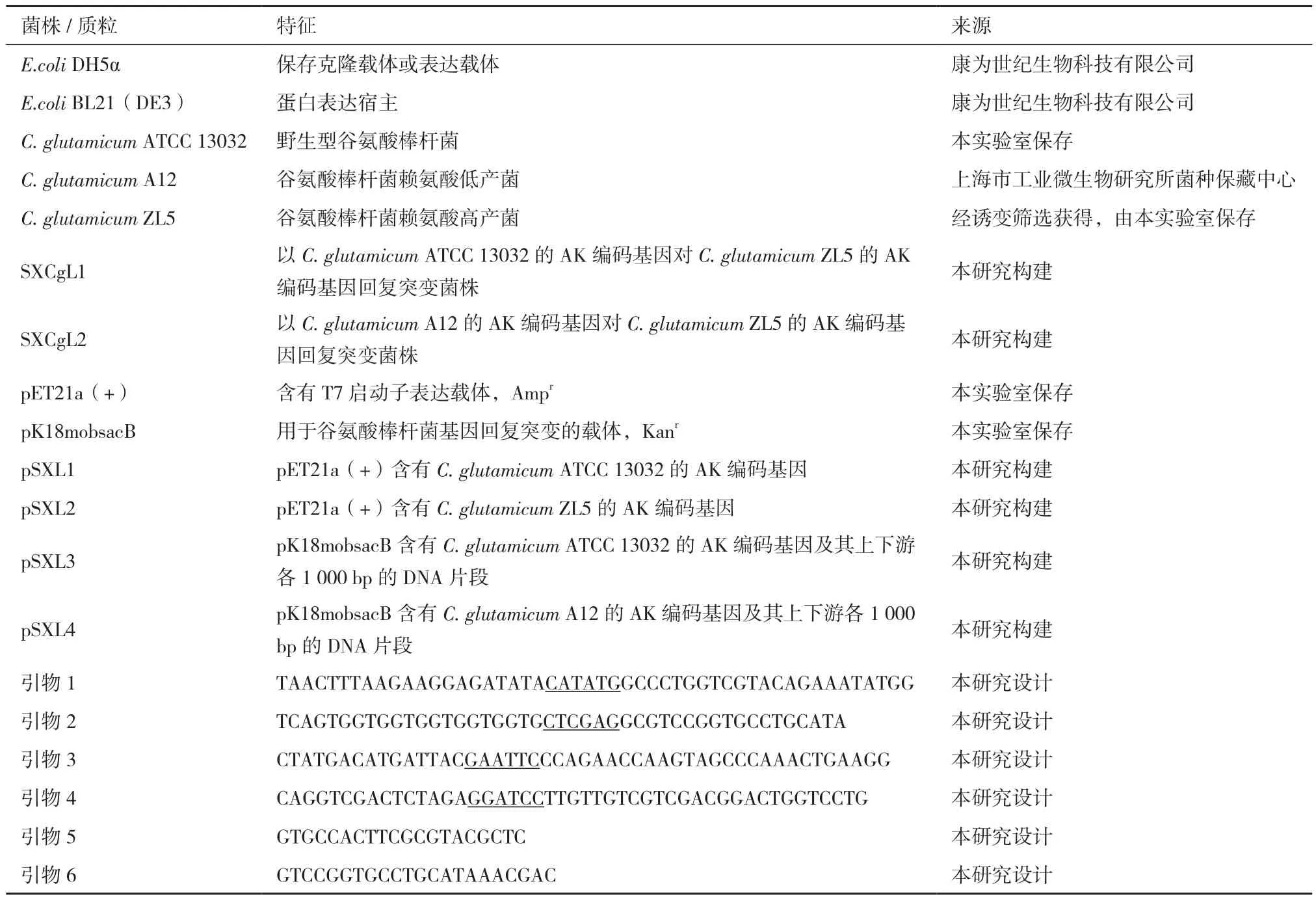
表1 试验所用的菌株、质粒与引物
1.1.3 谷氨酸棒杆菌赖氨酸摇瓶发酵培养基 种子培养基:葡萄糖10 g/L,酵母膏5 g/L,蛋白胨5 g/L,氯化钠5 g/L,牛肉膏10 g/L,麦芽糖提取物5 g/L,pH7.0;发酵培养基:糖蜜50 mL/L,玉米浆30 mL/L,硫酸铵36 g/L,硫酸镁0.27 g/L,磷酸(85%)0.225 mL/L,葡萄糖88 g/L,七水硫酸亚铁0.11 g/L,柠檬酸9.98×10-2g/L,维生素H 2.10×10-3g/L,维生素B1 3.50×10-3g/L,泛酸(维生素B5)1.40×10-2g/L,烟酰胺(维生素B3)4.20×10-2g/L,pH7.0。
1.2 方法
1.2.1 不同谷氨酸棒杆菌的天冬氨酸激酶氨基酸序列比对 本实验室前期利用第二代基因组测序技术对赖氨酸高产菌C. glutamicum ZL5进行了全基因组测序与拼接。利用NCBI的Genebank数据库中的C.glutamicum ATCC 13032天冬氨酸激酶序列(登录号为1021294)对C. glutamicum ZL5中的天冬氨酸激酶编码基因进行注释。利用Clone Manager软件将两个菌株的天冬氨酸激酶氨基酸序列进行比对。
1.2.2 天冬氨酸激酶编码基因克隆与重组表达质粒的构建 首先,采用oligo 7软件根据天冬氨酸激酶编码基因序列设计引物1和引物2(表1),以C.glutamicum ATCC 13032和C. glutamicum ZL5的基因组为模板,扩增两个菌株中的天冬氨酸激酶编码基因,借助引物序列在基因的上下游分别引入可用于与表达质粒pET21a(+)进行重组的同源臂序列。PCR条件为:预变性95℃ 10 min;95℃ 20 s;60℃30 s;72℃ 1.5 min,30个循环;72℃ 10 min。PCR产物用胶回收试剂盒纯化。用Nde I和Xho I限制性内切酶酶切pET21a(+)质粒,按ClonExpress® II One Step Cloning Kit说明书将PCR产物与线性化的pET21a(+)载体片段进行重组,转化E. coli DH5α感受态,阳性克隆培养后抽提质粒并测序验证。将正确重组的质粒分别命名为pSXL1和pSXL2,以用于C. glutamicum ATCC 13032和C. glutamicum ZL5天冬氨酸激酶的表达。
1.2.3 天冬氨酸激酶的表达与纯化 将验证正确的重组质粒pSXL1和pSXL2分别转化E. coli BL21(DE3)感受态。将转化菌株过夜活化后,以1%的接种量接种于100 mL含有50 μg/mL Kan的LB培养基,37℃培养至OD为0.6左右,加入终浓度为0.5 mmol/L的异丙基 -β-D-半乳糖苷(IPTG),20℃继续培养24 h诱导蛋白表达。将100 mL菌液高速离心收集菌体,用8 mL缓冲液A(组成:20 mmol/L Tris-HCl,pH7.5,10%(V/V)甘油)重悬细胞,在冰浴条件下用超声波破碎细胞,然后4℃ 12 000 r/min离心15 min,上清即可溶性的酶液。
重组蛋白的纯化利用His标签采用Ni2+-树脂亲和层析进行。首先,用600 μL缓冲液A平衡Ni2+-树脂亲和层析柱,然后加入含有酶液的上清,再分别用600 μL 20 mmol/L与500 mmol/L咪唑 Tris-HCl缓冲液(pH7.5)洗脱杂蛋白和目的蛋白,收集洗脱液。高浓度的咪唑洗脱液在10 kD超滤管中超滤3次,超滤液为缓冲液B(组成:100 mmol/L Tris-HCl,pH7.5,20%(V/V)甘油),超滤条件为4℃条件下7 500 × g离心30 min,除去洗脱液中的咪唑。对纯化后的蛋白进行浓度为12% SDS-PAGE 电泳分析。用 Pierce®BCA Protein Assay Kit对蛋白进行定量[15]。
1.2.4 不同抑制剂对天冬氨酸激酶酶活影响 AK酶活检测方法参照文献[8,16]。标准反应体系为1 mL混合液中包含200 mmol/L Tris-HCl(pH7.5),10 mmol/L MgSO4·6H2O,10 mmol/L Aspa-rtate,10 mmol/L ATP,160 mmol/L NH2OH·HCl(用KOH中和),以及适量的酶。30℃下反应25 min后,在反应体系中加入等体积的5%(W/V)FeCl3溶液终止反应,酶标仪540 nm检测反应体系中生成的天冬氨酸异羟肟酸的吸光值。一个单位天冬氨酸激酶活力定义为:30℃反应条件下,反应体系中每分钟生成1 μmol天冬氨酸异羟肟酸所需的酶量。在30℃,pH7.5条件下,在反应体系中加入不同浓度的赖氨酸或/和苏氨酸,检测残留的酶活力,从而检测酶活的抑制情况。不同抑制剂母液先用KOH调pH到7.5。
1.2.5 赖氨酸高产菌中的天冬氨酸激酶基因回复突变菌株的构建 用于C. glutamicum ZL5菌株基因组上对其天冬氨酸激酶编码基因回复突变的质粒构建过程如图2-A所示。首先,以C. glutamicum ATCC 13032基因组为模板,用引物3和引物4(表1)扩增包含天冬氨酸激酶编码基因及其上下游各1 000 bp的DNA片段,按上述相同的方法将含有C.glutamicum ATCC 13032天冬氨酸激酶编码基因的打靶片段重组至谷氨酸棒杆菌自杀质粒pK18mobsacB中,以构建用于在C. glutamicum ZL5菌株基因组上对其天冬氨酸激酶基因进行回复突变的打靶质粒,测序正确后的质粒命名为pSXL3。
C. glutamicum ZL5感受态的制备参考余秉琦等的方法[17-19]。电击转化条件为2 500 V,2 mm电击杯,电击两次。用电转仪将pSXL3质粒转入ZL5菌株感受态后,经过两次同源臂重组,分别在Kan抗性平板及15%蔗糖平板上进行两次筛选(图2-B),PCR扩增包含突变位点的DNA片段并测序验证,成功回复突变的菌株命名为SXCgL1。
1.2.6 赖氨酸高产菌株与回复突变菌株的赖氨酸摇瓶发酵检测 将C. glutamicum ZL5菌株和回复突变菌株SXCgL1甘油菌在LBG固体平板上划线活化,30℃培养约36 h;将挑取适量菌种转接种到装有20 mL液体LBG培养基的100 mL摇瓶中,30℃,220 r/min培养10-12 h;将活化的菌株按初始OD为0.1接到装有10 mL种子培养基的250 mL摇瓶中,30℃,220 r/min培养12 h;将种子液按5%接种量分别接到装有28.5 mL发酵培养基的500 mL摇瓶中,每个菌株3个平行,30℃,220 r/min培养60 h。每隔一段时间测发酵液的pH,用50%的尿素调节pH在7.0左右。发酵结束后,取1 mL发酵液于1.5 mL离心管中,12 000 r/min离心5 min,取50 μL发酵液稀释20倍,用山东省科学院生产的SBA-40D 生物传感分析仪检测发酵液中残糖和赖氨酸含量。发酵液稀释50倍,用紫外分光光度计检测菌体OD值。
1.2.7 天冬氨酸激酶突变体同源建模与成键分析 以C. glutamicum ATCC 13032来源的天冬氨酸激酶的两种晶体结构为模板,具体模板为无活性的CgAK晶体结构(PDB ID:3AAW)和有活性的CgAK晶体结构(PDB ID:3AB4),利用YASARA软件分别对天冬氨酸激酶野生型和突变体进行同源建模。然后利用PyMOL软件将天冬氨酸激酶突变体G359D的三维结构与C. glutamicum ATCC 13032的天冬氨酸激酶的结构进行拟合与成键分析。

图2 质粒pSXL3构建过程与回复突变菌株SXCgL1菌株构建示意图
2 结果
2.1 天冬氨酸激酶氨基酸突变位点的发现
本实验室前期对一系列野生型与赖氨酸生产谷氨酸棒杆菌进行了二代基因组测序与比较基因组分析。这些菌株中包括从上海市工业微生物研究所菌种保藏中心购买的赖氨酸低产菌C. glutamicum A12和经诱变筛选获得的赖氨酸高产菌C. glutamicum ZL5。由于野生型C. glutamicum ATCC 13032的基因组通常作为参考基因组用于比较基因组分析,因此本研究也从NCBI中提取野生型C. glutamicum ATCC 13032的天冬氨酸激酶编码基因进行分析。通过多序列比对发现C. glutamicum ZL5的天冬氨酸激酶编码氨基酸序列与另外两个来源于野生型C. glutamicum ATCC 13032与C. glutamicum A12天冬氨酸激酶氨基酸序列的一致性为99.3%,仅在359位点存在差异,即赖氨酸高产菌C. glutamicum ZL5中的天冬氨酸激酶存在G359D的单点突变,而C. glutamicum ATCC 13032和C. glutamicum A12的天冬氨酸激酶氨基酸序列完全相同,如图3所示。为方便下文的论述,将C. glutamicum ATCC 13032的天冬氨酸激酶命名为AK-WT,将C. glutamicum ZL5的天冬氨酸激酶命名为AK-G359D。
2.2 天冬氨酸激酶在大肠杆菌中的重组表达与蛋白纯化
为研究G359D点突变的功能,将AK-WT与AK-G359D在大肠杆菌中进行重组表达。首先,利用PCR扩增天冬氨酸激酶AK-WT与AK-G359D编码基因,DNA片段长度1 300 bp左右(图4-A),条带大小与预期相符。然后用ClonExpress® II试剂盒成功将各基因分别重组至表达质粒pET21a(+),构建各重组表达质粒pSXL1和pSXL2。将蛋白表达质粒转入E. coli BL21(DE3)中IPTG诱导表达与蛋白亲和纯化。如图4-B所示,在大肠杆菌中,成功表达并纯化了AK-WT与AK-G359D,其α亚基和β亚基分子质量分别为45.55 kD和19.33 kD,与理论值相符。

图4 天冬氨酸激酶编码基因的PCR(A)与纯化蛋白的SDS-PAGE(B)电泳图
2.3 赖氨酸与苏氨酸对重组天冬氨酸激酶酶活的影响
为研究G359D的突变对天冬氨酸激酶酶活的影响,检测了不同苏氨酸或/和赖氨酸浓度下AK-WT与AK-G359D酶活。结果(图5)显示,AK-WT受苏氨酸与赖氨酸的协同抑制,而AK-G359D则可有效解除苏氨酸与赖氨酸的协同抑制。具体而言,在仅存在苏氨酸时,AK-WT的酶活不受显著抑制,苏氨酸浓度从2 mmol/L提高到10 mmol/L,其酶活维持在92.63%±1.55%与96.22%±2.18%之间;而AK-G359D酶活随着苏氨酸浓度的提高有所下降,由2 mmol/L时的86.10%±3.55%下降到10 mmol/L时的76.46%±3.70%。而仅存在赖氨酸时,AK-WT与AK-G359D酶活受抑制影响情况一致,随着赖氨酸浓度提高酶活都有所下降,10 mmol/L时剩余酶活分别为58.13%±2.60%和59.46%±5.17%。但在苏氨酸与赖氨酸同时存在时,AK-WT与AK-G359D酶活表现出明显差异,AK-WT在两者同时存在时酶活显著下降,2 mmol/L时其酶活仅剩余4.77%±0.81%,10 mmol/L时为4.38%±1.28%,而AK-G359D的酶活则维持在较高的水平,在两者浓度为2 mmol/L时其酶活仍保留了87.23%±0.94%,甚至在两者浓度高达10 mmol/L时其酶活仍剩余76.94%±1.61%。实验结果说明了G359D的突变使天冬氨酸激酶有效解除了苏氨酸和赖氨酸的协同抑制。

图5 不同抑制剂存在时天冬氨酸激酶野生型与突变体的酶活检测结果
2.4 天冬氨酸激酶突变体G359D的回复突变对赖氨酸发酵的影响
为在谷氨酸棒杆菌体内进一步验证G359D的作用,在赖氨酸高产菌C. glutamicum ZL5的基因组上利用同源重组对AK-G359D编码基因进行了回复突变。首先,以C. glutamicum ATCC 13032基因组为模板,扩增了天冬氨酸激酶AK-G359D编码基因回复突变的打靶片段,其理论长度约3 300 bp,如图6-A所示,其条带大小与预期相符。然后重组至自杀性质粒pK18mobsacB中,成功获得回复突变的打靶质粒pSXL3。将该质粒电击转化至赖氨酸高产菌C.glutamicum ZL5中,经过两次同源臂交换后,随机选取4个转化子提取基因组。利用基因组验证引物5与引物6(表1)进行基因组的PCR验证,结果扩增得到600 bp的目的片段,条带大小与预期相符,如图6-B所示。将扩增出的基因片段进行测序发现,lysC-1与lysC-3成功发生回复突变(图6-C),即由天冬氨酸激酶AK-G359D编码基因回复成天冬氨酸激酶AK-WT编码基因。选择将转化子lysC-1命名为SXCgL1,进行后续的赖氨酸发酵验证。发酵结果(图7)所示,赖氨酸高产菌C. glutamicum ZL5与回复突变菌株SXCgL1在生长上没有差异,但在赖氨酸的产量上具有显著差异。具体而言,在摇瓶发酵60 h后,高产菌C. glutamicum ZL5与回复突变菌株SXCgL1的OD分别为31.43±1.35与31.35±0.21,但赖氨酸产量分别为13.16±0.29 g/L与11.11±0.13 g/L,即天冬氨酸激酶AK-G359编码基因的回复突变导致赖氨酸的产量下降15.57%。
由于C. glutamicum A12的天冬氨酸激酶基因与C.glutamicum ZL5天冬氨酸激酶基因仅存在1个碱基的突变,即导致G359D的突变,无其它碱基的突变。而C.glutamicum ATCC13032的天冬氨酸激酶基因与C. glutamicum ZL5天冬氨酸激酶基因存在15个碱基的突变,除导致G359D的突变,还存在其他14个位点的同义突变。因此,本研究进一步以C. glutamicum A12基因组为模板,扩增了其基因序列(图6-A),并对C. glutamicum ZL5天冬氨酸激酶基因进行替换,以进行回复突变。经基因组PCR验证与测序可见,成功获得仅对G359D相对应的1076碱基进行了回复突变,由A突变为G,而其他位点并未发生突变(图6-D)。将单点回复突变的转化子lysC*-2命名为SXCgL2,并进行后续赖氨酸发酵验证。发酵结果如图7所示,回复突变菌株SXCgL2的验证结果与SXCgL1无明显差异。在摇瓶发酵60 h后,其OD为30.77±0.99,赖氨酸产量为11.33±0.12 g/L,与SXCgL1的生长与赖氨酸产量相差不大。
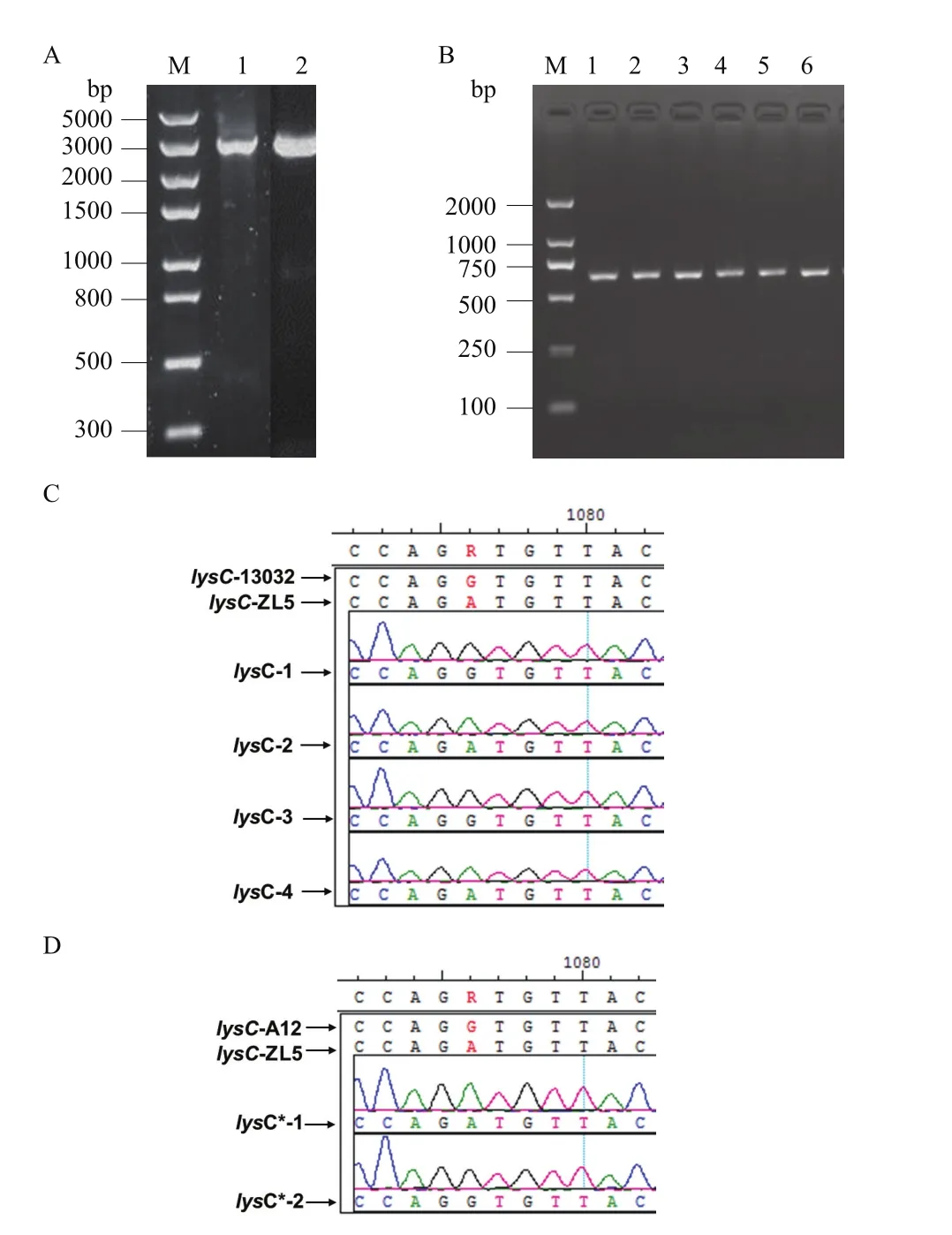
图6 C. glutamicum ZL5菌株天冬氨酸激酶回复突变菌株构建构建过程中电泳验证与测序结果
2.5 天冬氨酸激酶突变体G359D的结构分析
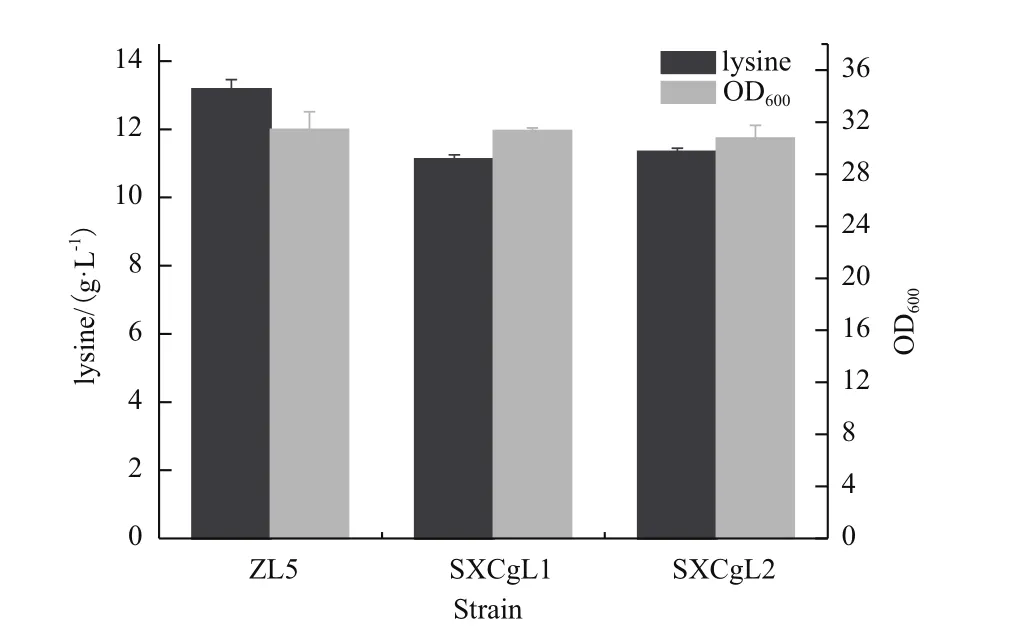
图7 C. glutamicum ZL5和回复突变菌株SXCgL1菌株赖氨酸摇瓶发酵结果
为进一步揭示AK-G359D解除赖氨酸和苏氨酸协同抑制的机制,首先以C. glutamicum ATCC 13032来源的天冬氨酸激酶的晶体结构3AAW与3AB4为模板,模拟构建了AK-G359D与赖氨酸和苏氨酸结合状态的三维结构,如图8-A中品红色结构所示。然后,将该结构与AK-WT的结构(如图8-A中蓝绿色结构所示)进行了拟合分析,结果表明,虽然两个酶均可结合赖氨酸和苏氨酸,但其在活性位点附近的β-折叠的组成和构象存在显著差异。具体而言,在AK-WT中,赖氨酸的结合使来源于α亚基的一个β-折叠(β5(α),其C末端有Arg151)靠近活性中心,导致Arg151与底物结合位点Glu74之间形成双配位基离子键,阻止了底物进入,使酶处于非活性状态,如图8-B与图9-A所示。而在AK-G359D中,该区域存在两个β-折叠,且偏离活性中心的位置,其Arg151与Glu74之间无法形成离子键,使酶处于开放状态,允许底物分子的结合,如图8-B与图9-B所示。
3 讨论
天冬氨酸激酶是天冬氨酸家族氨基酸合成中非常关键的限速酶,但其往往受到代谢产物的反馈抑制。天冬氨酸激酶解除抑制的突变改造与机制解析对提升相关氨基酸的合成具有重要的指导意义。在前期研究中,Yoshida等[12]通过结构分析发现Ile44(β)、Val360(α)、Thr361(α)、Thr361(α)、Gly359(α)和Ile42(β)直接或间接参与了赖氨酸的结合,这些位点的突变可能有效解除抑制。但当Yoshida等[13]将CgAK中Gly359突变为丙氨酸,发现该突变体却未能解除苏氨酸和赖氨酸的协同抑制,其原因可能在于丙氨酸与甘氨酸结构较为相近,其侧链结构的改变对解除抑制的贡献较小。而朱运明等[14]将来自于北京棒杆菌CpAK中与CgAK的Gly359对应的Gly377位点进行突变后,发现CpAK-G377F突变体解除苏氨酸和赖氨酸的协同抑制,但在高浓度下其解除效果不太理想,至10 mmol/L时其酶活仅残留24.81%。针对上述问题,本文通过对来自野生型C. glutamicum ATCC 13032和赖氨酸高产菌C.glutamicum ZL5的AK蛋白比较发现,高产菌株AK中存在G359D的单点突变。体外重组酶的酶活检测发现,该G359D突变体可有效解除苏氨酸和赖氨酸的协同抑制,即使高浓度至10 mmol/L时,其酶活可保留至76.94%。对比可见,本研究发现的突变体解除苏氨酸和赖氨酸协同抑制的能力显著高于目前已报道的水平,即Gly359突变为酸性氨基酸时可高效解除协同抑制。

A:AK-WT与AK-G359D的三维结构拟合图(蓝绿色的为AK-WT的三维结构,品红色的为AK-G359D的三维结构,红圈圈出的为AK的活性中心);B:天冬氨酸激酶活性中心放大效果图(大的虚线红圈及红色弧形箭头标明了AK-WT与AK-G359D的α亚基中β5(含Arg151)所在loop的变化,小的虚线红圈及红色弧形箭头标明了AK-WT与AK-G359D的α亚基中Arg151侧链方向的变化;红色箭头指向的是AK-WT中α亚基上Arg151与Glu74之间形成的双配位基离子键)
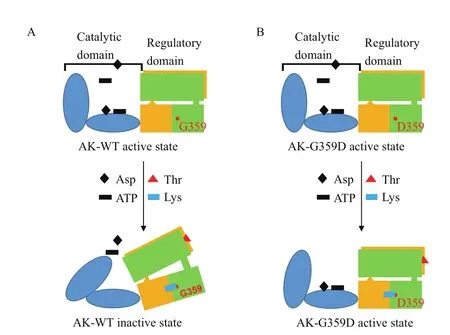
图8 天冬氨酸激酶野生型和突变体的三维结构比对结果
为什么G359D突变体能有效解除赖氨酸和苏氨酸的协同抑制呢?Yoshida等[12]通过对CgAKs几种晶体结构的解析发现,当CgAK不与抑制剂结合时,其结构比较松弛(CgAK-R),处于有活性状态;而当在苏氨酸和赖氨酸同时存在时,将发生明显的别构而处于紧张状态(CgAK-T),处于无活性构象。具体机制为苏氨酸的结合后引起α亚基的调节区域和β亚基的相互作用,然后赖氨酸的结合将进一步导致β-折叠构象的改变,使β5(α)C末端的Arg151与底物结合位点Glu74之间形成双配位基离子键,最终导致酶变为CgAK-T状态。Arg151与Glu74之间的离子键既阻止了底物天冬氨酸的结合,又稳定了CgAK-T的非活性状态。本实验室在野生酶的研究基础上,重建了AK-G359D突变体与赖氨酸和苏氨酸结合状态时的三维结构,发现了AK-G359D突变体解除协同抑制的分子机理。虽然G359D的突变并没有解除其与赖氨酸之间的非共价键结合,但是与野生型酶相比,其活性中心附近的结构发生了明显变化。比如,β5(α)处于远离活性中心的位置从而使位于β5(α)C末端的Arg151无法与底物结合位点Glu74之间离子键,因此有效阻断了赖氨酸与苏氨酸结合引起催化中心的别构变化而解除其协同抑制。
为什么AK-G359D没有解除赖氨酸单独存在时的抑制,但能有效解除赖氨酸与苏氨酸的协同抑制?类似情况也在Chen等[9]通过协同共进化发现的E328A突变体出现。结合酶活测定结果与蛋白结构的比对分析发现,野生型CgAK是α2β2异源四聚体[12],α亚基的N-末端为催化域,β亚基与α亚基的C-末端构成了调节域。当赖氨酸单独存在时,赖氨酸会与α亚基的C-末端结合并使其发生别构效应,而与催化域之间形成了弱结合,导致赖氨酸对酶的部分抑制。Yoshida等[12]还发现赖氨酸是天冬氨酸的竞争性抑制剂。另外,G359位点本身是通过水分子的氢键作用而稳定赖氨酸的结合。因此,在AKG359D中,虽然解除了G359与水分子间形成的氢键,但仍然可与赖氨酸结合,赖氨酸导致的别构抑制与竞争性抑制仍然存在,因此赖氨酸对AK-G359D和AK-WT的抑制效果无显著差异。当苏氨酸单独存在时,苏氨酸使β亚基与α亚基的C-末端结合而形成完整的调节域,但并不引起别构变化,使酶仍为活性状态。但AK-G359D在高浓度苏氨酸存在时酶活却略有下降,表明该位点的突变可能导致苏氨酸结合后调节域的构象改变而影响催化域的结构。但是当苏氨酸与赖氨酸共同存在时,野生型AK-WT中,苏氨酸结合后将强化赖氨酸与酶的结合,使调节域与N端的催化域之间形成强结合,酶处于完全抑制状态,使酶活非常低。但在AK-G359D突变体中,苏氨酸结合后调节域构象改变,而无法加强赖氨酸的结合,使调节域不能再与催化域间形成强结合,而有效解除由赖氨酸导致的别构抑制。
4 结论
本研究通过对野生型C. glutamicum ATCC 13032和赖氨酸高产菌C. glutamicum ZL5天冬氨酸激酶氨基酸序列进行比对,发现C. glutamicum ZL5的天冬氨酸激酶中仅存在G359D的单点突变。体外纯酶酶活检测发现天冬氨酸激酶G359D突变体有效解除了赖氨酸与苏氨酸协同抑制;在C. glutamicum ZL5体内将天冬氨酸激酶G359D回复突变后发现赖氨酸产量下降,再次证明了天冬氨酸激酶G359D的突变是非常重要的突变。突变体和野生型的天冬氨酸激酶的三维结构比对和成键分析发现,G359D的突变使Arg151与底物结合位点Glu74之间无法形成离子键,从而解除抑制剂的协同抑制。
致谢:衷心感谢曹国强老师与李庆刚老师在实验设计方面给的帮助,感谢刘娇老师在酶学性质研究过程中给予的指导。
[1]Bommareddy RR, Chen Z, Rappert S, et al. A de novo NADPH generation pathway for improving lysine production of Corynebacterium glutamicum by rational design of the coenzyme specificity of glyceraldehyde 3-phosphate dehydrogenase[J].Metab Eng, 2014, 25:30-37.
[2]Pérez-García F, Peters-Wendisch P, Wendisch F. Engineering Corynebacterium glutamicum for fast production of L-lysine and L-pipecolic acid[J]. Appl Microbiol Biot, 2016, 100(18):8075-8090.
[3]Jakobsen M, Brautaset T, Degnes KF, et al. Overexpression of wild-type aspartokinase increases L-lysine production in the thermotolerant methylotrophic bacterium Bacillus methanolicus[J]. Appl Environ Microbiol, 2009, 75(3):652-661.
[4]Xu J, Zhang J, Guo Y, et al. Improvement of cell growth and L-lysine production by genetically modified Corynebacterium glutamicum during growth on molasses[J]. J Ind Microbiol Biot, 2013, 40(12):1423-1432.
[5]Sagong HY, Kim KJ. Structural basis for redox sensitivity in Corynebacterium glutamicum diaminopimelate epimerase:an enzyme involved in l-lysine biosynthesis[J]. Sci Rep-UK, 2017, 7:1-13.
[6]Eggeling L, Bott M. A giant market and a powerful metabolism:L-lysine provided by Corynebacterium glutamicum[J]. Appl Microbiol Biot, 2015, 99(8):3387-3394.
[7]Takeno S, Hori K, Ohtani S, et al. L-Lysine production independent of the oxidative pentose phosphate pathway by Corynebacterium glutamicum with the Streptococcus mutans gapN gene[J]. Metab Eng, 2016, 37:1-10.
[8]Dong X, Zhao Y, Zhao J, et al. Characterization of aspartate kinase and homoserine dehydrogenase from Corynebacterium glutamicum IWJ001 and systematic investigation of l-isoleucine biosynthesis[J]. J Ind Microbiol Biot, 2016, 43(6):873-885.
[9]Chen Z, Meyer W, Rappert S, et al. Coevolutionary analysis enabled rational deregulation of allosteric enzyme inhibition in Corynebacterium glutamicum for lysine production[J]. Appl Environ Microb, 2011, 77(13):4352-4360.
[10]Ma CW, Xiu ZL, Zeng AP. Exploring signal transduction in heteromultimeric protein based on energy dissipation model[J].J Biomol Struct Dyn, 2015, 33(1):134-146.
[11]Tsujimoto M, Yoshida A, Shimizu T, et al. Aspartate kinase involved in 4-hydroxy-3-nitrosobenzamide biosynthesis in Streptomyces murayamaensis[J]. Biosci Biotech Bioch, 2016, 80(11):2255-2263.
[12]Yoshida A, Tomita T, Kuzuyama T, et al. Mechanism of concerted inhibition of α2β2-type hetero-oligomeric aspartate kinase from Corynebacterium glutamicum[J]. J Biol Chem, 2010, 285(35):27477-27486.
[13]Yoshida A, Tomita T, et al. Structural Insight into concerted inhibition of alpha 2 beta 2-type aspartate kinase from Corynebacterium glutamicum[J]. J Mol Biol, 2007, 368(2):521-536.
[14]朱运明, 王晓飞, 等. 北京棒杆菌天冬氨酸激酶 G377 定点突变及酶学性质表征[J]. 食品科学, 2014, 35(9):192-197.
[15]Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of proteindye binding[J]. Anal Biochem, 1976, 72:248-254.
[16]Min W, Li H, Li H, et al. Characterization of Aspartate Kinase from Corynebacterium pekinense and the Critical Site of Arg169[J].Int J Mol Sci, 2015, 16(12):28270-28284.
[17]余秉琦, 沈微, 诸葛健. 适用于异源DNA高效整合转化的谷氨酸棒杆菌电转化法[J]. 中国生物工程杂志, 2005(2):78-81.
[18]Van der Rest ME, Lange C, Molenaar D. A heat shock following electroporation induces highly efficient transformation of Corynebacterium glutamicum with xenogeneic plasmid DNA[J].Appl Microbiol Biot, 1999, 52(4):541-545.
[19]Anusree M, Wendisch VF, Nampoothiri KM. Co-expression of endoglucanase and β-glucosidase in Corynebacterium glutamicum DM1729 towards direct lysine fermentation from cellulose[J].Bioresource Technol, 2016, 213:239-244.
Aspartokinase G359D from Corynebacterium glutamicum Relieves the Synergistic Inhibition of Lysine and Threonine
XU De-yu1,2ZHENG Xiao-mei2ZHAO Jing2ZHENG Ping2ZHAO Shu-xin1
(1. College of Biotechnology,Tianjin University of Science and Technology,Tianjin 300457;2. Tianjin Institute of Industrial Biotechnology,Chinese Academy of Sciences,Tianjin 300308)
Aspartokinase(AK)is a key enzyme involved in the lysine biosynthesis,but its activity is synergistically inhibited by end-products such as lysine and threonine. This study focuses on finding new mutation to relieve the synergistic inhibition and unveiling this molecular mechanism form protein structure analysis. The amino acid sequences of AK from high lysine-producing strain Corynebacterium glutamicum ZL5 and wild-type(WT)C. glutamicum ATCC 13032 were aligned,and it was shown that G359D mutation was detected in aspartokinase from C. glutamicum ZL5. To discovery the biological function of this mutation,these WT and G359D Aks were expressed in Escherichia coli and were purified by the affinity chromatography;then,the purified recombined proteins were used for enzymatic activity detection when added the inhibitors lysine and threonine. The G359D protein displayed high resistance against the synergistic inhibition of lysine and threonine. The enzymatic activity of G359D remained at 76.94%±1.61% when the concentration of lysine and threonine reached 10 mmol/L,but the WT protein only was in 4.38%±1.28%. The similar result was also verified by the reverse mutation of G359D in the genome of C. glutamicum ZL5 producing high lysine yield,and the lysine yield decreased by 15.57%. From the further homology modeling and protein structure analysis,the G359D still interacted with lysine and threonine,but it enabled to relieve the allosteric effect of lysine,resulting from that the Arg151and Glu74in the catalytic active site of G359D could not form the ionic bond,thus allow the substrate into the active site. The aspartokinases G359D enabled to relieve the synergistic inhibition of lysine and threonine by blocking the allosteric effect of lysine.
Corynebacterium glutamicum;aspartokinase;lysine;threonine;synergistic inhibition
10.13560/j.cnki.biotech.bull.1985.2017-0387
2017-05-11
中国科学院重点部署项目(ZDRW-ZS-2016-2)
徐德雨,男,硕士研究生,研究方向:轻工技术与工程(发酵工程);E-mail:xu_dy@tib.cas.cn
郑平,女,研究员,硕士生导师,研究方向:细菌的代谢工程改造和系统生物学;E-mail:zheng_p@tib.cas.cn赵树欣,女,教授,硕士生导师,研究方向:色素红曲以及现代酿造技术;E-mail:zsx999@tust.edu.cn
(责任编辑 朱琳峰)

