基于粪便DNA的小麂亲权鉴定和婚配制研究
任 鹏,龚 堃,鲍毅新,黄相相, 周 晓, 韩金巧
浙江师范大学生态研究所, 金华 321004
基于粪便DNA的小麂亲权鉴定和婚配制研究
任 鹏,龚 堃,鲍毅新*,黄相相, 周 晓, 韩金巧
浙江师范大学生态研究所, 金华 321004
2014年4月至2015年1月,在古田山国家级自然保护区内共收集634份粪便样本,2份肌肉样本。通过严格筛选,最终获得390份可用于PCR扩增的样本。用多态性较高的8个微卫星位点进行基因型分型,共识别出177个小麂个体。SRY基因性别鉴定显示研究样本中雄性84只,雌性93只。所使用的8个微卫星位点在177个样本中,平均等位基因数(A)为11,平均观测杂合度(Ho)在0.960—1.000之间,平均值为0.9685,平均期望杂合度(He)在0.799—0.887之间,平均值为0.8429,多态信息含量(PIC)在0.766—0.872之间,平均多态信息含量为0.8214,基因杂合度水平较高,为遗传多样性丰富的种群。采用Cervus3.0进行亲权分析,当置信度为95%和80%时,8个微卫星位点的鉴定率均达到100%。共鉴定出父-母-子24对,母-子23对,父-子19对,涉及到104只个体。根据亲缘关系分析小麂的婚配制,结果发现小麂的婚配制属于1雄多雌,但并不是目前所知的亚型,而可能是一种被称作“检查策略”的一雄多雌制。
小麂;亲权鉴定;遗传多样性;婚配制
小麂(Muntiacusreevesi)属偶蹄目(Artiodactyla)鹿科(Cervidae)麂属(Muntiacus),是我国特有的小型鹿科动物,广泛分布于中国中部和南部地区[1],有重要的经济价值。2008年国际自然与自然资源保护联盟(IUCN)濒危物种红色名录将其列入低危(Least concerned,LC)级[2]。由于营独居生活的鹿科动物不仅活动范围大,且多生活在密林中,在野外难以直接观察,所以鹿科动物的社会结构和婚配制的研究较少,且主要是在圈养状态下直接观察[3],或通过无线电遥测技术进行研究[4]。目前对小麂的研究主要集中在宏观生态学[5]、细胞遗传学[6]和进化生物学[7]等方面。近年来,随着野生动物非损伤性取样(noninvasive sampling)技术的应用,粪便样品以其易于采集的特点而成为最具潜在价值的研究材料[8]。利用粪便DNA结合微卫星标记在进行鹿科动物个体识别[9]、亲权鉴定[10]、遗传多样性[11]、家域[12]等方面得到了广泛应用。目前通过微卫星标记来研究婚配制的报道有很多,如北极熊(Ursusmaritimus)[13]、宽吻凯门鳄(Caimanlatirostris)[14]、扇尾噪刺莺(Gerygoneflavolateralis)[15]等。用粪便DNA来研究婚配制的报道较少,仅见黑犀牛(Dicerosbicornis)[16]、黑麂(Muntiacuscrinifrons)[17]。这使得分子生物学的手段成为研究小麂婚配制的新视角,同时可以进一步分析小麂种群的遗传多样性、种群内成员之间的关系、物种的进化潜力和抵抗不良环境的能力、繁殖策略等。
本文基于小麂粪便样本,通过微卫星标记进行个体识别和性别鉴定,对小麂进行亲权鉴定,分析小麂种群的遗传多样性,探讨小麂的婚配制度,以及婚配制对维持种群遗传多样性的作用,为其它物种进行婚配制的研究提供了新的研究视角,对制定有效的保护和管理策略有重要意义。
1 材料和方法
1.1 样本的采集
本研究的采样点是古田山国家级自然保护区,位于浙江省衢州市开化县苏庄镇西北,与江西婺源、德兴毗邻,约在118°03′49.7″—118°11′12.2″E,29°10′19.4″—29°17′41.4″N之间,总面积为81.07 km2。主峰青尖海拔1258 m,属中亚热带季风候带,有明显的季节变化。年均降水量1963.7 mm,年均温度15.3℃,无霜期约为250 d。分布着典型的中亚热带常绿阔叶林,小麂种群资源丰富,具有一定的代表性。为了最大限度地收集保护区内的小麂新鲜粪便,分别于2014年4月(春)、7月(夏)、10月(秋)和2015年1月(冬)进行了4次重复采样,采样样线的设置参考郑祥[16]等的研究,在核心区(5条)、缓冲区(2条)、实验区(4条)共设置11条样带。样带单侧宽度为10 m,长度在3—6 km不等,相互之间的间距大于1500 m,避免了单次采样获得小麂的粪便样本较少。在调查区域内,成年黑麂和小麂的粪便较易区分,容易混淆的是小麂和黑麂幼龄个体排出的粪便,由于幼龄个体的肠道发育不成熟,小麂和黑麂排出的粪便差异不大,很容易误判,在采样时,对于那些幼龄个体的粪便,如果完全无法判断,则舍弃,不采集。采样时使用一次性灭菌手套,将粪便样品放入盛有无水乙醇的灭菌封口袋中,标注采集时间、地点、海拔和经纬度等,带回实验室-20℃保存。4次采样共获得634份粪便样本,并从当地获得两份肌肉样本(一雌一雄)。
1.2 基因组DNA的提取和PCR扩增
1.2.1 DNA的提取
粪便和肌肉组织分别使用OMEGA-Stool DNA Kit试剂盒和酚氯仿抽提法[18],提取的DNA样本保存于-20℃,用于后期的PCR实验。在根据粪便形态初步鉴定的基础上,利用线粒体细胞色素b基因(Cytb)进行验证[19],作为物种鉴定的补充,该方法作为分子标记进行物种鉴定具有简单、快速、准确等优点,已在哺乳动物中广泛应用[20- 21]。
1.2.2 微卫星引物的确定和PCR扩增
本研究所用引物和PCR反应体系参照Wang等[22-23]的研究,经多次实验后选定8个扩增稳定且多态信息含量高的微卫星位点(表1),进行小麂的微卫星DNA分型分析,分别选用TAMRA、FAM和HEX进行荧光标记(由上海生工合成)。对SRY性别决定基因进行扩增时,为减少假阴性对实验的影响,对每个样本的SRY基因进行3次扩增,出现条带在2次以上的判断为雄性,SRY引物及扩增体系参考鲁晓瑄等[24]的研究。

表1 8个微卫星引物序列
1.3 数据分析
微卫星位点的扩增产物,用ABI 3700 DNA序列测定仪进行分型(上海生工)。分型结果使用GeneMarker 1.91软件读取,通过对比由8个微卫星标记基因型组合成的指纹图谱,对小麂进行个体识别[25]。利用Cervus 3.0计算等位基因数(A)、平均观测杂合度(Ho)、平均期望杂合度(He)、亲本排除概率(E- 1P;E- 2P;E-PP)、多态信息含量(PIC)和Hardy-Weinberg平衡检测,并进行亲权鉴定[26]。由于实验样本的父本和母本均未知,因此以所有识别出的雄性为候选父本、所有雌性为候选母本,取样概率均为1.0,使用Kingroup v2[19,27]计算个体间的亲缘系数(r),作为亲权鉴定的补充,运用POPGENE 32[28]计算种群的近交系数Fis值。
2 结果
2.1 个体识别和亲权鉴定
2.1.1 性别鉴定和个体识别
643份小麂粪便样本中有442份可以多次稳定提取DNA(重复次数≥3次)且能扩增获得PCR产物,经1.0%琼脂糖凝胶电泳检测,部分结果如图1所示。通过对比可知,等体积的粪便DNA和肌肉DNA电泳条带亮度差异较小,粪便中DNA的获得量能满足后续PCR实验的需求。为进一步证实采集的样本为小麂,随机抽取100份粪便DNA样本,PCR扩增线粒体细胞b基因(Cytb),将PCR产物进行测序,结果表明,除3份样本测序失败外,97%的样本均与数据库中的小麂Cytb基因片段有极高的相似度,均达到99%以上。
对442份粪便DNA样本进行微卫星位点的扩增R产物进行检测,舍弃那些位点数≤5的样本,最终保留390份样本进行Sry性别决定基因的扩增,同时以两份肌肉样本(性别已知)作为对照,用2%的琼糖凝胶电泳检测,目标片段大小为220bp[24],部分结果如图1。共鉴定出177个小麂个体,其中雄性84只,雌性93只。
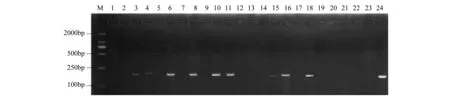
图1 小麂Sry基因PCR结果Fig.1 The PCR amplification results of Sry gene in Muntiacus reevesi1—22:不同粪便样本DNA;23:雌性小麂肌肉DNA;24:雄性小麂肌肉DN A
2.1.2 微卫星位点遗传多样性分析
分析8个微卫星位点在177个个体中的遗传信息参数(表 2),结果表明,平均等位基因数(A)为11.00,多态信息含量(PIC)在0.766—0.872之间,平均多态信息含量为0.8214,属于高度多态位点(PIC>0.5);观测杂合度(Ho)在0.960—1.000之间,平均值为0.9685,期望杂合度(He)在0.799—0.887之间,平均值为0.8429,Ho>He。Fis均为负值,平均值为-0.1886;基因杂合度水平较高,无效等位基因(F(Null))频率极低,平均为-0.0779。8个位点的累积双亲排除概率大于99.99%。Hardy-Weinberg平衡检测结果显示微卫星位点Mreg260、Mre61和Mreg283均偏离平衡,其它5个位点均符合。

表2 8个微卫星位点在177个小麂中的遗传信息参数分析
A:位点等位基因数allele number of 1oci;N:样本数sample sizes;Ho:位点观测杂合度observed heterozygosiry;He:位点期望杂合度heterozygosiry;PIC:位点多态信息含量polymorphic informafion content;E- 1P:第1个亲本的排除概率exclusion probability of the first parent;E- 2P:第2个亲本的排除概率exclusion probability of the second parent;E-PP:双亲的排除概率exclusion probability of parent pair;Fis:近交系数的F-统计量检测值inbreeding coefficient with F-statistics;HWE:哈迪温伯格平衡Hardy-Weinberg equilibrium;F(Null):无效等位基因频率frequency with null;NS:不显著偏离no significant deviation;*显著偏离the locus which showed deviation from Hardy-Weinberg equilibrium (P<0.05)
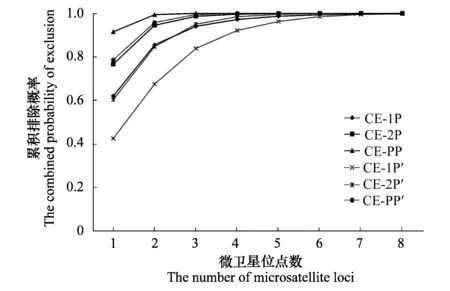
图2 微卫星位点数与累积排除概率之间的关系 Fig.2 Relationship between the combined probability of exculotion and the number of microsatellite lociCE- 1P,CE- 2P,CE-PP:PIC由高到低排列的累积排除概率;CE- 1P′,CE- 2P′,CE-PP′:PIC由低到高排列的累积排除概率
2.1.3 微卫星位点的亲权排除率分析
根据8个微卫星位点的多态信息含量(PIC)大小,按由高到低和由低到高的顺序进行组合,依次增加微卫星位点数进行累积排除率分析,由图2可知,当微卫星位点达到6个时,不论PIC是由高到低还是或由低到高,累积排除概率除E- 1P′为0.9845外,其余均大于0.9900,说明所选的8个微卫星位点可以对亲权关系进行准确的判定。
2.1.4 亲权鉴定
通过Cervus3.0软件的Parent pair(Sex knowm)功能进行亲权鉴定,结果见附录1和附录2。共鉴定出父-母-子24对,母-子24对,父-子18对,所涉及到的104只个体隶属于26个系谱,另外73只个体没有鉴定出它们的亲子代关系。42对父-子的亲缘系数(r)值在0.5044—0.6332之间,平均值为0.5273,48对母-子在0.5044—0.6083之间,平均值为0.5359(表3)。由于系谱1、2和3较为复杂,画出了亲缘关系结构示意图(图3)。
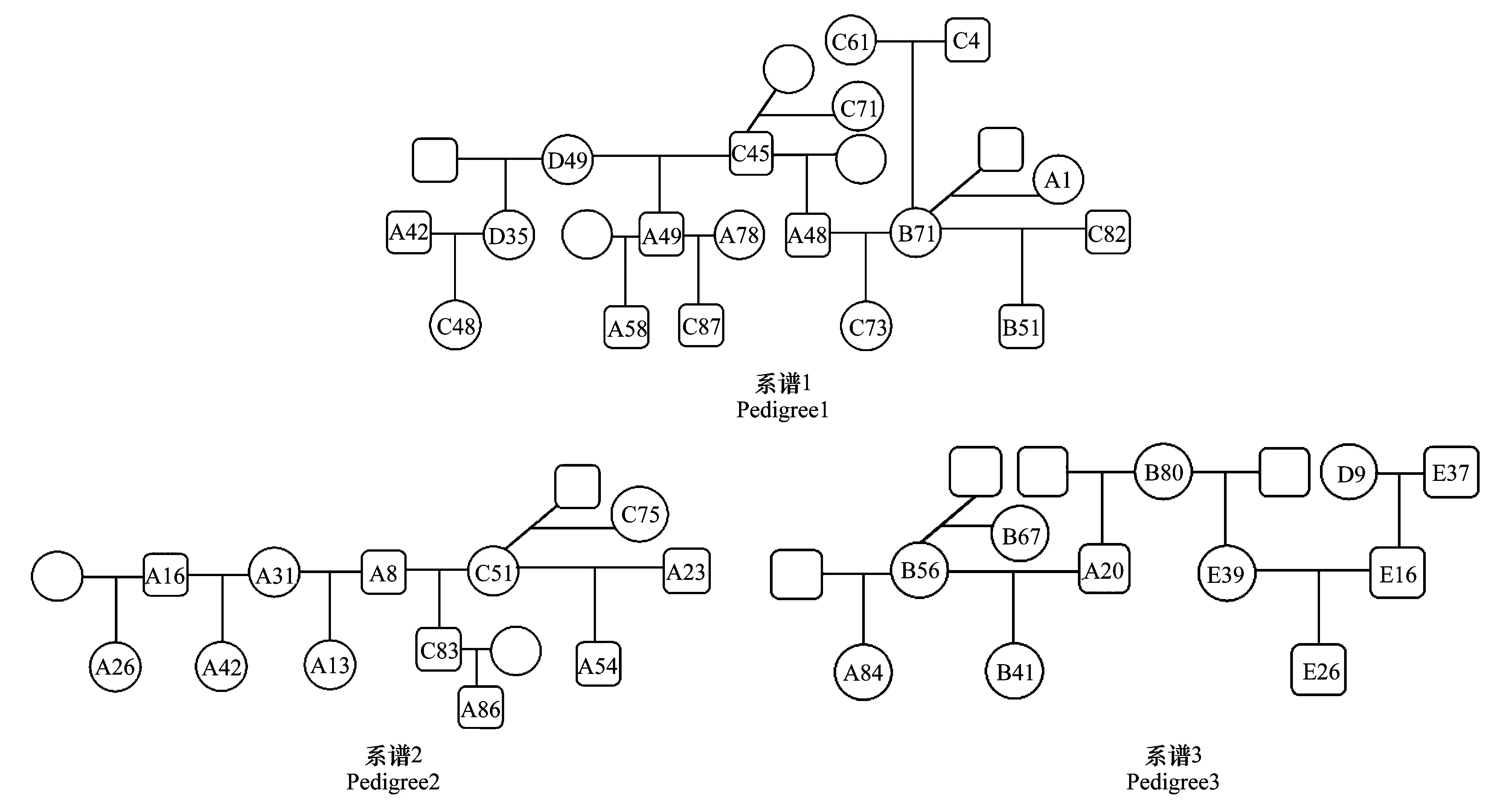
图3 系谱1、2和3的亲缘关系结构示意图Fig.3 Genetic pedigree chart of 1, 2 and 3
2.1.5 婚配制度
根据表3的系谱分析,可知小麂的婚配关系:1个雌性和3个雄性发生过交配的现象出现2次(家系1,3),1个雄性和3个雌性发生交配的现象出现3次(家系1,2,4),1个雌性和2个雄性发生过交配的现象出现9次(家系1,2,3,5,7,8,9,15),1个雄性和2个雌性发生交配的现象出现5次(家系1,2,6,16,18),其余均为1雄1雌。依据表3中的系谱,计算1个雌性和多个雄性交配以及1个雄性和多个雌性交配所产生后代之间的亲缘系数,结果表明,这些后代之间的亲缘系数(r)值在0.2759—0.3844之间,平均值为0.3064(表4)。由1个雄性和多个雌性发生交配的现象,可知小麂的婚配制仍属一雄多雌。
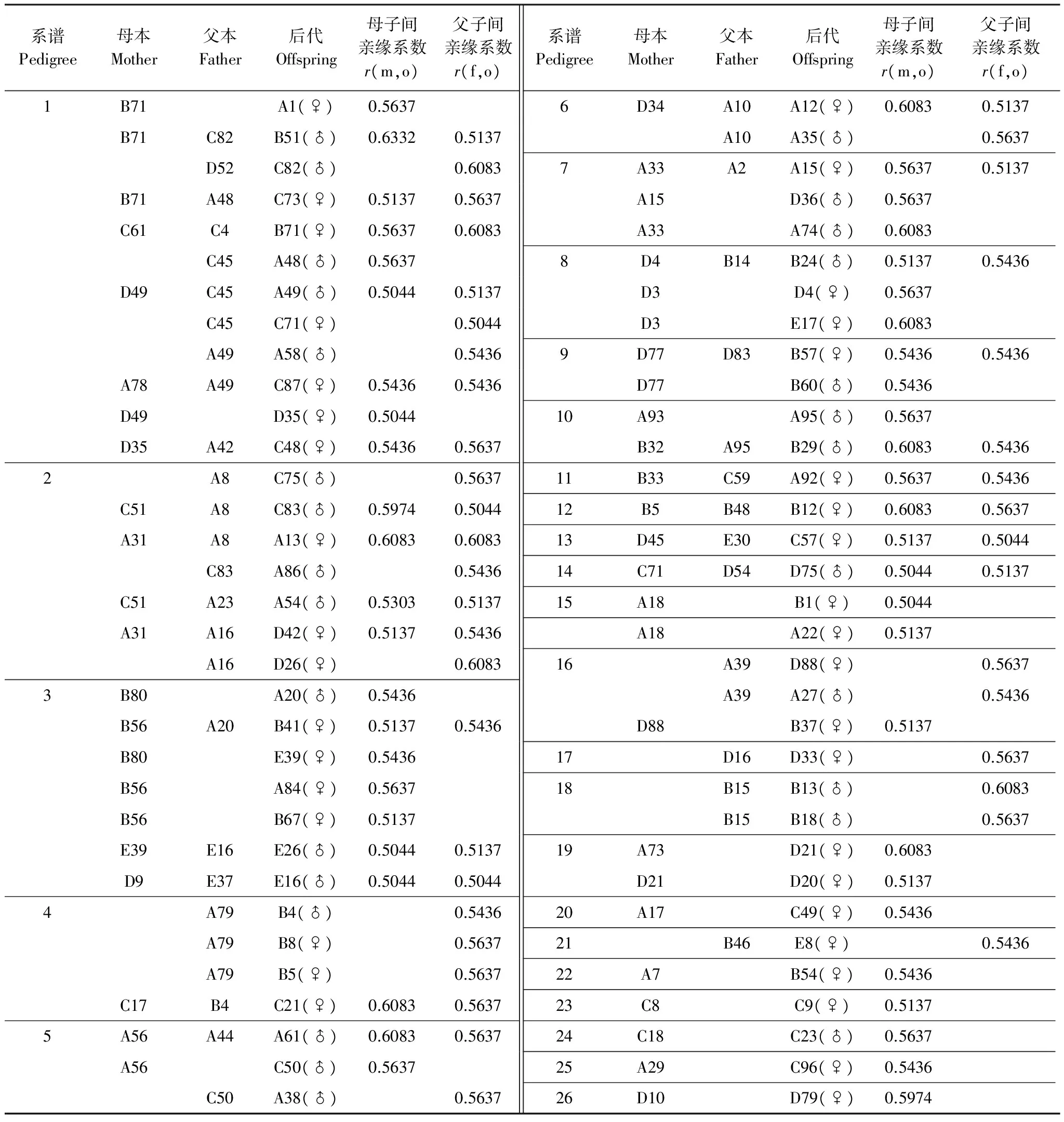
表3 小麂的亲子关系和亲缘系数
A,B,C,D,E为样本的采样编号;r(m,o):母子间亲缘系数the relationship coefficient between mother and offspring;r(f,o):父子间亲缘系数the relationship coefficient between father and offspring

表4 后代之间的亲缘系数
3 讨论
3.1 亲权鉴定和遗传多样性分析
研究表明,利用微卫星标记进行亲权鉴定时,其排除非亲本的能力依赖于微卫星位点数和等位基因的多样性,随着微卫星位点数的增多,排除概率随之增高[29]。本研究所选用的8个微卫星位点共获得88个等位基因位点,平均等位基因数(A)为11.00;多态信息含量PIC是衡量基因变异程度高低的指标,当PIC>0.5时,该基因座位为高度多态性位点[30],本研究中8个微卫星标记的多态信息含量范围在0.767—0.873之间,平均为0.823,均为高度多态位点。与其它鹿科动物相比,如麋鹿(Elaphurusdavidianus)[31]的平均等位基因数为2.11,PIC在0.053—0.519之间,平均为0.303、黑麂(Muntiacuscrinifrons)[32]的平均等位基因数为8.88,PIC在0.523—0.803之间,平均为0.661、马鹿(Cervuselaphus)[11]的平均等位基因数为9.00,PIC在0.53—0.78之间,平均值为0.69、白尾鹿(Odocoileusvirginianus)[33]的平均等位基因数为8.38,PIC在0.356—0.898之间,平均值为0.684、梅花鹿(Cervusnippon)[10]的平均等位基因数为5.93,PIC在0.217—0.825之间、平均值为0.606。遗传多样性是指种内基因的变化,包括种内显著不同种群和同一种群内不同个体之间的遗传变异,是物种进化的潜力[11]。本研究选用的8个微卫星位点在古田山小麂种群中检测出丰富的遗传多样性水平。基因杂合度又称基因多样度,一般认为它是度量群体遗传变异的一个最适参数[34],其值越大则说明群体遗传变异越大。在鹿科动物中麋鹿(Elaphurusdavidianus)[31]的He为0.056—0.598,平均为0.374;黑麂(Muntiacuscrinifrons)[32]为0.680—0.836,平均为0.78;马鹿(Cervuselaphus)[11]为0.61—0.81,平均为0.74;白尾鹿(Odocoileusvirginianus)[33]为0.289—0.919,平均为0.723;梅花鹿[10](Cervusnippon)为0.257—0.863,平均为0.685。而研究中的8个微卫星位点,平均期望杂合度(He)在0.799—0.887之间,平均为0.8429,与其它鹿科动物相比,杂合度较高。由此可知,和上述5种鹿科动物相比,小麂的等位基因数和PIC值均较高,表明本研究所选的8个微卫星位点具有较高的使用价值,同时也说明古田山小麂种群含有丰富的遗传多样性。小麂的遗传多样性高于其他鹿科动物,这也是其分布广泛,生存能力和适应性强的原因所在。
无效等位基因是应用微卫星DNA进行亲权鉴定中影响其准确性的主要因素[10],本研究所使用的8个微卫星位点,杂合子丰富,其无效等位基因频率极低,所有实验样品,包括3个偏离Hardy-Weinberg 平衡的位点,至少均能稳定扩增出5个等位基因,因此无效等位基因的频率不会影响到鉴定的准确性。在Cervus的模拟分析中,从8个微卫星位点中随机选取,当位点数达到6个时,从93个雌性小麂中找到其母本和从84个雄性中找出其父本的概率均超过99%,为提高鉴定的准确性, 将8个位点全部用于亲权鉴定分析。在实际种群中,利用微卫星数据得到的亲缘系数通常会在理论值上下浮动,本研究所有父-子,母-子单元中,父子和母子间的平均亲缘系数为0.5316,接近理论值0.50,子代间的亲缘系数平均为0.3064,接近理论值0.25[9,35],进一步验证了亲权鉴定的准确性。本试验采用X2检验和G统计检验Hardy-Weinberg平衡,结果发现,共3个微卫星位点出现偏离Hardy-Weinberg平衡的现象。Hardy-Weinberg平衡种群的实质是指针对于一个较大的、随机交配的群体来说,在无外界选择,没有个体迁入、迁出和无突变位点产生的假设基础之上。Ardren等[36]指出,在濒危物种种群中出现不符合Hardy-Weinberg平衡的现象,主要原因有近亲繁殖、种群亚结构和无效等位基因导致的杂合度不足[37]。Fis是评价种群是否存在近亲繁殖的一个重要参数,当Fis为正值时,表示群体内存在近交,如果是负值,则群体内不存在近交[38]。本研究中古田山小麂群体的Fis为-0.1886,可知种群之间不存在近交现象,排除由近交导致的种群偏离Hardy-Weinberg平衡。小麂具有较大的活动范围,在古田山范围内没有任何地理障碍阻隔个体间的迁移和基因交流,小麂种群不会存在亚结构,无效等位基因频率极低,杂合子丰富,因此排除种群亚结构和无效等位基因导致其偏离Hardy-Weinberg平衡的可能。但小麂大范围的迁移使得保护区范围内可能存在一定的迁入和迁出,使采样点的小麂种群不符合理想种群的条件,导致其偏离Hardy-Weinberg平衡。同时保护区内小麂的栖息地存在片段化,破碎的栖息地使得小麂不能随机交配,这是导致微卫星位点偏离Hardy-Weinberg平衡的原因。
3.2 小麂的婚配制度
婚配制度是一种进化稳定策略(evolutionarily stable strategy,ESS)[39],动物在繁殖行为中采取哪一种婚配制度,取决于动物个体之间相互作用和对外界环境的适应,影响动物婚配制度的主要生态因素可能是食物和营巢地在时间和空间上的分布情况[40],鹿科动物的婚配制度多是一雄多雌[41]。一雄多雌制主要有三种亚型[42]:资源保卫型一雄多雌制(resource-defense polygyny),特点是雄性个体之间经过激烈的竞争建立各自的领域,占有繁殖必需的资源,吸引处于繁殖期雌性群体的到来,从而获得与多个雌性交配的机会;雌性或后宫保卫型一雄多雌制(female or harem defense),特点是雄性直接占有多个配偶,雌性通常集群生活;雄性优势型或求偶场一雄多雌制(male dominance or lek polypyny),特点是与雄性是否占用繁殖必需的资源无关,雄性之间通过确立社会等级或求偶炫耀来竞争。小麂、黑麂、赤麂(Muntiacusmuntjak)等麂属动物的繁殖特点是没有季节性[41],全年均可繁殖,没有固定的发情期和交配期,同时小麂喜独居,个体之间的交往频次不高[43],因此,小麂的婚配制仍属于一雄多雌,但并不是目前所知的亚型。Odden[44]和Wegge[4]对赤麂婚配制的研究认为,赤麂的婚配类型不是一雄一雌,也不是目前所知的一雄多雌中的任何一种亚型,而是一种被称作“检查或搜索策略(inspection or roaming strategy)”下的一雄多雌制,即雄性通过定期检查是否有经过自己领域的的发情雌性,以期与多个雌性发生交配。小麂和赤麂不仅在进化上属于近亲,而且行为和生活方式也极为相似,两者在野外都是营独居生活的,发情期和交配期均不固定,雄性个体有各自的领域[4,45],由此可以推断,小麂的婚配制应和赤麂相似。小麂在发情期内有明显的标记行为,尤其是进入发情期时雄性个体的标记行为变得更为频繁[46],雄性个体在留下自身信息的同时,也通过检查自己领域内雌性的信息,以便和更多的雌性发生交配。在雌性的繁殖期内,雌性个体一旦受孕,便不再和其它雄性交配[47],但这种婚配关系并不固定,在下一个繁殖期,雌性个体又可以和另外一个雄性交配,因此本研究中出现的一个雌性和多个雄性交配的现象应出现在雌性不同的繁殖期之间。小麂的这种婚配制度,使得搜索能力强的雄性能和多个在繁殖期的雌性进行交配,增加了营独居生活的小麂个体在繁殖期成功交配的概率。对于雌性来说,不固定的婚配关系,促进了雌雄之间的基因交流,使得子代的杂合性增加,是古田山小麂种群遗传多样性较高的重要原因。
3.3 保护对策
为了更好地发挥古田山保护区的作用,建议每5a复查1次,开展常规化的小麂种群数量动态监测,这有利于掌握小麂种群数量的变化规律,同时加强对保护区内小麂栖息环境质量的监测,包括食物(种类、可利用量、质量)、隐蔽所(类型、数量和质量)等变化特征,估算保护区内小麂的环境容纳量,以及时制定和调整相应的保护对策和措施。应继续加强当地居民对野生动物的保护意识,减少对小麂的捕猎,使小麂种群进一步扩大。同时应加强对小麂的科研力度,对影响保护区内小麂生存和繁殖的因素有较全面的理解和把握。对小麂进行人工繁育,在自然保护区的实验区,可以考虑建立以小麂为主的养殖场,驯养和繁殖人工种群,为小麂的就地保护和异地保护以及资源利用开拓新的途径。
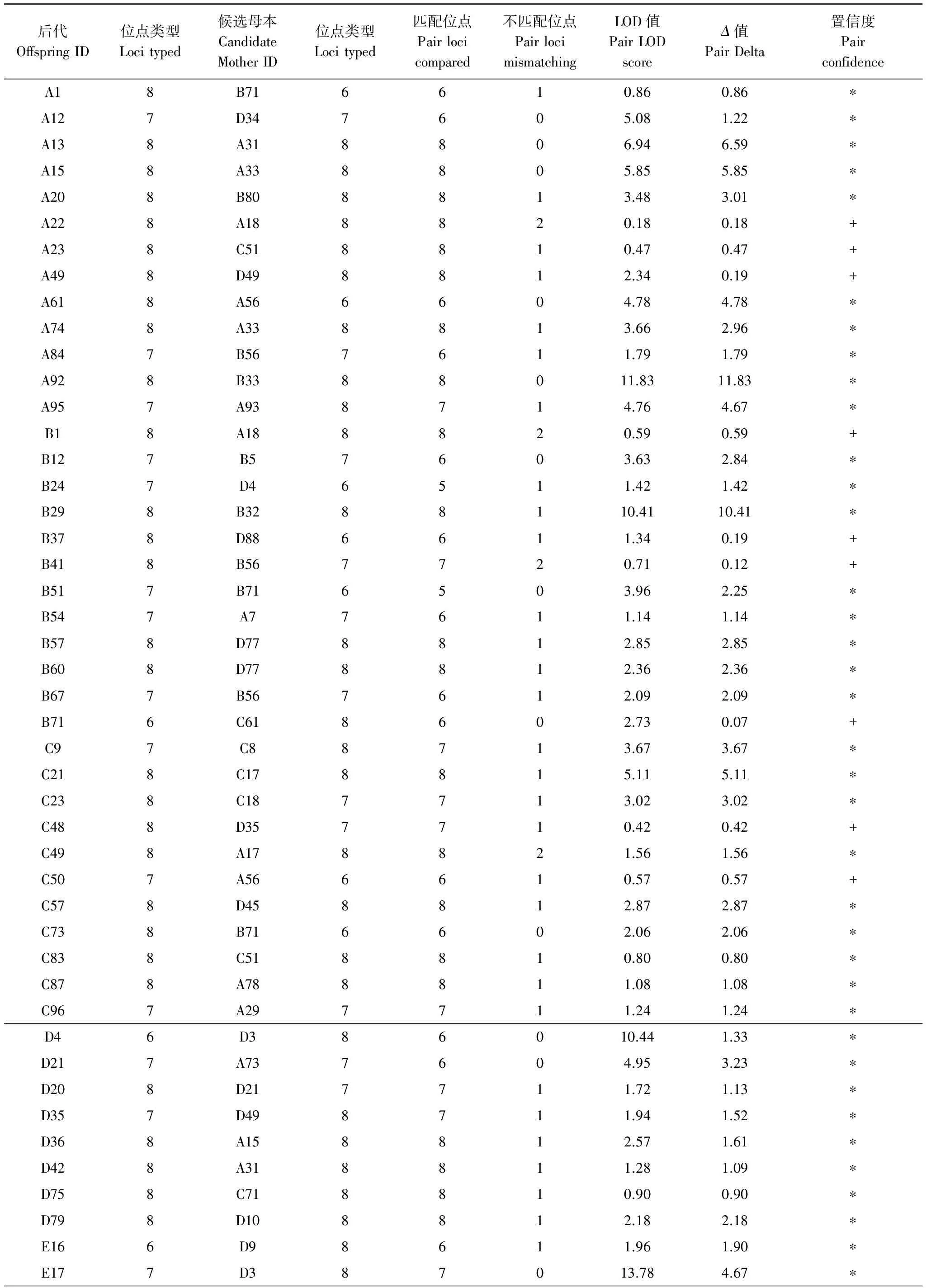
附录1 Cervus 3.0软件分析的母子鉴定结果
*:Δ值>标准Δ值(95%水平下)=0.62;+:0.62>Δ值>标准Δ值(80%水平下)=0.00

附录2 Cervus 3.0软件分析的父子鉴定结果
*:Δ值>标准Δ值(95%水平下)=0.51;+:0.51>Δ值>标准Δ值(80%水平下)=0.00
[1] 辜永河, 徐龙辉. 小麂一新亚种——江口亚种(偶蹄目, 鹿科). 动物学报, 1998, 44(3): 264- 270.
[2] 杨奇森, 岩崑. 中国兽类彩色图谱. 北京: 科学出版社, 2007.
[3] Miura S. Social Behavior and Territoriality in Male Sika Deer (CervusnipponTemminck 1838) during the Rut. Ethology, 1984, 64(1): 33- 73.
[4] Wegge P, Mosand HM. Can the mating system of the size-monomorphic Indian muntjac (Muntiacusmuntjak) be inferred from its social structure, spacing behaviour and habitat? A case study from lowland Nepal. Ethology Ecology & Evolution, 2015, 27(2): 220- 232.
[5] Hemami M R, Watkinson A R, Gill R M A, Dolman P M. Estimating abundance of introduced Chinese muntjacMuntiacusreevesiand native roe deerCapreoluscapreolususing portable thermal imaging equipment. Mammal Review, 2007, 37(3): 246- 254.
[6] Yang F, O′Brien P C M, Wienberg J, Neitzel H, Lin C C, Ferguson-Smith M A. Chromosomal evolution of the Chinese muntjac (Muntiacusreevesi). Chromosoma, 1997, 106(1): 37- 43.
[7] Huang L, Jing M, Nie W, Robinson T J, Yang F. Chromosome homologies between tsessebe (Damaliscuslunatus) and Chinese muntjac (Muntiacusreevesi) facilitate tracing the evolutionary history of Damaliscus (Bovidae, Antilopinae, Alcelaphini). Cytogenetic & Genome Research, 2011, 132(4): 264- 270.
[8] 魏辅文, 饶刚, 李明, 方盛国, 冯祚建. 分子粪便学及其应用—可靠性、局限性和展望.兽类学报, 2001, 21(2): 143- 152.
[9] Blouin M S, Parsons M, Lacaille V, Lotz S. Use of microsatellite loci to classify individuals by relatedness. Molecular Ecology, 1996, 5(3): 393- 401.
[10] Castro J, Bouza C, Presa P, Pino-Querido A, Riaza A, Ferreiro I, Sánchez L, Martínez P. Potential sources of error in parentage assessment of turbot (Scophthalmusmaximus) using microsatellite loci. Aquaculture, 2004, 242(1/4): 119- 135.
[11] 田新民,张明海,张辉, 杨春文, 金志民. 黑龙江省完达山东部林区马鹿种群遗传多样性的微卫星分析.生态学杂志, 2010, 29(3): 543- 548.
[12] 周璨林,艾斯卡尔·买买提,日沙来提·吐尔地,马合木提·哈力克.非损伤技术研究天山马鹿性别比与冬季家域.科技导报, 2015, 33(4): 91- 96.
[13] Zeyl E, Aars J, Ehrich D, Bachmann L, Wiig Ø. The mating system of polar bears: a genetic approach. Canadian Journal of Zoology, 2009, 87(12): 1195- 1209.
[14] Amavet P S, Vilardi J C, Rueda E C, Larriera A, Saidman B O. Mating system and population analysis of the broad-snouted caiman (Caimanlatirostris) using microsatellite markers. Amphibia-Reptilia, 2012, 33(1): 83- 93.
[15] Gazda G, Kuehn R, Sato N J, Keita D Tanaka, Okahisa Y, Ueda K. Establishment of microsatellite markers to assess the mating system of the fan-tailed gerygone (Gerygoneflavolateralis) for studying cuckoo-host arms race. Annales Zoologici Fennici, 2016, 52(5/6): 280- 284.
[16] Garnier J N, Bruford M W, Goossens B. Mating system and reproductive skew in the black rhinoceros. Molecular Ecology, 2001, 10(8): 2031- 2041.
[17] Chen X, Jiang K, Bao Y, Wang H, Shi W, Zheng W. The mating system study of black muntjac (Muntiacuscrinifrons) based on fecal DNA. Acta Ecologica Sinica, 2015, 35(5): 137- 141.
[18] 程宏毅,鲍毅新,郑荣泉, 陈良, 葛宝明. 黑麂粪便DNA提取及其PCR检测.生态科学, 2006, 25(2): 158- 161.
[19] 李晋,陶艾艾,孟凯, 吴海龙. 一种鹿科动物标本的分子鉴定.兽类学报, 2009, 29(1): 106- 108.
[20] 华育平,张琼,徐艳春,郑冬.虎物种特异性鉴定的PCR方法研究.兽类学报, 2004, 24(2): 103- 108.
[21] Yan P,Wu X B,Shi Y, Gu C M, Wang R P, Wang C L. Identification of Chinese alligators (Alligatorsinensis) meat by diagnostic PCR of the mitochondrial cytochromebgene. Biological Conservation, 2005, 121(1): 45- 51.
[22] Wang H, Luo X, Shi W B, Zhang B W. Development and characterization of fourteen novel microsatellite loci in Chinese muntjac (Muntiacusreevesi). Conservation Genetics Resources, 2013, 5(4): 1083- 1085.
[23] Wang H, Luo X, Shi W B, Zhang B W. Isolation and characterization of polymorphic microsatellite loci of the Chinese muntjac (Muntiacusreevesi). Genetics & Molecular Research, 2014, 13(1): 1905- 1908.
[24] 鲁晓瑄, 张悦, 单祥年. 小麂Sry基因的克隆和测序. 遗传, 2003, 25(3): 299- 301.
[25] Bellemain E, Swenson J E, Tallmon D, Brunberg S, Taberlet P. Estimating population size of elusive animals with DNA from hunter-collected feces: four methods for brown bears. Conservation Biology, 2005, 19(1): 150- 161.
[26] Kalinowski S T, Taper M L, Marshall T C. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 2007, 16(5): 1099- 1106.
[27] Konovalov D A, Manning C, Henshaw M T. KINGROUP: a program for pedigree relationship reconstruction and kin group assignments using genetic markers. Molecular Ecology Notes, 2004, 4(4): 779- 782.
[28] Raymond M, Rousset F. GENEPOP (Version 1.2): population genetics software for exact tests and ecumenicism. Journal of Heredity, 1995, 86(3): 248- 249.
[29] Louis Bernatchez P D. Individual-based genotype analysis in studies of parentage and population assignment: how many loci, how many alleles?. Canadian Journal of Fisheries & Aquatic Sciences, 2000, 57(1): 1- 12.
[30] Botstein D, White R L, Skolnick M, Davis R W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314- 331.
[31] Wu H L, Ni X W, Zhang L Y, Xia J S, Zhong Z Y, Zhu G P, Wan Q H. Eighteen novel polymorphic microsatellite loci developed from the Père David′s deer (Elaphurusdavidianus). Conservation Genetics, 2008, 9(6): 1679- 1682.
[32] Wu H L, Wan Q H, Fang S G. Microsatellite analysis of genetic variation and population subdivision for the black muntjac,Muntiacuscrinifrons. Biochemical Genetics, 2008, 45(11- 12): 775- 788.
[33] Anderson J D, Honeycutt R L, Gonzales R A, Gee K L, Skow L C, Gallagher R L, Honeycutt D A, DeYoung R W. Development of microsatellite DNA markers for the automated genetic characterization of white-tailed deer populations. Journal of Wildlife Management, 2002, 66(1): 67- 74.
[34] Nei M. Molecular population genetics and evolution. Frontiers of Biology, 1975, 40: 1-208.
[35] Queller D C, Goodnight K F. Estimating relatedness using genetic markers. Evolution, 1989, 43(2): 258- 275.
[36] Ardren W R, Borer S, Thrower F, Joyce J E, Kapuscinski A R. Inheritance of 12 microsatellite loci inOncorhynchusmykiss. Journal of Heredity, 1999, 90(5): 529- 536.
[37] Lade J A, Murray N D, Marks C A, Robinson N A. Microsatellite differentiation between Phillip Island and mainland Australian populations of the red foxVulpesvulpes. Molecular Ecology, 1996, 5(1): 81-87.
[38] Weir B S, Cockerham C C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358- 1370.
[39] Odng L W. Avian mating system//Famer D S, King J R ed. Avinn Biology (VI). New York: Academic Press, 1982: 1- 92.
[40] 张建军,张知彬.动物的婚配制度.动物学杂志, 2004, 38(2): 84- 89.
[41] 盛和林.中国鹿科动物.生物学通报, 1992, (5): 4- 7.
[42] Krasnec M O, Cook C N, Breed M D. Mating systems in sexual animals. Nature Education Knowledge, 2012, 3(10): 72- 72.
[43] 徐宏发,周自力,盛和林.黄麂习性的初步观察.野生动物学报, 1990, (2): 18- 19.
[44] Odden M, Wegge P. Predicting spacing behavior and mating systems of solitary cervids: A study of hog deer and Indian muntjac. Zoology, 2007, 110(4): 261- 270.
[45] Mccullough D R, Pei K C J, Wang Y. Home range, activity patterns, and habitat relations of Reeves′ muntjacs in Taiwan. The Journal of Wildlife Management, 2000, 64(2): 430- 441.
[46] 吴玥,康蔼黎,苏铁,张恩迪. 黄麂标记行为初探.四川动物, 2004, 22(2): 73- 75.
[47] Yahner R H. Temporal patterns in male mating behavior of captive reeve′s Muntjac (Muntiacusreevesi). Journal of Mammalogy, 1979, 60(3): 560- 567.
ParentageverificationandmatingsystemofChinesemuntjac(Muntiacusreevesi)basedonfecalDNA
REN Peng,GONG Kun,BAO Yixin*,HUANG Xiangxiang,ZHOU Xiao,HAN Jinqiao
InstituteofEcology,ZhejiangNormalUniversity,Jinhua321004,China
Microsatellite technology was utilized in the present study to investigate paternity testing, mating system, genetic diversity, and population quantity of Chinese muntjac (Muntiacusreevesi) to increase the knowledge of relationships between members of the species, reproductive strategies, and evolution potential. Therefore, the present study aims to assist the facilitation of in-depth research and protection of the species. We collected 634 fecal samples and 2 muscle samples from Chinese muntjac (M.reevesi) in Gutianshan National Nature Reserve in January 2013 and April 2014. After repeated experiments, 8 microsatellite loci were selected, which had stable amplification and high polymorphism information, and 442 samples were utilized for polymerase chain reaction (PCR) amplification. Based on the preliminary identification in terms of the fecal morphology, a verification test was conducted with the mitochondrial cytochrome b gene (Cyt b), which was regarded as a supplement to the species identification. Individual identification for Chinese muntjac was conducted by contrasting the fingerprints synthesized by the eight microsatellite marker genotypes. The SRY gene was amplified at three times to reduce any false negative influence on the fecal samples, where the target band was identified as male when it appeared more than two times. The SRY gene and 8 microsatellite loci were utilized for sexual and individual identification, respectively, and 177 individuals, including 94 females and 83 males, were identified. The results showed that the total number of microsatellite alleles were 88, and the mean number of alleles was 11, ranging from 8 to 16, and the mean observed and expected heterozygosities were 0.9685 (0.960—1.000) and 0.8429 (0.799—0.887), respectively. The polymorphism information content ranged from 0.766 to 0.872, with an average of 0.821. Based on the above data, the Chinese muntjac population in Gutianshan National Nature Reserve has a high level of genetic diversity. The identification of 8 microsatellite loci identification rate from Cervus3.0 was 100%, when the confidence level was 95% and 80%. Among 24 parentage groups, 19 father-child relationships and 23 mother-child relationships were obtained among 104 individuals. The result of the relationship analysis showed that the mating system of Chinese muntjac belongs to a polygyny. However, it does not belong to any known subtypes, and this polygyny might be called a “check strategy”. During the female reproductive period, once the female individual is pregnant she will not mate with any other males. However, the relationship is not fixed and the female might still mate with another male in the next breeding season. Therefore, in the present study, the mating phenomenon of a female and several males appeared in different breeding periods.
Chinese muntjac (Muntiacusreevesi); parentage identification; genetic diversity; mating system
古田山国家级自然保护区资助项目
2016- 08- 02; < class="emphasis_bold">网络出版日期
日期:2017- 06- 01
*通讯作者Corresponding author.E-mail: baoyix@21cn.com
10.5846/stxb201608021591
任鹏,龚堃,鲍毅新,黄相相, 周晓, 韩金巧.基于粪便DNA的小麂亲权鉴定和婚配制研究.生态学报,2017,37(20):6933- 6944.
Ren P,Gong K,Bao Y X,Huang X X,Zhou X,Han J Q.Parentage verification and mating system of Chinese muntjac (Muntiacusreevesi) based on fecal DNA.Acta Ecologica Sinica,2017,37(20):6933- 6944.
——从虐童事例切入

