利福平对毛泡桐长链非编码RNA表达谱变化的影响
李永生,李冰冰,曹亚兵,王 哲,董焱鹏,邓敏捷,赵振利,范国强
(河南农业大学泡桐研究所,河南 郑州 450002)
利福平对毛泡桐长链非编码RNA表达谱变化的影响
李永生,李冰冰,曹亚兵,王 哲,董焱鹏,邓敏捷,赵振利,范国强
(河南农业大学泡桐研究所,河南 郑州 450002)
为阐明lncRNAs对泡桐生长发育的调控作用,以利福平处理前后毛泡桐健康苗为试验材料,利用高通量测序技术研究利福平处理前后毛泡桐长链非编码RNA及其靶基因的表达变化情况。共鉴定出129个差异表达的lncRNAs,其中有83个差异lncRNAs对应289个靶基因,对这些靶基因进行GO功能分类,发现这些差异的靶基因共参与9个GO分类,其中细胞过程、代谢过程及涉及单有机体的靶基因数量最多。KEGG代谢通路分析发现,这些差异靶基因参与了植物激素信号转导、次生代谢生物合成等代谢通路。并采用qRT-PCR技术验证了随机挑选的6个差异表达长链非编码RNA及靶基因,结果与高通量测序数据一致。
毛泡桐;利福平处理;长链非编码RNA
lncRNA (Long non-coding RNA)是指长度在200 nt以上,没有长的开放阅读框(ORF),不具备编码蛋白质的功能的非编码RNA[1],但在特定的条件下有些 lncRNAs 可以编码功能性寡肽[2]。基于在基因组的位置,lncRNA 可分为:正义 lncRNAs,反义 lncRNAs,内含子 lncRNAs,基因间区 lncRNAs 和双向型 lncRNAs[3]。在人基因组中,大约只有 2% 的蛋白编码基因(约 20 000个),98% 的基因组区域不编码蛋白质,这些非编码序列曾经被认为是基因组在进化过程中产生的“junk DNA”[4]。自在哺乳动物中发现lncRNA以来[5],随着测序技术的发展,人们发现了大量非编码 RNA (non-coding RNA,ncRNA),它们在生物生长、发育和分化以及胁迫反应中具有重要作用[6-9]。目前,在拟南芥中共鉴定 6 480个 lncRNAs,其中 22 个与盐和水分胁迫相关;在玉米中鉴定出3 819 个cis-NATs,其中 503 个 cis-NATs 与特定的生物学功能相关[10-11];在小麦中鉴定出响应白粉病和热胁迫的 lncRNAs共125个[12]。此外,还在水稻中鉴定出了大量与生殖相关的 lncRNAs[13]。对木本植物来说,也只在毛果杨和毛白杨中进行了 lncRNAs 的鉴定及分析[14-15]。相关研究表明,抗生素不仅可以作为一种抑菌杀菌物质在植物组培及病害防治中起重要作用,并且对植物的生长发育也有显著的影响[16-17],例如医用生长素能够诱导禾本科植物种子萌发,促进幼苗生长,促进叶片中叶绿素的合成及降解[18];适当浓度抗生素可以显著提高大豆组培过程中的出愈率并降低褐化率等[19]。而利福平作为一种半合成抗生素可以通过与 RNA 聚合酶β亚基的特异性结合来抑制RNA的合成,其作用原理成为各种生化过程中研究细胞发生机制的遗传分析和生物化学工具,并可用于重组蛋白的研究[20]。但是作为半合成抗生素的利福平对木本植物的生长影响还鲜有报道,因此,为了更好地了解泡桐在生长发育过程中的分子机制并进一步培育出优良品种,本研究利用利福平处理的毛泡桐幼苗,采用高通量测序技术对泡桐lncRNAs进行鉴定,并通过预测 lncRNAs 的靶基因来了解泡桐中与利福平处理相关的 lncRNAs 功能,以期为泡桐种质资源创新提供理论基础,同时为更深入了解泡桐的分子生物学特征和培育满足国民经济发展需求的优良品种奠定坚实基础。
1 材料与方法
1.1试验材料
以河南农业大学林木生物技术实验室培养的毛泡桐(Paulowniatomentosa)健康苗(PT)为材料。材料在温度为(25 ± 2)℃,光照度为130 μmol·m-2·s-1,光照周期为16 h/8 h(光/暗)下培育30 d。取长势均匀、长度约1.5 cm的上述泡桐顶芽接种于盛有50 mL含100 mg·L-1利福平的1/2 MS培养基(100 mL三角瓶)中进行利福平处理(PTL-100),同时将顶芽分别转入不含利福平的1/2 MS培养基中培养,作为对照试验(PT)。每个三角瓶中培养 3 个外植体,每个样品培养30瓶,培养方法和条件参照翟晓巧等[21]的方法。待上述幼苗培养 30 d 后,分别剪取顶芽,用液氮冷冻后置于-80 ℃冰箱内,为 RNA 提取做准备。
1.2总RNA的提取、lncRNA文库构建和高通量测序
利用TRLzol试剂参照试剂盒使用说明书进行总RNA提取,使用Agilent 2100生物分析仪和NanoDrop 2000超微量分光光度计进行质量检测。按照TruSeq Stranded标准进行RNA-Seq文库的构建。文库构建之后使用Illumina HiSeq 2000平台进行测序,将测序得到的raw reads去除含adapter、含N和低质量的reads,最终得到clean reads。利用SOAP 2软件将clean reads比对到SILVA rRNA数据库中(http://www.arb-silva.de/)以去除比对上核糖体的reads,将保留下来的数据用于后续分析。最后,利用TopHat 2将过滤完核糖体的reads比对到泡桐参考基因组上进行分析。
1.3lncRNA鉴定
使用Cuffcompare软件将组装好的转录本比对到泡桐基因组上,以鉴定出具有编码功能的蛋白质转录本,然后删除剩余长度小于200 bp的转录本,通过Coding Poteintial Calculator(CPC)对剩余转录本进行编码能力的预测,将CPC的值小于零的转录本认为是没有编码蛋白质的能力,这样的转录本是非编码的即lncRNA。
1.4lncRNA保守性分析
根据LI等[22]的研究方法确定已鉴定的 lncRNAs 在泡桐基因组染色体上的分布情况,并基于 lncRNA在基因组的分布位点将 lncRNAs 分为以下5类:“i”,“j”,“u”,“x”,“o”[23]。并使用BLASTN软件(E值<1e-5)将所鉴定的lncRNAs比对到CANTATAdb数据库中 (http://yeti.aum.edu.pl/CANTATA),以对 lncRNAs的保守性进行分析。将泡桐 lncRNAs 与其他物种 lncRNAs 进行比对,覆盖度大于等于10%认为是保守的lncRNAs。为了进一步分析 lncRNAs 是否为 miRNA 的前体序列,将 lncRNAs 的序列与 miRBase数据库进行比对(Release 21,http://www.mirbase.org/),并使用 RNAfold 软件预测 lncRNAs 和 miRNAs 前体的二级结构。
1.5lncRNA的靶基因预测
基于lncRNAs与靶基因不同作用方式,利用cis和trans两种方法对 lncRNAs 进行靶基因预测。cis作用是lncRNAs的一种靶基因作用方式,预测范围是lncRNAs上游10 kb,下游20 kb内的基因。trans作用靶基因的预测不依赖于和编码基因的位置关系,主要依赖于与其共表达的mRNA之间的关系。利用BLAST软件(E<1e-5,identity≥95%)选择与 lncRNAs 互补的靶基因序列,最后使用RNAplex软件来计算lncRNAs和靶基因序列之间的折叠能,根据折叠能来判断lncRNAs潜在的trans靶基因。
1.6差异lncRNA的鉴定、分析
采用FPKM(Fragments per kilobase of transcript per million fragments mapped)作为衡量lncRNAs及mRNA表达水平的指标,在差异表达lncRNAs及mRNA的鉴定过程中,将差异倍数(Fold Change)≥2且校正后的FDR<0.001 作为筛选标准。Fold Change表示PTL-100和PT两样品的表达量的比值。FDR值越小,差异倍数越大,表明表达差异越显著。为进一步了解这些差异mRNA的功能,对其进行Nr注释,之后进行GO功能分类和差异靶基因的KEGG pathway分析。
1.7qRT-PCR验证
总RNA提取方法参照1.2的方法,首先需要将所验证 lncRNAs和靶基因利用Primer Express 3.0(Applied Biosystems,Stockholm,Sweden)进行引物设计(表1),总RNA反转录合成cDNA的第一条链,整个反应体系为:反应体系总体积为20 μL,每20 μL中含有1 μL的模板,正向和反向引物各0.4 μmol ,cDNA 1 μL,7.0 μL ddH2O。扩增程序为:95 ℃,1 min;95 ℃,15 s,然后57 ℃,15 s;40个循环。每个样品3次重复。以18S rRNA作为内参进行校正,数据处理使用2-△△Ct法[24]计算相对表达量。
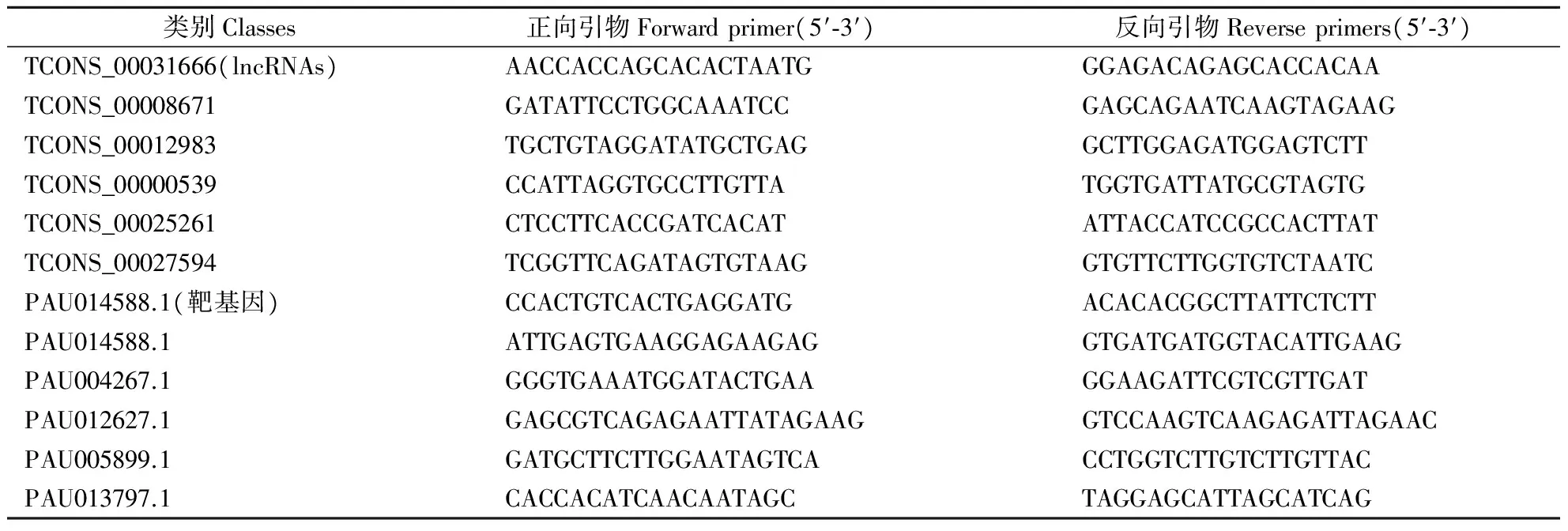
表1 QRT-PCR验证的差异lncRNAs和差异靶基因的引物序列Table 1 Primers of quantitative RT-PCR analysis of differentially expressed lncRNAs and target genes
2 结果与分析
2.1泡桐中lncRNA的鉴定及保守性分析
利用高通量测序对PT和PTL-100进行Illumina测序,总共获得了2 598个 lncRNAs,在PT,PTL-100文库中分别鉴定了2 581、2 552个lncRNAs。依据 lncRNA 在基因组上的位置,对2 598个lncRNAs 进行分类。结果表明,有 23个lncRNAs 属于“j”类(至少有1个junction位点与参考基因一致,为潜在的新转录本);有 38 个为与参考基因外显子有一定交集的“o”类;2 136个属于基因间区的“u”类;401个属于在参考基因的反义链与外显子有交集的“x”。该结果表明,大部分lncRNA属于基因间区的 lncRNAs。
使用BLAST软件将鉴定出的lncRNAs比对到拟南芥、水稻、大豆、卷柏、水稻等10种模式物种的lncRNA序列上。结果表明,毛泡桐中只有少数lncRNAs是保守的(表2)。其中,与葡萄中 lncRNAs 的匹配数量最高。为了确定lncRNAs是否为miRNAs 前体,将2 598个lncRNAs与miRBase 数据库中 miRNAs的前体序列进行比对。结果(表3)表明,有8个lncRNAs作为13个已知 miRNAs的前体序列,说明lncRNA 经过Dicer酶的连续切割加工可以形成多个miRNA,而且每个miRNA具有不同的亚细胞定位和独特的功能。对 lncRNA 进行家族分析表明,有412个lncRNAs属于158个保守的lncRNAs家族,其中miRNA家族和snoRNA家族数量最多。
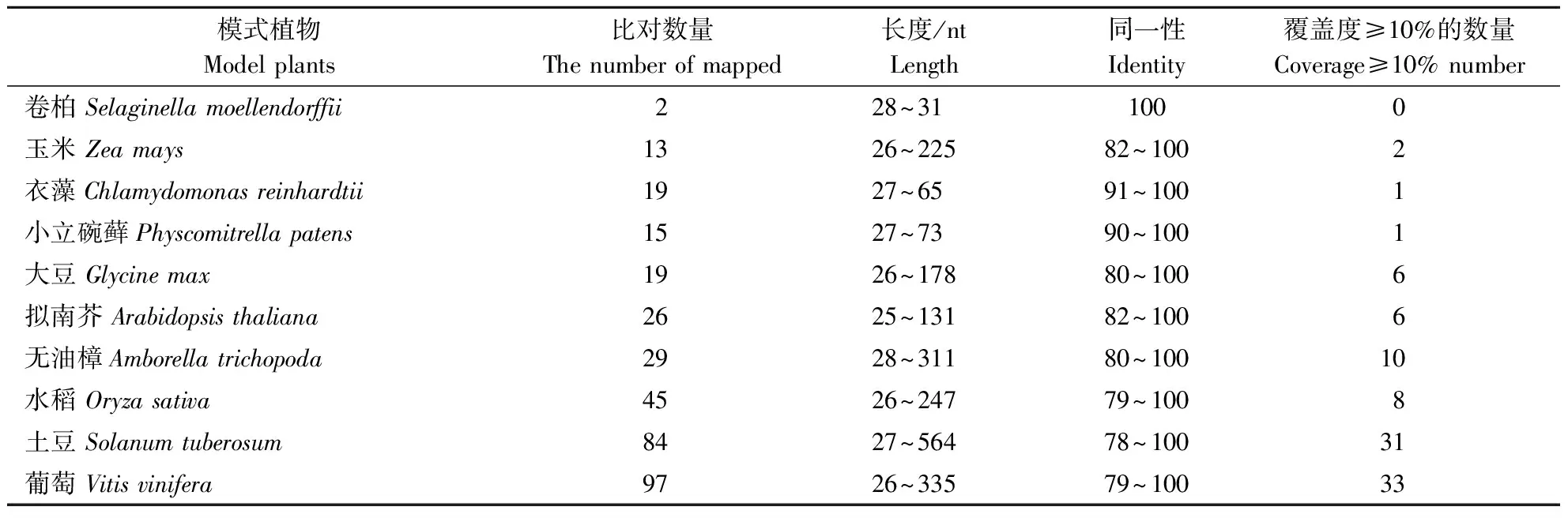
表2 毛泡桐lncRNA的保守性分析Table 2 Conservative Analysis of lncRNA of Paulownia tomentosa

表3 作为miRNAs前体的lncRNAs序列Table 3 LncRNAs as precursors to miRNAs
2.2lncRNA靶基因的预测
lncRNAs作为新发现的基因表达调控因子,广泛参与植物生长发育的调节,并在胁迫响应中起着重要的作用。因此,进一步鉴定和分析其靶基因有助于了解毛泡桐lncRNAs的功能。使用RNAplex软件来预测LncRNAs的靶基因,共鉴定出2 395个lncRNAs,共有6 414个靶基因,其中2 220个lncRNAs 通过cis作用调控4 803个基因,2119个lncRNAs 通过trans 作用调控2 014个基因。由此可以看出,1个lncRNA可以有多个靶基因,而1个靶基因也可以对应多个lncRNAs。在这些测序得出的lncRNAs中,249个lncRNAs只分别对应1个靶基因,8个lncRNAs分别对应10个和11个靶基因,1个lncRNA(TCONS_00011451)最多对应12个靶基因。
2.3差异lncRNAs的鉴定,注释和富集分析
本研究共鉴定出129个差异lncRNAs,其中上调的59个,下调的70个。大部分lncRNAs的差异表达倍数小于5倍,其中分别有20和24个lncRNAs的差异表达倍数大于10倍。在2个文库中共鉴定出特有的lncRNAs个数分别为15个(PTL-100)和12个(PT)。为了更好了解差异lncRNAs的功能,对其靶基因进行了预测。结果表明,在129个lncRNAs中,共鉴定出了289个靶基因,其中214个靶基因认为是75个lncRNAs的顺式调控基因,62个靶基因被认为是62个lncRNAs的反式调控基因。并且在289个预测的靶基因中,有52个是差异表达的,其中41个靶基因认为是32个lncRNAs的顺式调控基因,10个靶基因是10个lncRNAs的反式调控基因。
为了找出在PT,PTL-100文库中与毛泡桐生长发育密切相关的lncRNAs,对52个差异表达的靶基因进行GO功能分类和功能注释。结果表明,这些差异的靶基因共参与9个GO分类(图1),其中细胞过程、代谢过程及涉及单有机体的靶基因数量最多,细胞、细胞组分等数量最少。KEGG pathway分析表明这些差异靶基因共参与28个KEGG代谢通路。其中,植物激素信号转导、次生代谢生物合成、代谢途径和RNA转运等高度富集(表4)。
2.4qRT-PCR对测序结果的验证
为了验证高通量测序结果的可靠性,本试验在PT和PTL-100两个文库中随机挑选了6个lncRNAs及其相应的靶基因进行qRT-PCR验证(图2)。结果表明,挑选验证的lncRNAs及其靶基因的表达趋势与高通量测序结果保持一致,证实了RNA-Seq测序数据真实可靠。同时进一步分析发现大部分lncRNAs的表达趋势与靶基因的表达趋势是相反的,只有1个是一致的,这可能是由于lncRNAs调控机制的复杂性引起的。

A1:细胞过程, A2:发育过程, A3:代谢过程, A4:刺激响应, A5:单有机体过程;B1:细胞, B2:细胞组分,B3:细胞器, C:结合。 A1: cellular process, A2: developmental process, A3: metabolic process, A4: response to stimulus, A5: single-organism process;B1: cell, B2: cell part, B3: organelle, C: binding.

代谢通路Pathway有通路注释的DEGsDEGswithpathwayannotation代谢通路IDPathwayID托品烷、哌啶和嘧啶碱的生物合成Tropane,piperidineandpyridinealkaloidbiosynthesis1ko00960光合生物固氮Carbonfixationinphotosyntheticorganisms1ko00710同源重组Homologousrecombination1ko03440糖链的生物合成N⁃Glycanbiosynthesis1ko00510磷酸肌醇代谢Inositolphosphatemetabolism1ko00562甘油酯代谢Glycerolipidmetabolism1ko00561谷胱甘肽代谢Glutathionemetabolism1ko00480萜类骨干Terpenoidbackbonebiosynthesis1ko00900丙酮酸代谢Pyruvatemetabolism1ko00620细胞自噬调节Regulationofautophagy1ko04140半乳糖代谢Galactosemetabolism1ko00052昼夜节律-植物Circadianrhythm⁃plant1ko04712类黄酮生物合成Flavonoidbiosynthesis1ko00941碳代谢Carbonmetabolism1ko01200内质网蛋白加工Proteinprocessinginendoplasmicreticulum1ko04141剪接小体Spliceosome1ko03040代谢途径Metabolicpathways12ko01100半胱氨酸和蛋氨酸代谢Cysteineandmethioninemetabolism2ko00270氨基糖和核苷酸糖代谢Aminosugarandnucleotidesugarmetabolism2ko00520泛素介导的蛋白降解Ubiquitinmediatedproteolysis2ko04120核糖体Ribosome2ko03010醚脂代谢Etherlipidmetabolism2ko00565脂代谢Glycerophospholipidmetabolism2ko00564胞吞作用Endocytosis2ko04144磷脂酰肌醇信号系统Phosphatidylinositolsignalingsystem1ko04070RNA转运RNAtransport2ko03013植物激素信号转导Planthormonesignaltransduction2ko04075次生代谢物的生物合成Biosynthesisofsecondarymetabolites7ko01110

A: lncRNAs A1:TCONS_00031666 A2:TCONS_00008671 A3:TCONS_00012983 A4: TCONS_00000539 A5: 00025261 A6: 00027594 B:靶基因 B1: PAU019746.1 B2:PAU007418.1 B3: PAU000476.1 B4:PAU017667.1 B5:PAU029158.1 B6:PAU023015.1
3 结论与讨论
LncRNAs 最初被认为是基因之间的贫瘠区域或由于低 RNA 聚合酶保真度导致的“转录噪声”。但越来越多的研究表明,lncRNAs 的调节作用几乎存在于基因表达的每个阶段,从调节染色质结构到调控 mRNAs生物发生、稳定性、降解和翻译过程[25-26]。本研究获得了2 598个 lncRNAs,其中23个属于“j”类,38个为 “o”类;2 136个属于“u”类;401个属于“x”类;其表达水平低于编码蛋白的转录本,在进化水平上表现出保守性,与葡萄中 lncRNAs 的亲缘关系最近。鉴定出的大部分 lncRNA 位于基因间区,表明 lncRNAs 主要控制靶基因的表达与抑制,以及帮助 RNA 识别基因。而本研究共鉴定出2 395个 lncRNAs 对应6 414个靶基因,其中差异表达的 lncRNAs 共129个对应 289 个靶基因(52个为差异表达)。这些差异表达 IncRNA的靶基因共参与 9个GO分类与 28个 KEGG 代谢通路,其中细胞过程、代谢过程及涉及单有机体的靶基因数量最多,细胞、细胞组分等数量最少。代谢通路中参与植物激素信号转导、次生代谢生物合成、代谢途径和 RNA 转运等高度富集具有组织和细胞特异性表达模式,并通过多种复杂机制调节基因表达。这表明 lncRNAs 的调节作用广泛,几乎存在于基因表达的各个阶段,验证了前人的研究。
随着生物信息学技术的发展,已有大量关于lncRNA的研究工作,但关于其在植物生长发育过程中具体功能的研究相对较少,而本研究主要针对于调节激素合成及参与光合作用相关靶基因的 LncRNA 进行了筛选。调控激素合成方面,本研究表明 lncRNAs 与油菜素内酯(BR)的激发和合成相关。BR被定义为第6大类激素,是广泛存在于植物中。SZEKERES等[27]研究表明,与拟南芥野生型相比,BR诱导基因突变体表现为叶片变小,根长和下胚轴变短,这表明BR对植物生长调节起着正调控作用。而细胞膜上的类受体蛋白激酶BRI1是植物感知BR信号并将信号进一步传递到下游的关键复合物[28-31]。WANG等[32]发现,BRI1 的过表达能够使植物的叶柄和叶片变大。本研究中,LncRNA(TCONS_00023723)所对应的靶基因(PAU021885.1)具有编码 BRI1 的功能,并且在利福平处理后的毛泡桐中呈现显著上调趋势,表明 LncRNA(TCONS_00023723)在一定程度上对泡桐苗的生长起着正调控作用,即可以调节靶基因(PAU021885.1)的上调,从而导致泡桐苗出现叶片变大,植株生长加快的现象。而在调节光合作用,气孔运动方面,lncRNA 同样具有重要的调节作用。此过程中,磷酸烯醇式丙酮酸羧化酶(Phosphoenolpyruvate carboxylase,PEPC) 尤其对C4途径而言是一种关键的细胞质酶。具有提高植物对光合、水分等利用率,提高生物产量等作用。有研究表明,将玉米 PEPC 基因导入水稻中,能提高光合速率及产量[33]。同时 PEPC 可以通过调节细胞pH,保持离子平衡,调节气孔保卫细胞的渗透压及运动等方式提高植物抗逆性[34]。在本研究中,LncRNA(TCONS_00002733)所对应的编码 PEPC 的靶基因PAU002506.1在利福平处理后是显著上调的,表明了LncRNA(TCONS_00002733)的靶基因可能通过参与光合作用、调控气孔运动等过程来调节泡桐生长以及响应抗逆性,在一定程度上为培育出生长更加优良的泡桐种质提供理论基础,但其分子机制较为复杂,需更深入的研究进行阐明。
总之,本研究通过高通量测序技术,对毛泡桐 PT 及对应的 PTL-100 进行 lncRNAs 变化研究,鉴定出了与一些差异靶基因,通过对其功能注释分析,为更好地了解泡桐生长发育奠定了一定的理论基础。
[1] MERCER T R,DINGER M E,MATTICK J S.Long non-coding RNAs: insights into functions[J].Nat Rev Genet,2009,10(3):155-159.
[2] AMOR B B,WIRTH S,MERCHAN F,et al.Novel long non-protein coding RNAs involved in Arabidopsis differentiation and stress responses [J].Genome Res,2009,19(1): 57-69.
[3] PONTING,CHRIS P,OLIVER,et al.Evolution and functions of long non-coding RNAs[J].Cell,2009,136(4):629-41.
[4] CLAMP M,FRY B,KAMAL M,et al.Distinguishing protein-coding and noncoding genes in the human genome[J].Proc Natl Acad Sci USA,2007,104(49): 19428-19433.
[5] LUKIW W J,HANDLEY P,WONG L,et al.BC 200 RNA in normal human neocortex,non-Alzheimer dementia (NAD),and senile dementia of the Alzheimer type (AD)[J].Neurochem Res 1992,17:591-597.
[6] WILUSZ J E,SUNWOO H,SPECTOR D L.Long noncoding RNAs: functional surprises from the RNA world[J].Gene Dev,2009,23(13):1494.
[7] MERCER T R,DINGER M E,MATTICK J S.Long non-coding RNAs:insights into functions[J].Nat Rev Genet,2009,10(3):155.
[8] KIM E D,SUNG S.Long noncoding RNA:unveiling hidden layer of gene regulatory networks[J].Trends in Plant Science,2012,17(1):16-21.
[9] ZHU Q H,WANG M B.Molecular functions of long non-Coding RNAs in plants[J].Genes,2012,3(1):176.
[10] WANG H,CHUNG P J,LIU J,et al.Genome wide identification of long non-coding natural antisense transcripts and their responses to light in Arabidopsis[J].Genome Res,2014,24:444-453.
[11] LU T,ZHU C,LU G,et al.Strand specific RNA-Seq reveals widespread occurrence of novel cis-natural antisense transcripts in rice [J].BMC Genomics,2012,13:721-735.
[12] PENG H,XIE C,QIN D,et al.Identification and characterization of wheat long non-protein coding RNAs responsive to powdery mildew infection and heat stress by using microarray analysis and SBS sequencing[J].BMC Plant Biol,2011,11(1):61.
[13] ZHANG Y C,LIAO J Y,LI Z Y,et al.Genome-wide screening and functional analysis identify a large number of long noncoding RNAs involved in the sexual reproduction of rice[J].Genome Biol,2014,15(12):512.
[14] SHUAI P,LIANG D,TANG S,et al.Genome-wide identification and functional prediction of novel and drought-responsive lincRNAs in Populus trichocarpa[J].J Exp Bot,2014,65(17):4975-4983.
[15] TIAN J,SONG Y,DU Q,et al.Population genomic analysis of gibberellin-responsive long non-coding RNAs in Populus [J].J Exp Bot,2016,67(8):2467.
[16] 李文琴,贺长征,阎富英,等.植物生长调节剂与抗生素在种子处理中的应用[J] .天津农业科学,2002,8(2):31 -33.
[17] 谭小力,戚存扣,张丽丽,等.不同抗生素对油菜种子萌发的影响[J] .江苏农业科学,2006(5):27 -29.
[18] 刘萍,齐付国,丁义峰,等.青霉素和氨苄青霉素对小麦种子萌发及幼苗生理生化的影响 [J].华北农学报,2004,19(3):66-68.
[19] 王萍,吴颖,季静,等.抗生素对大豆愈伤组织的诱导和生长的影响[J].遗传,2001,23(4):321-324.
[21] 翟晓巧,曹喜兵,范国强.甲基磺酸甲酯处理的豫杂一号泡桐丛枝病幼苗的生长及SSR分析[J].林业科学,2010,46(12):176-181.
[22] LI H,WANG Y,CHEN M,et al.Genome-wide long non-coding RNA screening,identification and characterization in a model microorganism Chlamydomonas reinhardtii[J].Sci Rep-UK,2016,6 :34109.
[23] ROBERTS A,PIMENTEL H,TRAPNELL C,et al.Identification of novel transcripts in annotated genomes using RNA-Seq [J].Bioinformatics,2011,27(17):2325-2329.
[24] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method[J].Methods,2012,25(4):402-408.
[25] JE-HYUN Y,KOTB A,MYRIAM G.Posttranscriptional gene regulation by long noncoding RNA[J].J Mol Biol,2013,425(19): 3723-30.
[26] WILUSZ J E.Long noncoding RNAs: Re-writing dogmas of RNA processing and stability[J].Biochim Biophys Acta,2015,1859(1):128-138.
[27] SZEKERES M,NEMETH K,KONCEZ-KALMAZ Z,et al.Brassinosteroids rescue the deficiency of CYP90,a cytochrome P450,controlling cell elongation and de-etiolation in Arabidopsis [J].Cell,1996,85(2):171-182.
[28] 关钰,王海娇,王学路.BKI1通过其22个氨基酸区域与BRI1的互作来调控油菜素甾醇信号 [J].复旦学报(自然科学版),2011(3):320-327.
[29] NAM K H,LI J.BRI1/ BAK1,a receptor kinase pair media ting brassinosteroid signaling [J].Cell,2002,110(2):203-212.
[30] TANG W,KIM T W,OSES-PRIETO J A,et al.BSKs mediate signal transduction from the receptor kinase BRI1 in Arabidopsis [J].Science,2008,321(5888):557-560.
[31] WANG X,CHORY J.Brassinosteroids regulate dissociation of BKI1,a negative regulator of BRI1 signaling from the Plasma Membrane[J].Science,2006,313(5790):1118-1122.
[32] WANG Z Y,SETO H,FUJIOKA S,et al.BRI1 is a critical component of a plasma-membrane receptor for plant steroids [J].Nature.2001,410(6826):380-383.
[33] AGARIE S,MIURA A,SUMIKURA R,et al.Over expression of C4 PEPC caused O2-insensitive photosynthesis in transgenic rice plant [J].Plant Sci,2002,162 : 257-265.
[34] 周宝元,丁在松,赵明.PEPC过表达可以减轻干旱胁迫对水稻光合的抑制作用 [J].作物学报,2011,(1):112-118.
(责任编辑:李莹)
Changesofexpressionoflongnon-codingRNAsinPaulowniatomentosatreatedwithrifampin
LI Yongsheng, LI Bingbing, CAO Yabing, WANG Zhe, DONG Yanpeng, DENG Minjie, ZHAO Zhenli, FAN Guoqiang
(Institute of Paulownia,Henan Agricultural University,Zhengzhou 450002,China)
To elucidate the regulation of lncRNAs in the growth and development ofPaulownia,the high-throughput sequencing technology was used to explore the expression of long non-coding RNA and its target genes after being treated with rifampin inPaulowniatomentosa.A total of 129 differentially expressed long non-coding RNAs and 289 corresponding target genes were identified.GO analysis showed that these target genes involved in 9 categories,and most of them involved in cellular process,metabolic process,single-organism process and other classifications.KEGG pathway analysis indicated that these differentially expressed target genes were involved in plant hormone signal transduction and secondary metabolic biosynthesis.To confirm the expression of Paulownia lncRNAs and their target genes,qRT-PCR analysis was performed to verify the results of the RNA-Seq.Six lncRNAs and their corresponding target genes were selected randomly from the PT and PTL-100 libraries for quantitative PCR.The result suggested that the RNA-seq dataset was dependable.
Paulowniatomentosa; rifampin treatment; long non-coding RNA
2017-07-03
国家公益性林业行业科研专项基金项目(201004002)
李永生(1981-),男,河南南阳人,讲师,博士,主要从事泡桐生物技术研究工作。
范国强(1964-),男,河南禹州人,教授,博士,博士生导师。
1000-2340(2017)05-0640-07
S792.43
A

