甘菊DlNAC1转录因子基因提高烟草耐高温能力
朱 凯 武 剑 杨艳芳 陈发棣 喻德跃
(1.南京农业大学大豆研究所,国家大豆改良中心,作物遗传与种质创新国家重点实验室,南京 210095; 2.中国林业科学研究院林业研究所,林木遗传育种国家重点实验室,国家林业局林木培育重点实验室,北京 100091; 3.南京农业大学园艺学院观赏园艺系,南京 210095)
甘菊DlNAC1转录因子基因提高烟草耐高温能力
朱 凯1武 剑1杨艳芳2*陈发棣3喻德跃1
(1.南京农业大学大豆研究所,国家大豆改良中心,作物遗传与种质创新国家重点实验室,南京 210095;2.中国林业科学研究院林业研究所,林木遗传育种国家重点实验室,国家林业局林木培育重点实验室,北京 100091;3.南京农业大学园艺学院观赏园艺系,南京 210095)
研究针对从甘菊中克隆获得的DlNAC1基因(GenBank登录号为EF602305)进行生物信息学分析,并利用根癌农杆菌介导的叶盘转化法将该基因在烟草中进行过表达研究。结果发现DlNAC1蛋白具有较高亲水性,二级结构中占比最高的为无规则卷曲,并具有N糖基化位点等6类潜在的模体结构和典型的由一个扭曲的反平行β片层和α螺旋组成的NAC结构域。将DlNAC1基因在烟草中过表达后,通过PCR方法从55株转化植株中鉴定出36株为阳性植株,并且转基因烟草T0代植株在45℃高温胁迫后,转35S:DlNAC1基因阳性植株生长状况良好,而对照植株发生萎蔫,并且转基因植株叶片含水量显著高于对照植株。然而,在4℃低温胁迫后,发现转基因烟草T1代植株没有提高耐低温能力。甘菊DlNAC1基因能够提高烟草植株耐高温能力,为今后菊花抗逆育种提供了科学依据。
菊花;NAC转录因子;生物信息学;高温胁迫;转基因烟草
转录因子(Transcription factor,TF)通常是由基因编码的一类蛋白质,能够与位于基因启动子区域的顺式作用元件发生特异性结合,通过和某些辅助调控因子发生作用而影响转录复合体的形成,从而调节植物基因的特异性表达[1]。目前研究证明,AP2/ERF、MYB、bHLH、WRKY、bZIP和NAC等多种转录因子,在植物生长发育、病原威胁以及低温、干旱等的逆境胁迫中起到非常重要的作用[2~6]。
NAC转录因子广泛分布于陆生植物中,并且数量众多。自首先从矮牵牛中发现NAC基因以来[7],还从拟南芥等多种植物中发现了NAC转录因子[8~10]。例如,通过全基因组分析发现,在模式植物拟南芥和水稻中分别存在117个和151个NAC基因[11~12]。研究表明NAC基因家族在植物生长发育、激素调控和信号转导等过程中发挥作用,还与光反应、细胞的程序性凋亡以及衰老有关[13~20]。此外,研究还发现多个NAC转录因子与植物的非生物胁迫相关。例如,Hong等[21]将水稻来源的ONAC022过量表达,提高了转基因水稻的耐干旱和耐盐能力。
甘菊(Dendranthemalavandulifolium(Fisch. ex Trautv.) Makino)为菊科(Asteraceae)多年生植物,不仅具有较高的观赏价值,还拥有良好的抗逆性。目前,也有一些有关菊花NAC转录因子基因的相关研究报道。Huang等[22]利用RNA-Seq技术,在菊花(Chrysanthemumlavandulifolium)中发现了44个NAC同源基因的表达在高盐胁迫下表达量升高,并且其中的15个NAC基因受到ABA诱导调控。本研究组前期工作中从甘菊中克隆得到一个NAC转录因子同源基因——DlNAC1,并初步确定这个基因与菊花的干旱和高盐等非生物胁迫的耐受性相关[23]。本研究通过生物信息学方法,对DlNAC1基因所编码的蛋白序列进行了分析,并利用根癌农杆菌介导法将DlNAC1基因转入烟草,分析了转基因植株在高温胁迫下的含水量和低温胁迫下的存活率,为今后深入研究DlNAC1基因功能和利用其改良植物抗逆性提供理论基础。
1 材料与方法
1.1 实验材料
烟草品种为‘三生烟’(Nicotianatabacum‘Xantbine’),用培养45 d的烟草的幼嫩叶片作转化的外植体。
1.2 实验方法
1.2.1 生物信息学分析
分别利用ExPaSy网站的Protparam(http://web.expasy.org/protparam/)进行基本理化性质分析,利用ProtScale(http://web.expasy.org/protscale/)进行蛋白质疏水性的计算,利用SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行二级结构分析,利用ProScan(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_proscan.html)进行模体分析,利用Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/)进行三维结构模拟分析。
1.2.2 烟草转化及再生
研究中植物表达载体35S:DlNAC1为研究组前期构建并保存,利用液氮速冻法将植物表达载体35S:DlNAC1导入根癌农杆菌菌株EHA105中。以培养45 d的烟草倒数第4、5片叶作为转化受体,采用叶盘法[24]转化烟草并再生获得转基因烟草。
1.2.3T0代转基因阳性植株的鉴定
CTAB法提取移栽至营养钵中抗卡那霉素转基因烟草的基因组DNA,以此作为模板,利用扩增DlNAC1 cDNA全长序列为模板分别设计特异性引物:NAC-P1,5′-GCTCTAGAATGGAGGATACTCGGTTA-3′;NAC-P2,5′-CGGGATCCGTTTAAGAAATTAAGCAT-3′,进行PCR扩增鉴定。反应体系为25 μL,反应条件为95℃预变性5 min,95℃ 30 s,58℃ 40 s,72℃ 60 s,32个循环。野生型烟草作为阴性对照。
1.2.4热胁迫处理下转35S:DlNAC1烟草T0代叶片相对含水量的测定
选取从生根培养基中移栽到含营养土∶蛭石(1∶1)的花盆中继续生长2个月的转35S:DlNAC1烟草T0代阳性植株为研究对象,以野生型烟草为对照,45℃条件下高温胁迫处理6 h,分别在处理的第0、2、4、6 h将叶片从植株上取下,立即称鲜重(Wf),然后将叶片浸入去离子水中4℃过夜,取出后用滤纸吸去表面水分,称饱和鲜重(Wt),然后将叶片放入烘箱于70℃烘干至恒重,称干重(Wd),3次重复。相对含水量计算公式如下:
相对含水量(Relative water content,RWC)(%)=(Wf-Wd)/(Wt-Wd)×100%
(1)
1.2.5转35S:DlNAC1烟草T1代阳性植株筛选及遗传分析
为了鉴定转35S:DlNAC1烟草的耐逆性,选择经PCR检测为转35S:DlNAC1阳性株系(L-31、L-32和L-33)的T0代种子和野生型烟草种子,用70%酒精(乙醇)和10%H2O2进行表面消毒,无菌水冲洗5~6次后播种于1/2MS培养基上。转基因烟草种子中加100 mg·L-1卡那霉素,非转基因对照不加。置于光照16 h/黑暗8 h交替的25℃温室中筛选培养30 d,统计卡那霉素抗性的分离比。
用卡方检测进行显著性分析,以此来判断基因后代卡那霉素抗性苗和敏感苗的比例是否符合孟德尔遗传规律。
1.2.6转35S:DlNAC1烟草T1代低温胁迫下存活率分析
将在1/2MS培养基上经卡那霉素抗性筛选后正常生长30 d的转35S:DlNAC1烟草T1代植株移栽到装有营养土∶蛭石(1∶1)的花盆中,于25℃的环境下继续生长20 d。将烟草植株在(-4±1)℃条件胁迫处理8 h,然后置于室温下恢复7 d。3次重复,每个重复10株烟草。以上处理,以同等生长期的野生型烟草作为对照。
1.2.7 数据统计分析
利用SAS(ver.9.0,SAS Institute,Inc)软件统计,采用Duncan(P<0.05)多重比较法对数据进行分析。
2 结果与分析
2.1 DlNAC1蛋白理化性质分析
研究组前期工作中,从甘菊中克隆获得一个NAC-like基因——DlNAC1,其cDNA序列长度为934 bp,编码一个262个氨基酸的开放阅读框(Open read frame,ORF)。其编码的蛋白DlNAC1分子量为29.9 kD,等电点pI为~9.24,运用ExPaSy-Protparam工具对该蛋白其他理化性质进行了预测分析,结果如表1所示:DlNAC1负电荷残基数为30,正电荷残基数均为35,不稳定指数为45.63,脂肪指数为70.30,亲水性为-0.585,表明此蛋白为亲水性蛋白。
2.2 蛋白亲水性/疏水性预测
本研究利用ExPaSy-ProtScale对DlNAC1蛋白进行了亲水性/疏水性分析(图1),根据氨基酸亲水性越强分值越低,疏水性越强分值越高的规律,从整体结果看,DlNAC1蛋白编码的氨基酸序列中,亲水性氨基酸分布多于疏水性氨基酸,表明这个蛋白属于亲水性蛋白。
表1DlNAC1蛋白理化性质分析
Table1ThebasicphysicochemicalpropertiesofDlNAC1
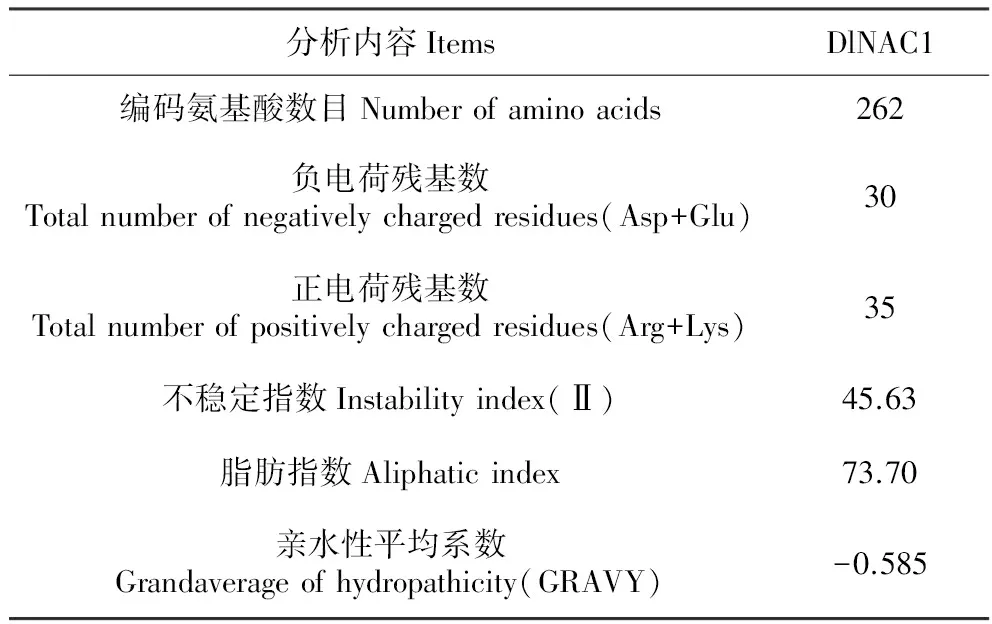
分析内容ItemsDlNAC1编码氨基酸数目Numberofaminoacids262负电荷残基数Totalnumberofnegativelychargedresidues(Asp+Glu)30正电荷残基数Totalnumberofpositivelychargedresidues(Arg+Lys)35不稳定指数Instabilityindex(Ⅱ)45.63脂肪指数Aliphaticindex73.70亲水性平均系数Grandaverageofhydropathicity(GRAVY)-0.585
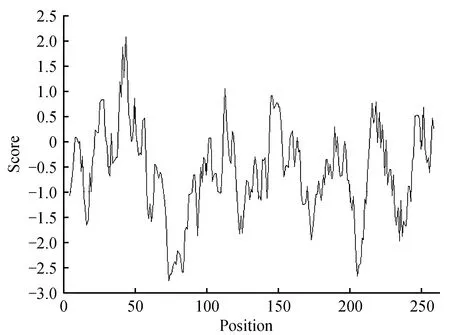
图1 DlNAC1蛋白亲水性/疏水性分析Fig.1 Hydrophobic analysis of DlNAC1
2.3 DlNAC1二级结构分析
利用ExPaSy-SOPMA对DlNAC1蛋白的二级结构进行分析,发现其主要由4种形式组成,并且以无规则卷曲(R.coil)占比最高,为53.44%,其次为α-螺旋(21.37%)和延伸链(19.47%);而β-转角的占比最低,为5.73%。同时,DlNAC1蛋白的N端主要分布为α-螺旋和延伸链,C端主要分布为α-螺旋和无规则卷曲,N末端和C末端都是以α-螺旋的形式存在(图2)。
2.4 模体分析
利用NPS提供的Proscan对DlNAC1蛋白进行模体扫描,表1显示,DlNAC1蛋白具有6类模体结构,其中包含3个潜在的N糖基化位点、1个潜在的cAMP-和cGMP依赖的蛋白激酶磷酸化位点、5个潜在的蛋白激酶C磷酸化位点、7个潜在的酪蛋白激酶II磷酸化位点、1个潜在的酪氨酸激酶磷酸化位点和3个N-豆蔻酰化位点(表2)。
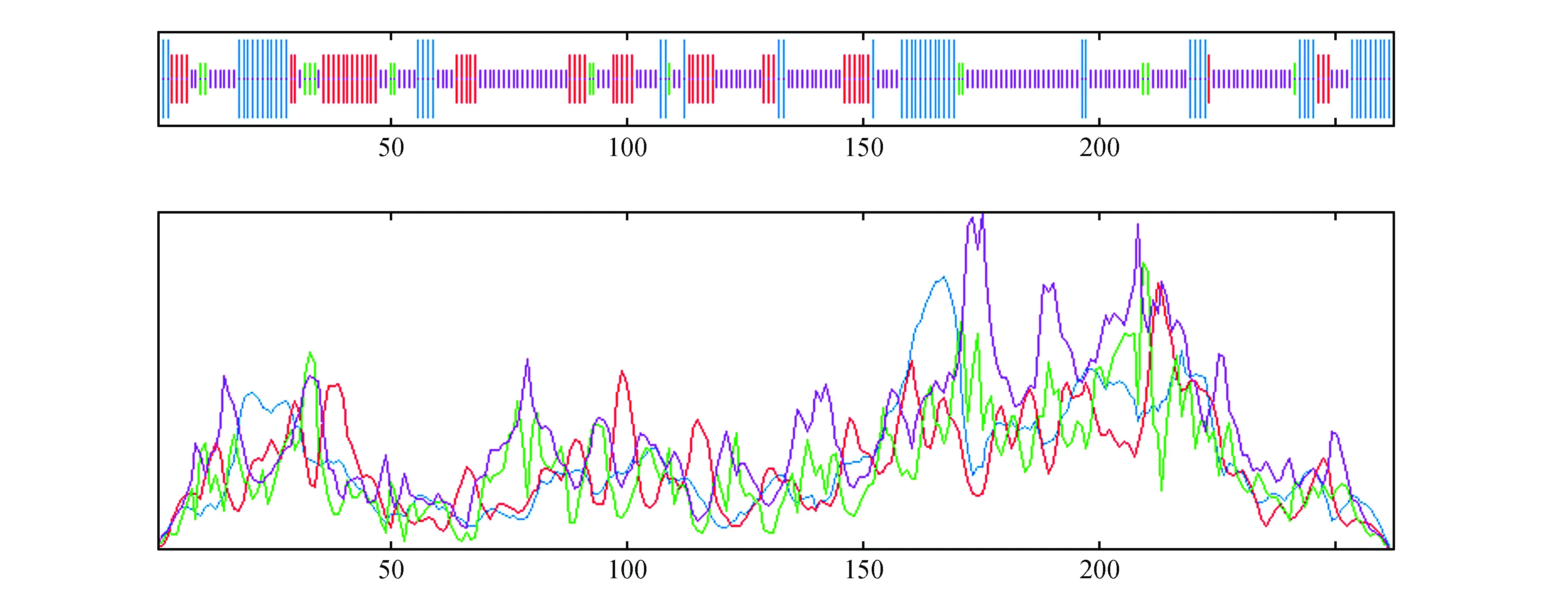
图2 DlNAC1蛋白二级结构预测分析 蓝色:α螺旋;红色:延伸链;紫色:无规则卷曲;绿色:β-转角Fig.2 The predicated secondary structure of DlNAC1 protein Blue:Alpha helix; Red:Extended strand; Purple:Random coil; Green:Beta turn
Table2ScanningofDlNAC1proteinforsite/signatureswithProScansoftware
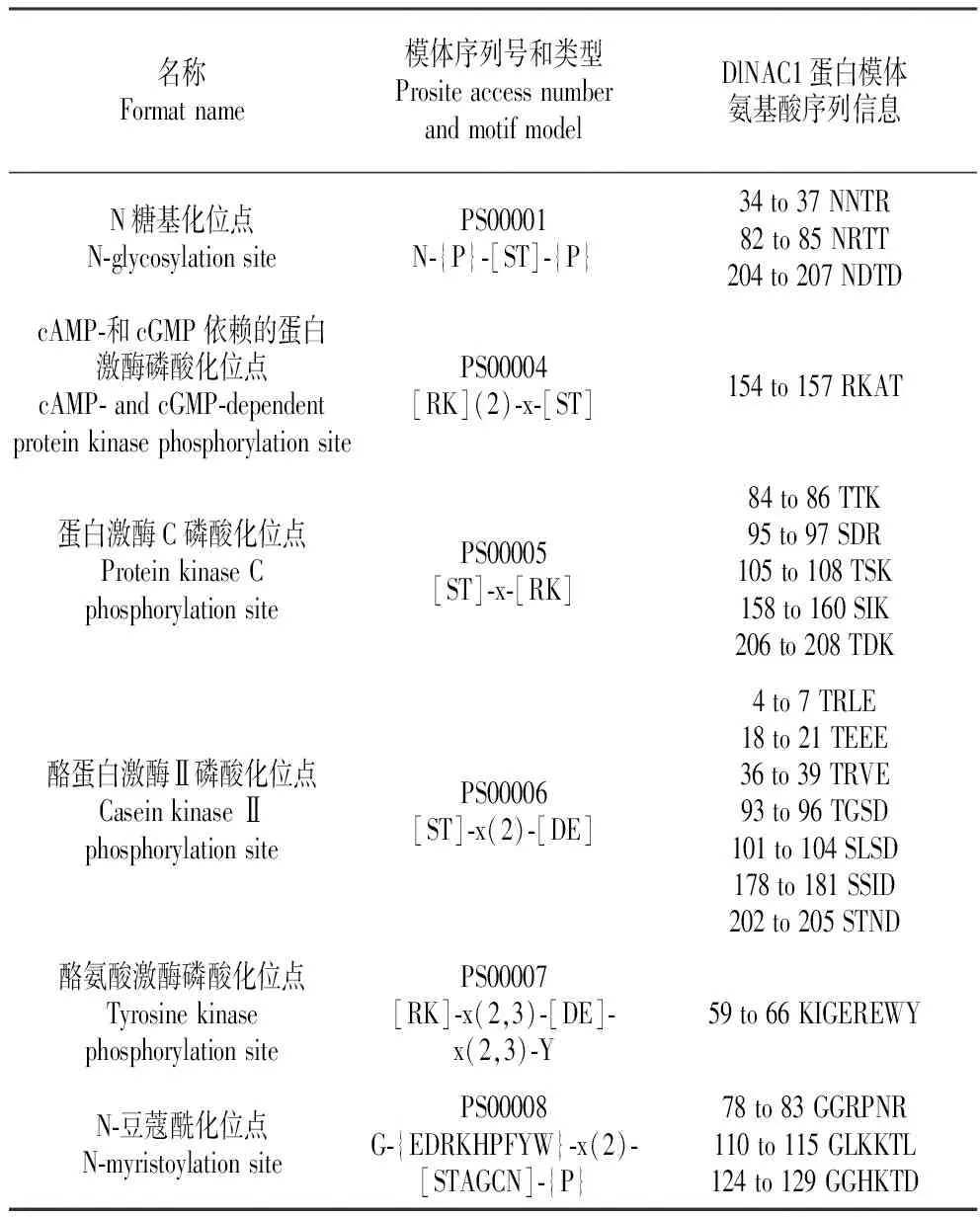
名称Formatname模体序列号和类型PrositeaccessnumberandmotifmodelDlNAC1蛋白模体氨基酸序列信息N糖基化位点N⁃glycosylationsitePS00001N⁃{P}⁃[ST]⁃{P}34to37NNTR82to85NRTT204to207NDTDcAMP⁃和cGMP依赖的蛋白激酶磷酸化位点cAMP⁃andcGMP⁃dependentproteinkinasephosphorylationsitePS00004[RK](2)⁃x⁃[ST]154to157RKAT蛋白激酶C磷酸化位点ProteinkinaseCphosphorylationsitePS00005[ST]⁃x⁃[RK]84to86TTK95to97SDR105to108TSK158to160SIK206to208TDK酪蛋白激酶Ⅱ磷酸化位点CaseinkinaseⅡphosphorylationsitePS00006[ST]⁃x(2)⁃[DE]4to7TRLE18to21TEEE36to39TRVE93to96TGSD101to104SLSD178to181SSID202to205STND酪氨酸激酶磷酸化位点TyrosinekinasephosphorylationsitePS00007[RK]⁃x(2,3)⁃[DE]⁃x(2,3)⁃Y59to66KIGEREWYN⁃豆蔻酰化位点N⁃myristoylationsitePS00008G⁃{EDRKHPFYW}⁃x(2)⁃[STAGCN]⁃{P}78to83GGRPNR110to115GLKKTL124to129GGHKTD
2.5 DlNAC1转录因子三维结构分析
利用ExPaSy-phyre2在线预测DlNAC1转录因子的三维结构,可以看出DlNAC1蛋白具有NAC转录因子家族的典型特征(图3),具有由一个扭曲的反平行β片层和α螺旋组成的NAC结构域。
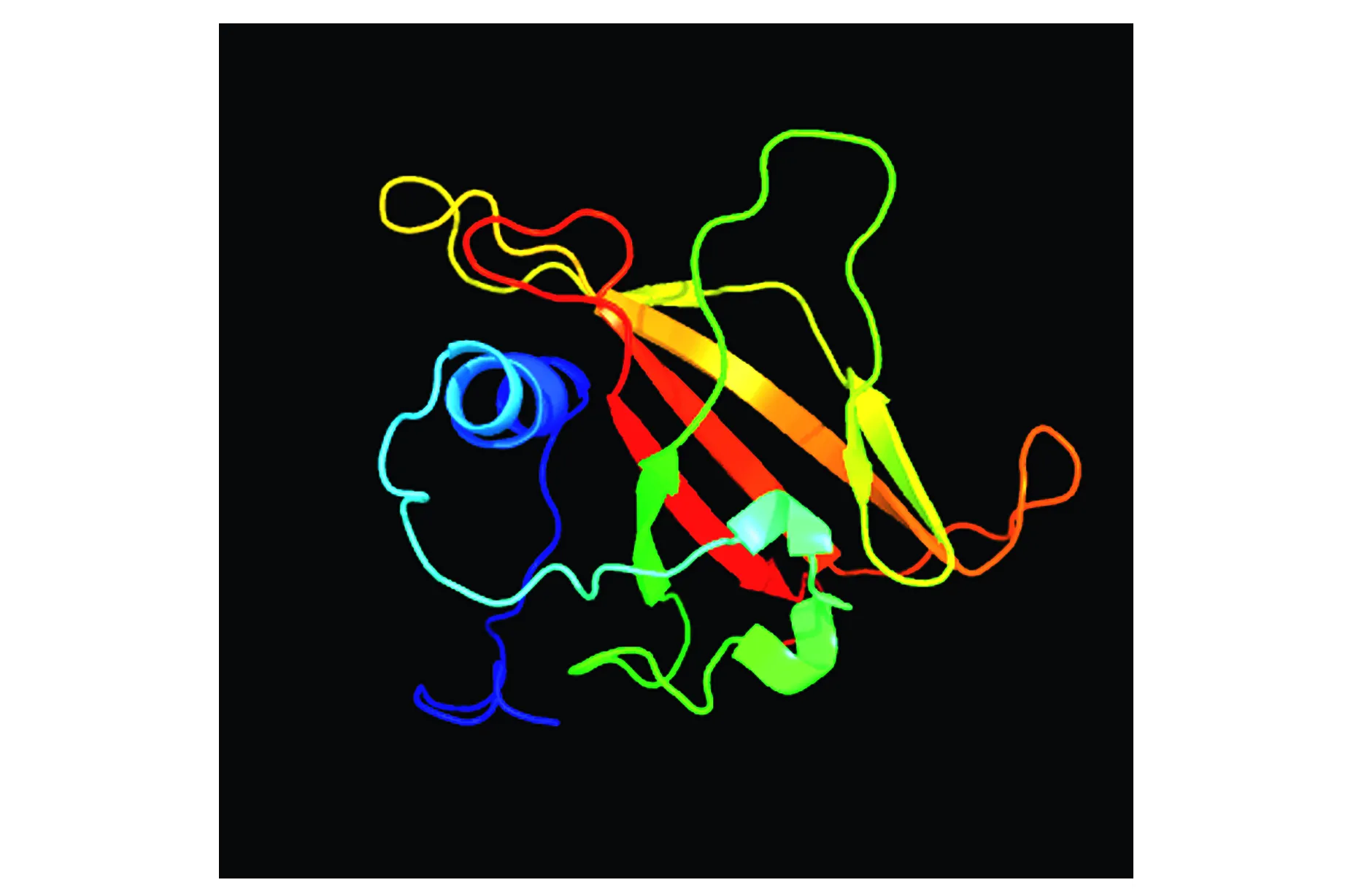
图3 DlNAC1蛋白三维结构分析Fig.3 Tertiary structure of DlNAC1 protein
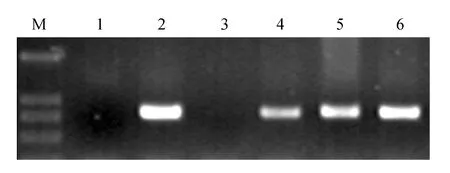
图4 转35S:DlNAC1烟草植株PCR检测(部分结果)M.DL2000;1.野生型野草(阴性对照);2. 35S:DlNAC1质粒(阳性对照);3~6.转基因植株Fig.4 The PCR identification of 35S:DlNAC1 transgenic plants(part results) M. DL2000; 1. Wild type(negative control); 2. 35S:DlNAC1 plasmid(positive control); 3-6. Transgenic plants
2.6 转35S:DlNAC1基因烟草阳性检测
选取55株经卡那霉素鉴定为阳性的烟草植株,提取其基因组DNA,并以野生型烟草和35S:DlNAC1质粒分别作为阴性对照和阳性对照,进行PCR鉴定。结果发现36株转35S:DlNAC1植株中扩增出与阳性质粒PCR产物相同的条带,而作为阴性对照的非转基因烟草样品未扩增出任何条带(图4)。
2.7转35S:DlNAC1烟草T0代耐高温能力分析
将转35S:DlNAC1烟草T0代植株和对照植株进行45℃高温处理,由图5A所示,高温胁迫处理6 h后,对照植株叶片明显萎蔫,植株倒伏,转35S:DlNAC1烟草T0代L-6和L-11植株则能保持一定的含水量,叶片没有发生明显萎蔫现象,L-15植株下部部分叶片出现萎蔫,而顶部叶片未发生萎蔫现象。叶片相对含水量分析表明,转DlNAC1基因植株与对照植株在45℃处理前两者叶片相对含水量无明显差异,但在高温胁迫处理后,转35S:DlNAC1植株叶片相对含水量与对照相比下降较慢,在胁迫处理6 h时,转35S:DlNAC1植株叶片相对含水量显著高于对照(图5B)。以上结果均表明,转35S:DlNAC1植株对高温胁迫的抵抗能力与对照相比有所增强。
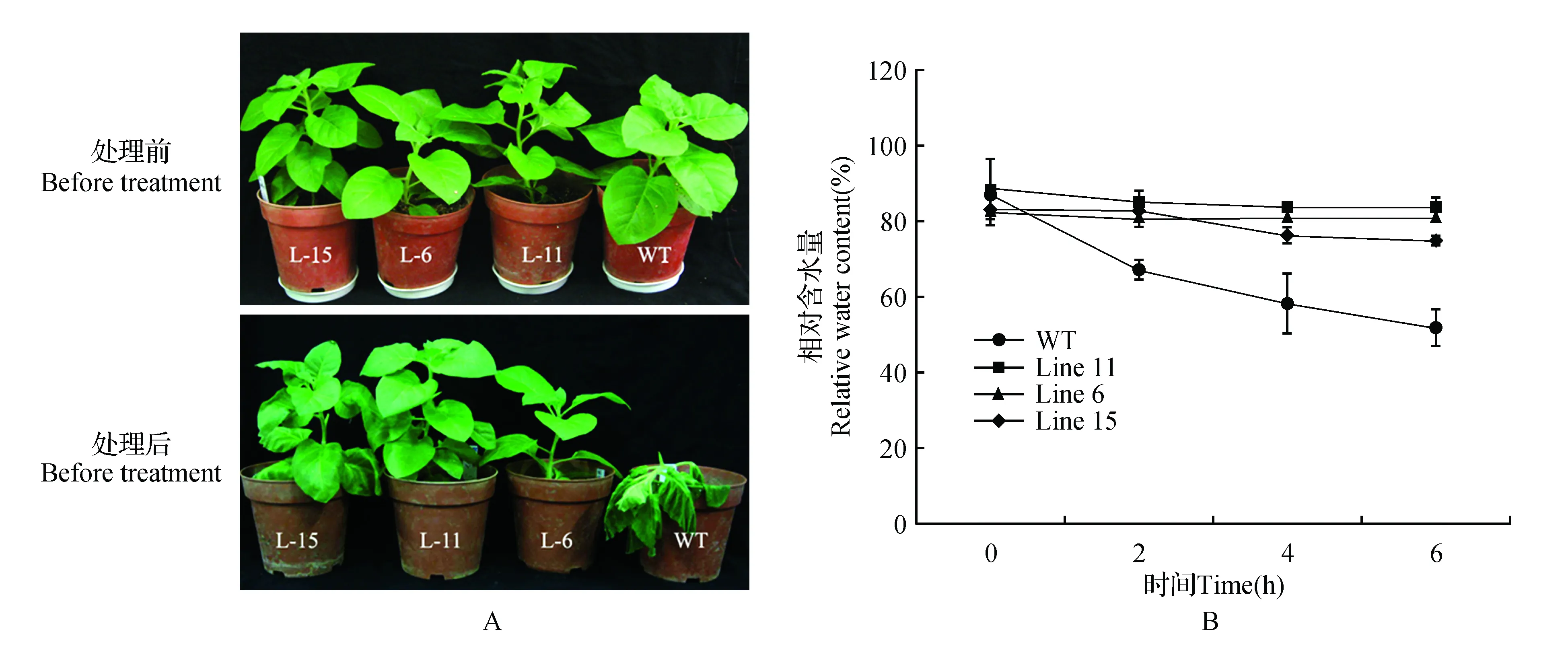
图5 高温胁迫对转35S:DlNAC1植株相对含水量的影响Fig.5 Relative water content of 35S:DlNAC1 plants exposed to heat stress
2.8转35S:DlNAC1烟草T1代阳性植株筛选及遗传分析
利用PCR方法检测的转35S:DlNAC1烟草阳性植株当代(T0)自交后所得的T1代种子(L-31、L-32、L-33)为研究材料,进行卡那霉素抗性分离比测定。结果表明:转基因种子播种于含有100 mg·L-1卡那霉素的1/2MS培养基上,15 d长成约0.8 cm高的小苗,根长可达到2~3 cm,30 d长成约高3 cm的小苗;而未转基因种子和卡那霉素抗性阴性的种子虽能萌发,但不长根,最后黄化死亡。根据种子萌发总数、长根并能生长与不长根不生长的总数计算比值(表3)。由表3可知,转35S:DlNAC1阳性株系L-31、L-32和L-33的分离比基本符合孟德尔3∶1的遗传规律。
2.9 转35S:DlNAC1烟草低温胁迫下存活率分析
为了研究转35S:DlNAC1烟草的耐冷性,选取生长了50 d的转35S:DlNAC1阳性株系L-31、L-32和L-33为研究对象,以野生型烟草为对照,进行了(-4±1)℃胁迫处理。如图6A所示,转35S:DlNAC1烟草L-31和L-33株系和对照在低温胁迫处理后存活率没有明显差异,而L-32株系的存活率与前三者相比较低。由图6B也可以看出,低温处理后,L-31和L-33株系存活植株数目与长势与对照无明显差异,而L-32株系存活植株和长势则明显不如对照和L-31和L-33株系。该结果表明将DlNAC1基因转入烟草没有提高转基因植株耐低温的能力。
表3T1代种子萌发率及卡那霉素抗性检测
Table3T1seeds’germinationrateandkanarmycinselection
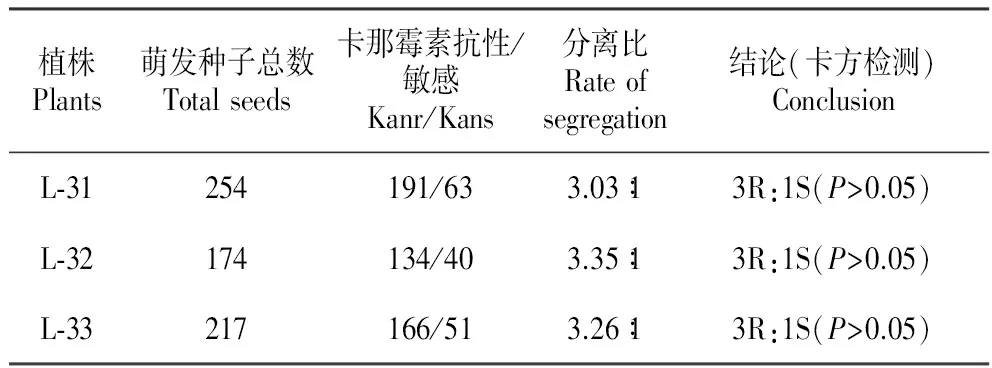
植株Plants萌发种子总数Totalseeds卡那霉素抗性/敏感Kanr/Kans分离比Rateofsegregation结论(卡方检测)ConclusionL⁃31254191/633.03∶13R:1S(P>0.05)L⁃32174134/403.35∶13R:1S(P>0.05)L⁃33217166/513.26∶13R:1S(P>0.05)
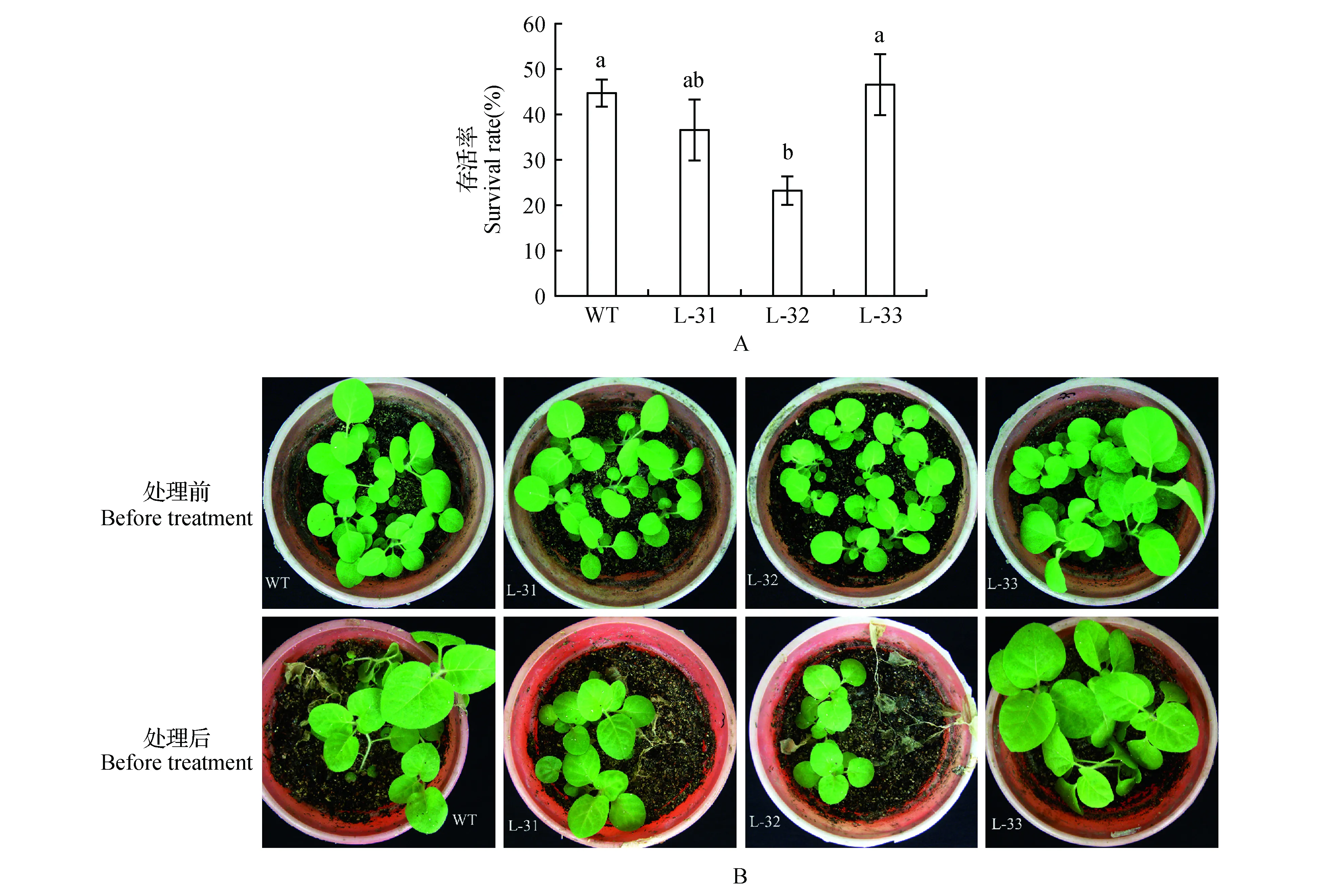
图6 低温胁迫对转35S:DlNAC1植株存活率的影响Fig.6 Survival rates of 35S:DlNAC1 plants exposed to freezing stress
3 讨论
蛋白质的序列信息是预测其结构的基础,并且蛋白质的低级结构决定了高级结构和功能。因此,分析蛋白质的氨基酸序列具有重要意义。本文中通过生物信息手段针对研究组前期从甘菊中所获得的DlNAC1基因编码的氨基酸序列进行了分析,发现该基因编码一个亲水蛋白,该蛋白二级结构主要为无规则卷曲,占比高达53.44%,三维结构分析发现该蛋白具有一个典型的NAC家族结构域。这与前人研究结果较为一致[25~26]。黎帮勇等[25]报道毛竹中NAC基因家族的125氨基酸序列大部分为亲水蛋白,其中有65条亲疏水性值小于-0.5,并且这125条毛竹NAC家族蛋白质二级结构也几乎都以无规则卷曲占比最高。
NAC转录因子的一个重要的生物学功能就是参与植物对非生物胁迫应答反应。高温是植物生长发育过程中的主要逆境之一,目前,研究报道已经有多个NAC转录因子参与调控植物的高温胁迫[8,27~31]。例如,在拟南芥中,NAC078转录因子就与热激反应有关[8]。此外,水稻中的SNAC3基因强烈被高温诱导,过表达该基因后,对转基因水稻植株进行42℃高温胁迫,发现40%转基因植株存活下来,而野生型只有20%存活[28]。研究发现NAC转录因子调控AP2/ERF家族的DREB2A转录因子,进而调控了包含热激蛋白HSP(heat stress protein)基因在内的众多下游基因的表达[27,31]。在本研究中,高温胁迫下过表达DlNAC1烟草T0代植株长势明显好于野生型,且转基因烟草叶片较野生型烟草叶片能够维持较好的相对含水量,表明过表达DlNAC1烟草提高了抵抗高温胁迫的能力。然而,有关DlNAC1是否调控DREB2A而提高耐高温能力的分子机理,尚需要进一步研究去揭示。
菊花是我国十大名花之一,栽培范围广泛,具有较强的抗寒和抗旱能力。目前,已经从菊花中分离克隆了多个与逆境相关的转录因子基因,如,DREB、锌指蛋白基因、WRKY和NAC等,研究也证实这些转录因子基因确实能够提高转基因植株的耐逆能力[22,32~36]。例如,在拟南芥中过表达菊花来源的ChiMYB基因,提高了转基因植株的耐旱性和耐盐能力[37]。本研究组前期研究发现,在烟草中过量表达菊花来源的DmDREBa基因能够提高烟草的耐低温和耐干旱能力[38~39],而DmDREBb基因则更积极响应高盐胁迫[39]。此外,我们还发现过表达DlNAC1能够增加烟草的耐盐和耐旱能力[23]。然而在本文中,却发现过表达DlNAC1基因的烟草植株并没有提高抗低温能力。推测原因,可能是受到寄主基因的影响,也有可能与基因插入位置、拷贝数等有关。由此可见,转录因子调控下游耐逆基因的表达是一个非常复杂的过程。这些针对菊花转录因子基因展开的耐逆性研究,都为今后利用NAC等转录因子基因,改良提高菊花的耐逆性提供了基因资源和理论基础。
1.Yamaguchi-shinozaki K,Shinozaki K.Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses[J].Annual Review of Plant Biology,2006,57(1):781-803.
2.Du M M,Zhai Q Z,Deng L,et al.Closely related NAC transcription factors of tomato differentially regulate stomatal closure and reopening during pathogen attack[J].The Plant Cell,2014,26(7):3167-3184.
3.Ko S S,Li M J,Ku M S B,et al.The bHLH142 transcription factor coordinates with TDR1 to modulate the expression ofEAT1 and regulate pollen development in rice[J].The Plant Cell,2014,26(6):2486-2504.
4.Vermeirssen V,De Clercq I,Van Parys T,et al.Arabidopsisensemble reverse-engineered gene regulatory network discloses interconnected transcription factors in oxidative stress[J].The Plant Cell,2014,26(12):4656-4679.
5.Xu Z Y,Kim S Y,Hyeon D Y,et al.TheArabidopsisNAC transcription factor ANAC096 cooperates with bZIP-Type transcription factors in dehydration and osmotic stress responses[J].The Plant Cell,2013,25(11):4708-4724.
6.Pautler M,Eveland A L,Larue T,et al.FASCIATEDEAR4 encodes a bZIP transcription factor that regulates shoot meristem size in maize[J].The Plant Cell,2015,27(1):104-120.
7.Souer E,Vanhouwelingen A,Kloos D,et al.The no apical meristem gene ofPetuniais required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J].Cell,1996,85(2):159-170.
8.Morishita T,Kojima Y,Maruta T,et al.ArabidopsisNAC transcription factor,ANAC078,regulates flavonoid biosynthesis under high-light[J].Plant and Cell Physiology,2009,50(12):2210-2222.
9.申玉华,徐振军,唐立红,等.紫花苜蓿NAC类转录因子基因MsNAC2的克隆及其功能分析[J].中国农业科学,2015,48(15):2925-2938.
Shen Y H,Xu Z J,Tang L H,et al.Cloning and function analysis of theMsNAC2 gene with NAC transcription factor from Alfalfa[J].Scientia Agricultura Sinica,2015,48(15):2925-2938.
10.姜秀明,牛义岭,许向阳.番茄NAC基因家族的系统进化及表达分析[J].分子植物育种,2016,14(8):1948-1964.
Jiang X M,Niu Y L,Xu X Y.Phylogenetic evolution and expression analysis ofNACgene family in tomato(Solanumlycopersicum)[J].Molecular Plant Breeding,2016,14(8):1948-1964.
11.Nuruzzaman M,Manimekalair,Sharoni A M,et al.Genome-wide analysis of NAC transcription factor family in rice[J].Gene,2010,465(1-2):30-44.
12.Le D T,Nishiyama R,Watanabe Y,et al.Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J].DNA Research,2011,18(4):263-276.
13.Duval I,Lachance D,Giguère I,et al.Large-scale screening of transcription factor-promoter interactions in spruce reveals a transcriptional network involved in vascular development[J].Journal of Experimental Botany,2014,65(9):2319-2333.
14.Shibuya K,Shimizu K,Niki T,et al.Identification of a NAC transcription factor,EPHEMERAL1,that controls petal senescence in Japanese morning glory[J].The Plant Journal,2014,79(6):1044-1051.
15.Xu B,Ohtani M,Yamaguchi M,et al.Contribution of NAC transcription factors to plant adaptation to land[J].Science,2014,343(6178):1505-1508.
16.Jensen M K,Hagedorn P H,De Torres-zabala M,et al.Transcriptional regulation by an NAC(NAM-ATAF1,2-CUC2) transcription factor attenuates ABA signalling for efficient basal defence towardsBlumeriagraminisf.sp.hordeiinArabidopsis[J].The Plant Journal,2008,56(6):867-880.
17.杨晓娜,田云,卢向阳.NAC转录因子在植物生长发育中的调控作用[J].化学与生物工程,2014,31(1):1-5,25.
Yang X N,Tian Y,Lu X Y.The regulation role of NAC transcription factors in plant growth and development[J].Chemistry & Bioengineering,2014,31(1):1-5,25.
18.Peng H,Zhao J F,Neff M M.ATAF2 integratesArabidopsisbrassinosteroid inactivation and seedling photomorphogenesis[J].Development,2015,142(23):4129-4138.
19.Pimenta M R,Silva P A,Mendes G C,et al.The stress-induced soybean NAC transcription factor gmNAC81 plays a positive role in developmentally programmed leaf senescence[J].Plant and Cell Physiology,2016,57(5):1098-1114.
20.Takasaki H,Maruyama K,Takahashi F,et al.SNAC-As,stress-responsive NAC transcription factors,mediate ABA-inducible leaf senescence[J].The Plant Journal,2015,84(6):1114-1123.
21.Hong Y B,Zhang H J,Huang L,et al.Overexpression of a stress-responsive NAC transcription factor geneONAC022 improves drought and salt tolerance in rice[J].Frontiers in Plant Science,2016,7:4.
22.Huang H,Wang Y,Wang S L,et al.Transcriptome-wide survey and expression analysis of stress-responsiveNACgenes inChrysanthemumlavandulifolium[J].Plant Science,2012,193-194:18-27.
23.Yang Y F,Zhu K,Wu J,et al.Identification and characterization of a novelNAC-like gene in chrysanthemum(Dendranthemalavandulifolium)[J].Plant Cell Reports,2016,35(8):1783-1798.
24.高越峰,朱祯,肖桂芳,等.大豆Kunitz型胰蛋白酶抑制剂基因的分离及其在抗虫植物基因工程中的应用[J].植物学报,1998,40(5):405-411.
Gao Y F,Zhu Z,Xiao G F,et al.Isolation of soybean Kunitz trypsin inhibitor gene and its application in plant insect-resistant genetic engineering[J].Acta Botinica Sinica,1998,40(5):405-411.
25.黎帮勇,胡尚连,曹颖,等.毛竹NAC转录因子家族生物信息学分析[J].基因组学与应用生物学,2015,34(8):1769-1777.
Li B Y,Hu S L,Cao Y,et al.Bioinformatics analysis ofNACgene family in moso bamboo[J].Genomics and Applied Biology,2015,34(8):1769-1777.
26.岳俊燕,岳文冉,杨杞,等.中间锦鸡儿转录因子基因CiNAC1的克隆及功能分析[J].西北植物学报,2016,36(7):1285-1293.
Yue J Y,Yue W R,Yang Q,et al.Cloning and functional analysis ofCiNAC1 fromCaraganaintermedia[J].Acta Botanica Boreali-Occidentalia Sinica,2016,36(7):1285-1293.
27.Wu A H,Allu A D,Garapati P,et al.JUNGBRUNNEN1,a reactive oxygen species-responsive NAC transcription factor,regulates longevity inArabidopsis[J].The Plant Cell,2012,24(2):482-506.
28.Fang Y J,Liao K F,Du H,et al.A stress-responsive NAC transcription factor SNAC3 confers heat and drought tolerance through modulation of reactive oxygen species in rice[J].Journal of Experimental Botany,2015,66(21):6803-6817.
29.Niu F F,Wang C,Yan J L,et al.Functional characterization of NAC55 transcription factor from oilseed rape(BrassicanapusL.) as a novel transcriptional activator modulating reactive oxygen species accumulation and cell death[J].Plant Molecular Biology,2016,92(1-2):89-104.
30.Wang A H,Hu J H,Huang X X,et al.Comparative transcriptome analysis reveals heat-responsive genes in Chinese Cabbage(Brassicarapassp.chinensis)[J].Frontiers in Plant Science,2016,7:939.
31.Sakuma Y,Maruyama K,Qin F,et al.Dual function of anArabidopsistranscription factor DREB2A in water-stress-responsive and heat-stress-responsive gene expression[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(49):18822-18827.
32.Liu L Q,Zhu K,Yang Y F,et al.Molecular cloning,expression profiling and trans-activation property studies of aDREB2-like gene from chrysanthemum(Dendranthemavestitum)[J].Journal of Plant Research,2008,121(2):215-226.
33.Yang Y F,Wu J,Zhu K,et al.Identification and characterization of two chrysanthemum(Dendronthema×moriforlium)DREBgenes,belonging to the AP2/EREBP family[J].Molecular Biology Reports,2009,36(1):71-81.
34.Yang Y J,Ma C,Xu Y J,et al.A zinc finger protein regulates flowering time and abiotic stress tolerance in chrysanthemum by modulating gibberellin biosynthesis[J].The Plant Cell,2014,26(5):2038-2054.
35.Liu Q L,Xu K D,Zhao L J,et al.Overexpression of a novel chrysanthemum NAC transcription factor gene enhances salt tolerance in tobacco[J].Biotechnology Letters,2011,33(10):2073-2082.
36.Liu Q L,Zhong M,Li S,et al.Overexpression of a chrysanthemum transcription factor gene,DgWRKY3,in tobacco enhances tolerance to salt stress[J].Plant Physiology and Biochemistry,2013,69:27-33.
37.He M,Wang H,Liu Y Z,et al.Cloning and characterization ofChiMYBinChrysanthemumindicumwith an emphasis on salinity stress tolerance[J].Geneticsand Molecular Research,2016,15(3):gmr.15038985.
38.杨艳芳,武剑,朱凯,等.过量表达菊花DmDREBa基因提高转化烟草耐低温能力[J].植物研究,2016,36(5):721-729.
Yang Y F,Wu J,Zhu K,et al.Overexpression ofDmDREBagene significantly enhances low temperature tolerance in transgenic tobacco[J].Bulletin of Botanical Research,2016,36(5):721-729.
39.武剑,杨艳芳,王慧,等.菊花DmDREBa/b基因提高转基因烟草耐逆性研究[J].分子植物育种,2016,14(11):3063-3072.
Wu J,Yang Y F,Wang H,et al.Study ofDmDREBa/bgene enhanced stress tolerance in transgenic tobacco[J].Molecular Plant Breeding,2016,14(11):3063-3072.
Partly supported by National Natural Science Foundation of China(31570675);National non-profit Research Institutions of Chinese Academy of Forestry(CAFYBB2014QB001)
introduction:ZHU Kai(1982—),male,M.Agr.,major in plant molecular biology.
date:2017-01-03
OverexpressionofDlNAC1GeneIsolatedfromDendranthemalavandulifoliuminEnhancingHeatStressToleranceinTransgenicTobacco
ZHU Kai1WU Jian1YANG Yan-Fang2*CHEN Fa-Di3YU De-Yue1
(1.National Center for Soybean Improvement,National Key Laboratory of Crop Genetics and Germplasm Enhancement,Nanjing Agricultural University,Nanjing 210095;2.Research Institute of Forestry,Chinese Academy of Forestry,State Key Laboratory of Tree Genetics and Breeding,Key Laboratory of Tree Breeding and Cultivation of State Forestry Administration,Beijing 100091;3.College of Horticulture,Nanjing Agricultural University,Nanjing 210095)
We studied the amino sequenceofDlNAC1 gene(GenBank accession number EF602304) cloned fromDendranthemalavandulifoliumby bioinformatics methods, and theDlNAC1 gene was transformed tobacco plants byAgrobacteriumtumefaciensmediation method. Thepredicted DlNAC1 proteinhad hydrophilicity, and the second structure of this protein was mainly random coil. The NAC protein had six motif models and one NAC domain. The 36 of 55 transgenic tobacco plants were positive by PCR analysis. The heat stress tolerance of PCR positive tobacco plants was increased comparing with the wild plants. With 45℃ treatment for 6 h, the transgenic plants looked much stronger, whereas the wild type looked very weak with wilting leaves. The water content of transgenic plants was more than wild plants. However, the low temperature tolerance of the transgenic plants was not improvedafter 4℃ stress. All these results indicated thatDlNAC1 gene improved the tolerance ability to the high temperature stress of transgenic tobacco plants.
chrysanthemum;NAC transcription factor;bioinformatics;heat stress;transgenic tobacco
国家自然科学基金项目(31570675);中央级公益性科研院所基本科研业务费专项(CAFYBB2014QB001)
朱凯(1982—),男,硕士,主要从事植物分子生物学研究。
* 通信作者:E-mail:echoyyf@caf.ac.cn
2017-01-03
* Corresponding author:E-mail:echoyyf@caf.ac.cn
Q943.2
A
10.7525/j.issn.1673-5102.2017.03.014

