甘蓝型油菜SHR蛋白基因鉴定与结构分析
刘慧萍,陈永勤,黄毅,顿小玲
(1. 湖北大学生命科学学院,湖北 武汉 430062;2. 中国农业科学院油料作物研究所,农业部油料作物生物学与遗传育种重点实验室,湖北 武汉 430062)
甘蓝型油菜SHR蛋白基因鉴定与结构分析
刘慧萍1,2,陈永勤1,黄毅2,顿小玲2
(1. 湖北大学生命科学学院,湖北 武汉 430062;2. 中国农业科学院油料作物研究所,
农业部油料作物生物学与遗传育种重点实验室,湖北 武汉 430062)
植物SHR蛋白属于GRAS蛋白转录因子家族,保守羧基端含有VHIID结构域,氨基端不保守,参与调控植物物质合成和生长发育.参照拟南芥AtSHR基因的CDS序列,采用生物信息学的方法在甘蓝型油菜数据库中鉴定出3个BnSHR基因,进而分析其基因结构、生化特性、进化关系和蛋白结构.研究结果显示3个BnSHR基因中有2个位于C基因组(来源于甘蓝)上,1个位于A基因组(来源于白菜);3个BnSHR基因的氨基酸序列一致性为88.25%,与AtSHR基因在进化上也高度同源,SHR基因在结构和功能上保守;亚细胞定位预测显示BnSHR蛋白可能定位于过氧化物酶体,参与种子萌发过程中脂肪转变为糖分的过程;PHYRE2预测了BnSHR蛋白的三级结构,由于序列的差异性导致了氨基末端处结构相差较大,但VHIID保守区结构类似,为β-折叠.
甘蓝型油菜;SHR基因;基因结构;氨基酸序列;三级结构
0 引言
GRAS蛋白家族是高等植物特有的转录因子,具有高度保守的羧基末端,参与植物的生长发育、信号转导、解毒作用、生物胁迫和非生物胁迫相关的应答过程[1].2004年,Bolle等通过系统进化树的分析,鉴定出至少33个蛋白家族成员,可被分为7个分支,分别为:DELLA、SCR、Ls、HAM、PAT1、SHR、SCL9[2].其保守的羧基末端一般由几个典型的结构组成: LXXLL、7个亮氨酸重复区I(LHRI)、NLS、VHIID、7个亮氨酸重复区II(LHRII)、PFYRE和SAW[3].其中,SHORT-ROOT(SHR)蛋白因子与家族其他GRAS蛋白序列相似性相对较小,其保守羧基端仅含有VHIID 基序和PFYRE 基序[3,4],导致其在功能上与其他成员也有所不同,成为了进化树上的单独分支[5].
1993年Benfey PN等通过T-DNA插入突变经筛选后得到了拟南芥shr突变体,首次发现SHR基因与拟南芥根的伸长生长和辐射形态形成相关[6].后来拟南芥的SHORT-ROOT基因(AtSHR)在2000年被成功克隆,发现其编码区全长1 593 bp,在根和茎中都能检测到其转录产物,并且证实了SHR在拟南芥根基本组织(内皮和皮层)形成的不对称平周分裂和使内皮层细胞规则排列中的必要性[4].2011年,Lucas等研究shr突变体时发现,由于主根基本组织发生变化,中柱鞘细胞减少,导致从中发生的侧根数减少,且侧根形态也发生巨大缺陷[7].
此外,进一步研究表明SHR蛋白作为转录因子在中柱细胞内被表达,后移动到内皮细胞核中发挥作用,例如在SCR上游起作用,激活和维持SCR的表达,正调控根辐射形态的形成等生理活动[8-10];且SHR蛋白移动范围也决定了皮层细胞层的数量[11].关于SHR蛋白的表达特性,有GFP荧光标记实验表明SHR最早在球形胚晚期的前形成层中表达,SCR也在球形胚时期的基本组织细胞中开始表达,表明SHR与SCR的相互作用很可能在胚胎发育早期建立,以非细胞自主方式发挥作用[4,12].另外,Levesque等(2006)在对一系列彼此独立的基因芯片结果(包括在shr突变体背景下直接诱导表达SHR,SHR功能缺失和异位表达SHR)进行生物信息学研究后,发现其下游可能有495个基因受其调控,其中106个基因上调表达,另外389个基因下调表达,其中又有8个基因的调控情况具有高度置信性,极可能是直接被SHR诱导表达的;另一方面,按照功能可将受SHR间接调控的基因划分为转录因子(46个)、蛋白质磷酸化酶(40个)、物质代谢类(111个)和激酶(37个)[13].
SHR基因是一个功能进化比较保守的基因,作为植物组成型表达产物,在除拟南芥的其他植物中也发挥着重要作用.目前已在水稻(Oryzasativa)、白杨(Populustremula)、蒺藜苜蓿(Medicagotruncatula)和辐射松(Pinusradiata)等植物中都发现了SHR基因[14-17].其中水稻SHR基因(OsSHR)在根部的作用与拟南芥(AtSHR)类似[7],但OsSHR基因家族还有特定的成员参与水稻叶片气孔的形成[14].白杨SHR基因(PttSHR)在芽的形成层表达,蒺藜苜蓿SHR基因(MtSHR)是在组织培养诱导的不定根中发现的,而辐射松SHR基因(PrSHR)参与了根分生组织的形成和维持,同时在下胚轴形成层区也有表达.而甘蓝型油菜的BnSHR基因特征与表达模式尚少见报道.
本研究利用拟南芥AtSHR序列在油菜全基因组数据库中进行比对,找到甘蓝、白菜及甘蓝型油菜的SHR基因与蛋白序列,采用生物信息学方法加以鉴定,并对其氨基酸组成、结构和进化关系展开分析,为研究甘蓝型油菜SHR蛋白功能提供研究方向和参考依据.
1 材料与方法
1.1材料采用的拟南芥(Arabidopsis)SHR基因序列和氨基酸序列从TAIR10(http://www.arabidopsis.org/)下载,白菜(Brassicarapa)、甘蓝(Brassicaoleracea)和甘蓝型油菜(Brassicanapus)的SHR基因CDS序列和氨基酸序列从Brassica Datebase (http://brassicadb.org/)下载.
1.2甘蓝型油菜SHR蛋白基因鉴别以拟南芥SHR基因的CDS序列在Brassica Database本地数据库中进行BLASTn检索,并在NCBI(https://www.ncbi.nlm.nih.gov/)和油菜全基因组注释信息网站(http://www.genoscope.cns.fr/blat-server/cgi-bin/colza/webBlat)上验证,筛选到白菜、甘蓝和甘蓝型油菜的全部SHR基因,经SMART(http://smart.embl-heidelberg.de/)[18]分析,确定其GRAS结构域所在位点.
采用ProtParam(http://web.expasy.org/protparam/)[19]预测蛋白质理论分子量(MW)、等电点(pI)、不稳定系数(Instability index)、脂肪系数(Aliphatic index)和GRAVY.利用PSORT预测甘蓝型油菜SHR蛋白的亚细胞定位位置.
1.3甘蓝型油菜SHR蛋白基因系统发生与motif分析为了详细地了解各SHR蛋白之间的进化关系,利用Clustal W(MEGA6)[20]对甘蓝型油菜、甘蓝、白菜和拟南芥的SHR基因的CDS序列进行了比对,并以拟南芥的SHR基因(AtSHR)做为外类群,利用MEGA6中的邻接法(Neighbor-Joining,Psssion模型)构建了系统进化树,其中bootstrap值设为1 000次重复.
将甘蓝型油菜、白菜和甘蓝SHR蛋白的氨基酸序列保存为一个FASTA格式文档,用MEME4.10.2(http://meme-suite.org/tools/meme)[21]在线软件对其蛋白序列进行motif分析,参数设置:最优宽度6-50个氨基酸,一次或0次出现,最多预测10个motif.
1.4甘蓝型油菜SHR蛋白氨基酸序列分析和三级结构预测利用DNAMAN软件(DNAMAN6.0.3.99简体中文版)预测甘蓝型油菜3个SHR蛋白和拟南芥SHR蛋白的二级结构并进行氨基酸序列分析,比较它们的同源性和GRAS蛋白家族的特征位点.DNAMAN软件同时预测了这4个蛋白的疏水性,生成图谱.为了更加详细地了解甘蓝型油菜SHR蛋白的立体构象,采用PHYRE2软件进行蛋白三级结构的在线预测(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)[22],同时用蛋白质画图工具PYMOL(http://www.pymol.org/)优化蛋白结构图,找出重要结构域.
2 结果与分析
2.1甘蓝型油菜SHR基因鉴定在TAIR网站上搜索到拟南芥SHR基因AT4G37650,按习惯称为AtSHR,下载其基因序列和蛋白质序列,经比对发现拟南芥上没有其他同源SHR基因.通过在NCBI、油菜全基因组注释信息网站和Brassica Datebase上的BLASTn比对,获得了同样的甘蓝型油菜SHR蛋白基因3个,白菜SHR蛋白基因3个,以及甘蓝SHR蛋白基因3个,如表1.通过本地数据库下载上述9个基因的CDS序列和蛋白质序列,并将甘蓝型油菜的3个SHR基因分别记为BnSHR1、BnSHR2、BnSHR3,下同.
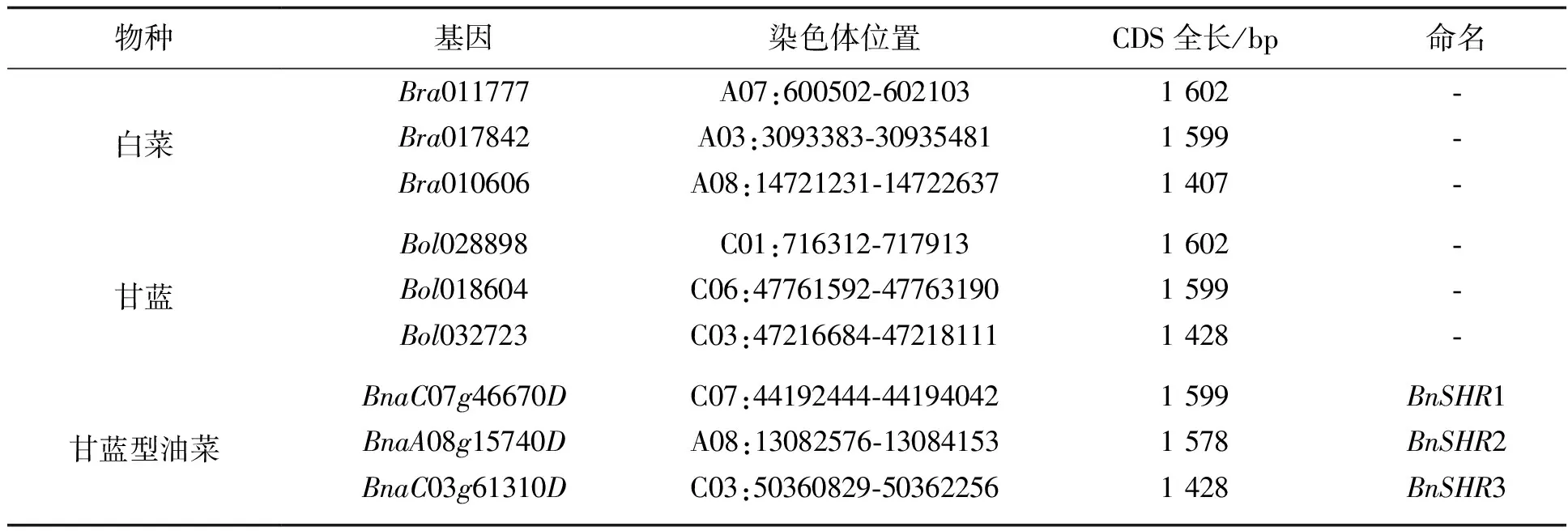
表1 AtSHR在白菜、甘蓝和甘蓝型油菜中的同源基因的相关信息
2.2甘蓝型油菜SHR蛋白基因系统发生及保守性目前SHR基因甚至GRAS蛋白家族都是在拟南芥和水稻中研究的最多,GRAS蛋白家族的系统发生关系已有研究[2,17].但SHR蛋白作为其中一个亚族,其同源基因少且多数物种的SHR基因序列并未准确测定和正式公布.所以本研究主要针对性地讨论BnSHR与其来源物种和模式生物拟南芥中SHR的进化关系.根据上述获得的基因的CDS序列分析它们的进化关系,以AtSHR为外类群,结果如图1.从进化树上可以看出,各基因间同源性较高,且明显分为四类:A、B、C、D.其中A类包括BnSHR1、Bol018604、Bra017842三个基因,BnSHR1位于C07染色体,而Bol018604位于C06染色体.B类中的两个基因分别为甘蓝的Bol028898和白菜的Bra011777,它们在甘蓝型油菜中没有同源拷贝.C类的BnSHR2和Bra010606都定位于A08染色体上,初步认定甘蓝型油菜中的BnSHR2应该来源于白菜的A08染色体.D类中的基因是BnSHR3和Bol032723,位于染色体C03,来源于甘蓝.由此可以看出甘蓝型油菜的SHR基因来源于其亲本组合白菜和甘蓝,且2个来自甘蓝,1个来自白菜,序列比对分系析表明其同源性都达到了100%,在AtSHR为外类群的条件下,各个BnSHR与AtSHR在进化上依然非常相近,这表明了SHR蛋白基因在进化上的保守性和严谨性,其中VHIID为SHR蛋白含有的GRAS结构域中的一类保守基序.这些结果暗示了BnSHR在基因功能上可能与AtSHR相似.
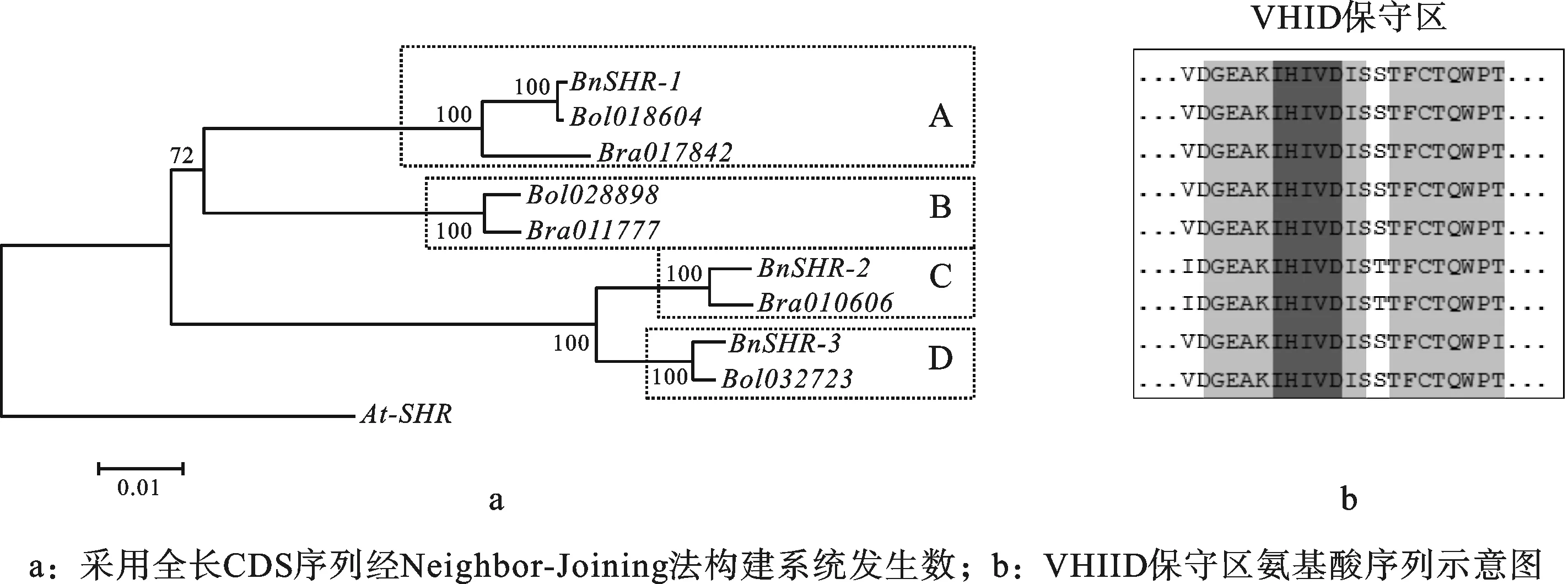
图1 甘蓝型油菜、白菜、甘蓝和拟南芥SHR蛋白基因的系统进化树分析
利用MEME4.10.2软件分析3个甘蓝型油菜SHR蛋白和其亲本蛋白全长氨基酸序列发现10个保守基序(motif),一次命名为motif1-10(图2).motif3为VHIID保守基序,存在于每一个SHR蛋白质中;motif7、motif8和motif10处于高度变异的氨基末端,其中motif10只存在于BnSHR1、BnSHR2和Bol018604中,但总体一致性较高;余下的6个motif都存在于保守的羧基末端.比较BnSHR2和Bra010606发现,BnSHR2上多了一个Bra010606没有的motif10,这可能是由于在甘蓝型油菜中A、C基因组发生了同源性重组的原因[23].

图2 甘蓝型油菜SHR蛋白的保守motif 组成分析
2.3BnSHR1、BnSHR2、BnSHR3的理化性质分析蛋白质的功能通常与它的理化性质密不可分.从表2中可以看出甘蓝型油菜SHR蛋白所含氨基酸残基数在500左右,其中N端380个左右的氨基酸残基组成了GRAS结构域,属于GRAS蛋白家族;BnSHR蛋白质保守结构域GRAS中组成基序与其功能密切相关.3个蛋白的不稳定系数均大于40,为不稳定蛋白质.在生化特性上BnSHR1、BnSHR2和BnSHR3等电点均小于7,带负电荷且都为亲水性蛋白质,这可能是其作为转录因子与DNA结合的理化基础.此外,蛋白质的脂肪系数代表其所含脂肪族氨基酸的含量,BnSHR1(脂肪系数:66.58)、BnSHR2(脂肪系数:68.23)和BnSHR3(脂肪系数:69.07)在氨基酸组成成分上的相似性极高.利用PSORT对BnSHR的亚细胞定位预测发现,该蛋白很可能位于过氧化物酶体和线粒体.其氨基酸序列比较及蛋白功能的研究将会在下面通过生物信息学进行进一步的探索.
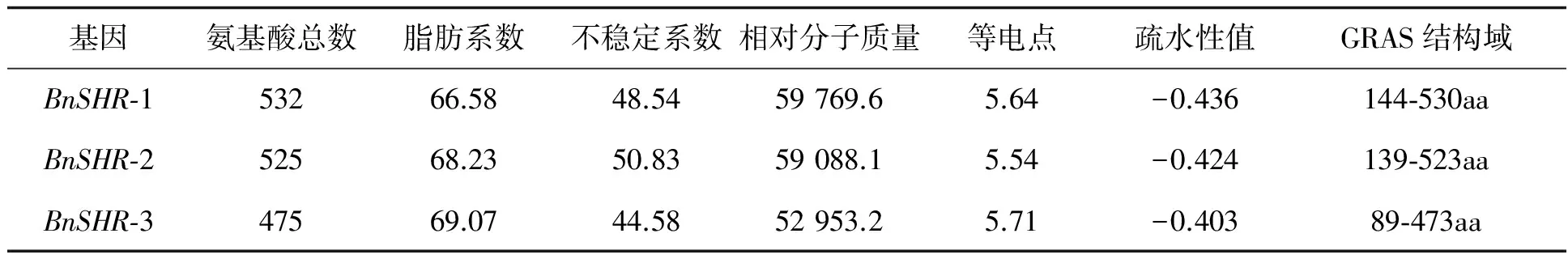
表2 甘蓝型油菜中3种SHR蛋白理化性质
2.4甘蓝型油菜SHR蛋白序列和构型分析比对分析甘蓝型油菜和拟南芥的SHR蛋白质的氨基酸序列发现,他们的相似性达到了90.12%.pfam分析也表明3个甘蓝型油菜的SHR蛋白具有典型的GRAS结构域,属于GRAS蛋白家族.在C-末端有和拟南芥一样高度保守的基序和氨基酸,比如SAW基序以及附近的WX7G和WX10W,还有与SHR蛋白核定位和在细胞间的移动密切相关的苏氨酸磷酸化位点[24].在不保守的N-末端,发现BnSHR含有谷氨酰胺聚合物、苏氨酸聚合物、丝氨酸聚合物和丙氨酸聚合物(图3),同样的聚合物也在AtSHR和OsSHR中出现[4,14].在经过多序列比对后,预测甘蓝型油菜和拟南芥的SHR蛋白的疏水性,如图4.根据每个位点的氨基酸的疏水性绘制图谱,从图中可以看出4个蛋白质的疏水性图谱几乎重合,证明了BnSHR与AtSHR在生理特性上的高度相似性.

图3 甘蓝型油菜和拟南芥的SHR蛋白质氨基酸序列分析

图4 甘蓝型油菜和拟南芥SHR蛋白氨基酸序列的疏水性

表3 SHR蛋白质二级结构预测
DNAMAN对甘蓝型油菜SHR蛋白和AtSHR蛋白的二级结构预测结果显示如表3.BnSHR与AtSHR在蛋白质的二级结构组成上非常相似,大部分由无规则卷曲和α-螺旋组成,也有显著的β-折叠结构.为了进一步分析SHR蛋白质的三级结构,本研究采用PHYRE2软件进行蛋白三级结构的在线预测,可信度最高的为同一个模板c4kifB(来源于PDB),以此模板进行同源建模结果如图5.从图中可以看出拟南芥和甘蓝型油菜的SHR蛋白的空间构象十分相似,除了由于少量的一级结构的不同而引起的差异,其余各二级结构的走向和空间位置都相当一致.图中箭头所指的第三个β-折叠在图5中有单独显示,是SHR蛋白质具有的GRAS蛋白家族的典型结构VHIID,左图中标有氨基酸名称,在图5中可以看出VHIID保守基序所在的空间位置和其前后结构都几乎一致,并且β-折叠的走向全部一致,为平行折叠片,活性更高.以上结果从蛋白质的一级结构,空间结构和理化性质说明了BnSHR与AtSHR的在各方面的高度相似性,可以推测BnSHR可能与AtSHR一样地在根部表达并发挥功能.
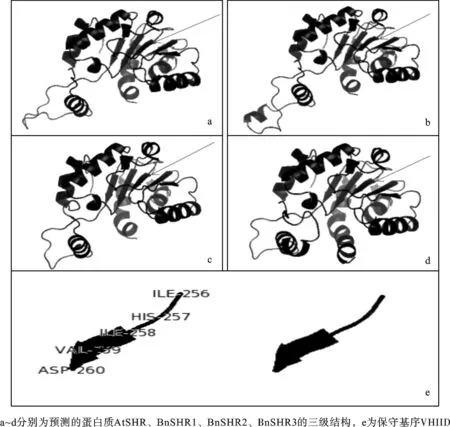
图5 同源建模预测的SHR蛋白质空间结
3 讨论
本研究采用生物信息学分析方法,参考拟南芥SHR蛋白基因AtSHR的CDS序列和GRAS蛋白结构域序列,从甘蓝型油菜数据库中鉴定了3个BnSHR基因,比较分析了基因结构、生化特性、进化关系和蛋白结构,发掘出甘蓝型油菜SHR蛋白基因包含的重要信息.
甘蓝型油菜是来源于白菜和甘蓝自然杂交和加倍的异源四倍体,含有两套遗传信息,分别位于A基因组和C基因组.通过比对鉴别出的甘蓝型油菜BnSHR基因有两个位于C基因组,一个位于A基因组.而在白菜和甘蓝的比对结果中发现这两个基因组上分别有3个SHR蛋白基因,我们推测一种可能是由于甘蓝型油菜形成过程中的种间杂交和染色体加倍打破了物种间的界限,导致基因组间遗传物质的重新调整与分配,从而在甘蓝型油菜中只保留了3个SHR蛋白基因;另一种可能是甘蓝型油菜的进化过程中染色体变异导致部分SHR蛋白基因改变或消失.通过分析甘蓝型油菜和拟南芥、白菜以及甘蓝的SHR蛋白基因的进化关系后发现,3个BnSHR基因虽然位于不同的分支,但它们的相似性极高,且都与拟南芥AtSHR基因高度同源,这说明了SHR基因在进化上的保守,BnSHR属于SHR蛋白亚家族.比较分析甘蓝型油菜和拟南芥的SHR蛋白的一级结构、二级结构和构型构象,发现均有较高的相似性.蛋白质的一级结构决定它的高级结构,在4个蛋白质的氨基酸序列的一致性达到90.12%的情况下,他们的高级结构也应有所相似,而通过各种软件预测的二级结构和三级结构也恰好证明了这一点.蛋白质的结构决定蛋白质的功能,所以BnSHR也可能和OsSHR、MtSHR、PrSHR[16,18,19]一样,与AtSHR[8-10]相似的在根部表达且有重要功能,即甘蓝型油菜的SHR基因的功能可能也与根的生长和形态建成相关,但有可能在作用机制上有所不同[25].另外,3个BnSHR基因表达的氨基酸序列一致性为88.25%,不是互为拷贝,这也说明这3个蛋白质的时空表达和功能上可能有所不同.亚细胞定位预测显示BnSHR蛋白可能定位于过氧化物酶体,植物中过氧化物酶体除了参与氧化乙醇酸帮助植物避免氧化毒害外,在油料植物中有一种特有的过氧化物酶体(乙醛酸循环体),参与种子萌发过程中脂肪转变为糖分的过程[26-27],BnSHR蛋白可能也参与了这个过程[28].
迄今为止,拟南芥SHR蛋白基因的研究较为丰富.AtSHR在拟南芥上的表达相当多样化,TAIR网站上记录的表达位点有心皮、茎叶、集体叶结构、花、花梗、保卫细胞、下胚轴、花序分生组织、叶尖、花瓣、叶柄、植物胚胎、根、根中柱、种子、花萼、茎尖、茎轴系统、茎杆、脉管叶和脉管系统.除了上述与根相关的一些功能外,有研究发现SHR蛋白同样也可以影响叶的发育,SHR和SCR基因的不表达直接影响叶片细胞的分裂活性而抑制了叶片的生长,而不是SHR在根部作用后的二级作用结果[29];同时SHR作为植物维管束鞘细胞光合作用的分子基础之一,具有高保守性,影响了维管束鞘细胞的数量和生理机能,是改造C3植物光合效率的有效切入点之一[30].油菜也为C3植物,提高光合作用效率有利于植物生长,增加产量,且由于SHR蛋白的保守性,BnSHR可以是改造油菜光合作用方式的分子基础.
本研究通过生物信息学方法分析了3个甘蓝型油菜SHR蛋白的结构和基因信息,根据它们序列和结构的相似性以及在进化上的保守性,初步预测了BnSHR的相关功能和可研究性.并已经根据比对得到的BnSHR 基因序列设计了不同的引物,接下来会通过定量PCR进行组织特异性表达的分析,进一步辨别这3个基因的功能,结合本实验室油菜根系转录组研究结果分析BnSHR基因对油菜根系发育和功能的影响.目前对于蓝型油菜SHR蛋白的研究远没有水稻等作物透彻,本文中为甘蓝型油菜中这一蛋白的研究提供了相关的生物信息学基础,同时也为甘蓝型油菜遗传改良提供了可用基因资源和分子调控理论指导.
[1] 李桂英,填玉富,杨成君.植物GRAS家族转录因子的现状研究[J]. 安徽农业科学, 2014,42(14): 4207-4210.
[2]Bolle C. The role of GRAS proteins in plant signal transduction and development[J]. Planta, 2004, 218(5): 683-692.
[3] Pysh L D, Wysocka-Diller J W, Camilleri C, et al. The GRAS gene family in Arabidopsis: sequence characterization and basic expression analysis of the SCARECROW-LIKE genes[J]. Plant J,1999, 18(1): 111-119.
[4] Helariutta Y, Fukaki H, Wysocka-Diller J. The SHORT-ROOT gene controls radial patterning of the Arabidopsis root through radial signaling[J]. Cell, 2000, 101(5): 555-567.
[5] Czikkel B E, Maxwell D P. NtGRAS1, a novel stress-induced member of the GRAS family in tobacco, localizes to the nucleus[J]. J Plant Physiol, 2007, 164(9): 1220-1230.
[6] Benfey P N, Linstead P J, Keith R. Root development in Arabidopsis: four mutants with dramatically altered root morphogenesis[J]. Development, 1993, 119(1): 57-70.
[7] Mika⊇l L, Ranjan S, Ivan A, et al. SHORT-ROOT regulates primary, lateral, and adventitious root development in Arabidopsis[J]. Plant Physiology, 2011, 155 (1): 384-398.
[8] Cui H, Levesque M P, Vernoux T. An evolutionarily conserved mechanism delimiting SHR movement defines a single layer of endodermis in plants[J]. Science, 2007, 316: 421-425.
[9] Koizumi K, Hayashi T, Gallagher K L. SCARECROW reinforces SHORT-ROOT signaling and inhibitspericlinal cell divisions in the ground tissue by maintaining SHR at high levels in the endodermis[J]. Plant Signaling and Behavior, 2012, 7: 1573-1577.
[10] Della Rovere F, Fattorini L, D'Angeli S. Arabidopsis SHR and SCR transcription factors and AUX1 auxin influx carrier control the switch between adventitious rooting and xylogenesis in planta and in in vitro cultured thin cell layers[J]. Annals of Botany, 2015, 115(4): 617-628.
[11] Wu S, Lee C M, Hayashi T. A plausible mechanism, based upon SHORT-ROOT movement, for regulating the number of cortex cell layers in roots[J]. PNAS, 2014, 111(45):16184-16189.
[12] Wysocka-Diller J W, Helariutta Y, Fukaki H, et al. Molecular analysis of SCARECROW function reveals a radial patterning mechanism common to root and shoot[J]. Development, 2000, 127: 595-603.
[13] Levesque M P, Vernoux T, Busch W. Whole-genome analysis of the SHORT-ROOT developmental pathway in Arabidopsis[J]. PLoS Biol, 2006, 4(5): 739-752.
[14] Kamiya N,Itoh J I, Morikami A. The SCARECROW gene’s role in asymmetric cell divisions in rice plants[J]. Plant J, 2003, 36: 45-54.
[15] Schrader J, Nilsson J, Mellerowicz E. A high-resolution transcript profile across the wood-forming meristem of poplar identifies potential regulators of cambial stem cell identity[J]. Plant Cell, 2004, 16: 2278-2292.
[16] Imin N, Nizamidin N, Wu T. Factors involved in root formation in Medicago truncatula[J]. J Exp Bot, 2007, 58: 439-451.
[17] Alicia Solé. Characterization and expression of a Pinus radiata putative ortholog to the Arabidopsis SHORT-ROOT gene[J]. Tree Physiology, 2008, 28: 1629-1639.
[18] Ivica L, Tobias D, Peer B. SMART: recent updates, new developments and status in 2015[J]. Nucleic Acids Research, 2014, 4(1): D257-D260.
[19] Walker J M. The proteomics protocols handbook[M]. Clifton, USA:Humana Press, 2005: 571-607.
[20] Tamura K,Stecher G, Peterson D. MEGE6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30: 2725-2729.
[21] Bailey T L, Bodén M, Buske F A, et al. MEME SUITE: tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37: W202-W208.
[22] Kelley L A, Mezulis S, Yates C M, et al. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nature Protocols, 2015, 10: 845-858.
[23] Chèvre A M.甘蓝型油菜染色体的同源和部分同源重组[A].第十二届国际油菜大会论文集[C].第十二届国际油菜大会筹备委员会, 2007, 5:85, 1137-1140.
[24] Gallagher K L, Paquette A J, Nakajima K. Mechanisms regulating SHORT-ROOT intercellular movement[J]. Curr Biol, 2004, 14(20): 1847-1851.
[25] Long Y,Smet W, Cruz-Ramírez A. Arabidopsis BIRD zinc finger proteins jointly stabilize tissue boundaries by confining the cell fate regulator SHORT-ROOT and contributing to fate specification[J]. The Plant Cell, 2015, 27(4): 1185-1199.
[26] Beevers H. Microbodies in higher plants[J]. Plant Physiol, 1979, 30: 159-193.
[27] Pracharoenwattana I, Smith SM. When is a peroxisome not a peroxisome?[J]. Trends Plant Sci, 2008, 13(10): 522-525.
[28] Markus S, Timothy S D, Stefan R H, et al. A gene expression map of Arabidopsis thaliana development[J]. Nature Genetics, 2005, 37(5): 501-506.
[29] Dhondt S, Coppens F, De W F, et al. SHORT-ROOT and SCARECROW regulate leaf growth in Arabidopsis by stimulating S-phase progression of the cell cycle[J]. Plant Physiol, 2010, 154(3): 1183-1195
[30] Cui H, Kong D, Liu X. SCARECROW, SCR-LIKE 23 and SHORT-ROOT control bundle sheath cell fate and function inArabidopsis thaliana[J]. Plant J, 2014, 8(2): 319-327.
IdentificationandstructuralanalysisofSHRgenesinBrassicanapus
LIU Huiping1,2,CHEN Yongqin1,HUANG Yi2,DUN Xiaoling2
(1. College of Life Science, Hubei University, Wuhan 430062, China;
2. Oil Crops Research Institute of the Chinese Academy of Agricultural Sciences,Key Laboratory of Biology and Genetic
Improvement of Oil Crops, Ministry of Agriculture, Wuhan 430062, China)
Plant SHR proteins belong to GRAS protein family of transcription factors, whose conservative C-terminus contains the VHIID domain, while the N-terminus are not conserved, play crucial role in biosynthesis and development regulation. To investigate the homologs inBrassicanapus, 3BnSHRgenes inBrassicanapuswere identified through amino acid sequence ofAtSHRagainst Brassica Database, then analyzed their gene structure, biochemical characteristics, evolutionary relationships and protein structure. The results show that 2BnSHRlocate in C genome fromBrassicaoleracea, 1BnSHRlocates in A genome fromBrassicarapa; the consistency of this amino acid squences is 88.25%, also have highly homology withAtSHRin evolution, may result in the conserved functions; subcellular localization prediction of BnSHR proteins are in peroxysome, may involve in transforming fat into sugar during the germination; the predicted tertiary structure of proteins display the architectural difference during N-terminus, otherwise, show the similar β-plated sheet with VHIID conserved region.
Brassicanapus;SHRgene; gene structure; amino acid sequence; tertiary structure
2017-02-22
国家自然科学基金(31501820)资助
刘慧萍(1993-),女,硕士生,E-mail:huiping.l@139.com;顿小玲,通信作者,助理研究员,E-mail:dunxiaoling@163.com
1000-2375(2017)06-0620-08
S565.4
A
10.3969/j.issn.1000-2375.2017.06.011
(责任编辑 游俊)

