苦荞C4H基因的cDNA克隆及生物信息学分析
刘荣华,王丽,孙朝霞,侯思宇,2*,李红英,2,3
(1.山西农业大学 农学院,山西 太谷 030801; 2.山西农业大学 农业生物工程研究所,山西 太谷 030801;3.特色杂粮种质资源发掘与育种山西省重点实验室,山西 太原 030031)
苦荞C4H基因的cDNA克隆及生物信息学分析
刘荣华1,王丽1,孙朝霞1,侯思宇1,2*,李红英1,2,3
(1.山西农业大学 农学院,山西 太谷 030801; 2.山西农业大学 农业生物工程研究所,山西 太谷 030801;3.特色杂粮种质资源发掘与育种山西省重点实验室,山西 太原 030031)
[目的]为了解植物苯丙烷类代谢途径中关键酶——肉桂酸-4-羟基化酶基因结构特征及系统进化,拟克隆苦荞肉桂酸-4-羟基化酶(Cinnamate-4-hydroxylase,C4H)基因并进行生物信息学分析。[方法]本研究通过RT-PCR技术,克隆2个苦荞C4H基因cDNA和DNA序列,并通过生物信息学分析其基因结构及蛋白理化性质,最大似然树法构建系统进化树。[结果]2个基因cDNA序列长度分别为1 812 bp和1 476 bp,各编码504和491个氨基酸残基。其DNA序列分别为2 774 bp和2 364 bp,均包含3个外显子和2个内含子,第1个外显子和2个内含子序列长度存在明显差异。蛋白分子量分别为58.002 kDa和56.401 kDa,等电点分别为9.18和9.09。2个基因编码氨基酸与苦荞肉桂酸-4-羟基化酶同源性分别为99%和86%,故暂且命名为FtC4H1和FtC4H2。FtC4H1和FtC4H2与其他物种直系同源蛋白聚为两类,FtC4H1 和FtC4H2与莴苣和丹参同源蛋白亲缘关系较近,氨基酸序列同源性分别为88%和84%。[结论]本研究通过对苦荞2个C4H基因的核酸、氨基酸序列、蛋白结构及系统进化树进行分析,为后续苯丙烷代谢途径及荞麦基因挖掘利用提供理论基础。
苦荞; 肉桂酸-4-羟基化酶; 电子克隆; 生物信息学分析
荞麦(FagopyrumMiller)系蓼科(Polygonaceae)荞麦属(FagypyrumGaerth),多为一年生双子叶小杂粮作物。荞麦在世界各地均有栽培,其栽培的主要地区有中国、俄罗斯、日本、加拿大、美国等,我国大多种植于东北、华北、西北和西南,及云、贵、川一带的高寒山区或丘陵地区[1]。荞麦富含多种人体必需氨基酸、微量元素钙和硒、次生代谢物黄酮类物质—芦丁,具有开胃宽肠、消热利湿、降低“三高”的功效,是集营养、保健和药用为一体的作物,已被营养学家称为21世纪最有前途的绿色食品,具有很高的研究和利用价值[2~4]。与其他大宗粮食作物相比,荞麦产量偏低,种植区域零散,作为小宗作物中的一种,在栽培管理、食品加工及分子生物学领域研究报道较少,因此,国际植物遗传资源研究所(International Plant Genetic Resources Institute, IPGRI)将荞麦归为“未被充分利用的作物”之一[5]。另外,我国对荞麦方面的研究相对落后,与日本、韩国和俄罗斯等国还存在着较大的差距,尤其在荞麦种质资源遗传多样性、分子育种及基因资源挖掘等研究方面。
肉桂酸-4-羟基化酶(C4H),又称反式肉桂酸-4-单氧化酶,催化肉桂酸羟化作用产生4-香豆酸盐,是苯丙烷途径中继L-苯丙氨酸解氨酶(PAL)之后的第2个关键酶[6,7],该酶在植物细胞中的含量可以影响木质素和黄酮类物质合成等多条代谢支路[8]。当前,已有许多物种克隆到该基因的序列已经初步阐明其功能。王安娜等[9]通过RT-PCR技术从大豆中克隆到一个长度为1 766 bp,编码506个氨基酸残基的C4H基因,从分子水平上揭示了大豆的结构特点,为提高其表达活性提供了理论依据。赵乐等[10]以独行菜为材料,通过分析转录组数据,设计特异性引物,克隆了独行菜C4H基因(LaC4H)的cDNA序列,并进行生物信息学分析和组织特异性分析,为进一步研究LaC4H基因在独行菜黄酮类化合物生物合成途径中的功能奠定基础。尽管前人已有苦荞C4H基因克隆及生物信息学分析的报道[8],但不同苦荞品种间该基因序列结构变异及基因家族成员等相关信息尚不清楚,因此需进一步深入研究该基因序列信息及结构功能,为荞麦芦丁生物合成途径相关分子机制及分子育种研究奠定理论基础。
本研究拟克隆一个肉桂酸-4-羟基化酶基因,对其核酸和氨基酸序列,蛋白质结构及系统进化树进行相关生物信息学分析,旨在为荞麦基因资源挖掘利用及分子调控提供一定理论基础。
1 材料和方法
1.1 材料和试剂
本试验所用材料为“黑丰1号”,由山西省农业科学院提供,种植于山西农业大学农作站,待种子萌发到长出2~3片真叶时取材。选取0.1 g叶片组织放入1.5 mL的离心管中,液氮速冻后-80 ℃保存备用。RNA 提取试剂、植物 RNAOut 试剂盒购自天恩泽基因工程有限公司;逆转录试剂盒购自TaKaRa公司;其它化学试剂均为国产或进口分析纯。
1.2 RNA提取及基因克隆
取出保存在-80 ℃冰箱中的材料。参照北京艾德莱生物科技有限公司PLANTpure通用植物总RNA快速提取试剂盒提供的操作说明进行提取(略有改动)。最后将提取好的RNA放入-80 ℃冰箱中保存备用。cDNA第一链合成总反应体系为20 μL,其中Total RNA 5 μL,Anchored Oligo(dT) 1 μL,2×TS Reaction Mix 10 μL,TransScriptTMRT/RI Enzyme Mix 1 μL,RNase free water 3 μL。反转录合成cDNA第一链的PCR程序设置为:42 ℃,30 min(反转录反应);85 ℃,5 min(反转录酶的失活反应)。根据苦荞叶片转录组注释数据(未发表),找到2个包含完整CDS序列的C4H基因,基于RT-PCR法,从苦荞叶片cDNA文库和基因组中,克隆C4H基因的cDNA和DNA序列,设计FtC4H1基因的上游引物序列是CAACTCAACAAACTTACACC,下游引物序列是ATGGGAAAGCCTTCATTCAT;FtC4H2基因的上游引物序列为ATGGATATAGTTCTCCTTC,下游引物序列为TCAGTTCCTGGGCTTCA;PCR反应体系为总体积25 μL,1 μL cDNA模板,上下游引物各1 μL,高保真Taq酶1 μL(2.5 U),10×PCR 反应缓冲液2.5 μL,最后加ddH2O补足。PCR程序设置为:94 ℃ 1 min,60 ℃ 45 s,72 ℃ 30 s,25个循环。最后试验所获得的基因序列提交至NCBI中GenBank数据库中,得到登录号(C4H1,GenBank accession:KY797634;C4H2,GenBank accession:KY860362)。启动子序列通过本地BLAST苦荞简化基因组测序数据[11],搜索基因转录起始位点上游1 500 bp的核酸序列,基于PLARE软件在线预测启动子所包含的顺式作用元件。
1.3 生物信息学分析
采用DNAstar 7.0和Vector NTI Advance 11软件分析核酸序列及绘制基因结构图谱。利用ExPasy在线蛋白质专家数据库(http://www.expasy.org/proteomics),预测编码蛋白一级结构序列特征、蛋白亲水/疏水性、蛋白二级结构、信号肽、跨膜区及亚细胞定位,SWISS-MODEL 预测蛋白三级结构。根据FtC4H1和FtC4H2编码的氨基酸序列,从NCBI网站分别下载拟南芥(Arabidopsisthaliana,登录号:NM_128601.3)、大豆(Glycinemax,登录号:FJ968526.2)、马铃薯(Solanumtuberosum,登录号:DQ341174.1)、丹参(Salviamiltiorrhiza,登录号:DQ355979.1)、黄芩(Scutellariabaicalensis,登录号:HM062778.1)、莴苣(Lactucasative,登录号:KF981868.1)、白桑(Morusalba,登录号:KJ616396.1)和橄榄(Canariumalbum,登录号:FJ821504.1)8种植物的同源氨基酸序列,采用MEGA7.0软件中最大似然树法构建系统进化树,Bootstrap值设置为1 000次。
2 结果与分析
2.1 苦荞FtC4H基因序列特征分析
从苦荞叶片cDNA文库中克隆到2条C4H基因cDNA序列,核酸长度分别为1 812 bp和1 476 bp,各自编码504和491个氨基酸残基,暂且命名为FtC4H1和FtC4H2,分别进行基因序列特征分析(图1A和图1B),FtC4H1基因序列全长2 774 bp,其中3个外显子长度分别为785 bp,134 bp和596 bp,2个内含子分别为660 bp和302 bp;FtC4H2基因序列全长236 4bp,3个外显子分别为746 bp,134 bp和596 bp,2个内含子分别为469 bp和161 bp,第1个外显子和2个内含子长度存在明显差异。FtC4H1和FtC4H2基因序列常见酶切位点有HindⅢ、BamHⅠ、PstⅠ和EcoRⅠ。利用ExPASY ProtParam 在线预测FtC4H基因编码的氨基酸序列组成和理化性质(表1),根据其疏水性平均值(GRAVY)推测FtC4H1和FtC4H2蛋白均为亲水性蛋白。通过Signal P4.0 Server 预测信号肽,结果表明,FtC4H1和FtC4H2蛋白信号肽存在几率为0,推测该基因编码的蛋白可能为分泌型蛋白。FtC4H1和FtC4H2蛋白质二级结构的预测结果表明,α-螺旋比例最高分别为43.06%和42.97%,无规则卷曲分别为31.35%和29.74%,延伸链分别为16.67%和18.33%,β-转角分别为8.93%和8.96%。三级结构预测表明(图1C和图1D):二者均由7个结构域组成,均有25个α螺旋和34个无规则卷曲,但延伸链的数目分别为9个和8个。两者的结构组成相似,部分结构域空间构像不同。同源序列比对如图2所示,FtC4H2在第250至262位缺失13个氨基酸(LKLFKDYFVDERK),推测FtC4H2基因缺失的这一段氨基酸序列就是导致FtC4H1和FtC4H2蛋白三维空间结构不完全相同的原因。

表1 FtC4H1和FtC4H2氨基酸理化性质Table 1 The physical and chemical characteristics of FtC4H1 and FtC4H2 amino acid sequences
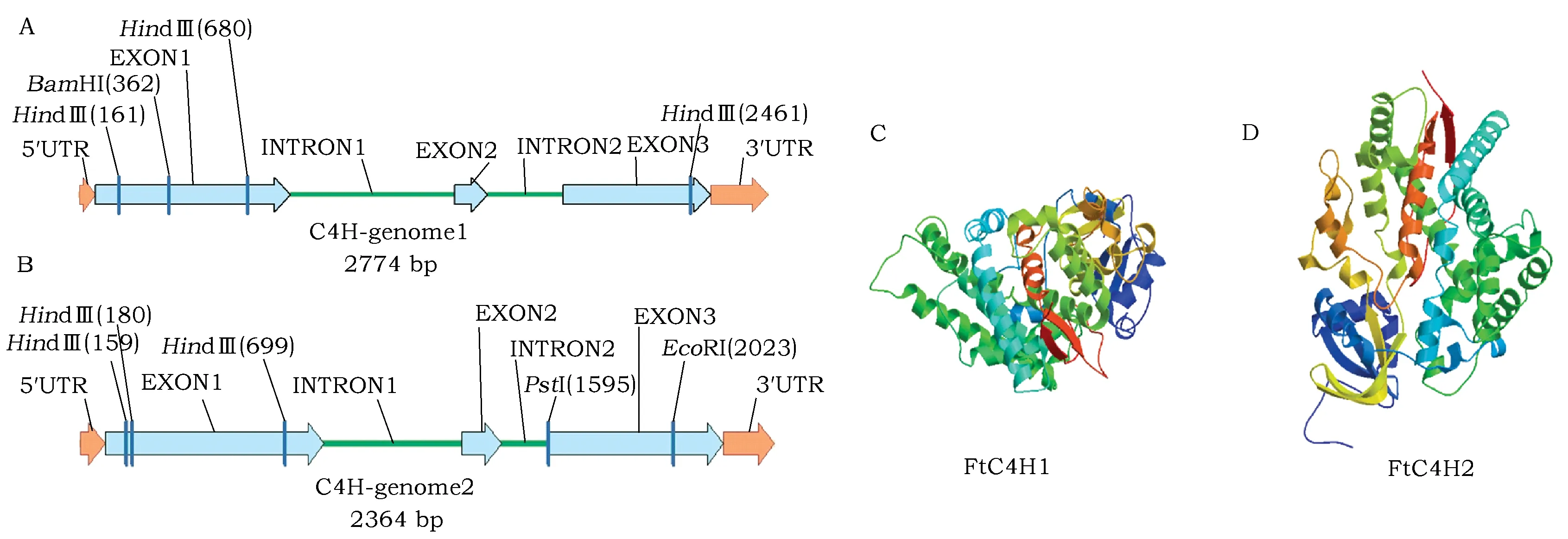
图1 FtC4H1和FtC4H2基因序列特征分析Fig.1 The gene sequence characterization analysis of FtC4H1 and FtC4H2 注:A和B分别为FtC4H1 和FtC4H2基因结构图;C和D分别为FtC4H1 和FtC4H2蛋白三级结构Note: A and B are gene structure of FtC4H1 and FtC4H2, respectively; C and D are the tertiary structure of FtC4H1 and FtC4H2 proteins, respectively

图2 FtC4H蛋白同源序列分析Fig.2 FtC4H protein homologous sequence analysis
2.2 启动子序列分析
用PlantCARE软件在线预测FtC4H1和FtC4H2基因转录起始位点上游序列所包含的启动子元件,结果见表2:FtC4H1启动子区域含有多种环境胁迫响应元件,其中包含有6个防御和胁迫应答元件TATA-box、1个厌氧感应顺式作用元件ARE、2个参与低温反应性的顺式作用元件LTR;2种植物激素应答元件,分别为赤霉素响应元件p-box和3个茉莉酸甲酯响应元件CGTCA-motif;还含有参与生理调控、胚乳表达所必须的顺式作用元件circadian、Skn-1_motif等细胞特异表达元件。FtC4H2启动子区域也含有多种环境胁迫响应元件,即12个防御和胁迫应答元件TATA-box、1个热激响应元件HSE等;2种激素应答元件,分别为赤霉素响应元件p-box和水杨酸响应元件TCA-element。
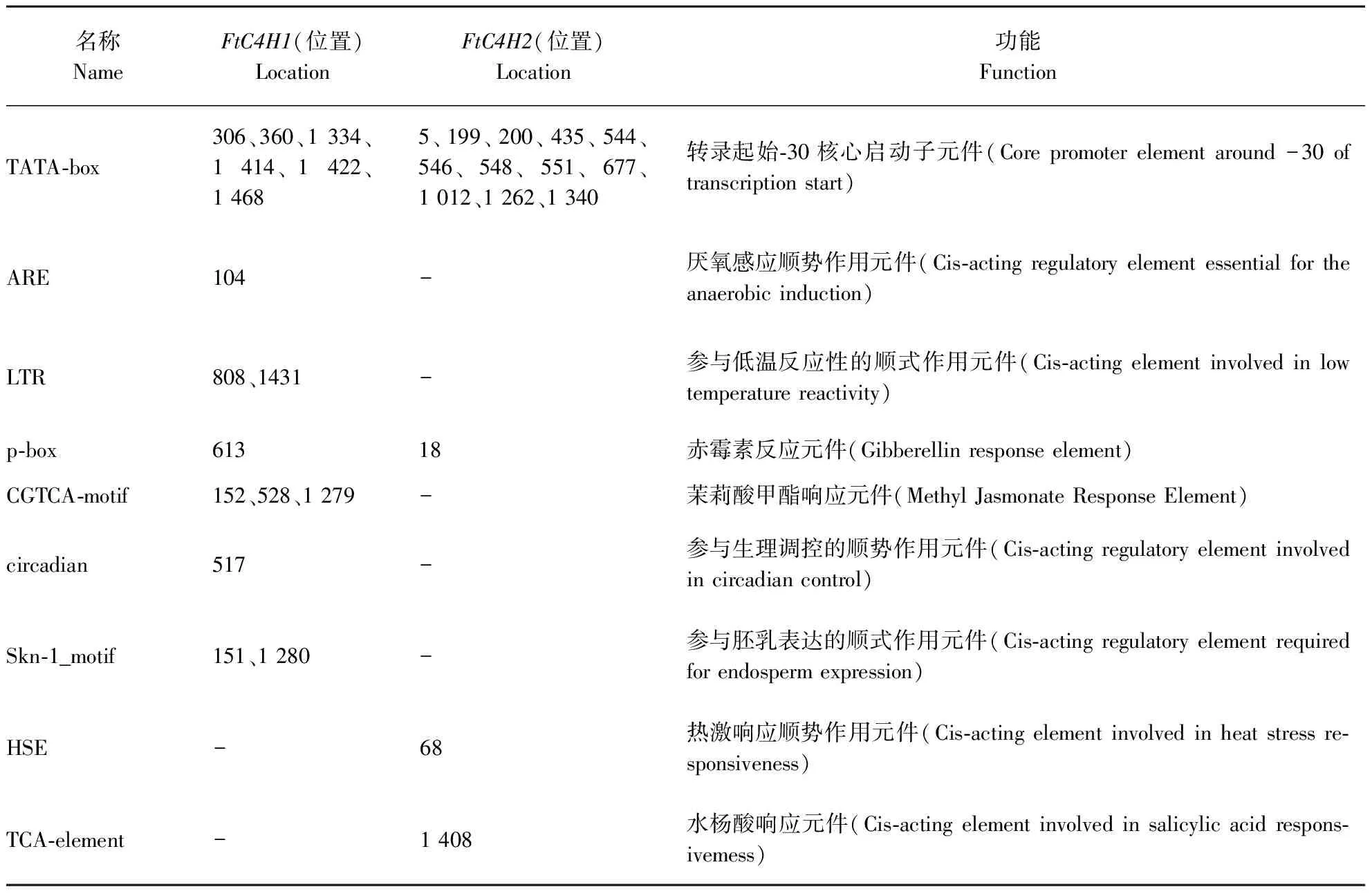
表2 FtC4H1和FtC4H2启动子元件Table 2 Promoter elements of FtC4H1 and FtC4H2
2.3 基因系统进化树分析
用MEGA7.0软件,采用最大似然树法构建系统进化树(图3)。结果表明:进化树聚为两类,其中本研究克隆的2个苦荞C4H基因与大豆、黄芩、丹参、莴苣、拟南芥和马铃薯聚为一个大类,但两者各为一个独立的分支;另外白桑和橄榄聚为一类。FtC4H1编码的氨基酸序列与上述8种植物的同源序列的相似性在85%~88%之间,其中与莴苣亲缘关系较近;FtC4H2编码的氨基酸序列与上述8种植物的同源序列相似性在82%~84%之间,其中与丹参的亲缘关系较近。
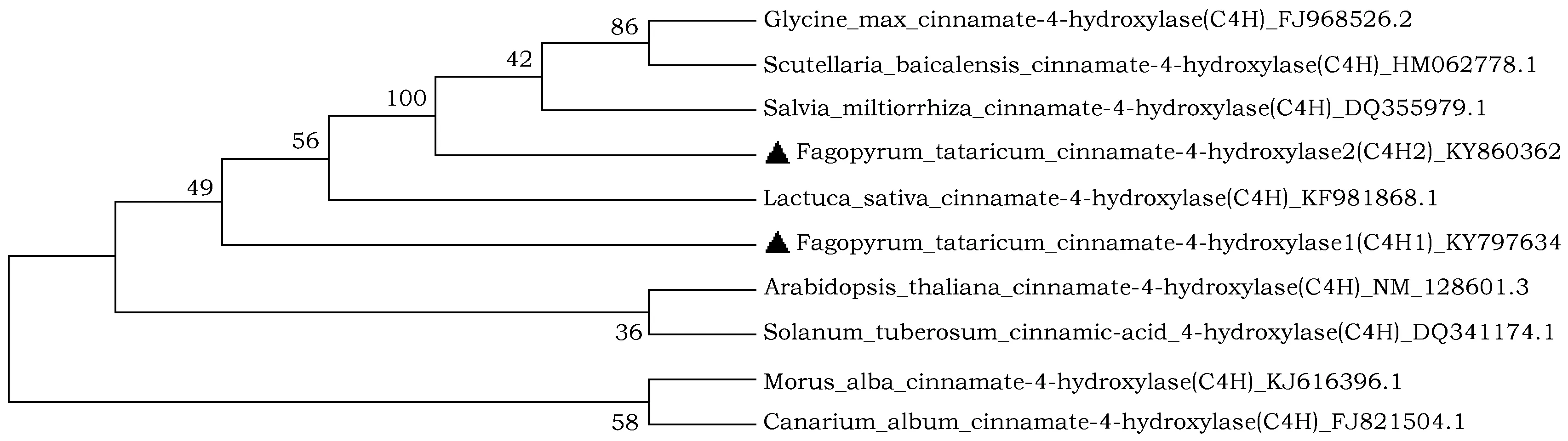
图3 不同物种基于FtC4H氨基酸序列的系统进化树Fig.3 Phylogenetic tree of different plants based on amino acid FtC4H
3 讨论
苯丙烷类代谢途径是植物重要的次生代谢途径之一,该途径以苯丙氨酸为底物,在一系列酶的催化作用下,合成黄酮、木质素、香豆素等多种次生代谢产物,参与植物的生长发育、形态建成及抵抗逆境胁迫等多种重要的生命过程[12]。肉桂酸-4-羟基化酶(C4H)是植物苯丙烷代谢通路中的第2个酶,该酶在植物细胞中的含量可以影响黄酮等多种次生代谢物质的合成,基于此功能,国内外已有较多相关报道。Beak等通过对黑莓C4H基因的研究,发现C4H基因的表达量与黄酮积累量,在果实发育的不同时期表现出同步增加或减少的变化趋势[13];张东雪等通过对异黄酮含量合成差异较大的2个大豆品种豆27和九农20为材料,分别克隆控制大豆异黄酮合成的C4H基因,运用生物信息学分析对其基因功能进行分析预测,从而发掘潜在的功能性SNP[14]。但是,关于荞麦中C4H基因的报道却很少,陈翰鸿等以高黄酮苦荞栽培种西荞2号为材料,对其肉桂酸羟基化酶(FtC4H)cDNA进行克隆和序列分析,结果表明,其克隆得到的FtC4H基因的ORF全长为1 515 bp,编码504个氨基酸并具有C4H的所有活性位点,同时分析了芽期苦荞期在UV-B胁迫下子叶和胚轴中FtC4H表达水平与总黄酮含量具有显著的相关性[8]。李铁柱等[15]为了探明生物合成杜仲绿原酸的分子调控机制,对杜仲C4H基因的cDNA全长序列进行特征分析,结果鉴定出了2个基因家族成员,分别为EuC4H1和EuC4H2,全长分别为1 641 bp和1 611 bp,编码547和537个氨基酸,均为疏水性不稳定蛋白质。而本研究从苦荞叶片中成功克隆到2个C4H基因cDNA序列,长度分别为1 812 bp和1 476 bp,各自编码504和491个氨基酸,同时,与前人克隆的杜仲C4H基因编码区及氨基酸序列长度均不相同,且蛋白判别为亲水性不稳定蛋白质,推测C4H基因结构的不同导致功能也有所差异,杜仲C4H基因可能与杜仲绿原酸合成相关[16],而苦荞C4H基因与苯丙烷类代谢黄酮醇分支途径相关。2个基因启动子元件包含多个激素和环境胁迫响应元件,表明这2个基因的表达其可能受到外源激素信号分子调控及响应外界环境胁迫,推测苦荞中黄酮醇类物质合成可能受到上述外源激素和环境胁迫的影响。进一步分析其氨基酸序列理化性质及结构预测,表明2者的理化性质接近,一级结构基本一致;其蛋白质二级结构总体趋势大致相似,但其二级结构域组成所占比例出现差异,而三级结构预测进一步证实存在明显差异。系统进化树结果表明,C4H基因编码氨基酸序列在菊类分支植物物种间具有相对较高的保守性,推测菊类分支植物中存在苯丙烷类代谢黄酮类分支途径的保守进化趋势,可能与此类植物未经大规模人类的驯化相关,尽管荞麦经历人类耕种文明和驯化,但其仍然保留了苯丙烷类代谢黄酮类分支途径中的关键基因,且前人报道莴苣[17]和丹参[18]中同样含有高含量的黄酮类物质,具有较强的抗氧化性。
综上所述,应用生物信息学等相关技术手段对FtC4H基因的启动子元件、氨基酸结构、亲疏水性、同源序列比对及进化树等进行分析、预测,为进一步研究FtC4H基因的功能提供依据。
[1]赵丽娟.荞麦种质资源遗传多样性分析[D].北京:中国农业科学院,2006.
[2]向达兵,彭镰心,赵钢,等.荞麦栽培研究进展[J].作物杂志,2013(3):1-6.
[3]黎瑞源,石桃雄,刘筱嘉,等.荞麦分子遗传学研究进展[J].黑龙江农业科学,2014(11):151-156.
[4]樊冬丽.山西省荞麦品种资源的遗传多样性研究[D].太谷:山西农业大学,2003.
[5]王健胜.荞麦栽培品种的遗传多样性分析[D].杨凌:西北农林科技大学,2005.
[6]Fahrendorf T,Dixon R A.Stress responses in alfalfa (Medicago sativa L.) X VIII.Molecular cloning and expression of the elicitor-in-ducible cinnamic acid 4-hydroxylase cytochrome P450[J].Archives of biochemistry and biophysics,1993,305(2):509-515.
[7]李波,梁颖,柴友荣.植物肉桂酰辅酶A还原酶(CCR)基因的研究进展[J].分子植物育种,2006,4(3S):55-65.
[8]陈鸿翰,袁梦求,李双江,等.苦荞肉桂酸羟化酶基因(FtC4H)的克隆及其UV-B胁迫下的组织表达[J].农业生物技术学报,2013,21(2):137-147.
[9]王安娜,王婵婵,吴蕾,等.大豆C4H基因克隆及生物信息学分析[J].东北农业大学学报,2010,41(4):12-16.
[10]赵乐,马利刚,杨泽岸,等.独行菜C4H基因克隆与表达分析[J].药学学报,2017,52(5):821-831.
[11]Hou S,Sun Z,Linghu B,et al.Genetic diversity of buckwheat cultivars (Fagopyrum tartaricum Gaertn.) assessed with SSR markers developed from genome survey sequences[J].Plant molecular biology reporter,2016,34(1):233-241.
[12]黄满芬,王恒,方荣俊,等.桑树肉桂酸-4-羟化酶基因(MmC4H)的克隆及在不同桑品种间的表达差异[J].蚕业科学,2014,40(4):592-600.
[13]Baek M H,Chung B Y,Kim J H,et al.cDNA cloning and expression pattern of cinnamate-4-hydroxylase in the Korean black raspberry[J].BMB Reports,2008,41(7):529-536.
[14]张东雪,王艳,井妍,等.大豆GmC4H基因的克隆及同源基因的生物信息学分析[J].基因组学与应用生物学,2016,35(10):2768-2774.
[15]李铁柱,杜红岩,王淋.杜仲C4H基因cDNA全长序列特征分析[J].经济林研究,2014,32(1):34-39.
[16]杜红岩.杜仲活性成分与药理研究的新进展[J].经济林研究,2003,21(2):58-61,82.
[17]杜鹃.莴苣黄酮类化合物的抗氧化性研究[J].吉林医药学院学报,2013,34(3):176-178.
[18]董顺福,韩丽琴,赵文秀,等.六种中药黄酮与钙镁铜锌铁含量分析及其药效机理的研究[J].辽宁中医杂志,2007,34(10):1447-1448.
CloningandbioinformaticsanalysisofC4HgeneinTataryBuckwheat
LiuRonghua1,WangLi1,SunZhaoxia1,HouSiyu1,2*,LiHongying1,2,3
(1.CollegeofAgriculture,ShanxiAgriculturalUniversity,Taigu030801,China;2.InstituteofAgriculturalBioengineer,ShanxiAgriculturalUniversity,Taigu030801,China;3.ShanxiKeyLaboratoryofGeneticResourcesandBreedinginMinorCrops,Taiyuan030031,China)
[Objective]In order to understand the structural characteristics and phylogenetic evolution of the key enzyme—cinnamate-4-hydroxylase gene in plant phenylpropane metabolic pathway, we wanted to clone the cinnamate-4-hydroxylase (C4H) gene and analyze it by bioinformatics.[Methods]In this study, we used RT-PCR to clone the cDNA and DNA sequences ofC4Hgene from tartary buckwheat, the genetic structure and protein physicochemical properties were analyzed by bioinformatics, and constructed the phylogenetic tree by maximum likelihood method .[Result]The cDNA sequences of the two genes were 1 812 bp and 1 476 bp, and respectively encoding 504 and 491 amino acid residues. The DNA sequence of two genes were 2 774 bp and 2 364 bp, including three exons and two introns, there were significant differences in the first exon and two introns sequence length. Protein molecular weight were 58.002kDa and 56.401kDa, respectively, isoelectric point were 9.18 and 9.09. The homology of the two genes encoding the amino acid and the tartary buckwheat cinnamate-4-hydroxylase was 99% and 86% respectively, so it was namedFtC4H1 andFtC4H2 temporarily.FtC4H1 andFtC4H2 were into two classes with orthologous proteins of other species.FtC4H1 andFtC4H2 were closed to the homologous proteins ofLettuceandSalviamiltiorrhiza, and the amino acid sequence homology was 88% and 84%.[Conclusion]In this study, we analyzed the nucleic acid, amino acid sequence, protein structure and phylogenetic tree of twoC4Hgenes in tartary buckwheat, and provided the theoretical basis for the subsequent extraction of phenylpropane and the utilization of buckwheat gene.
Tartary buckwheat, Cinnamate-4-hydroxylase, Electronic cloning, Bioinformatics analysis
2017-07-06
2017-08-10
刘荣华(1993-),女(汉),山西大同人,硕士,研究方向:分子遗传育种
*通信作者:侯思宇,副教授,硕士生导师,Tel:18635068055;E-mail:bragren123@126.com
国家自然科学基金(NSFC:31301385);山西省科技攻关项目(20150311007-1)
S515;Q94
A
1671-8151(2017)11-0767-07
(编辑:韩志强)

