冷冻电镜技术:从原子尺度看生命——2017年诺贝尔化学奖简介
杨 慧 李慎涛 薛 冰
(首都医科大学中心实验室,北京 100069)
·诺贝尔奖·
冷冻电镜技术:从原子尺度看生命——2017年诺贝尔化学奖简介
杨 慧 李慎涛 薛 冰*
(首都医科大学中心实验室,北京 100069)
瑞典皇家科学院将2017年度诺贝尔化学奖授予雅克·迪波什(Jacques Dubochet)、约阿希姆·弗兰克(Joachim Frank)和理查德·亨德森(Richard Henderson),以表彰他们在“开发冷冻电镜技术解析溶液中生物大分子高分辨率结构方面”的贡献。
诺贝尔化学奖;冷冻电镜;生物大分子高分辨率结构
北京时间2017年10月4日,瑞典皇家科学院将2017年度诺贝尔化学奖授予瑞士的雅克·迪波什(Jacques Dubochet)、美国的约阿希姆·弗兰克(Joachim Frank)和德国的科学家理查德·亨德森(Richard Henderson),以表彰他们在“开发冷冻电镜技术解析溶液中生物大分子高分辨率结构方面”的贡献。他们建立了一种高效的方法,可将运动中的分子快速冷冻并借助冷冻电镜图像和计算机数据处理绘出其原子水平分辨率的三维结构。这一技术将生物化学的发展推进了全新的时代,三位科学家获得这一奖项可谓实至名归。
1933年,德国物理学家Ernst Ruska用电子束作光源、电磁线圈作透镜,设计并制造出第一台电子显微镜,放大倍数达到12 000倍。1954年西门子公司研制出有电子衍射功能的高分辨电镜Elmiskop I,被全球1 200家研究单位用于研究工作。此后的30年,电镜技术得到飞速发展,分辨率已达到单个原子的水平[1]。1986年,Ruska因发明电镜获得诺贝尔物理奖[2]。
电镜技术最初被认为只适用于无机物质的研究[3],在生物领域的应用受到了诸多不利因素的制约:第一,生物样品含有丰富的水分,而透射电镜工作的条件是高度真空环境;第二,高能电子束会对样品造成破坏;第三,生物样品主要组分是碳、氢、氧、氮等轻元素,对电子的反射和散射作用与背景相比效果相似,所获图像衬度低;第四,观察过程中蛋白分子与电子发生相互作用或由于温度变化而发生漂移,导致图像模糊。直至冷冻电镜技术的出现和发展才在一定程度上解决了上述问题,结构生物学因此而得到突飞猛进的发展[4]。
冷冻电子显微镜,简称冷冻电镜(cryo-electron microscopy,cryo-EM),是将生物大分子快速冷冻后,在低温环境下利用透射电子显微镜对样品进行成像,再经图像处理和重构计算获得样品的三维结构[5-6]。经过30年的发展,冷冻电镜技术已经成为重要的结构生物学研究方法,与X线晶体学(X-ray crystallography)和磁共振(nuclear magnetic resonance,NMR)一起构成了高分辨率结构生物学研究的基础。不过另外两种技术都有各自的局限性,比如X线晶体学只能对生长极为有序的三维结晶进行观察,而磁共振技术则要求被检测样品颗粒小,纯度非常高,不能够有重叠峰出现。但是,分子蛋白的特点是难于结晶,很多生物大分子相互结合、组装之后形成较大复合体,或者不稳定,难以用上述这两种技术进行分析和检测。单颗粒冷冻电镜技术的优势则是能够观察少量非结晶生物样品,获得高分辨率的结构图像,因此,冷冻电镜技术已成为生物大分子的结构研究的重要手段,这项技术极大地推动了生物学的发展[5-6]。
1 获奖者简介
1.1雅克·迪波什(JacquesDubochet)
Jacques Dubochet(图1),1942年生于瑞士,1973年博士毕业于日内瓦大学和瑞士巴塞尔大学,现为瑞士洛桑大学生物物理学荣誉教授。20世纪80年代,Jacques Dubochet就职于海德堡欧洲分子生物学实验室,带领一个课题组发现了水的玻璃态,成功地建立了单颗粒样品快速冷冻制样方法以及可以应用于细胞和组织等体积较大样品制备的高压冷冻方法,冷冻电镜样品制备问题的解决为冷冻电镜技术的发展提供了先决条件。时至今日,冷冻电镜领域的研究者仍然应用该方法来制备样品。

图1 雅克·迪波什(Jacques Dubochet)
1.2约阿基姆·弗兰克(JoachimFrank)
Joachim Frank(图2),1940年出生于德国,现在哥伦比亚大学任教,生物物理学家,美国科学院院士和美国人文与科学院院士。2014年获得本杰明·富兰克林生命科学奖(Benjamin Franklin Medal in Life Science of the Franklin Institute),2017年获威利生物医学科学奖(Wiley Prize in Biomedical Sciences)。Frank 长期以来从事电子显微镜成像方面的研究,并在发明冷冻电镜的过程中做出了重要贡献。1978年,Frank开始研究冷冻电镜单颗粒三维重构方法学,1995年,他用自己建立的方法成功解析出大肠杆菌核糖体的三维结构。现今大部分生物大分子冷冻电镜结构都应用单颗粒三维重构技术解析出来。

图2 约阿基姆·弗兰克(Joachim Frank)
1.3理查德·亨德森(RichardHenderson)
Richard Henderson(图3),生于1945年,苏格兰分子生物学家和生物物理学家,就职于剑桥大学英国医学研究会分子生物学实验室(MRC Laboratory of Molecular Biology, Cambridge, UK)。2016年获得英国最高科学奖皇家科普利奖章)( Copley Medal of the Royal Society)和美国国家研究院颁发的亚历山大·霍兰德生物物理学奖(Alexander Hollaender Award in Biophysics)。他是利用电子显微镜研究生物分子领域的开创者之一。1990年,Henderson发表了利用冷冻电镜技术获得了细菌视紫红质蛋白原子水平的三维结构模型,这是第一个用冷冻电镜解析出来的膜蛋白结构。他的研究证实了用冷冻电镜技术可以确定分子蛋白的近原子分辨率的三维结构,被视作冷冻电镜发展史上的里程碑。

图3 理查德·亨德森(Richard Henderson)
2 主要科学贡献
冷冻电镜技术发展经过了数十年的历程,科学家们从提出设想到实践探索,终于将这一技术发展至相对成熟,现已成为结构生物学研究的利器,用于解析大分子蛋白和蛋白复合体的结构,揭示蛋白生物学功能的分子基础。总的来说,冷冻电镜技术由低温制样、低剂量电镜成像和计算机图像处理三部分组成。
1)冷冻样品制备
生物样品的特点是不耐高能电子辐射,并含有大量水分。电子束对样品的破坏作用迫使科学家们寻找新的样品制备方法。早在1934年Marton就提出了冷却样品的方法[3]。20世纪50年代,Humberto Fernández-Morán开始尝试用冷却样品的方法以减少水分的蒸发和保护样品免受辐照损伤。他制备了薄层冷冻切片,发现当生物样品被冷冻后,水分会产生冰晶,冰晶产生强烈的电子衍射,掩盖了生物样品本身的信号,而且冰晶可改变样品的结构。因此,在电镜观察之前必须想方设法去除样品中的水分。然而,经过脱水处理的大分子蛋白结构发生改变,或聚集,或塌陷。显然,最理想的状态是在能保留水分的情况下观察生物样品[7]。1974年,Kenneth A. Taylor 和Robert M. Glaeser在约-120 ℃(该温度高于水的玻璃态向冰晶转化的温度)的低温下,观察到含水生物样品的衍射图像,但没有观察到冰晶的形成,并将此原因归结于水分子和蛋白表面相互作用所致。同时,他们发现冷冻样品不但可以耐受更长时间和更大剂量的电子辐照,并且衬度也有一定程度的提高[8-9]。在此基础上,Dubochet研究组继续尝试利用冷冻电镜观察含水生物样品,他们将液氮换成液态乙烷,发现了水的玻璃态。玻璃态的水和冰不一样,它无固定的形状,不存在晶体结构,与固态相比,它更像一种极端黏滞、呈现固态的液体。当Dubochet把这一发现撰写成论文向期刊投稿时,发现Mayer 和Bruggeler已抢先一步使用X线证实在快速冷冻情况下小水滴可以呈现玻璃态。Dubochet的论文被认为存在“扭曲自然”的争议而被拒稿。1981年,JournalofMicroscopy同意为他们刊登一篇简短的通讯。而在此之后,这一技术的发展出乎意料地顺利,顺理成章地成为冷冻电镜的制样方法[10]。其基本流程是把载有样品(如缓冲液中的病毒、蛋白质和DNA组装的颗粒等)的电镜载网快速投入经液氮/液氦冷却的液态乙烷中,在载网孔或支持膜上形成玻璃态的薄冰,样品颗粒(粒径大约十几到过百纳米)分散包埋在其中而形成冷冻样品(图4)。薄层水玻璃化法简单快速,适用于单颗粒生物样品的研究,此制样技术被广泛用于病毒、大分子及其复合体的研究[11]。

图4 冷冻电镜样品制备示意图[12]
值得一提的是,对于厚度大于1μm的样品,例如细胞和组织,不能被电子束穿过,不适合用该方法制备样品。Dubochet研究组意识到唯一能够克服薄层水玻璃化技术缺陷的方法是先对较大样品进行玻璃化处理,然后制备超薄切片,这就是CEMOVIS(Cryo-EM of Vitreous Sections)技术。经过25年的努力,Dubochet研究组建立了高压冷冻技术(high pressure freezing,HPF),他们发现,在常压下对生物样品进行快速冷冻,玻璃化深度只有10μm,而当压力达到2 kPa时,玻璃化深度则可达到常压下的10倍[13-14]。目前,应用高压冷冻技术制备的样品玻璃化深度可以达到200μm。对玻璃化生物样品可以进一步制备超薄切片,在冷冻电镜下可观察到接近活体状态的超微结构。以往,传统生物组织样品超薄切片制备技术采用固定、脱水、包埋等一系列化学处理,并使用重金属染色帮助提高图像的衬度,这些化学处理会破坏观察对象,在电镜下形成假象。高压冷冻制样技术避免了样品中可溶性成分的抽取、流失和移位,使研究者可以直接观察到接近生活状态的细胞和组织的天然形态[14]。另外,应用冷冻电子断层成像技术(cryo-electron tomography)可以对高压冷冻样品的蛋白分子甚至细胞器进行原位分析和三维重构,对了解蛋白的天然构象和功能具有重要意义[15-16]。
2012年,Dubochet在“冷冻电镜——第一个三十”评述中所说:“只要处理得当,水就可以成为电镜的好朋友”。Dubochet的研究改变了长期以来人们对水的状态的传统认识,玻璃态水的发现为冷冻电镜解决了样品制备的问题[10]。
2)低剂量电子成像技术(low-dose techniques)
有史以来,图像一直都是人类探索未知事物的关键。在冷冻电镜技术诞生之前,科学家们利用X线衍射和磁共振解析了上千种生物分子的结构,并将这些成果应用于基础研究和药物的开发。Henderson最初的专业是晶体衍射技术,他深知X线衍射和磁共振技术的弊端,使他果断放弃了X线衍射技术,转向冷冻电镜技术,并于1975年重构出细菌视紫红质蛋白模型,尽管这个模型现在看起来还远不够精细,但在当时已是冷冻电镜技术解析出的最好结构(图5)[17]。

图5 1975年,Henderson第一次用冷冻电镜技术得到的粗糙的细菌视紫红质三维结构[17]
1990年,Henderson研究组第一次使用冷冻电镜技术对同一样品的多个拷贝进行平均计算得到了细菌视紫红质蛋白的高分辨结构(图6),历时15年,他最终证实了利用冷冻电镜技术可以获得生物大分子的高分辨结构的可行性。他们发现,在冷冻温度下,可以用尽量低的电子剂量成像对样品进行数据采集,获得能够满足研究需求的蛋白氨基酸侧链信息。这一突破性进展被认为是冷冻电镜技术应用于大分子结构解析的里程碑[18]。
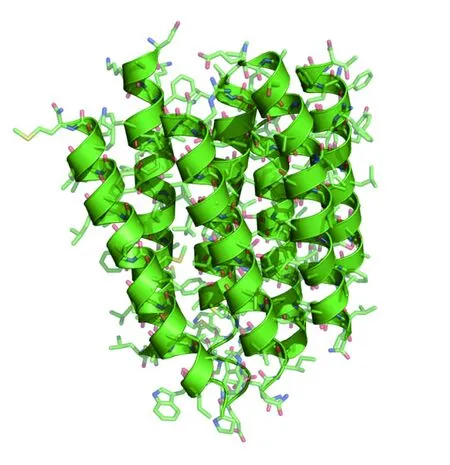
图6 1990年,Henderson用冷冻电镜解析出细菌视紫红质近原子分辨率的三维结构[18]
在研究过程中,Henderson研究组试用了全世界范围内的数台电镜以期提高数据质量,但发现了这些电镜的技术局限以及样品制备方法的不足,制约了图像分辨率的提高[18]。尽管当时情况不容乐观,但Henderson仍断言利用低电子密度、无损电子辐照在高衬度电镜下可以确定单个颗粒的二维位置和三维结构。他预言:随着电镜技术的发展和样品制备水平的提高,冷冻电镜技术一定能成为研究疑难样品和非结晶生物大分子的有力工具,并会发展成为常规、快捷的研究手段[18]。
事实上,电子束照射导致的样品损伤取决于电子剂量、样品的温度和电子束能量,生物样品的性质决定了必须采用低剂量电子成像技术。在超低温环境下观察样品,既解决了样品含有水分的问题,也减少了电子辐照和温度升高对生物样品的损伤。可以根据研究目的来综合考虑拍摄条件,例如:100 kV电压,液氮温度下适用的电子剂量一般是5~10 e-/ Å2;如果要获得近原子级别的高分辨三维结构,则需要加速电压达到300 kV,电子剂量一般要达到15~30 e-/ Å2[19]。 然而这样的低剂量成像带来的最大问题就是单张电镜图像的信噪比非常低,照片的衬度很差,导致获得的图像模糊。因此,新型的成像设备和强大的图像处理系统对于冷冻电镜技术的发展至关重要。
3)图像处理技术
试想,在20世纪70年代的冷冻电镜下观察不染色、非结晶、非对称、在溶解状态下随机分布的颗粒样品会看到什么结果?Frank[20]在一篇研究报告里描述如下:“在充满噪声的背景中仅能看到模模糊糊的样品”。他的研究工作正是从这里开始。
Frank等[21]提出了一种交叉相关函数的方法来校准低剂量电子辐照下拍摄的单分子图像。定量分析结果表明,利用对样品无损的电子辐照剂量获取的信息可以对随机分布的颗粒进行定位,通过对颗粒样品图像进行叠加和平均计算得到高分辨数据。这一方法的可行性在对负染的谷氨酰胺合成酶的研究过程中得以阐明。
对于均一非结晶样品的图像分析,难点在于每个颗粒的定位与定向,通常需要通过确定5个参数来判断二维平面图像的位置和三维结构的方向。然而,生物样品大多含有杂质,异质性高,这就要求分析系统具备可以识别样品潜在亚结构的能力以区分非均质样品中不同类别的颗粒;在颗粒染色的情况下,还要能够识别出负染的结构。1981年,Frank 和Marin van Heel提出了一种方法,可以根据颗粒的方向和结构特点对颗粒图像进行分类[22-23]。计算机可以根据细微的差异将图像自动分类,把颗粒分成不同的集群,每个集群代表一个具有特定位置和结构的颗粒的二维投影。在同一集群里不同颗粒被假定为具有足够的相似性,允许进入叠加和平均值的计算,使信噪比得以提高。1986-1987年,Frank和Michael Radermacher建立了非对称颗粒从二维投影到确定相应三维结构的方法——随机圆锥倾斜法(Random Conical Tilt)[24-25]。可以说,Frank建立了初步的方法学体系,奠定了冷冻电镜单颗粒三维重构的基本原理[26](图7)。此外,Frank还发展了多种用于图像分析的数学工具,将其汇总到一起开发出一系列计算机程序——SPIDER,用于单颗粒冷冻电镜结构分析,并得到广泛使用[27]。
综上所述,经过30年的不断改进和发展,冷冻电镜技术取得了长足的进展,在样品制备、成像和计算机处理等方面建立了理论基础,完成了方法学和技术准备,图像的分辨率也有了很大程度的提高。然而,研究者可以观察到的二维投影图像仍处于模糊状态,这成为冷冻电镜技术发展的瓶颈,限制了该技术的广泛应用。
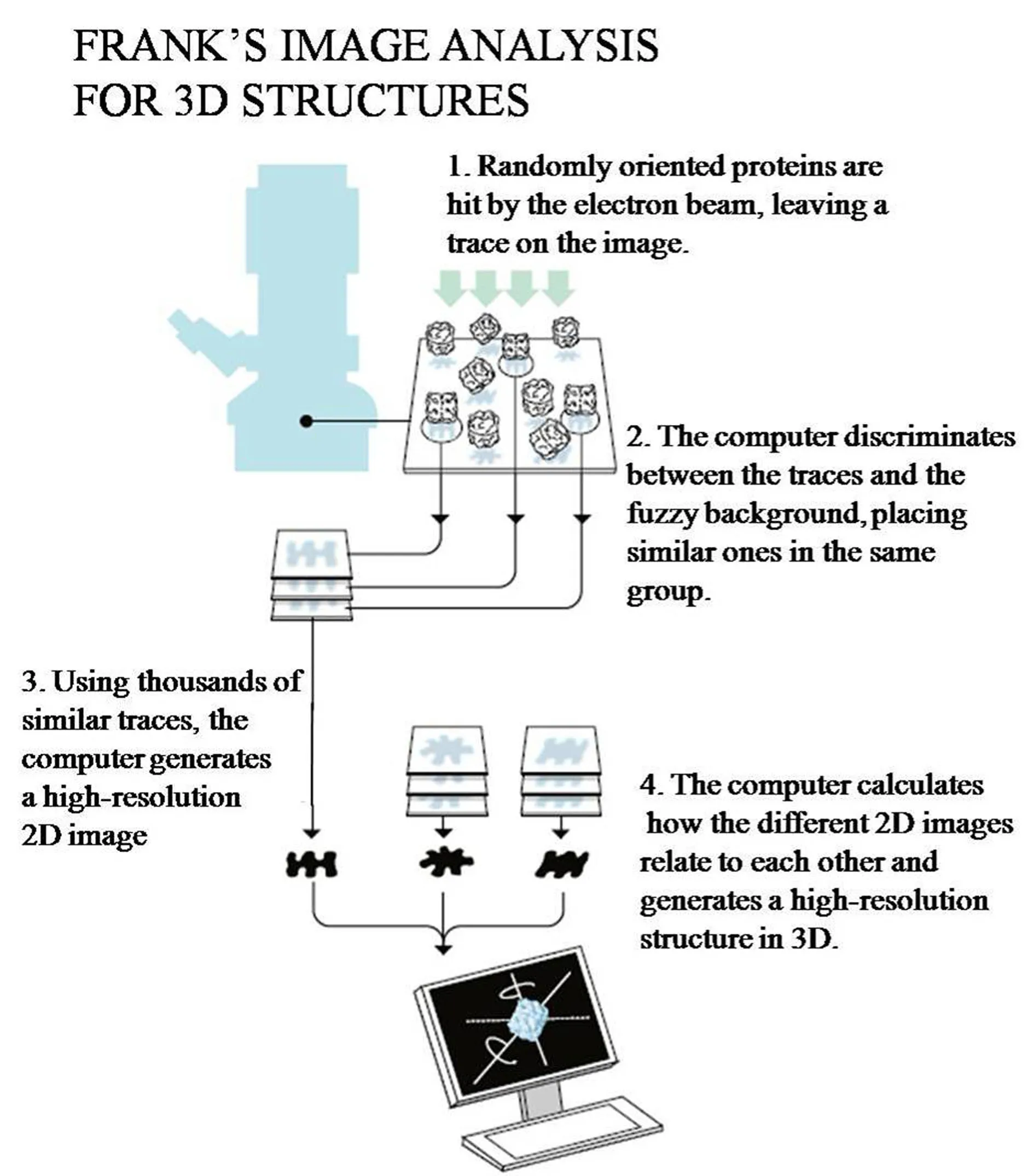
图7 Frank建立的三维结构图像分析技术示意图[26]
2012-2013年间,电子直接探测相机(electron direct detection device,DDD)的应用,使冷冻电镜技术突破了技术瓶颈,如虎添翼。DDD相机可以直接探测到高能电子,使信噪比和空间分辨率有了飞跃性的提高[28-29](图8)。DDD相机的另一个优势是采集信号速度快,某些情况下竟可以录制“电影”,这一功能可以补偿和矫正被观察中的样品由于电子辐照和热漂移效应产生的位移[29]。
过去的几年中电镜的分辨率得到显著提高,过去仅能看到蛋白分子模糊呈泡状结构,现在则可以看到原子水平分辨率的精细结构[29]。
成像技术上取得突破后,在短时间内解析出了多个蛋白的高分辨结构。2013年,美国加州大学旧金山分校程亦凡实验室率先利用直接电子探测器解析了约700 000(~15 nm)的蛋白酶体的结构,分辨率达3.3 Å[30];同年解析了约300 000的膜蛋白辣椒素受体(transient receptor potential vanilloid type-1,TRPV-1)四聚体结构,分辨率达3.4 Å[31]。美国国立卫生研究院Subramaniam实验室解析了334 000的谷氨酸脱氢酶的结构,分辨率达1.8 Å,这是到目前为止用单颗粒分析法解析的最高分辨率的蛋白结构[29]。
2015年,清华大学施一公研究团队报道了分辨率高达3.4 Å的人γ-分泌酶的三维结构,并且基于结构分析研究了γ-分泌酶致病突变体的功能,为理解γ-分泌酶的工作机制以及阿尔茨海默症的发病机制提供了重要依据[32]。随后他们又发表了“3.6 Å分辨率的酵母剪接体结构”和“前体信使RNA剪接的结构基础”两篇论文。由于动态性强,剪接体的研究一直以来被认为是极为困难的课题,然而借助冷冻电镜技术,能够看到冻结在运动中的生物分子,并且将其运动过程视觉化呈现出来,这是人类前所未见的成果[33-34]。因此,该研究在国内引起了不小的轰动。
2016和2017年,清华大学杨茂君课题组用冷冻电镜方法成功解析了线粒体呼吸链复合体的三维结构,为进一步理解其生物学功能奠定了基础[35-36]。
如今,冷冻电镜技术正如Henderson在20年前所预言,应用范围越来越广,解析的蛋白越来越小,并帮助科学家们解决了越来越多历史性的难题。
3 意义、启示与展望
冷冻电镜技术的发展历经了30年。1990年,用此技术解析出第一个蛋白高分辨结构;2012至2013年,DDD相机的应用使这项技术突破技术瓶颈,解析出更小分子质量蛋白的高辨率三维结构。本技术的建立、发展和广泛应用该归功于Jacques Dubochet、Joachim Frank和Richard Henderson在各自领域为此做出的贡献,他们的工作人们使对生命分子的研究进入原子水平的时代。
与X线晶体衍射和磁共振相比,单颗粒冷冻电镜技术具有无可比拟的优势,这项技术不需要蛋白结晶,只需要少量的蛋白样品,并且适合于不同大小的样品,例如小到64 000的血红蛋白,大到几兆道尔顿的大颗粒。冷冻电子断层技术甚至可用于更大的生物样品,例如组织器官和细胞,可获得大分子及其复合物在细胞或组织中原位的高分辨图像和结构[37-38]。
冷冻电镜技术的另一优势是,它呈现的不仅仅是静态结构。冷冻电镜的样品制备可在瞬间完成,可以捕捉到蛋白分子动态的变化[39-40]。或许将来人们可以在细胞或组织器官内原位观察到分子间相互作用及其动态变化过程。
尽管这次诺贝尔化学奖主要是褒奖3位科学家在单颗粒冷冻电镜技术方面所作出的贡献,但并不意味着这项技术已经非常成熟或完善。事实上,还有很多问题需要继续思考和探索,例如:冷冻样品制备方案的优化、“分子机器”动态结构的解析、解析后大分子的功能研究等。另外,冷冻电镜技术应用于细胞、组织乃至器官领域还有很多空白,例如生物大分子甚至细胞器在细胞原位的三维结构、相互作用、活动等等。通过对处于不同状态的蛋白分子结构解析来研究和阐明其功能,可以从原子水平揭示生命的密码,从而推动基础医学研究、疾病预防及治疗和高效药物的开发,造福人类。
[1] 郭可信. 金相学史话(6):电子显微镜在材料科学中的应用[J]. 材料科学与工程, 2002,20(1): 5-10.
[2] Ruska E. Nobel Lectures, Physics 1981-1990[M]. Singapore: World Scientific Publishing,1993 .
[3] Marton L. Electron microscopy of biological objects [J].Nature, 1934, 133, 911-911.
[4] Scientific Background on the Nobel Prize in Chemistry 2017[EB/OL]. (2017-10-05)[2017-10-13].https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/advanced.html
[5] Nogales E. The development of cryo-EM into a mainstream structural biology technique [J]. Nat Methods, 2016, 13(1):24-27.
[6] Kühlbrandt W. Cryo-EM enters a new era [J]. Elife, 2014, 3:e03678.
[7] http://onlinelibrary.wiley.com/doi/10.1111/j.1749-6632.1960.tb49990.x/abstract;jsessionid=27B4652D0FF81771784BD08341B32B95.f04t04.
[8] Taylor K A, Glaeser R M. Electron diffraction of frozen, hydrated protein crystals[J]. Science, 1974, 186(4168):1036-1037.
[9] Taylor K A, Glaeser R M. Electron microscopy of frozen hydrated biological specimens[J]. J Ultrastruct Res, 1976, 55(3):448-456.
[10] Dubochet J. Cryo-EM-the first thirty years [J].J Microsc, 2012, 245(3):221-224.
[11] Dubochet J, Lepault J, Freeman R, et al. Electron microscopy of frozen water and aqueous solutions [J].J Microsc, 1982, 128(3): 219-237.
[12] Image-Dubochet’s preparation method (948 kB) [EB/OL].( 2017-10-05)[2017-10-13]. https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/fig_ke_en_17_dubochetspreparationmethod.pdf.
[13] Sartori N, Richter K, Dubochet J. Vitrification depth can be increased more than 10 fold by high pressure freezing [J]. J Microsc, 1993, 172(1):55-61.
[14] Al-Amoudi A, Chang J J, Leforestier A, et al. Cryo-electron microscopy of vitreous sections [J]. EMBO J, 2004, 23(18): 3583-3588.
[15] Beck M, Baumeister W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail [J]. Trends Cell Biol, 2016, 26(11):825-837.
[16] Oikonomou C M, Jensen G J. Cellular electron cryotomography: toward structural biology in situ [J]. Annu Rev Biochem, 2017, 86: 873-896.
[17] Henderson R, Unwin P N. Three-dimensional model of purple membrane obtained by electron microscopy [J]. Nature, 1975, 257(5521): 28-32.
[18] Henderson R, Baldwin J M, Ceska T A, et al. Model for the structure of bacteriorhodopsin based on high-resolution electron cryo-microscopy [J]. J Mol Biol, 1990, 213(4):899-929.
[19] Grigorieff N, Harrison S C. Near- atomic resolution reconstructions of icosahedral viruses from electron cryo-microscopy [J]. Curr Opin Struct Biol, 2011, 21(2): 265-273.
[20] Frank J. Averaging of low exposure electron micrographs of non-periodic objects [J]. Ultramicroscopy, 1975, 1(2):159-162.
[21] Frank J, Goldfarb W, Eisenberg D, et al. Reconstruction of glutamine synthetase using computer averaging[J]. Ultramicroscopy, 1978, 3(3): 283-290
[22] Van Heel M, Frank J. Use of multivariates statistics in analysing the images of biological macromolecules [J]. Ultramicroscopy, 1981, 6(2):187-194.
[23] Frank J, Van Heel M. Correspondence analysis of aligned images of biological particles [J]. J Mol Biol, 1982, 161(1):134-137.
[24] Radermacher M, Wagenknecht T, Verschoor A, et al. A new 3-D reconstruction scheme applied to the 50s ribosomal subunit of E. coli [J]. J Microsc, 1986, 141(Pt 1): RP1-2.
[25] Radermacher M, Wagenknecht T, Verschoor A, et al. Three-dimensional reconstruction from a single-exposure, random conical tilt series applied to the 50S ribosomal subunit of Escherichia coli [J]. J Microsc, 1987, 146(Pt 2): 113-136.
[26] Image-Frank’s image analysis (pdf 1 MB) [EB/OL]. (2017-10-05) [2017-10-13]. https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/fig_ke_en_17_franksimageanalysis.pdf.
[27] Frank J, Shimkin B, Dowse H. Spider-A modular software system for electron image processing [J]. Ultramicroscopy, 1981, 6(1): 343-357.
[28] Bammes BE, Rochat R H, Jakana J, et al. Direct electron detection yields cryo-EM reconstructions at resolutions beyond 3/4 Nyquist frequency [J]. J Struct Biol, 2012, 177(3):589-601.
[29] Meyerson J R, Chittori S, Merk A, et al. Structural basis of kainate subtype glutamate receptor desensitization [J]. Nat, 2016, 537(7621): 567-571.
[30] Li X, Mooney P, Zheng S, et al. Electron counting and beam-induced motion correction enable near-atomic-resolution single-particle cryo-EM [J]. Nat Methods, 2013, 10(6): 584-590.
[31] Liao M, Cao E, Julius D, et al. Structure of the TRPV1 ion channel determined by electron cryo-microscopy [J]. Nature, 2013, 504(7478): 107-112.
[32] Lu P, Bai X C, Ma D, et al.Three-dimensional structure of human γ-secretase [J]. Nature, 2014, 512(7513): 166-170.
[33] Yan C, Hang J, Wan R, et al. Structure of a yeast spliceosome at 3.6-angstrom resolution [J]. Science, 2015, 349(6253): 1182-1191.
[34] Hang J, Wan R, Yan C, et al. Structural basis of pre-mRNA splicing [J]. Science, 2015, 349(6253): 1191-1198.
[35] Wu M, Gu J, Huang Y, et al. Structure of mammalian respiratory supercomplex I1III2IV1[J]. Cell, 2016, 167 (6):1598-1609.
[36] Guo R, Zong S, Wu M, et al. Architecture of human mitochondrial respiratory megacomplex I2III2IV2[J]. Cell, 2017, 170 (6): 1247-1257.
[37] Beck M, Baumeister W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail [J]. Trends Cell Biol, 2016, 26(11):825-837.
[38] Oikonomou C M, Jensen G J. Cellular electron cryotomography: toward structural biology in situ [J]. Annu Rev Biochem, 2017, 86: 873-896.
[39] Rubinstein J L. Cryo-EM captures the dynamics of ion channel opening[J]. Cell, 2017, 168(3): 341-343.
[40] Hite R K, Mackinnon R. Structural titration of Slo2.2, a Na+-dependent K+channel [J]. Cell, 2017, 168 (3): 390-399.
Cryo-electronmicroscopycapturelifeinatomicdetails—the2017NobelPrizeinChemistry
Yang Hui, Li Shentao, Xue Bing*
(CoreFacilityCenter,CapitalMedicalUniversity,Beijing100069,China)
The Royal Swedish Academy of Sciences has announced that the Nobel Prize in Chemistry 2017 was awarded to Jacques Dubochet, Joachim Frank and Richard Henderson for their developing cryo-electron microscopy for the high-resolution structure determination of biomolecules in solution.
the 2017 Nobel Prize in Chemistry;cryo-electron microscopy;high-resolution structure determination of biomolecules
*Corresponding authors, E-mail:xuebing_bj@126.com
时间:2017-10-14 16∶19
http://kns.cnki.net/kcms/detail/11.3662.R.20171014.1619.044.html
10.3969/j.issn.1006-7795.2017.05.027]
2017-10-12)
编辑 陈瑞芳

