茶树类黄酮3′-羟化酶基因的克隆与表达特性分析
王文丽,吴致君,刘志薇,王永鑫,李辉,崔新,庄静
茶树类黄酮3′-羟化酶基因的克隆与表达特性分析
王文丽,吴致君,刘志薇,王永鑫,李辉,崔新,庄静*
南京农业大学园艺学院,茶叶科学研究所,江苏南京210095
类黄酮3′-羟化酶(Flavonoid 3′-hydroxylase, F3′H)是细胞色素P450酶家族(Cytochrome P450,CYP450)的单加氧酶,在植物次生代谢和逆境调控方面起着重要的作用。本实验以茶树品种龙井43作为试验材料,利用RT-PCR方法,从cDNA中克隆得到茶树中编码类黄酮3′-羟化酶基因,命名为′。序列分析显示,′基因开放阅读框为1 530 bp,编码509个氨基酸,含有相对保守的P450酶系结合域。进化树分析表明,CsF3′H1与CsF3′H3的进化树关系相近,与CsF3′H2的进化树关系较远。序列多重比对显示,CsF3′H1蛋白具有F3′H蛋白的特征基序,并与高粱、猕猴桃、杨树、拟南芥和葡萄同源蛋白一致性为66.48%。氨基酸组分、理化性质、亲水性/疏水性和无序化分析显示,CsF3′H1是亲水性蛋白,无序化特征不明显。利用实时荧光定量PCR对′基因在茶树不同组织和不同激素处理下的表达谱进行检测,结果显示:不同组织中,′基因的表达水平在第一叶中最高,之后随着叶片的成熟逐渐下降,′基因在老叶和根中几乎不表达,表达水平从高到低依次为第一叶>第二叶>第三叶>第四叶>嫩茎>根>老叶;在不同激素处理中,′在SA激素处理下的表达量最高,为对照的2.24倍,在MeJA处理下表达量最低,为对照的0.43倍。
茶树;类黄酮3′-羟化酶;进化树分析;组织表达;激素处理
类黄酮3′-羟化酶是细胞色素P450(Cytochrome 450)酶家族中的单加氧酶[1]。细胞色素单加氧酶P450(CYP450)在生物界中广泛存在,是一大类定位在膜上的血红素蛋白[2]。Brugliera等从矮牵牛()中首次分离了′基因,被鉴定属于CYP75B2基因家族[3]。F3′H蛋白具有催化多种依赖NADPH或NADH的底物氧化反应的功能[3-5]。之后陆续在拟南芥()、玉米()、金鱼草()、矢车菊()、紫茎泽兰()、大豆()、葡萄()和苹果()等众多植物中分离和鉴定了′基因[6-11]。
茶具有抗癌、抗心血管疾病、抗辐射、抗氧化和减肥等功能。茶的这些功能与茶叶中富含多酚类次生代谢产物密切相关[6, 12-14]。类黄酮代谢途径是茶树中重要的次级代谢途径。类黄酮代谢途径中的中间产物能保护植物免受UV照射和昆虫的伤害,能通过调控花朵和种子的色素沉积,来吸引授粉者和种子传播者[15-16]。类黄酮3′-羟化酶(Flavonoid 3′-hydroxylase, F3′H)催化柚皮素(Naringenin)和二氢山奈素(2,3-dihydrokaempf, DHK)的B-环3′位置羟基化,分别生成圣草酚(Eriodictyol)和二氢槲皮素(2,3-dihydroquercetin, DHQ),是类黄酮代谢途径中的关键酶[17]。
本实验从茶树品种龙井43的cDNA中克隆得到1个′基因,命名为′。对该基因推导的氨基酸序列进行了序列比对、理化性质和亲/疏水性等方面的分析。本实验通过荧光定量PCR方法检测了′基因在龙井43茶树不同组织中和激素处理下的表达水平,为进一步研究茶树中类黄酮代谢途径及不同激素处理下′响应机制提供了实验基础。
1 材料与方法
1.1 植物材料
本实验材料选用种植在南京农业大学茶叶科学研究所的两年生龙井43(cv.)扦插盆栽幼苗。取龙井43扦插幼苗的幼嫩叶,提取RNA,再反转录成cDNA,作为′基因克隆的模板。
选取龙井43茶树两年生植株,对其叶片进行脱落酸(0.1 mmol·L-1ABA)、生长素(1 mmol·L-1IAA)、水杨酸(1 mmol·L-1SA)、赤霉素(1 mmol·L-1GA3)和茉莉酸甲酯(1 mmol·L-1MeJA)激素处理2 h。未做任何处理的茶树叶片作为对照。另外选取多株龙井43两年生植株的第一叶混样、第二叶混样、第三叶混样、第四叶混样、老叶混样、嫩茎混样和根混样作为不同组织基因表达材料。对上述取得的样品进行RNA提取,反转录为cDNA,用于实时荧光定量。
1.2 总RNA的提取及cDNA合成
使用Biotake Corporation(南京华普生物科技有限公司)试剂盒提取根组织的总RNA,使用Quick RNA Isolation Kit(北京华越洋公司)试剂盒提取叶和嫩茎组织的总RNA。用Nanodrop ND 1000(上海谱元仪器有限公司)检测提取的RNA样品浓度。用1.2%凝胶电泳检测RNA质量。利用PrimeScript RT reagent Kit with gDNA Eraser(大连TaKaRa公司)试剂盒将提取的总RNA样本反转录成cDNA。
1.3 茶树CsF3′H1基因克隆
基于本实验室课题组茶树转录组数据[18],得到1个茶树′转录本序列,并根据此序列设计1对克隆引物:
CsF3′H1-QF:ATGGAGACTCATTCCTGGATTGT;
CsF3′H1-QR:CTAATAAAGGCGACCTGAGAGC
扩增体系:2×Plus Master Mix酶10 μL、ddH2O 7 μL、模板1 μL和引物各1 μL。反应条件:94℃预变性5 min;94℃ 30 s,55℃ 30 s,72℃ 90 s,共32个循环;72℃ 10 min。之后进行PCR产物的分离和回收。以pMD 19-T作为载体进行连接,成功转化到大肠杆菌上后送测。委托南京金斯瑞生物科技有限公司进行测序。
1.4 生物信息学分析
核苷酸和蛋白质的BLAST比对和保守域预测在NCBI(美国国立生物技术中心)网站完成。氨基酸序列的多重比对和亲疏水性分析利用DNAMAN 6.0软件完成。利用在线工具包(SMS) (http://www.bio-soft.net/sms)和ExPASy- ProtParam (http://web.expasy.org/protparam)分析氨基酸的组成及理化性质。利用MEGA 5.0[19]软件构建系统发育树。荧光定量PCR数据的分析和图表绘制在Origin 6.0软件上进行。
1.5 茶树CsF3′H1基因的表达特性分析
实时荧光定量PCR(Quantitative real time RT-PCR)的步骤按照SYBR Premix试剂盒(大连TaKaRa公司)的操作说明进行。CFX96real-time PCR system作为荧光定量PCR平台,设计1对检测引物:
CsF3′H1-JF:CTATTGCAGCTTCTTGATGATCCGA
CsF3′H1-JR:GCTCTTTGGTTGCTTTGTTGATTAG
以茶树基因作为组织表达和激素处理的内参基因[20]:
TBP-JF:GGCGGATCAAGTGTTGGAAGGGAG
TBP-JR:ACGCTTGGGATTGTATTCGGCATTA
相对定量计算采用2–ΔΔT方法[21]。
2 结果与分析
2.1 茶树CsF3′H1基因的克隆
提取正常生长的龙井43扦插幼苗嫩叶的RNA,再反转录成cDNA,利用引物CsF3′H1-QF和CsF3′H1-QR进行PCR扩增,对扩增片段进行测序。结果如下:′基因开放阅读框长度为1 530 bp,编码509个氨基酸(图1)。
2.2 茶树CsF3′H1蛋白进化树分析
为了分析CsF3′H1蛋白在P450超基因家族蛋白的进化树关系。利用MEGA 5.0软件构建CsF3′H1蛋白与拟南芥P450超基因家族的进化树。结果显示(图2),CsF3′H1和CsF3′H3蛋白的进化树关系较接近,与CsF3′H2蛋白的进化树关系较疏远。
2.3 茶树CsF3′H1蛋白氨基酸序列比对
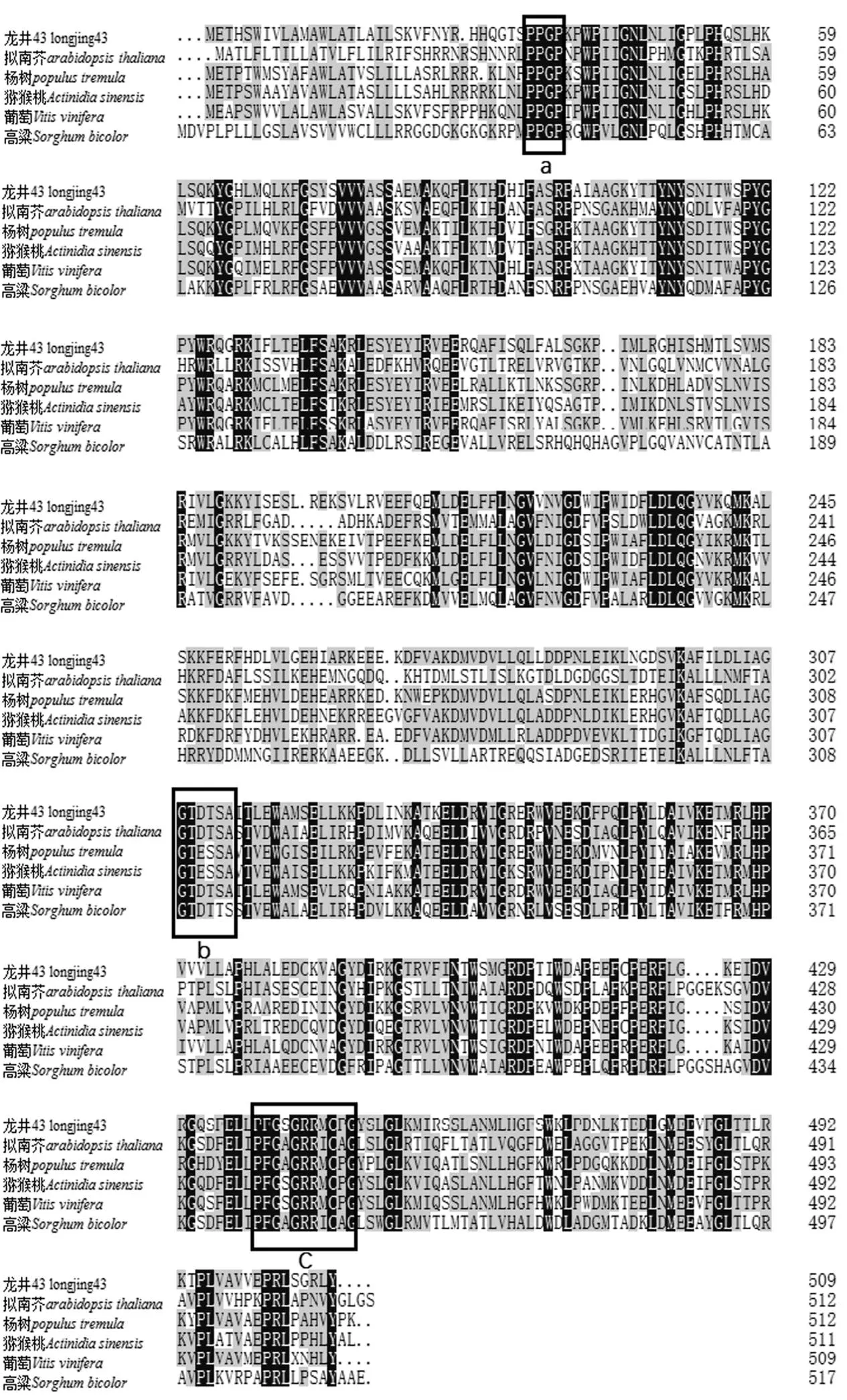
利用NCBI对CsF3′H1氨基酸序列进行BLAST同源检索与比对(图3)。结果显示,CsF3′H1蛋白含有P450家族典型的保守域,是P450超基因家族中的一员[22]。选取高粱、猕猴桃、拟南芥、葡萄和杨树4个代表性物种中的F3′H1同源蛋白与茶树的CsF3′H1蛋白进行序列的多重比对(图4)。结果显示,6个蛋白氨基酸序列的一致性为66.48%。在蛋白序列的N端含有膜的锚定位点基序,在C端含有与血红素结合区有关的氨基酸基序。

图1 茶树CsF3′H1基因核苷酸序列与推测的氨基酸序列
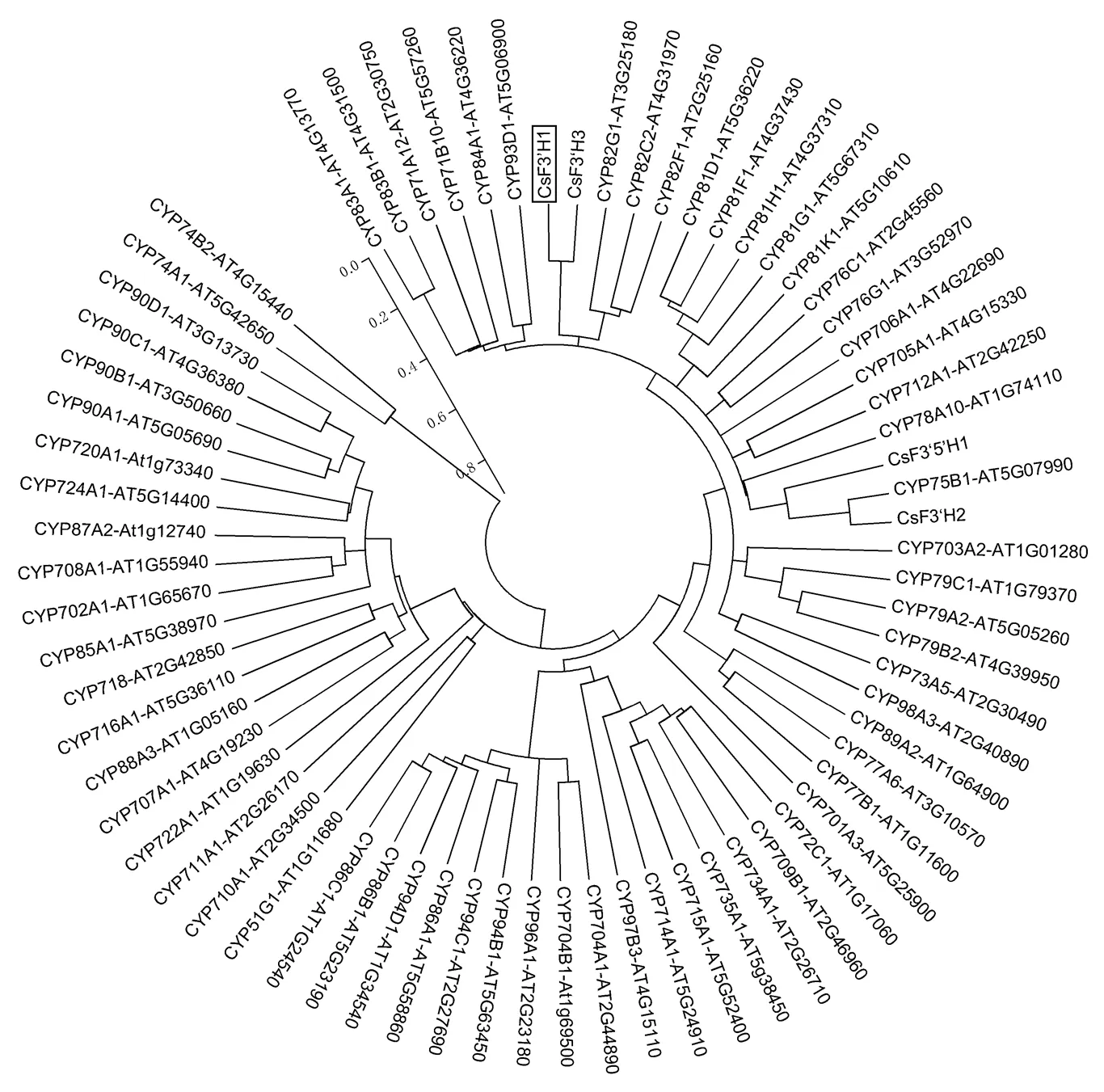
图2 茶树CsF3′H1蛋白的进化树分析

图3 茶树CsF3′H1蛋白的保守域预测
注:a:膜的锚定位点;b:与底物的选择和结合有关的氨基酸残基;c:与血红素结合区有关的氨基酸残基。
Note: a: The residues proposed to anchor. b: Residues of the active site hydrogen bond network. c: Residues of the heme-binding region.
图4 茶树′基因推导蛋白保守域及与其他物种氨基酸序列的多重比对
Fig. 4 The conserved domain and multiple alignment of CsF3′H1 protein
2.4 茶树CsF3′H1蛋白氨基酸的理化性质分析
F3′H1蛋白组成成分和理化性质的分析采用Gasteiger等[23]的方法(表1),结果显示上述植物的F3′H1蛋白氨基酸残基数在507~518之间,相对分子量为(5.59~5.83)×104。理论等电点()均在7~10之间。碱性氨基酸的平均含量为14%,酸性氨基酸的平均含量为10%左右,芳香族氨基酸和脂肪族氨基酸平均含量分别为7%和25%,总平均疏水性在蛋白质可溶性预测中处于-0.333~0.048之间。总之,不同的物种中F3′H1蛋白的理化性质存在一定差异,亲缘关系相近的物种差异相对较小。

表1 不同植物中F3′H1氨基酸组成成分及理化性质分析
2.5 茶树CsF3′H1蛋白推导的氨基酸亲/疏水性和无序化分析
利用DNAMAN 6.0软件对茶树′基因推导的氨基酸序列进行亲水性/疏水性分析。如图5显示,该蛋白疏水性最强的位点是在第373位的缬氨酸(Val),其次是第9位的亮氨酸(Leu);亲水性最强的位点是265位的谷氨酸(Glu),其次是第151的谷氨酸(Glu)。总的看来,大多数的氨基酸属于亲水性氨基酸。CsF3′H1蛋白氨基酸序列的折叠无序化分析结果(图6)显示,CsF3′H1无序化区域在整个氨基酸序列中占5个,最长的无序化区域含有氨基酸数为9个。无序化区域共含有37个氨基酸残基(图6中下划线部分),无序化比例为7.27%。总体而言,CsF3′H1蛋白氨基酸序列无序化程度不明显。
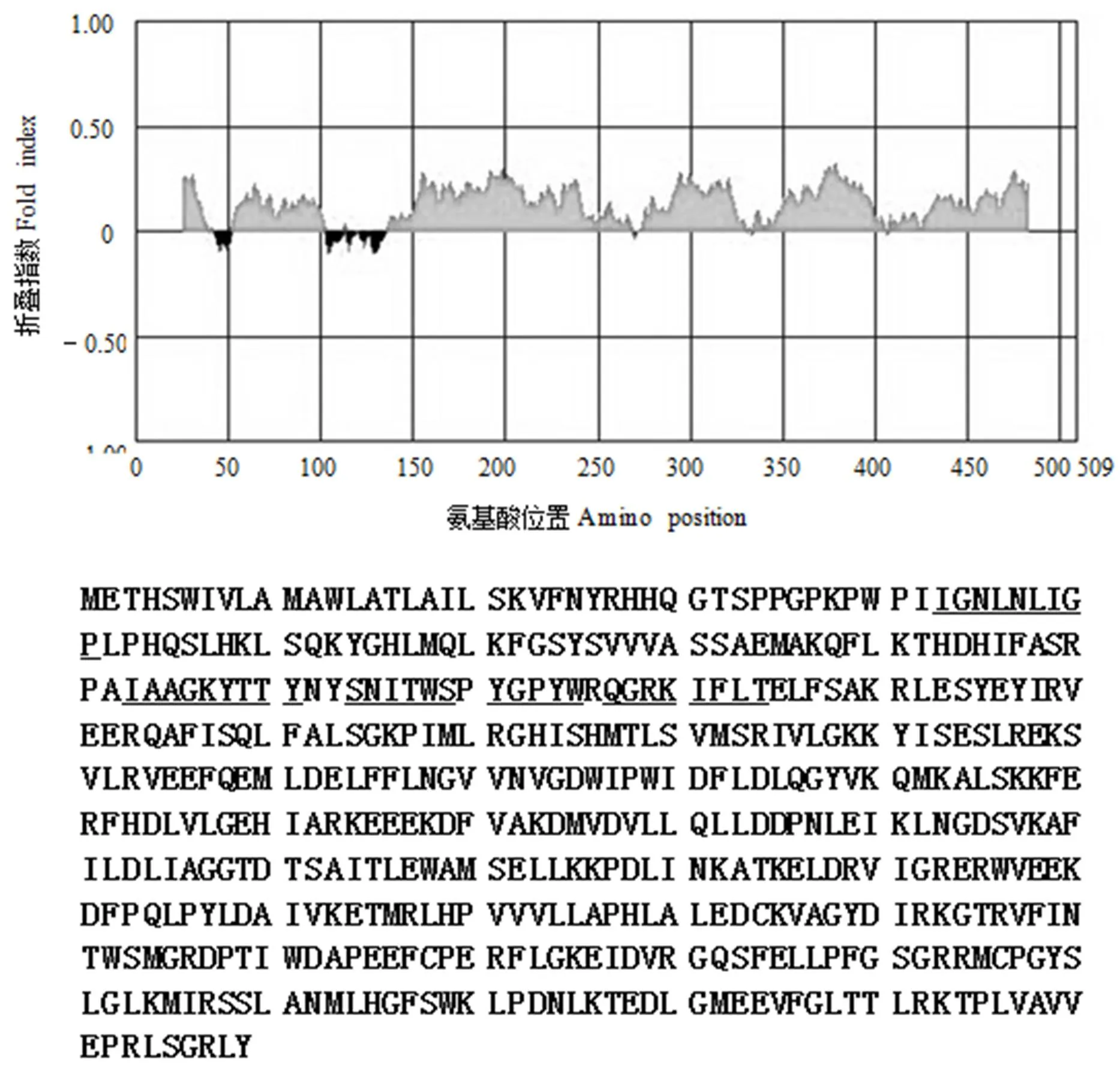
注:有下划线标注的是无序状态氨基酸,无下划线标注的是有序状态氨基酸。
Note: Amino acids represented non-ordered and ordered regions are showed in underline and non-underline, respectively.
图6 茶树CsF3′H1折叠状态的分析
Fig. 6 Analysis of the folding state of CsF3′H1
2.6 茶树CsF3′H1基因在不同组织中的表达分析
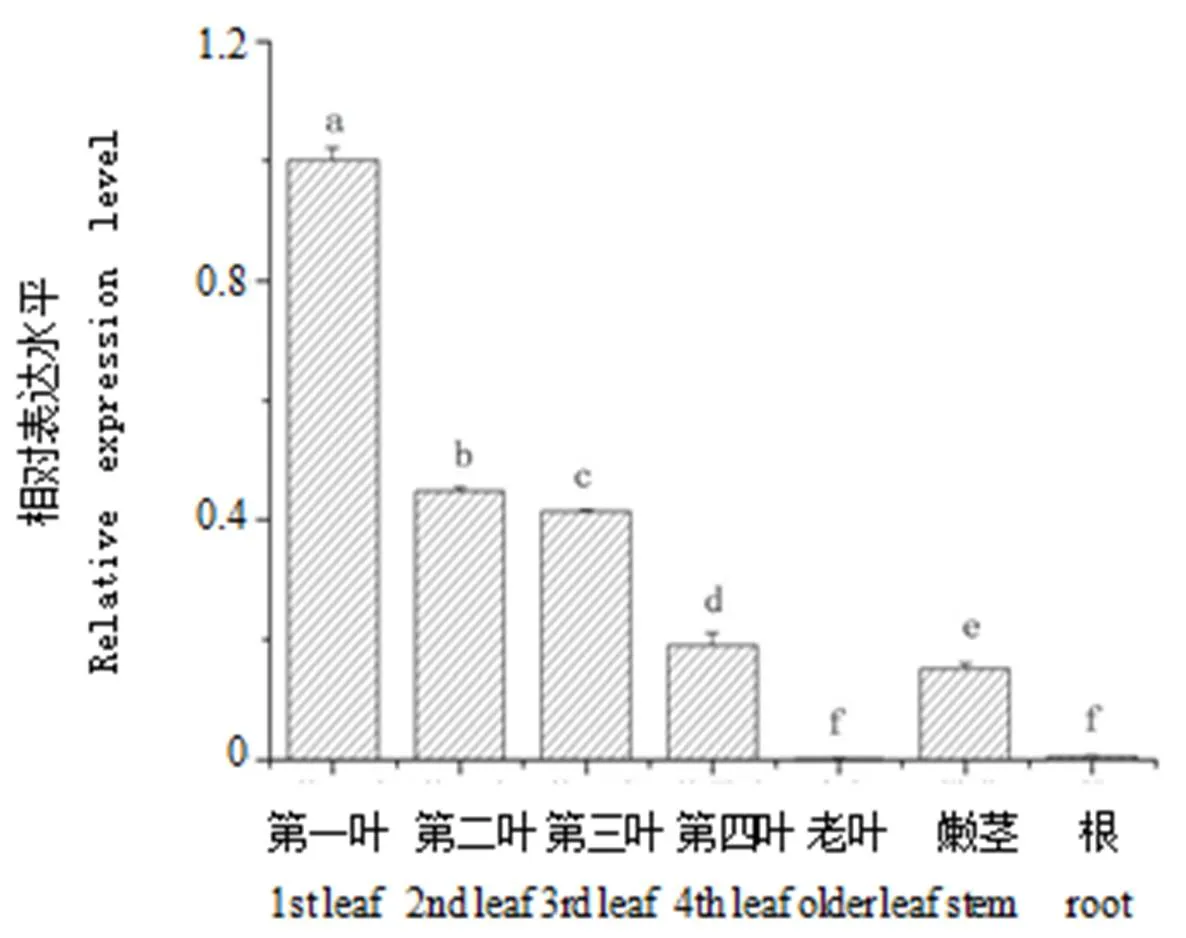
通过荧光定量PCR,检测了龙井43茶树中′基因在不同组织中的表达情况(图7)。结果显示,′基因在不同组织中表达差异很大,在第一叶中的表达量最高,随着叶片的发育,在第二、三、四叶中表达量逐渐下降,在根和老叶中的表达量几乎检测不到;′基因在第一叶中的表达量分别是根和老叶中的250倍和500倍;在不同组织中′基因的表达水平从高到低依次为:第一叶>第二叶>第三叶>第四叶>嫩茎>根>老叶。
2.7 茶树CsF3′H1基因在不同激素处理下的表达分析
用水杨酸、赤霉素、茉莉酸甲酯、脱落酸、吲哚乙酸5种激素分别处理茶树叶片2 h,结果(图8)显示,在水杨酸处理下′基因的表达量最高,为对照的2.24倍;′基因在茉莉酸甲酯处理下表达量最低,仅为对照的43%;与对照相比,′基因只有在水杨酸处理下表达量显著上调,在茉莉酸甲酯处理下表达量显著下调。
注:不同小写字母表示在0.05水平上差异显著。下同。
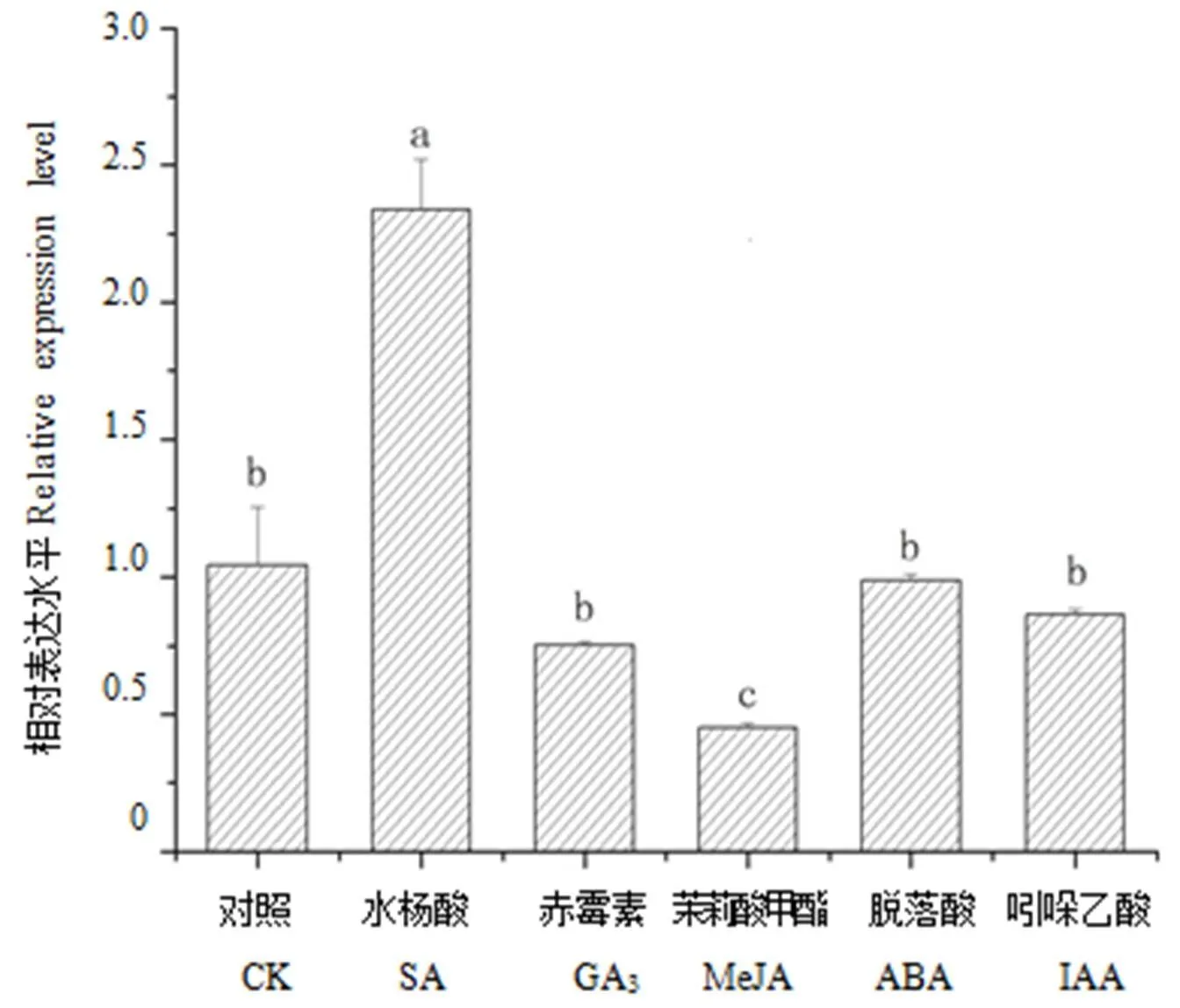
图8 CsF3′H1基因在茶树不同激素处理下的表达分析
3 讨论
类黄酮代谢途径是高等植物中重要的次级代谢途径之一。在植物中,′在决定植物花色、种皮和茎叶着色等性状方面发挥着重要的作用,′与′′的比例决定着葡萄果实着色的程度[17]。在茶树中,类黄酮途径中的代谢产物儿茶素能帮助人体提高抗癌、抗氧化、抗诱变、抗肿瘤和抗心血管疾病的能力[12-14]。类黄酮3′-羟化酶是类黄酮代谢途径中的关键酶,能催化柚皮素和二氢山奈素的B环3′位置羟基化。柚皮素和二氢山奈素被氧化形成一系列类黄酮途径中重要的中间产物,这些中间产物的结构稳定性和抗氧化功能与F3′H酶类密切相关[24]。Zhou等[25]的酶活实验中,鉴定了柚皮素是CsF3′H的最适底物[25]。
P450家族是植物中古老的超基因家族,按功能分类,主要分为参与生物合成和生物解毒两大类[26]。经鉴定,CsF3′H蛋白的F439GSGRRMCPG448血红蛋白结合区是P450超基因家族的典型特性基序,N端存在的P34PGP37保守位点是膜的锚定位点,G308TDTSA313基序一般是与底物选择相结合的残基位点[27-28]。进化树分析显示,CsF3′H1蛋白和CsF3′H3蛋白的进化树关系较接近,与CsF3′H2蛋白的进化树关系较远,CsF3′H2蛋白属于CYP75B1亚家族。这与Zhou等[25, 29]的实验结论相一致。
本实验从茶树中克隆获得1个编码F3′H酶的′基因,该基因开放阅读框长度为1 530 bp,编码509个氨基酸。大多数氨基酸属于亲水性氨基酸,无序化程度不明显。通过定量表达分析该基因在不同组织和不同激素下表达量的变化,不同组织中,荧光定量PCR分析显示′基因的表达水平在第一叶中表达量最高,从第一叶到老叶表达量依次递减,在老叶和根中的表达水平几乎检测不到,在嫩茎中的表达水平远小于鲜叶。推测′基因存在组织特异性,在鲜叶中的表达较高。Jiang等[18, 30]的研究表明茶树叶片中多酚类物质主要集中在嫩叶部位,推测′基因在类黄酮代谢途径中起正调控作用。
激素能通过信号转导启动或阻遏下游相关基因来发挥其作用。外源激素的喷施能明显地影响茶树的生长发育和鲜叶内含物质的生成[31]。响应不同激素刺激方面,′的表达水平除了在水杨酸处理下显著升高外,在其余激素处理下的表达量均处于下调状态。推测水杨酸能诱导′基因的表达,茉莉酸甲酯则抑制′基因的表达,赤霉素、脱落酸和吲哚乙酸对′的影响不大。
本实验研究了′基因在不同组织和激素刺激中的表达情况,推测′基因可能影响了儿茶素的合成。为进一步研究茶树中F3′H1蛋白的结构和功能奠定一定的理论基础。
[1] 侯杰, 佟玲, 崔国新, 等. 植物类黄酮3′羟化酶F3′H基因的研究进展[J]. 植物生理学报, 2011, 7(47): 641-647.
[2] De Vetten N, Ter Horst J, Van Schaik HP, et al. A cytochrome b5 is required for full activity of flavonoid 3′, 5′-hydroxylase, a cytochrome P450 involved in the formation of blue flower colors [J]. National Acad Sciences, 1999, 2(96): 778-783.
[3] Sandra E, Graham, Julian A. How similar are P450s and what can their differences teach us [J]. Archives of Biochemistry and Biophysics, 1999, 1(369): 24-29.
[4] Werck-Reichhart D, Feyereisen R. Cytochromes P450: a success story [J]. Genome Biology, 2000, 6(1): 30031-30039.
[5] Schuler M A, Werck-Reichhart D. Functional genomics of P450s [J]. Annual Review of Plant Biology, 2003, 1(54): 629-667.
[6] Soobrattee M A, Neergheen V. Phenolics as potential antioxidant therapeutic agents: mechanism and actions [J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2005, 1(597): 200-213.
[7] Schoenbohm C, Martens S, Eder C, et al. Identification of theflavonoid 3′-hydroxylase gene and functional expression of the encoded P450 enzyme [J]. Biology Chemistry, 2000, 8(381): 749-753.
[8] 刘海峰, 杨成君, 赵权, 等. 山葡萄中类黄酮3'-羟化酶基因(F3'H) cDNA的克隆和分析[J]. 植物生理学通讯, 2009, 45(12): 1186-1190.
[9] 黄文坤, 程红梅, 郭建英, 等. 紫茎泽兰类黄酮F3'H羟化酶基因的克隆、序列分析和原核表达[J]. 植物生理学通讯, 2007, 43(5) : 821- 826.
[10] Toda K, Akasaka M, Dubouzet E G, et al. Structure of flavonoid 3′-hydroxylase gene for pubescence color in soybean [J]. Crop Science, 2005, 6(45): 2212-2217.
[11] Han Y, Vimolmangkang S, Soria-Guerra RE, et al. Ectopic expression of apple F3′H genes contributes to anthocyanin accumulation in thett7 mutant grown under N stress [J]. Plant Physiology, 2010, 2(153): 806-820.
[12] Katiyar S, Mukhtar H. Tea in chemoprevention of cancer [J]. International Journal of Oncology, 1996, 2(8): 221-238.
[13] Hodgson J M. Tea flavonoids and cardiovascular disease [J]. Asia Pacific Journal Clinical Nutrition, 2008, 17(suppl1): 288-290.
[14] Wolfram S, Wang Y, Thielecke F, et al. Anti-obesity effects of green tea: from bedside to bench [J]. Molecular Nutrition & Food Research, 2006, 50(2): 176-187.
[15] Schaefer H M, Schaefer V, Levey D J, et al. How plant-animal interactions signal new insights in communication [J]. Trends in Ecology & Evolution, 2004, 19(11): 577-584.
[16] Regan B C, Julliot C, Simmen B, et al. Fruits, foliage and the evolution of primate colour vision [J]. Philosophical Transactions of the Royal Society B: Biolgical Sciences, 2001, 356 (1407): 229-283.
[17] Castellarin S, Gaspero G, Marconi R, et al. Colour variation in red grapevines (L.): genomic organisation, expression of flavonoid 3'-hydroxylase, flavonoid 3', 5'-hydroxylase genes and related metabolite profiling of red cyanidin-/blue delphinidin-based anthocyanins in berry skin [J]. BMC Genomics, 2006, 7 (12):1471-2164
[18] Wu Z J, Li X H, Liu Z W, et al. De novo assembly and transcriptome characterization: novel insights into catechins biosynthesis in[J]. BMC Plant Biology, 2014, 14(277): 1471-2229.
[19]Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Molecular Biology & Evolution, 2011, 28(10): 2731-2739.
[20] Wu Z J, Tian C, Jiang Q. Selection of suitable reference genes for qRT-PCR normalization during leaf development and hormonal stimuli in tea plant () [J]. Scientific Reports, 2016: 1-10. DOI: 1038/srep19748.
[21] Schäffer A, Aravind L, Madden T, et al. Improving the accuracy of PSI-BLAST protein database searches with composition-based statistics and other refinements [J]. Nucleic Acids Research, 2001, 14(29): 2994-3005.
[22] Zabala G, Vodkin L. Cloning of the pleiotropic T locus in soybean and two recessive alleles that differentially affect structure and expression of the encoded flavonoid 3′ hydroxylase [J]. Genetics, 2003, 1(163): 295-309.
[23] Gasteiger E, Gattiker A, Hoogland C, et al. EXPASY: the proteomics server for in-depth protein knowledge and analysis [J]. Nucleic Acids Research, 2003, 13(31): 3784-3788.
[24] 夏涛, 高丽萍. 类黄酮及茶儿茶素生物合成途径及其调控研究进展[J]. 中国农业科学, 2009, 42(8): 2899-2908.
[25] Zhou T S, Zhou R, Yu Y B, et al. Cloning and characterization of a flavonoid 3′-hydroxylase gene from tea plant ()[J]. International Journal of Molecular Sciences, 2016, 17(2): 261.
[26] 贺丽虹, 赵淑娟, 胡之璧. 植物细胞色素P450基因与功能研究进展[J]. 药物生物技术, 2008, 15(2): 142-147.
[27] Chapple C. Molecular-genetic analysis of plant cytochrome p450 dependent monooxygenases [J]. Annual Review of Plant Biology, 1998, 49(1): 311-342.
[28] Rupasinghe S, Schuler MA. Homology modeling of plant cytochrome P450s [J]. Phytochemistry Reviews, 2006, 5(2/3): 473-505.
[29] Wei K, Wang L , Zhang C, et al. Transcriptome analysis reveals key flavonoid 3′-hydroxylase and flavonoid 3′, 5′-hydroxylase genes in affecting the ratio of dihydroxylated to trihydroxylated catechins in[J]. PLoS One, 2015,10(9): e137925.
[30] Jiang X, Liu Y J, Li W W, et al. Tissue-specific, development-dependent phenolic compounds accumulation profile and gene expression pattern in tea plant () [J]. PLoS One, 2013, 8(4): e62315.
[31] 岳川, 曹红利, 曾建明, 等. 茶树中植物激素研究进展[J]. 茶叶科学, 2012, 32 (5): 382-392.
Cloning and Expression Analysis of the Gene Encoding Flavonoid 3′-Hydroxylase in Tea Plant ()
WANG Wenli, WU Zhijun, LIU Zhiwei, WANG Yongxin, LI Hui, CUI Xin, ZHUANG Jing*
Tea Science Research Institute, College of Horticulture, Nanjing Agricultural University, Nanjin 210095, China
Flavonoid 3′-hydroxylase (F3′H) belongs to the family of cytochrome P450 monooxygenases (CYP450) and plays important roles in plant secondary metabolism and stress reponses. In this study, a gene encoding F3’H-like protein was cloned by RT-PCR method using a cDNA template from tea () cultivar ‘Longjing43’. This gene is named as′. Sequence analysis showed that the open reading frame ofwas 1,530 bp length, encoding 509 amino acids. CsF3′H1 protein contains the conserved binding domain of P450 enzyme. Analysis of phylogenetic tree showed that CsF3'H1 protein has high similarity with CsF3'H3 protein and has low similarity with CsF3'H2 protein. Multiple alignments showed that CsF3'H1 protein has high similarity with the F3′H homologs from,,,, and(66.48% identity). CsF3'H1 protein contains the characterized motifs of F3'H-type protein. Analysis of amino acid composition, physical and chemical properties, hydrophilicity/hydrophobicity, and disordered residues of CsF3′H1protein showed that the disordered residues of CsF3′H1 protein are not obvious and most amino acids of CsF3′H1 protein are hydrophilic. The expression profiles of′gene in different tissues of tea plant or under hormonal treatments were detected using quantitativereal-time PCR analysis. Results showed that:gene has the highest expression level in the first leaf. The tissue expression profiles showed that the successive order ofgene expression levels was the first leaf > the second leaf > the third leaf > the fourth leaf > stem > roo > old leaf. The expression levelofgene was highest under SA treatment, which was 2.24 times high than the control.gene had the lowest expression level under MeJA treatment.
, flavonoid 3′-hydroxylase, phylogenetic tree, tissue expression, hormonal treatments
S571.1;Q52
A
1000-369X(2017)01-108-11
2016-08-22
2016-10-24
国家自然科学基金(31570691)
王文丽,女,硕士研究生,主要从事茶树分子生物学研究,E-mail:493930696@qq.com。
zhuangjing@njau.edu.cn

