DNA条形码在浙江沿海浮游鱼卵和仔鱼分类鉴定中的应用
陈永久,陈定标,蒋日进
(1.浙江海洋大学海洋科学与技术学院,国家海洋设施养殖工程技术研究中心,浙江舟山 316022;2.浙江海洋大学海洋与渔业研究所,浙江省海洋水产研究所,浙江舟山 316021)
DNA条形码在浙江沿海浮游鱼卵和仔鱼分类鉴定中的应用
陈永久1,陈定标1,蒋日进2
(1.浙江海洋大学海洋科学与技术学院,国家海洋设施养殖工程技术研究中心,浙江舟山 316022;2.浙江海洋大学海洋与渔业研究所,浙江省海洋水产研究所,浙江舟山 316021)
本研究的目的是对鱼卵和仔鱼进行分类鉴定,了解浙江沿海水域产卵区的物种组成。样品收集于2014年5月-2015年11月,在浙江沿海水域租用渔船采用大型浮游生物网(网长280 cm、网口内径80 cm、网口面积0.5 m2、网目0.5 mm、拖速2 n mile/h、拖网时间10 min)通过分析CO I DNA序列,结果显示从24个样本一共鉴定出10个鱼类物种,分属于7个科,即鳀鱼、小黄鱼、龙头鱼、康氏小公鱼、矛尾刺虾虎鱼、褐菖鲉和沙带鱼仔鱼,及凤鲚、蓝点马鲛、黄姑鱼鱼卵。种内平均K-2-P遗传距离0.47%,科内属间平均遗传距离21.7%,目内科间平均遗传距离24.6%,表明种内距离远小于种间距离,与绝大多数条形码研究结果一致。研究结果表明,DNA条形码技术是传统形态学强有力的补充。它是一种准确的,规范化的,有效的物种鉴定工具,在鱼类生物多样性研究中具有广阔的应用前景。
鱼卵;仔稚鱼;分类;DNA条形码;CO I;生物多样性
众所周知,对鱼卵、仔鱼的保护是实现渔业资源可持续开发利用的根本[1]。鱼类资源的世代强弱主要取决于鱼类早期生活史阶段的存活率。它的数量直接决定了成鱼资源的补充量。此外,鱼卵和仔鱼在生态系统中占有重要地位,对实现生态平衡具有重要意义[2]。通过对鱼类浮游生物群落生态的研究,可以有效地评估渔业资源的现状,确定鱼类产卵场和索饵场,了解鱼类浮游生物的补充机制,以及对环境的适应运转等。
尽管鱼类浮游生物具有重要的研究价值和生态地位,但是对其进行物种鉴定存在瓶颈。目前已鉴别的成鱼种类超过28 700余种,而能够鉴别的仔稚鱼仅仅占成鱼种类的1/10[3]。过去对鱼类浮游生物进行种类组成调查时一般采用形态学方法。但是由于海洋鱼类鱼卵发育变化复杂,不同种间形态差异不明显,仔稚鱼生长期形态变化快,仅仅通过形态学鉴定鱼类浮游生物是不适宜的[4-6]。KO et al[7]通过电镜扫描的方法在台湾五个实验室独立对鱼卵和仔鱼进行鉴定。结果鉴定到科的准确率为80.1%,鉴定到属的准确率为41.1%,鉴定到种的准确率仅为13.5%。也有学者也尝试过各种其他方法来鉴定鱼卵和仔鱼,例如单克隆抗体免疫染色技术[8]。这种方法不需要提取DNA,而且可以一次性鉴定大批鱼卵。但是它的成本很高。其他一些学者利用计算机科学辅助鉴定鱼卵和仔鱼[9-10]。这种方法适宜区分已知物种的大批鱼卵和仔鱼,但是对于多种鱼卵鉴定,需要提前把鉴别特征输入数据库。HEBERT et al[11]第一次提出利用线粒体DNA(mtDNA)特定编码区域进行物种分类,并期望赋予所有生物条形码。此后,国内外许多专家学者已经证明了利用DNA条形码技术进行海洋鱼类鉴别具有很高可信度[12],已经成为传统形态学鉴定强有力的补充。因为mtDNA结构简单,严格母系遗传,进化速率比核基因快[13]。其中细胞色素氧化酶第一亚基(CO I)基因即便在最初的发育阶段,在鱼卵中也有大量拷贝,作为鱼卵和仔鱼鉴定的分子标记是较合适的。因此通过DNA条形码技术实现鱼卵仔稚鱼的快速和准确鉴定在鱼类浮游生物多样性研究中具有广阔的应用前景。
本研究结合形态学和DNA条形码技术鉴定鱼卵和仔稚鱼。首先通过形态学方法将鱼卵和仔稚鱼鉴定到科或者属的水平;再分析CO I DNA序列鉴定到种的水平;最后对样本、形态及序列建立数据库。为下一步的鱼类生态学,生物多样性,及增殖放流等研究工作提供基础资料,以期最终实现浙江沿海渔业资源可持续开发利用。
1 材料与方法
1.1 样品采集
本文所采集的样品均来自浙江近海海域,具体如图1所示。采集时间为2014年5月-2015年11月。租用渔船,按照《海洋调查规范》采用大型浮游生物网(网长280 cm,网口内径80 cm,网口面积0.5 m2,网目0.5 mm)在海水表层进行水平拖网调查,拖速2 n mile/h、每个调查站位拖10 min。所采集的仔鱼和鱼卵立即保存于95%的酒精中,带回实验室后在显微镜下观察,进行形态学初步鉴定,并拍照保存。

图1 样品采集站点分布区域Fig.1 Sample collection sites
1.2 总DNA提取
使用QIAGEN公司DNeasy謼RBlood&Tissue Kit试剂盒提取样本DNA(具体操作方法参考QIAGEN的手册)。每尾仔鱼取整条或剪其尾部组织,稚鱼剪其尾鳍或胸鳍破碎消化,经过形态学鉴定为同一类的鱼卵取3~5颗进行消化。
1.3 PCR测序
线粒体CO I DNA的PCR引物为FishF1(5’-TCAACCAACCACAAAGACATTGGCAC-3’)和FishR1(5’-TAGACTTCTGGGTGGCCAAAGAATCA-3’)[12]。引物为上海生工生物有限公司合成。PCR扩增在一个25 μL 的反应体积中进行,反应体系为:ddH2O(8.5 μL)、2×Taq Mix(12.5 μL)、正向引物(1.0 μL、0.4 μM)、反向引物(1.0 μL、0.4 μM)、模板 DNA(2.0 μL)。PCR 扩增的程序为:95 ℃预变性 2 min;95 ℃变性 40 s;52℃退火1 min;72℃延伸1 min,共35个循环;最后72℃延伸5 min。PCR扩增产物通过琼脂糖凝胶(1.5%)电泳15 min,电泳完成后利用凝胶电泳成像仪观看图像。PCR产物检测阳性后送至上海生工生物技术有限公司进行序列测定,测序仪器为ABI-PRISM 3730。
1.4 序列分析
采用Sequencher 5.4(Gene Codes)和Clustal X2软件将测序结果进行编辑、人工校正和序列比对[14-15]。通过BLAST(http://www.ncbi.nlm.nih.gov/blast/blast.cgi),将所得DNA序列在Genbank中进行相似性检索,得出物种的分类地位。使用DnaSP 5.0软件[16]计算单倍型数。采用MEGA 5.0软件[17]计算序列Kimura双参数(K-2-P)遗传距离,再构建邻接法(NJ)聚类树,并通过Bootstrap检验聚类树各分支的置信度。
2 结果
2.1 DNA条形码鉴定结果
除去PCR扩增失败以及样品保存不良等问题,本研究一共获得了24条样本序列,共22个单倍型,包括3个鱼卵个体和21个仔鱼个体,编号见表1。序列长度为633 bp,均没有插入和缺失密码子。通过与Genbank数据库比对,如表1所示这24个样品分属7科10种,即鳀鱼Engraulis japonicus仔鱼、小黄鱼Larimichthys polyactis稚鱼、龙头鱼Harpadon nehereus仔鱼、康氏小公鱼Stolephorus commersonnii仔鱼、矛尾刺虾虎鱼Acanthogobius hasta仔鱼、褐菖鲉Sebastiscus marmoratus仔鱼、沙带鱼Lepturacanthus savala仔鱼,及凤鲚Coilia mystus鱼卵、蓝点马鲛Scomberomorus niphonius鱼卵、黄姑鱼Nibea albiflora鱼卵。
2.2 基于CO I序列构建的系统进化关系
基于CO I DNA序列构建的鱼卵、仔鱼及其参考物种的聚类关系树如图2所示。可以看出,鳀鱼聚为一支,最先与凤鲚和康氏小公鱼分享同一个祖先。小黄鱼与黄姑鱼在样品中的亲缘关系是最近的。此外,矛尾刺虾虎鱼聚为一支,其他种各自为一支。结果与传统形态学构建的演化关系基本相同。图中每一个编号代表一个单倍型。这也证明了DNA条形码技术在物种的分类鉴定研究上对于形态学是一个很好的补充。甚至能够克服形态学鉴定上的一些缺点。
2.3 K-2-P遗传距离分析
由表2可知,所有样本,同种内平均遗传距离为0.47%,同一科内属间平均遗传差异是21.7%,同一目内科间平均遗传差异是24.6%。根据相关报道[18],绝大多数鱼类的种内K-2-P距离不会超过2%,而且远小于种间距离。这24个样品与数据库对比结果显示,它们的相似度都在98%以上。因此我们认为,这24个样品都能够准确鉴定到种的水平。

图2 基于线粒体CO I DNA序列的24个鱼卵和幼仔鱼样本和参考物种的NJ聚类树Fig.2 NJ tree of 24 samples of fish eggs and larvae and reference species inferred from mitochondrial CO I DNA sequences.
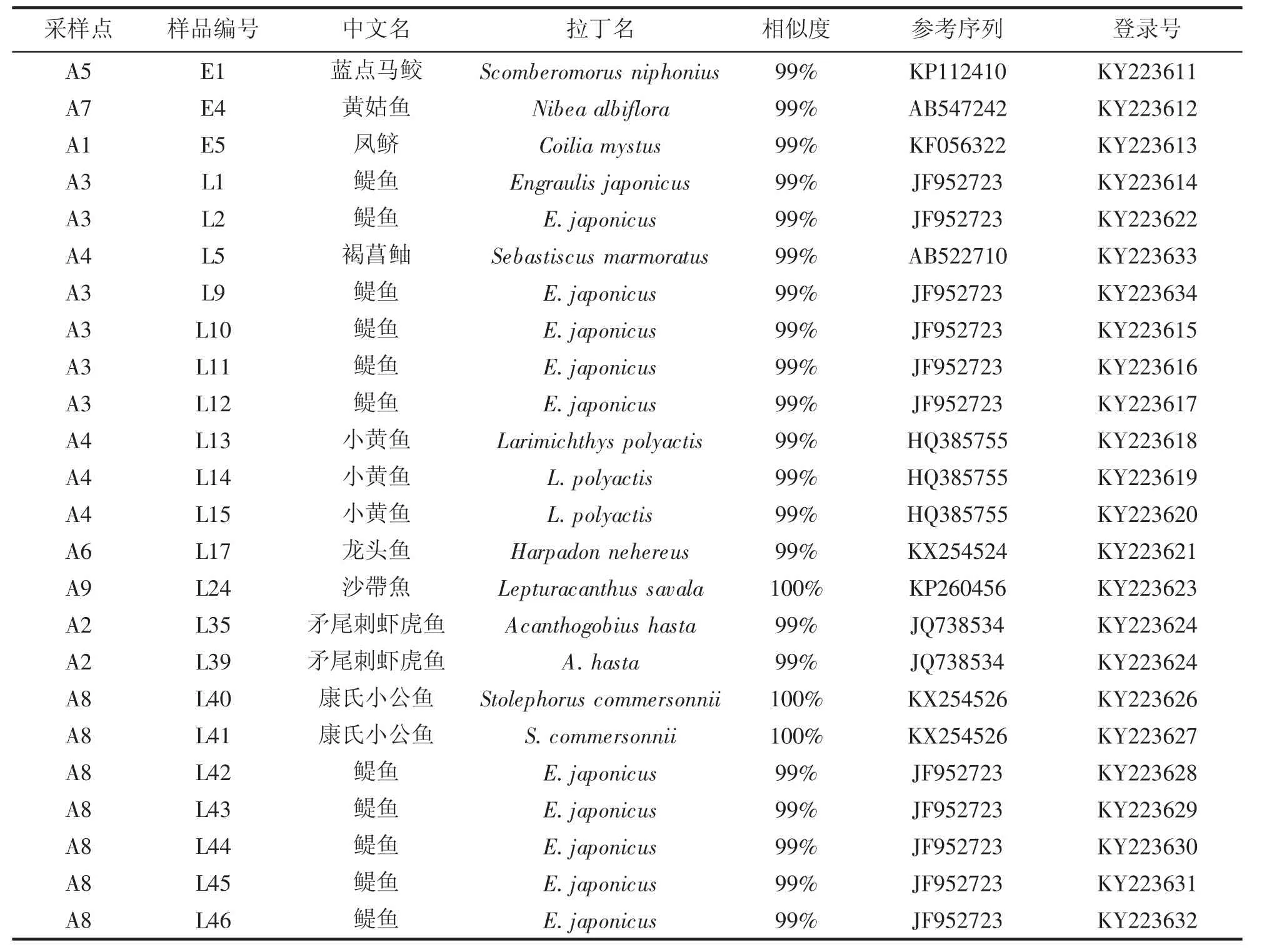
表1 浙江沿海鱼卵、仔鱼DNA序列比对及物种鉴定结果Tab.1 Results of DNA sequence comparison and species of fish eggs and larvae identified from the coastal sea of Zhejiang

表2 24个鱼卵、仔鱼样本及参考物种序列在不同分类级别的K-2-P遗传距离Tab.2 K-2-P distances for sequences of 24 samples of fish eggs and larvae,as well as reference species at different taxonomic levels
3 讨论
3.1 利用DNA条形码技术鉴定鱼卵、仔鱼的必要性
通过形态学建立的海洋鱼类分类以及鉴定系统较为完善,但是在仔稚鱼以及鱼卵形态鉴定的研究成果远远不及成鱼。研究表明,由于仔稚鱼的形态与成鱼的形态差异很大,亲缘关系远的物种的形态差异反而比亲缘关系近的要小,以及发育不完全等原因,形态学鉴定极为费时费力,并且鉴定到种水平的准确率不高。尽管不能否认形态学的基础作用,但要进一步将鱼卵、仔鱼鉴定到种水平需要借助DNA条形码技术。对鱼卵、仔鱼的鉴定仅仅依靠分子手段,只是属于一种技术过程。必须在提取DNA之前对其进行形态记录并且拍照保存,然后采用DNA条形码予以确认。此外,从条形码结果也可以反过来建立鱼卵、仔鱼的形态学鉴别特征,从而为传统的鱼卵、仔鱼分类鉴定的发展提供帮助。
3.2 DNA条形码技术的潜在问题
生物条形码数据库的完整性和准确性也极大的影响DNA条形码鉴定结果。如果数据库中没有该物种的成鱼序列,我们就无法鉴定该鱼卵或仔鱼的种类。KO et al[7]在类似的报道中也表明,数据库的不完整性是一个很严重的问题,特别是对一些非经济种类以及形态上很难区别的科或属,因为它们往往没有收录到数据库中。此外,错误的数据也会误导研究者。
基于我们的研究结果,不难看出,虽然利用COI单个基因能够解决大多数海洋硬骨鱼在种的水平的分类。然而,在高级阶元上的分类仍存在一定缺陷。VALDEZ-MORENO et al[19]人对墨西哥尤卡坦半岛附近海域的1 392个样品的K2P遗传距离分析发现,属内平均遗传距离是种内平均遗传距离的45倍左右,而科内平均遗传距离为属内的1.4倍,目内遗传距离仅为科内的1.19倍。由此可见,海洋硬骨鱼COI序列在高级阶元的差异并不明显。从表2可知,我们的结果有相似的趋势。王中铎等人也曾报道过,他们认为,这是由于COI序列仅为600 bp左右,信息位点不足,在高级阶元时碱基置换趋于饱和。导致海洋鱼类COI序列在高级阶元的分化明显变慢[20]。而在高级阶元的分类上,形态学已经构建了一个较为完善的系统。未来我们将通过新一代基因组测序技术以期获得更多信息位点加以验证[21]。
[1]李建生,胡 芬,程家骅.长江口水域春季鱼卵仔鱼分布及其与温度盐度的关系[J].海洋科学,2007,31(4):16-21.
[2]万瑞景,孙 珊.黄、东海生态系统中鱼卵、仔稚幼鱼种类组成与数量分布[J].动物学报,2006,52(1):28-44.
[3]SHAO K T,CHEN K C,WU J H.Identification of marine fish eggs in Taiwan using light microscopy,scanning electric microscopy and mtDNA sequencing[J].Marine&Freshwater Research,2002,53(2):355-365.
[4]卞晓东.鱼卵、仔稚鱼形态生态学基础研究[D].青岛:中国海洋大学,2010.
[5]CHEN I S,SHAO K T,HSU S L,et al.DNA barcoding of coastal larval fish communities of Dongsha Island,south China sea revealed by mitochondrial CO I sequences[J].Journal of Marine Science&Technology,2013,21(suppl):252-257.
[6]PEGG G G,SINCLAIR B,BRISKEY L,et al.MtDNA barcode identification of fish larvae in the southern Great Barrier Reef,Australia[J].Scientia Marina,2006,70(S2):7-12.
[7]KO H L,WANG Y T,CHIU T S,et al.Evaluating the accuracy of morphological identification of larval fishes by applying DNA barcoding[J].PLoS One,2013,8(1):p.e53451.
[8]HIROISHI,S,YUKI Y,YURUZUME E,et al.Identification of formalin-preserved eggs of red sea bream(Pagrus major)(Pisees:Sparidae)using monoelonal antibodies[J].Fishery Bulletin,2004,102(3):555-560.
[9]FROESE R,SCHFER W,RPKE A,et al.Computer aided approaches to identification.I:Expert systems[J].Fishbyte,1989,7(2):18-19.
[10]FROESE R,PAPASISSI C.The use of modern relational databases for identification of fish larvae[J].Journal of Applied Ichthyology,1990,6(1):37-45.
[11]HEBERT P D,CYWINSKA A,BALL S L,et al.Biological identifications through DNA barcodes.[J].Proceedings Biological Sciences,2003,270(1 512):313-321.
[12]WARD R D,ZEMLAK T S,INNES B H,et al.DNA barcoding Australia's fish species[J].Philosophical Transactions of the Royal Society of London B:Biological Sciences,2005,360(1 462):1 847-1 857.
[13]张春生,张晓龙,张雪莲,等.基于CO I基因序列的DNA条形码在中尼边境鼠类物种鉴定中的应用[J].西南国防医药,2012,22(1):22-24.
[14]HALL T A.BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT.[C]//Nucl Acids Symp Ser,1999:95-98.
[15]CHENNA R,SUGAWARA H,KOIKE T,et al.Multiple sequence alignment with the Clustal series of programs[J].Nucleic Acids Research,2003,31(13):3 497-3 500.
[16]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular,evolutionary,genetics,analysis,using maximum,like-lihood,evolutionary,distance,and maximum,parsimony,methods[J].Molecular Biology and Evolution,2011,28(10):2 731-2 739.
[17]LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1 451-1 452.
[18]周美玉,陈 骁,杨圣云.采用DNA条形码技术对厦门海域鱼卵、仔稚鱼种类的鉴定 [J].海洋环境科学,2015,34(1):120-125;135.
[19]VALDEZ-MORENO M,V魣SQUEZ-YEOMANS L,EL魱AS-GUTIRREZ M,et al.Using DNA barcodes to connect adults and early life stages of marine fishes from the Yucatan Peninsula,Mexico:potential in fisheries management[J].Marine and Freshwater Research,2010,61(6):655-671.
[20]王中铎,郭昱嵩,陈荣玲,等.南海常见硬骨鱼类CO I条码序列[J].海洋与湖沼,2009,40(5):608-614.
[21]RUBIN B E,REE R H,MOREAU C S.Inferring phylogenies from RAD sequence data[J].PLoS One,2012,7(4):95-98.
Application of DNA Barcoding in Classification and Identification of Fish Eggs and Larvae from Zhejiang Coastal Sea
CHEN Yong-jiu1,CHEN Ding-biao1,JIANG Ri-jin2
(1.School of Marine Science and Technology of Zhejiang Ocean University,National Engineering Research Center for Mariculture,Zhoushan 316022;2.Marine and Fishery Institute of Zhejiang Ocean University,Marine Fisheries Research Institute of Zhejiang Province,Zhoushan 316022,China)
This study aimed to identify and classify fish eggs and larvae and examine the species composition in spawning areas of Zhejiang Coastal waters.Samples were collected during May 2014-November 2015 using rental boats and plankton NETs(length 280 cm,internal diameter 80 cm,opening area 0.5 m2,mesh size 0.5 mm,trawl speed 2 n mile/h in the sea,trawling time 10 min).Via analysis of CO I DNA sequences,a total of ten species that belong in seven families were identified from 24 samples,i.e.the Japanese anchovy Engraulis japonicus,small yellow croaker Larimichthys polyactis,bummalo Harpadon nehereus,Commerson's anchovy Stolephorus commersonnii,yellowfin goby Acanthogobius hasta,sea ruffe Sebastiscus marmoratus,andLepturacanthus savala larvae,and Coilia mystus,Scomberomorus niphonius,and Nibea albiflora eggs.The average pairwise K-2-P distance is 0.47%within species,21.7%between genus within family,and 24.6%between families within order,indicating that the former is far smaller than the latter ones.Our results demonstrate that being a powerful complement to traditional morphology,DNA barcoding is an accurate,standardized,and efficient tool for species identification,and possesses a broad prospect of application in fish biodiversity research.
fish eggs;fish larvae;classification;DNA barcoding;CO I;biodiversity
S917.4
A
1008-830X(2017)03-0202-05
2016-12-01
浙江省海洋与渔业局项目(浙海渔计[2013]149号)”;浙江省海洋与渔业局项目(浙财农[2014]277号)”
陈永久(1969-),男,浙江普陀人,博士,副教授,研究方向:海洋生物遗传与进化.E-mail:yongjiu.chen@gmail.com

