陆地棉野生种系玛丽加朗特85基于SSR的遗传图谱
孔清泉,王玉红,周忠丽,王恒,蔡小彦,王星星,张振梅,王坤波,刘方
(棉花生物学国家重点实验室/中国农业科学院棉花研究所,河南安阳455000)
陆地棉野生种系玛丽加朗特85基于SSR的遗传图谱
孔清泉,王玉红,周忠丽,王恒,蔡小彦,王星星,张振梅,王坤波,刘方*
(棉花生物学国家重点实验室/中国农业科学院棉花研究所,河南安阳455000)
用陆地棉野生种系玛丽加朗特85与栽培种中棉所16杂交的188个F2单株,利用SSR标记构建了玛丽加朗特85的遗传图谱。图谱包含245个位点,26个连锁群,覆盖19条染色体,总长为1 866.05cM,标记间平均距离为7.62cM。其中79个标记偏离孟德尔分离比例,有5条染色体存在偏分离密集区 (Segregation distortion region,SDR):17号染色体上有2个SDR,3号、14号、15号和21号染色体上各有1个SDR。该研究为玛丽加朗特85优良性状基因定位、分子标记辅助育种奠定了工作基础。
半野生棉;玛丽加朗特85;遗传连锁图谱;SSR
棉花是我国重要的经济作物,然而随着“粮进棉退”趋势的加剧,1980年以后我国棉花种植区域逐渐向西北方向转移,其中向新疆集中转移的现象最为突出[1]。然而人们对当地水、土资源的不合理开发利用,导致新疆土壤次生盐渍化问题逐渐加重,并且已成为限制棉花持续发展的主要障碍性因子之一[2]。
陆地棉野生种系又称半野生棉,具有抗病虫、纤维强度高、品质优等特点,在干旱、盐碱含量较高的土壤中依然具有较强生存能力,是遗传育种研究及利用的重要种质资源[3]。近几年,科研工作者逐步开始挖掘半野生棉的优异基因。2016年郭展[4]利用渝棉1号与陆地棉野生种系阔叶棉(Gossypium hirsutumracelatifolium)TX43构建的F2群体,采用简单重复序列(Simple sequence repeat,SSR)标记,构建了1张总长度为1 367.9cM的遗传图谱。2015年李晓娜等[5]对半野生棉的棉仁含油量与SSR进行了关联分析,结果证明阔叶棉141是高油分品种。玛丽加朗特棉(G.hirsutumracemarie-galante)是7个半野生棉种系之一。本实验室的前期研究发现,玛丽加朗特85对新疆的复合盐碱具有高度抗性。中棉所16是1个陆地棉优良品种,前期鉴定中发现它对新疆复合盐碱的抗性较低[6]。本研究将中棉所16作为辅助材料,构建玛丽加朗特85的遗传图谱,有助于对其优良基因的定位、克隆、功能研究以及分子标记辅助育种的利用。
1 材料与方法
1.1 材料
利用半野生棉玛丽加朗特85为母本,陆地棉优质品种中棉所16作父本,配制杂交组合得F1,F1自交得F2。种植F2单株,随机选取188个单株用于遗传作图。
1.2 方法
1.2.1 棉花基因组DNA提取引物筛选F2群体基因型检测。从海南三亚野生棉种质圃取亲本、F1及188个F2单株的幼嫩叶片用改良的CTAB法[7]提取DNA。
从25 318对SSR引物中筛选在亲本玛丽加朗特85和中棉所16间具有多态性的引物,用于检测F2单株的基因型。基因扩增、电泳及染色参照陈浩东等[8]的方法。
1.2.2 群体基因分型数据读取及图谱构建。对于共显性引物:和母本玛丽加朗特85带型相同的单株数据代换为A,和父本中棉所16带型相同的单株数据代换为B,杂合单株记作H,缺失数据记作“-”。对于显性引物:母本玛丽加朗特85为显性(有带)以及分离群体中与母本玛丽加朗特85带型相同的单株记作D,纯合中棉所16带型单株记作B;父本中棉所16为显性(有带)以及分离群体中与父本中棉所16带型相同的单株记作C,纯合玛丽加朗特85带型单株记作A;缺失数据记作“-”。
参照陈浩东的方法[14],利用JoinMap 4.0作图软件,设置不同连锁群的LOD值设置为3~10。将连锁运算输出的结果,利用绘图软件MapChart 2.2绘制连锁图谱。根据使用的引物信息及已发表的遗传图谱上的共同标记结果确定连锁群所属的染色体。
2 结果与分析
2.1 引物筛选及F2群体基因型检测
本研究使用25 318对不同系列的SSR引物在双亲间进行多态性筛选,共筛选出多态性引物298对,其中59个显性标记,239个共显性标记,多态率为1.18%(表1)。不同种类的引物在两亲本上的多态性也不同。

表1 本研究使用的SSR引物情况
利用298对多态性引物对188个F2单株进行基因型检测,得到298个标记位点。图1是引物SWU10161对F2作图群体的基因分型电泳结果。
2.2 遗传连锁图谱的特征
利用多态性的引物对F2群体进行基因型检测后,获得298个位点,除53个标记没能进入任何连锁群,另外245个标记进入26个连锁群。根据已有的SSR遗传图谱[9-14]和SSR引物信息,将26个连锁群归入相应的染色体,覆盖19条染色体(图2、表2),平均每条染色体上有12.89个标记。图谱覆盖染色体长度为1 866.05cM,标记间平均距离为7.62cM,平均每条染色体的图谱长度为98.21cM。D2(17号染色体,Chr.17)染色体上标记数目最多,有25个;A6(6号染色体,Chr.6)最少,仅2个。遗传距离最长的是D3(14号染色体,Chr.14),最短的是D6(25号染色体,Chr.25);标记间平均距离最大的为A6(6号染色体,Chr.6),最小的为 D6(25号染色体,Chr.25)。At染色体组包含70个标记位点,图谱长度为604.87cM,Dt染色体组包含175个标记位点,图谱长度为1 261.18cM。
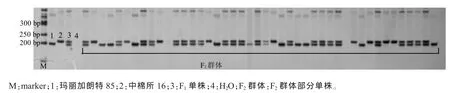
图1 引物SWU10161对F2群体基因分型电泳结果

图2 陆地棉野生种系玛丽加朗特85基于SSR的遗传图谱

表2 玛丽加朗特85×中棉所16构建的F2群体遗传连锁图谱信息
2.3 标记位点的偏分离检测
对进入遗传图谱的245个标记位点进行卡方检测,有79个位点不符合孟德尔分离比例 (P<0.05),其中35个偏向玛丽加朗特85,32个偏向中棉所16,有12个偏向杂合体。结果如表3。各染色体上的偏分离标记数相差较大。总的来说,Dt染色体组上的偏分离标记比例高于At染色体组上的偏分离比例。而且,部分偏分离位点成簇分布即偏分离热点区(Segregation distortion region,SDR)。据统计,图谱中有5条染色体存在SDR(≥3个偏分离标记),其中3、14、15、21号染色体上各存在1个SDR,17号染色体上有2个SDR。每个SDR内部不同标记偏分离方向基本一致,3和21号染色体上的SDR内部的标记大部分偏向杂合子,14号染色体上的SDR内部的标记大部分偏向父本中棉所16,15号染色体上的SDR内部的标记全部偏向母本玛丽加朗特85,17号染色体上的2个SDR,偏离方向不稳定。
3 讨论与结论
到目前为止,已发表的棉花高密度遗传图谱均为陆海种间遗传图谱,对半野生棉的研究较少,缺乏标记密度大、有代表性的半野生棉遗传图谱。
本研究构建了玛丽加朗特85遗传图谱,共包含245个SSR标记,图谱长度为1866.05cM。2016年郭展[4]构建了1张陆地棉优质品系渝棉1号×阔叶棉TX43的总长为1 367.9cM的图谱,包括At染色体组59个标记,Dt染色体组81个标记。宋宪亮[15]曾单独使用SSR标记构建的遗传图谱长度为5 644.3cM,后来又结合相关序列多态性(Sequence-related amplified polymorphism,SRAP)标记,使图谱长度增加近500cM,并且推测棉花遗传图谱长度不低于6200cM。本图谱中标记在染色体上分布不均匀(每条染色体上2~25个),而且多态性引物比例较低,这可能是因为分子标记类型单一,或者是因为所用的亲本在不同类型的引物间多态性差异较大。因此,在构建遗传图谱时,应该使用能扩增基因组不同区域的标记类型,充分利用不同类型的分子标记,以得到更多的多态性分子标记,提高对棉花基因组的覆盖范围和均匀性。
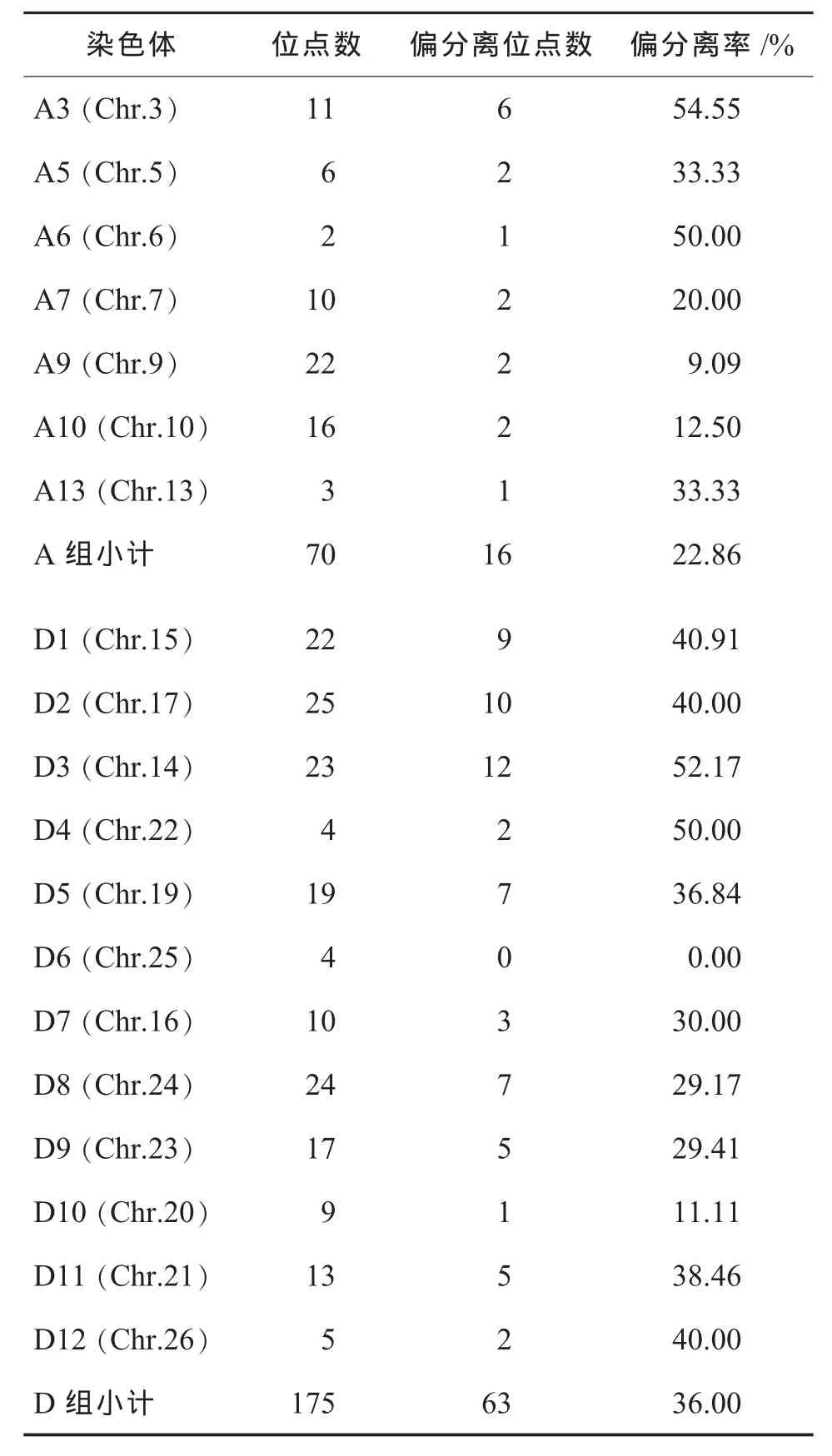
表3 本研究图谱中的偏分离情况
本研究为玛丽加朗特85优良基因或数量性状位点(Quantitative trait locus,QTL)的精细定位以及优良基因向栽培品种转育奠定了基础。
[1]朱会义.1980年以来中国棉花生产向新疆集中的主要原因[J].地理研究,2013,32(4):744-754.
[2]陈小兵,杨劲松,刘春卿,等.大农业条件下新疆土壤盐碱化及其调控对策[J].土壤,2007,39(3):347-353.
[3]胡绍安,崔荣霞,王春英,等.陆地棉野生种系的利用研究[J].棉花学报,1994,6(S1):15.
[4]郭展.陆地棉种内遗传图谱构建及衣分QTL定位[D].重庆:西南大学,2016.
[5]李晓娜,蔡小彦,Kiflom W O,等.半野生棉棉仁含油量与SSR标记的关联分析[J].棉花学报,2015,27(2):104-110.
[6]周忠丽,蔡小彦,王春英,等.半野生棉耐盐碱筛选初报[J].中国棉花,2015,42(1):15-18.
[7]宋国立,崔荣霞,王坤波,等.改良CTAB法快速提取棉花DNA[J].棉花学报,1998,10(5):273-275.
[8]陈浩东,刘方,王为,等.棉花多重PCR技术及其对杂交棉纯度鉴定的初步研究[J].棉花学报,2011,23(1):22-27.
[9]Guo W Z,Cai C P,Wang C B,et al.A preliminary analysis of genome structure and composition inGossypium hirsutum[J].BMC Genomics,2008,9(1):314.
[10]Yu J Z,Kohel R J,Fang D D,et al.A high-density simple sequence repeat and single nucleotide polymorphism genetic map of the tetraploid cotton genome[J].G3:(Bethesda),2012,2(1):43-58.
[11]Zhao Liang,Lü Y D,Cai Caiping,et al.Toward allotetraploid cotton genome assemble:integration ofa high-density molecular genetic linkage map with DNA sequence information[J].BMC Genomics,2012,13(1):539.
[12]Zhang Zhengsheng,Hu Meichun,Zhang Jian,et al.Construction of a comprehensive PCR-based marker linkage map and QTL mapping for fiber quality traits in upland cotton (Gossypium hirsutumL.)[J].Molecular Breeding, 2009,24(1):49-61.
[13]Zhang Ke,Zhang Jian,Ma Jing,et al.Genetic mapping and quantitative trait locus analysis of fiber quality traits using a three-parent composite population in upland cotton (Gossypium hirsutumL.)[J].Molecular Breeding, 2012,29(2):335-348.
[14]陈浩东.达尔文氏棉旱胁迫转录组测序、EST-SSR开发及高密度遗传图谱构建[D].北京:中国农业科学院,2013.
[15]宋宪亮.异源四倍体棉花栽培种分子遗传图谱的构建及部分性状QTL标记定位[D].泰安:山东农业大学,2004.
An SSR Genetic Map for AGossypium hirsutumracemarie-galante85
Kong Qingquan,Wang Yuhong,Zhou Zhongli,Wang Heng,Cai Xiaoyan,Wang Xingxing,Zhang Zhenmei, Wang Kunbo,Liu Fang*
S562.03
A
1000-632X(2017)08-0012-05
10.11963/1000-632X.kqqlf.20170811
2017-04-13 *通信作者:liufcri@163.cm
国家自然科学基金(31530053,31671745);国家重点研发计划(2016YFD0100203)

