一株抗蓝舌病病毒8型VP2蛋白单克隆抗体识别的线性抗原表位的鉴定
臧明鑫,李佳璇,谢双羽,崔文,姜艳平,徐义刚,乔薪瑗,王丽,周晗,刘敏,李一经,唐丽杰
东北农业大学 动物医学学院,黑龙江 哈尔滨 150030
一株抗蓝舌病病毒8型VP2蛋白单克隆抗体识别的线性抗原表位的鉴定
臧明鑫,李佳璇,谢双羽,崔文,姜艳平,徐义刚,乔薪瑗,王丽,周晗,刘敏,李一经,唐丽杰
东北农业大学 动物医学学院,黑龙江 哈尔滨 150030
臧明鑫, 李佳璇, 谢双羽, 等. 一株抗蓝舌病病毒8型VP2蛋白单克隆抗体识别的线性抗原表位的鉴定. 生物工程学报, 2017,33(8): 1244–1252.
Zang MX, Li JX, Xie SY, et al. Identification of the epitope recognized by a monoclonal antibody against the VP2 protein of bluetongue virus serotype 8. Chin J Biotech, 2017, 33(8): 1244–1252.
为研究本实验室制备的一株抗蓝舌病病毒8型 (BTV-8) VP2蛋白的单克隆抗体 (MAb) 3G11识别的B细胞抗原表位,利用噬菌体肽库展示技术对3G11识别的抗原表位进行筛选并鉴定。经过4轮淘选后挑取蓝斑测序,测序结果经分析后获得 KLLAT序列,与 BTV-8 VP2蛋白氨基酸序列比对后获得共同的短肽序列为283LL284;合成4种短肽序列:KLLAA、KALAT、KLAAT和KLLAT,与3G11细胞上清和腹水分别进行间接ELISA鉴定,结果表明,短肽KLLAA和KLLAT与3G11细胞上清及腹水具有较强的结合能力;与24种BTV标准阳性血清反应结果表明,这两种短肽都可与 BTV-8阳性血清发生特异性反应;序列分析结果可见,该表位的氨基酸序列283LL284在不同来源的BTV-8毒株间保守,确定283LL284为MAb3G11识别抗原表位的关键氨基酸。本研究为建立8型BTV特异性的免疫学检测方法和相关病毒蛋白的功能研究奠定了基础。
蓝舌病病毒8型,VP2蛋白,单克隆抗体,抗原表位
蓝舌病 (Bluetongue,BT) 是由蓝舌病病毒(Bluetongue virus,BTV) 引起,经库蠓、伊蚊等媒介昆虫进行传播的烈性非接触性传染病[1]。其主要感染山羊、绵羊、牛、骆驼等反刍类动物[2],感染动物的平均病死率为30%,其中绵羊的病死率可高达80%。世界卫生组织 (OIE) 规定其为必须呈报的动物疫病,中国将其列为一类动物传染病[3]。该病具有地域性、季节性和周期性的特点,迄今为止已发现 27种血清型的BTV,各型之间无交叉保护[4-6]。我国已检测出BTV-1、BTV-2、BTV-3、BTV-4、BTV-5、BTV-7、BTV-12、BTV-15、BTV-16和 BTV-24共 10种血清型,其中 BTV-1和 BTV-16为我国主要的致病血清型[7-9],但没有我国检测到BTV-8的报道。2006年8月,BTV-8在荷兰首次流行,后在欧洲大规模流行,该病在全球的暴发有扩大趋势,且血清型种类多、变异快,为有效控制其流行,早期诊断就显得尤为重要[10-12]。
BTV是迄今为止发现的分子量最大的RNA病毒[13],其基因组由10个双链RNA组成,这10个dsRNA包裹于双层蛋白质衣壳内,共编码7个结构蛋白 (VP1-7) 和 4种非结构蛋白(NS1、NS2、NS3/NS3a和 NS4)[14]。外壳蛋白VP2由L2基因编码,不同BTV血清型间VP2的氨基酸序列差异从 22.4%到 73%不等[15-17]。相同血清型的不同毒株间的VP2序列也存在较大差异,最大可达到30%,这表明VP2序列的变异与BTV血清型之间有密切联系[18-20]。本研究中应用噬菌体展示技术试剂盒筛选本实验室制备的一株抗 BTV-8 VP2蛋白的单克隆抗体(MAb) 3G11所识别的B细胞表位,并合成4种短肽作为抗原通过间接ELISA方法与BTV-8型标准阳性血清反应,对筛选出的抗原表位进行鉴定,本研究为建立8型BTV特异性的免疫学检测方法和相关病毒蛋白功能的研究奠定基础。
1 材料与方法
1.1 单克隆抗体和血清
抗BTV-8 VP2蛋白MAb 3G11 (抗体亚类IgG2b) 细胞株由本实验室制备并保存;1–24型BTV标准阳性血清及阴性血清购自蓝舌病参考实验室The Pirbright Institute;MAb 3G11纯化后腹水,效价为10–5(纯化后腹水SDS-PAGE鉴定结果见图1)。
1.2 主要试剂
Ph.D.-12TM13噬菌体肽库试剂盒购自美国New England BioLabs (NEB) 公司;HRP标记的山羊抗鼠IgG购自中杉金桥有限公司;TMB显色液购自北京博奥拓达科技有限公司。
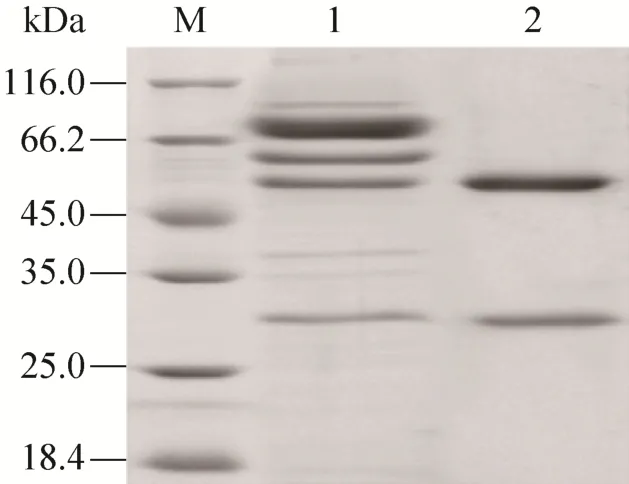
图1 纯化后的腹水 SDS-PAGE鉴定 (M:蛋白marker;1:纯化前腹水;2:纯化后腹水)Fig. 1 Analysis of purified ascitesby SDS-PAGE. M:marker; 1: ascites; 2: purified ascites.
1.3 间接免疫荧光试验
将BHK21细胞传至放置爬片的12孔板内,待细胞长满单层后接种8型BTV,同时设置对照孔。3%多聚甲醛固定液室温固定30 min;每孔加入3%的BSA 37 ℃封闭1 h;以保存的细胞上清液为一抗,同时设置细胞对照组,37 ℃孵育1 h;用PBS洗涤3次,5 min/次;加入1︰2 000稀释的FITC标记山羊抗鼠IgG,200 μL/孔,避光条件下37 ℃作用1 h;避光条件下用PBS洗涤3次,5 min/次;在倒置荧光显微镜下观察实验结果。
1.4 夹心ELISA鉴定MAb3G11特异性
兔抗BTV血清包被的ELISA板,每孔100 μL,37 ℃孵育2 h;用PBST洗3次,每次5 min;孔中加50 μL BTV-8型病毒,37 ℃ 1 h,洗板3次;1︰5 稀释单抗上清,每孔 50 μL。37 ℃ 1 h,洗板3次;加1︰8 000稀释羊抗鼠二抗 (HRP),每孔50 μL,37 ℃ 1 h,洗板3次;经TMB显色15 min,再加入终止液,读取OD450值。
1.5 抗原表位的筛选
参照 Ph.D.-12TM13噬菌体肽库说明书,将MAb腹水作为抗原,加入到微孔板中,对噬菌体肽库进行 4次淘选,将第4次淘选后的产物测序;将测序结果进行分析比对,获得表位序列KLLAT,为了验证该短肽的准确性,设计突变不同氨基酸的 4种短肽 KLLAA、KALAT、KLAAT和KLLAT,由武汉明皓生物科技股份有限公司合成。
1.6 利用间接ELISA鉴定短肽与MAb 3G11的反应性
用包被稀释液将 4种短肽 KLLAA、KALAT、KLAAT和KLLAT稀释成不同的浓度,包被酶标板,以MAb上清或纯化腹水为抗体,HRP标记的山羊抗鼠IgG作为酶标抗体,加入TMB显色液,显色10 min后加入终止液,鉴定短肽活性。
1.7 利用间接 ELISA鉴定短肽与标准血清的反应性
将短肽KLLAT作为包被抗原,一抗为1–24型BTV标准阳性血清及阴性血清,鉴定短肽与BTV阳性血清的反应活性。
1.8 抗原表位的保守性分析
利用 NCBI数据库中的 Blast软件对 MAb 3G11识别的抗原表位序列283LL284进行保守性分析。
2 结果与分析
2.1 间接免疫荧光鉴定结果
将 MAb 3G11细胞上清分别接种于 8型BTV感染的BHK21细胞和正常BHK21细胞,结果显示,接种BTV-8的BHK21细胞发出绿色荧光,对照细胞无荧光,表明MAb 3G11可以特异性识别8型BTV (图2)。
2.2 夹心ELISA法检测结果
夹心 ELISA检测结果如图 3所示,MAb 3G11能与 BTV-8发生阳性反应,而与其他 23种病毒不发生阳性反应。
2.3 抗原表位序列的筛选结果
将第4次筛选后的22个阳性克隆进行DNA测序并对短肽氨基酸序列进行比对,结果见表1,22个阳性克隆中共有18个展示的共同基因序列为KLLAT。
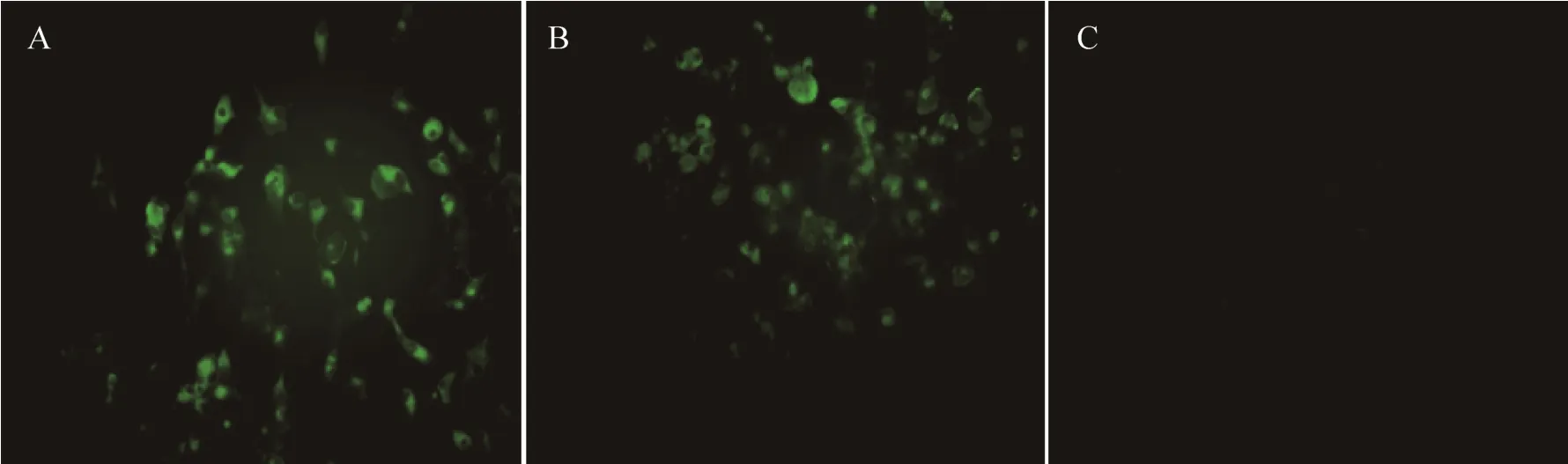
图2 单克隆抗体与BTV8的间接免疫荧光实验Fig. 2 IFA assay of MAb reaction with BTV8 infected cells. (A) IFA result of 3G11. (B) Positive control. (C) IFA result of SP2/0 supernatant and BHK-21 cells infected with BTV8.
2.4 4种合成短肽与MAb 3G11的反应性
用 4种合成短肽分别与单抗上清及腹水反应,结果如图4所示,短肽KLLAT和KLLAA与单抗上清及腹水均可发生特异性的反应,且S/N比值均大于2,表明该序列的短肽能与单抗上清和腹水中的抗体发生特异性结合,而KALAT、KLAAT短肽与上清及腹水的反应为弱阳性。可见283LL284为该表位序列的关键氨基酸。反应中腹水反应的值明显高于上清,与腹水中非特异性结合较多有关。当短肽包被量达到40 μg/mL时进入平台期,以其包被酶标板,将腹水分别进行 1×103、2×103、5×103、10×103、15×103、20×103、25×103、30×103倍的稀释后作为一抗,当稀释比例达到20×103倍后进入平台期。
图3 夹心ELISA鉴定MAb 3G11特异性Fig. 3 The specificity identification of MAb3G11 by AC-ELISA.
表1 噬菌体筛选序列测定结果Table 1 The sequencing results of phage display technology
2.5 短肽KLLAA和KLLAT与标准血清的反应性
合成短肽KLLAA和KLLAT与不同血清型BTV阳性血清的间接 ELISA反应结果基本一致,其中短肽KLLAA的反应结果如图5所示,可见短肽与 BTV-8、BTV-4、BTV-17、BTV-19标准阳性血清的反应呈较强阳性,短肽与BTV-1、BTV-3、BTV-7、BTV-10、BTV-12、BTV-13、BTV-15、BTV-20、BTV-21、BTV-23标准阳性血清的反应呈弱阳性。与BTV-8的反应值明显高于其他血清型 BTV。结果表明,283LL284序列为BTV-8的型异性抗原表位的关键氨基酸。与BTV-4、BTV-17、BTV-19有较强的阳性反应可能是由于短肽与标准阳性血清中的非特异性抗体结合导致。
2.6 表位序列的保守性分析
将短肽序列283LL284与GenBank中的10个BTV-8不同毒株及3个BTV-7不同毒株进行比对,结果如表2所示,13个毒株的第283和284位氨基酸处该序列高度保守;其他BTV1–24型VP2蛋白在该位置没有这两个氨基酸序列 (结果略)。
3 讨论
目前研究抗原 B细胞表位的常用方法有噬菌体展示技术和肽扫描技术[21-22]。噬菌体展示技术具有方法简便同时准确性更高等优点[23],因此本研究中应用噬菌体展示技术筛选 MAb 3G11识别的抗原表位。VP2蛋白为 BTV的型特异性抗原,可刺激机体产生中和抗体,不同血清型之间VP2蛋白的氨基酸序列差异较大。制备针对不同清血型VP2蛋白的单克隆抗体并建立血清型鉴定方法,可对流行BTV的血清型进行特异性检测[24-25]。
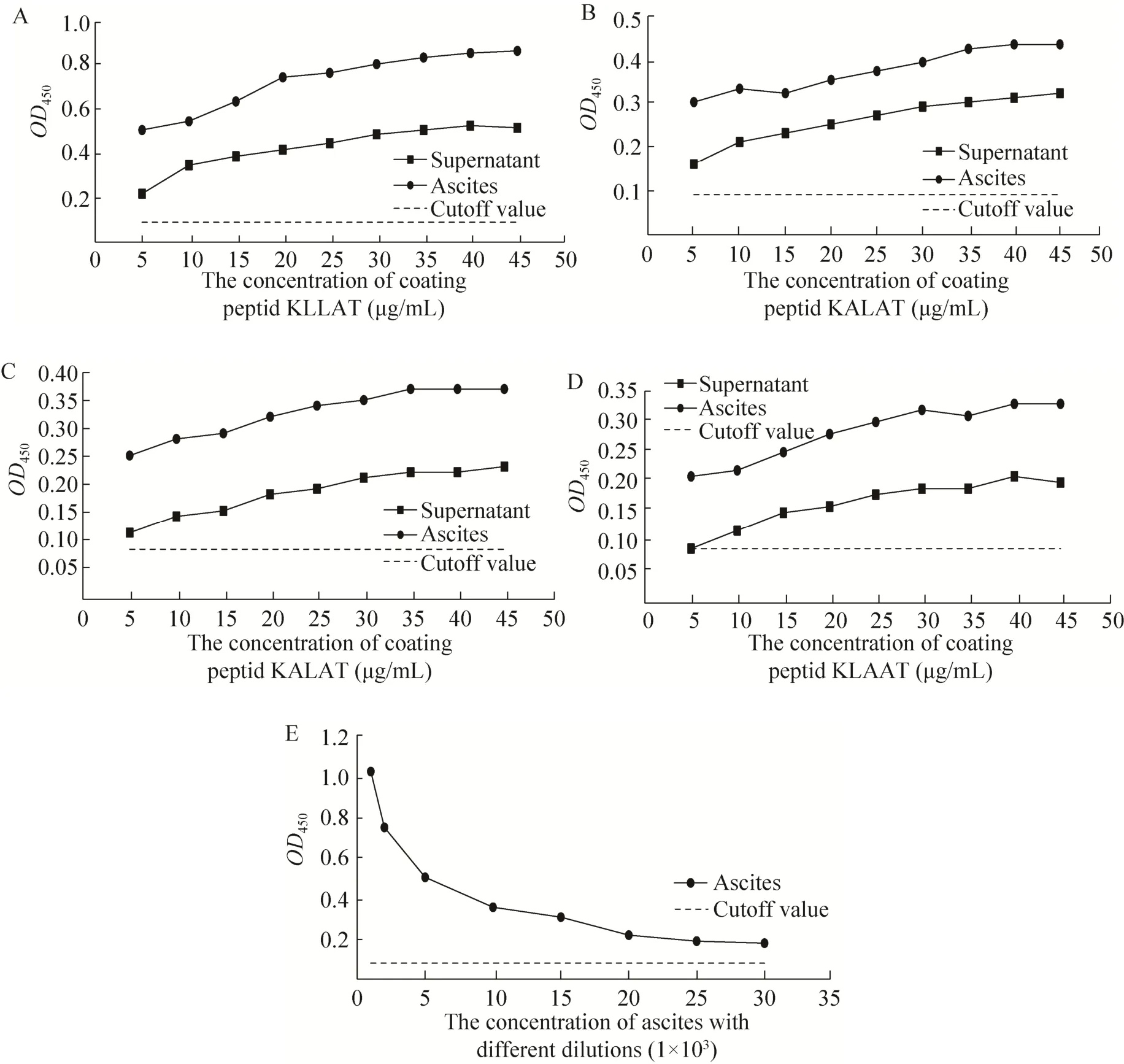
图4 4种短肽与MAb 3G11间接ELISA鉴定结果Fig. 4 Analysis of 4 short peptides reaction with the MAb3G11 by indirect ELISA. (A) Short peptide sequence KLLAT. (B) Short peptide sequence KLLAA. (C) Short peptide sequence KALAT. (D) Short peptide sequence KLAAT. (E) The concentration of ascites with different dilutions.
本研究以实验室制备的 BTV-8 VP2蛋白MAb 3G11经夹心ELISA检测能与BTV-8发生特异性反应,而不与 BTV1–24中其他 23个型病毒发生反应。应用噬菌体展示技术试剂盒筛选3G11所识别的抗原表位,实验中共进行了4轮淘选,其中第 3轮淘选的噬斑进行测序,结果中有3个样本为空样本,在肽库的结合区域没有任何氨基酸,这种结果可能由噬菌体展示肽库试剂盒有一定的局限性所导致。进行第4轮淘选后挑取阳性克隆的测序结果得到了 KLLAT序列,该序列的3个氨基酸LL和T与BTV-8 VP2的氨基酸残基283LLST286序列相符;席娜等[23]利用肽扫描技术鉴定 BTV-8 VP2的抗原表位为LCRLLSTIGRKMCNTE序列,该表位包含有283LL284序列,可见283LL284序列为BTV-8 VP2的抗原表位关键氨基酸;合成的短肽序列KLLAA、KALAT、KLAAT和KLLAT,与BTV-8标准阳性血清进行间接ELISA检测,可见含有LL序列的短肽与标准阳性血清结合能力较强,进一步验证283LL284序列为MAb 3G11识别的线性抗原表位的关键氨基酸。利用短肽分别与24种血清型的BTV标准阳性血清进行间接ELISA反应,结果可见,该短肽序列与BTV-8标准阳性血清有较强的结合能力,但与其他几种标准阳性血清的反应均呈阳性。这种结果可能由于合成短肽的序列过短,与其他标准阳性血清中抗体的非特异性结合较多所引起的[26]。同时不同标准阳性血清中抗体的浓度不同,进一步优化标准阳性血清的稀释比例可以降低短肽与血清中抗体的非特异性结合。
综上所述,本实验室制备针对 BTV-8的MAb及鉴定其抗原表位为BT的型特异性诊断方法的建立奠定了基础,为口岸检疫中新血清型检测方法的建立提供依据。
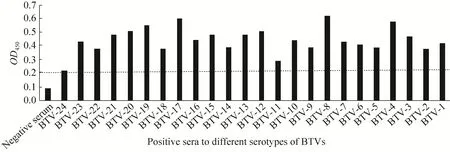
图5 短肽KLLAA与不同血清型BTV阳性血清间接ELISA鉴定结果Fig. 5 Analysis of the short peptide KLLAA’s reaction with positive sera against different BTV serotypes in indirect ELISA.

表2 短肽KLLAA与不同的BTV-7和BTV-8毒株氨基酸序列BLAST分析结果Table 2 BLAST analysis of the motif KLLAA with the VP2 amino acid sequences of BTV-7 and BTV-8 strains
REFERENCES:
[1] Wei T, Xu QY, Geng HW, et al. Identification of the group-specific conformational epitopein VP7 of bluetongue virus. Chin J PrevVet Med, 2014, 36(1):63–66 (in Chinese).魏天, 徐青元, 耿宏伟, 等. 蓝舌病病毒VP7蛋白竞争抑制群特异性构象抗原表位的初步鉴定.中国预防兽医学报, 2014, 36(1): 63–66.
[2] Tong L, Chai J, Zhang YF, et al. The gene product of the bluetongue virus and its biological function.China Anim Husb Vet Med, 2010, 37(7): 132–135(in Chinese).童亮, 柴俊, 张以芳, 等. 蓝舌病病毒基因产物及其生物学功能. 中国畜牧兽医, 2010, 37(7):132–135.
[3] Li N, Zhu JB, Xiao L, et al. Sequence analysis of bluetongue virus 1 M6 gene in Yunnan Province.China Anim Husb Vet Med, 2016, 43(2): 340–347(in Chinese).李楠, 朱建波, 肖雷, 等. 云南蓝舌病病毒 1型毒株 M6基因序列分析. 中国畜牧兽医, 2016,43(2): 340–347.
[4] Maan S, Maan NS, Belaganahalli MN, et al.Full-genome sequencing as a basis for molecular epidemiology studies of bluetongue virus in India.PLoS ONE, 2015, 10(6): e0131257.
[5] Schulz C, Bréard E, Sailleau C, et al. Bluetongue virus serotype 27: detection and characterization of two novel variants in Corsica, France. J General Virol, 2016, 97(9): 2073–2083.
[6] Yang H, Lü MN, Sun MF, et al. Complete genome sequence of the first bluetongue virus serotype 7 isolate from China: evidence for entry of Africanlineage strains and reassortment between the introduced and native strains. Arch Virol, 2016,161(1): 223–227.
[7] Sun J, Sun EC, Liu EZ, et al. Preparation of monoclonal antibody against VP5 of serotype 3 bluetongue virus and identification of the antigenicepitope. Chin J PrevVet Med, 2014, 36(4):387–390 (in Chinese).孙晶, 孙恩成, 刘二战, 等. 蓝舌病病毒 3型VP5蛋白单克隆抗体的制备及其抗原表位鉴定.中国预防兽医学报, 2014, 36(4): 387–390.
[8] Zhang YX. Virus neutralization test, insect vector investigation, isolation, identification and preliminary study on pathogenicity of bluetongue virus in Guangxi[D]. Nanning: Guangxi University,2016 (in Chinese).张怡轩. 广西BTV感染及传播媒介调查、病毒分离鉴定与致病性初步研究[D]. 南宁: 广西大学,2016.
[9] Franco Mahecha OL, Ogas Castells ML,Combessies G, et al. Single dilution avidity-blocking ELISA as an alternative to the bovine viral diarrhea virus neutralization test. J Virol Methods, 2011, 175(2): 228–235.
[10] Zhang SN, Li HC, Zhu JB, et al. The epidemiological survey and serotype identification of bluetongue disease and the epizootic hemorrhage disease in Inner Mongolia in 2015. Chin J Prev Vet Med, 2016, 38(12): 939–943 (in Chinese).张胜男,李华春,朱建波,等. 2015年内蒙古地区蓝舌病及流行性出血热流行病学调查及血清型鉴定. 中国预防兽医学报, 2016, 38(12):939–943.
[11] Yang H, Xiao L, Wang J, et al. Phylogenetic characterization genome segment 2 of bluetongue virus strains belonging to serotypes 5, 7 and 24 isolated for the first time in china during 2012 to 2014. Transbound Emerg Dis, 2017, 64(4):1317–1321.
[12] Balam D, Daggupati S, Maddireddy H. Studies of the antigenic relationships between bluetongue virus serotypes 2, 9 & 15 isolated in Andhra Pradesh, India. Vet World, 2011, 4(10): 444–448.
[13] Maan S, Maan NS, Nomikou K, et al. Novel bluetongue virus serotype from Kuwait[J]. Emerg Infect Dis, 2011, 17(5): 886–889.
[14] Lin HL, Ran DL, Wang W. Research progress on bluetongue disease. Anim Husbandr Feed Sci,2008, (1): 4–7 (in Chinese).林汉亮, 冉多良, 王文. 蓝舌病研究进展. 新疆畜牧业, 2008, (1): 4–7.
[15] Maan S, Maan NS, Batra K, et al. Reverse transcription loop-mediated isothermal amplification assays for rapid identification of eastern and western strains of bluetongue virus in India. J Virol Methods, 2016, 234: 65–74.
[16] Ayhan A, Velipasaoglu M, Salman MC, et al.Preparation and characterization of a monoclonal antibody against the core protein VP7 of the 25th serotype of bluetongue virus. Monoclon Antibod Immunodiag Immunother, 2015, 34(2): 116–121.
[17] Maan NS, Maan S, Belaganahalli MN, et al.Identification and differentiation of the twenty six bluetongue virus serotypes by RT-PCR amplification of the serotype-specific genome segment 2. PLoS ONE, 2012, 7(2): e32601.
[18] Zientara S, Sailleau C, Viarouge C, et al. Novel bluetongue virus in Goats, Corsica, France, 2014.Emerg Infect Dis, 2014, 20(12): 2123–2125.
[19] Matsuo E, Roy P. Minimum requirements for bluetongue virusprimary replication in vivo. J Virol, 2013, 87: 882–889.
[20] Mellor PS, Carpenter S, Harrup L, et al. Bluetongue in Europe and the Mediterranean Basin: history of occurrence prior to 2006. Prev Vet Med, 2008,87(1/2): 4–20.
[21] Zhang ZD, Cheng J, Zhang SL. Principle of phage display technique and its application. World Chin J Digestol, 2003, 11(4): 459–461 (in Chinese).张忠东, 成军, 张树林. 噬菌体展示技术的原理及应用. 世界华人消化杂志, 2003, 11(4): 459–461.
[22] Wang LF. Preparation of the monoclonal antibodies against VP2 protein of bluetongue virus serotype 17 and the VP2 B-cell epitopes identification[D].Beijing: Chinese Academy of Agricultural Sciences, 2011 (in Chinese).王凌凤. 蓝舌病病毒17型VP2蛋白单克隆抗体的制备及其抗原表位的鉴定[D]. 北京: 中国农业科学院, 2011.
[23] Xi N. Preparation of the monoclonal antibodies against VP2 protein of bluetongue virus serotype 8 and identification of B-cell epitopes[D].Changchun: Jilin Agricultural University, 2014 (in Chinese).席娜. 蓝舌病病毒 8型 VP2蛋白单克隆抗体的制备及 B细胞表位鉴定[D]. 长春: 吉林农业大学, 2014.
[24] Zhang Q, Sun EC, Xu QY, et al. Preparation of monoclonal antibody against VP2 of bluetongue virus serotype 15 and identification of B-cellepitope. Chin J Prev Vet Med, 2015, 37(5):392–396 (in Chinese).张沁, 孙恩成, 徐青元, 等. 蓝舌病病毒 15型VP2蛋白单克隆抗体的制备及其抗原表位鉴定.中国预防兽医学报, 2015, 37(5): 392–396.
[25] Geng HW, Qin YL, Li JP, et al. Development of monoclone competitive ELISA for detection of bluetongue virus antibodies. Chin J Prev Vet Med,2012, 34(5): 388–392 (in Chinese).耿宏伟, 秦永丽, 李俊平, 等. 蓝舌病病毒重组VP7蛋白单克隆抗体制备及竞争ELISA检测方法的建立. 中国预防兽医学报, 2012, 34(5):388–392.
[26] Liu ST. Comparative study of four techniques for the detection ofbluetongue virus[D]. Yangzhou:Yangzhou University, 2010 (in Chinese).刘松婷. 蓝舌病病毒四种检测技术比较研究[D].扬州: 扬州大学, 2010.
(本文责编 郝丽芳)
Identification of epitope recognized by a monoclonal antibody against VP2 protein of bluetongue virus serotype 8
Mingxin Zang, Jiaxuan Li, Shuangyu Xie, Wen Cui, Yanping Jiang, Yigang Xu,Xinyuan Qiao, Li Wang, Han Zhou, Min Liu, Yijing Li, and Lijie Tang
College of Veterinary Medicine, Northeast Agricultural University, Harbin 150030, Heilongjiang, China
To confirm the B cell epitope recognized by monoclonal antibody (MAb) 3G11 of bluetongue virus type 8(BTV-8) VP2 protein prepared in our laboratory, antigen epitopes recognized by 3G11 were screened and identified by phage display technology. KLLAT sequence was found by sequencing of blue spot after four rounds panning and283LL284of common short peptide sequence was obtained after comparison to amino acid sequence of BTV-8 VP2 protein. The peptide sequences KLLAA, KALAT, KLAAT and KLLAT were synthesized and identified by indirect ELISA. KLLAA and KLLAT bound strongly with supernatant and as cites of 3G11 cells and reacted specifically with BTV-8 positive standard sera. Further sequence analysis showed that amino acid sequence283LL284was conserved among different serotypes of BTV-8 strains, and283LL284was the key amino acids of antigen epitopes recognized by 3G11. This study laid the foundation to establish type 8 BTV specific immunological detection methods.
bluetongue virus serotype 8, VP2 protein, monoclonal antibody, antigen epitope
March 26, 2017; Accepted: July 28, 2017
Lijie Tang. Tel: +86-451-55190363; E-mail: tanglijie@163.com“十二五”国家科技支撑计划 (No. 2013BAD12B01) 资助。
Supported by: National Key Technology R&D Program of China (No. 2013BAD12B01).

