靶向沉默SSBP1基因对肝癌HepG2细胞增殖、侵袭转移的影响
马荣芬 陈秋英 刘艳芬
·论著·
靶向沉默SSBP1基因对肝癌HepG2细胞增殖、侵袭转移的影响
马荣芬 陈秋英 刘艳芬
目的 观察靶向沉默线粒体单链DNA结合蛋白(SSBP1)基因对肝癌HepG2细胞增殖、侵袭转移的影响。方法 设计并构建靶向SSBP1 基因的特异性siRNA,采用脂质体介导瞬时转染肝癌HepG2细胞,细胞分3组:对照组、空白转染组、转染组。Real time-PCR和Western-blot检测靶向干扰后SSBP1 mRNA和蛋白表达变化。CCK-8法检测细胞增殖。流式细胞仪检测细胞周期、凋亡率及线粒体膜电位。划痕实验及Transwell 侵袭实验检测细胞侵袭转移能力。Western-blot检测增殖、侵袭转移相关基因蛋白表达状况。结果 SSBP1 siRNA能够显著抑制SSBP1 mRNA和蛋白表达。与对照组和空白转染组比较,转染SSBP1 siRNA后HepG2细胞增殖能力、G2期和S期细胞比例、线粒体膜电位明显降低,G1期细胞比例、细胞凋亡率明显升高(P<0.05);同时细胞增殖相关基因PCNA、凋亡抑制基因Bcl-2、转移相关基因MMP-9蛋白表达显著下调,凋亡诱导基因Bax蛋白表达显著上调(P<0.05)。结论 靶向沉默SSBP1基因能够通过线粒体途径抑制肝癌HepG2细胞增殖、侵袭转移,并诱导其凋亡。
肝癌;线粒体单链DNA结合蛋白;细胞增殖;侵袭转移
近年来,我国肝癌发病率和病死率逐年增加,且呈年轻化趋势,肝癌发病是多因素、多步骤的复杂过程[1,2]。肝癌手术切除后易复发,且对放化疗不敏感,患者预后差[3]。肝癌细胞增殖活跃、凋亡减缓及侵袭转移是导致治疗失败、患者病死率高的主要原因[4-6]。因此,阐明肝癌发生发展的机制,研究抑制肿瘤细胞增殖、侵袭转移并诱导其凋亡的基因已经成为治疗恶性肿瘤的有效措施[7]。线粒体单链DNA结合蛋白(SSBP1)在真核生物进化过程中相对保守,主要通过参与线粒体DNA(mtDNA)的复制、转录、损伤后修复等作用与线粒体功能密切相关[8]。有研究发现,SSBP1在乳腺癌、卵巢癌、结直肠癌等多种恶性肿瘤组织表达上调[9-12]。但是,目前有关SSBP1是否参与调控肝癌细胞生物学行为的研究笔者尚未见报道。本研究通过RNA干扰技术探讨靶向沉默SSBP1基因对肝癌HepG2细胞增殖、侵袭转移的影响,旨在为寻找防治肝癌的有效靶点提供理论依据。
1 材料与方法
1.1 细胞、试剂与仪器 人肝癌细胞HepG2购自上海吉凯基因化学技术有限公司;RPMI 1640培养基、SSBP1 siRNA、LipofectamineTM 2000购自美国Invitrogen公司;Trizol、实时荧光定量试剂盒购自美国Promega公司;兔抗人PCNA、Bcl-2、Bax、MMP-9多克隆抗体购自美国Santa Cruz公司;细胞裂解液、辣根过氧化物酶标记的抗鼠IgG抗体、BCA蛋白浓度测定试剂盒、高灵敏度化学发光检测试剂盒购自日本Takara公司;Chemi DocTM XRS+化学发光凝胶成像系统购自美国Biorad公司;全自动7300型荧光定量PCR扩增仪购自美国ABI公司;FACS Calibur型流式细胞仪购自美国BD公司。
1.2 方法
1.2.1 细胞培养及转染:人肝癌细胞HepG2用含10% FBS、1%青/链霉素的DMEM培养基在37℃、5% CO2、饱和湿度条件下常规培养。待细胞达到90%融合时利用脂质体LipofectamineTM 2000试剂盒进行转染,具体操作步骤严格按照试剂盒说明书进行。细胞随机分为3组:对照组(不进行转染处理)、空白转染组(转染空质粒)、转染组(转染siRNA SSBP1)。
1.2.2 Real time-PCR:Trizol提取细胞总RNA,按照试剂盒说明书加样进行逆转录反应,ABI 7300型荧光定量PCR仪扩增。用仪器自带的分析软件得到各样本、各基因扩增的Ct值,以β-actin为内参照基因,目的基因表达相对水平RQ=2-ΔΔCt。
1.2.3 Western-blot:细胞裂解后提取总蛋白,BCA法测定蛋白浓度。取30 μg 总蛋白上样,SDS-PAGE分离后半干法电泳转移至PVDF膜,10%脱脂奶粉封闭2 h。加入兔抗人PCNA、Bcl-2、Bax、MMP-9多克隆抗体,4℃过夜,TBST洗3次。加入辣根过氧化物酶标记的羊抗鼠IgG,室温孵育2 h,化学发光法显色、定影。β-actin作为内参照,以目的蛋白与β-actin吸光度值的比值表示目的蛋白相对表达水平。
1.2.4 CCK-8实验:各组细胞接种于96孔板中,调整细胞接种为5×103个/孔。培养1 d、2 d、3 d、4 d后每孔加入10 μl CCK-8溶液,继续培养2 h,于酶标仪450 nm 波长处测定吸光度(A450)值,重复3次。
1.2.5 Annexin V-FITC/PI双染法检测细胞周期和凋亡率:选取对数生长期的细胞接种于6孔板,胰酶消化细胞,PBS洗2次,重悬细胞,调整细胞密度为1×109/L,加入FITC的Annexin V和PI染液,各10 μl混匀,室温下避光反应15 min,流式细胞仪检测细胞周期和凋亡率。
1.2.6 划痕实验:用10 μl枪头在6孔板底部做一划痕,加入无血清DMEM培养基培养24 h,测量划痕长度,计算细胞迁移抑制率,取均值。
1.2.7 Transwell 侵袭实验:胰酶消化细胞,调整细胞密度为1×105/L。Transwell 上室加入400 μl细胞悬液,下室加入600 μl 10%FBS的DMEM培养基,培养 24 h。 结晶紫染色30 min,光学显微镜拍照,随机选取5个视野,计数迁移至滤膜下表面的细胞数,即代表HepG2细胞侵袭力。
1.2.8 线粒体膜电位检测:选取对数生长期的细胞接种于6孔板,调整细胞密度为1×105个/孔,加入 0.5 ml JC-1染液,避光孵育20 min,PBS洗2次,流式细胞仪检测细胞线粒体膜电位。
2 结果
2.1 siRNA干扰后SSBP1在HepG2细胞的表达 Real time-PCR和Western blot结果显示,HepG2细胞转染SSBP1 siRNA后SSBP1 mRNA和蛋白表达显著低于对照组、空白转染组(P<0.05);对照组、空白转染组比较差异无统计学意义(P>0.05)。见表1。
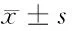

组别SSBP1表达水平mRNA蛋白SSBP1siRNA组0.33±0.050.29±0.04空白转染组2.11±0.27*1.99±0.25*对照组2.16±0.30*2.05±0.24*
注:与SSBP1 siRNA组比较,*P<0.05
2.2 SSBP1 siRNA对HepG2细胞增殖的影响 CCK-8实验结果显示,HepG2细胞转染SSBP1 siRNA后细胞A450值显著低于对照组、空白转染组,差异有统计学意义(P<0.05);对照组、空白转染组比较差异无统计学意义(P>0.05)。见表2。


组别A4501d2d3d4dSSBP1siRNA组0.29±0.040.34±0.030.49±0.070.60±0.09空白转染组0.30±0.050.41±0.05*0.69±0.09*0.93±0.11*对照组0.28±0.040.42±0.06*0.71±0.10*0.95±0.12*
注:与SSBP1 siRNA组比较,*P<0.05
2.3 SSBP1 siRNA对HepG2细胞周期的影响 流式细胞术结果显示,HepG2细胞转染SSBP1 siRNA后G1期细胞比例显著高于对照组、空白转染组,G2期、S期细胞比例显著低于对照组、空白转染组,差异有统计学意义(P<0.05);对照组、空白转染组比较差异无统计学意义(P>0.05)。见表3。


组别G1期G2期S期SSBP1siRNA组85.34±9.678.13±0.699.39±1.10空白转染组66.79±6.32*12.99±1.35*13.88±1.46*对照组67.12±6.34*13.65±1.46*14.24±1.62*
注:与SSBP1 siRNA组比较,*P<0.05
2.4 SSBP1 siRNA对HepG2细胞凋亡的影响 流式细胞术结果显示,HepG2细胞转染SSBP1 siRNA后细胞凋亡率显著高于对照组、空白转染组,差异有统计学意义(P<0.05);对照组、空白转染组比较差异无统计学意义(P>0.05)。见表4。
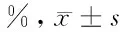

组别凋亡率1d2d3d4dSSBP1siRNA组2.03±0.134.90±0.637.02±0.119.26±0.12空白转染组2.01±0.123.92±0.43*4.96±0.68*6.02±0.08*对照组2.04±0.153.89±0.40*4.95±0.71*6.00±0.09*
注:与SSBP1 siRNA组比较,*P<0.05
2.5 SSBP1 siRNA对HepG2细胞迁移的影响 划痕实验结果显示,HepG2细胞转染SSBP1 siRNA后细胞划痕迁移抑制率显著高于对照组、空白转染组,差异有统计学意义(P<0.05);对照组、空白转染组比较差异无统计学意义(P>0.05)。见表5。


组别迁移抑制率SSBP1siRNA组35.06±4.14空白转染组12.21±1.11*对照组11.72±1.02*
注:与SSBP1 siRNA组比较,*P<0.05
2.6 SSBP1 siRNA对HepG2细胞侵袭力的影响 划痕实验结果显示,HepG2细胞转染SSBP1 siRNA后细胞划痕迁移抑制率显著高于对照组、空白转染组,差异有统计学意义(P<0.05);对照组、空白转染组比较差异无统计学意义(P>0.05)。见表6。
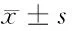
表6 SSBP1沉默对HepG2细胞侵袭力的影响 个,
注:与SSBP1 siRNA组比较,*P<0.05
2.7 SSBP1 siRNA对HepG2细胞线粒体膜电位的影响 流式细胞术检测结果显示,HepG2细胞转染SSBP1 siRNA后细胞线粒体膜电位显著低于对照组和空白转染组,差异均有统计学意义(P<0.05);对照组和空白转染组之间比较差异无统计学意义(P>0.05)。见表7。

组别MMP(FI)SSBP1siRNA组40.12±5.02空白转染组93.97±10.24*对照组95.56±8.53*
注:与SSBP1 siRNA组比较,*P<0.05
2.8 SSBP1 siRNA对HepG2细胞增殖、侵袭转移相关基因蛋白表达的影响 Western blot结果显示,HepG2细胞转染SSBP1 siRNA后细胞增殖相关基因PCNA、凋亡抑制基因Bcl-2蛋白表达显著低于对照组、空白转染组,凋亡诱导基因Bax蛋白表达显著高于对照组、空白转染组,差异有统计学意义(P<0.05);对照组、空白转染组比较差异无统计学意义(P>0.05)。见表8。
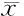
表8 SSBP1沉默对HepG2细胞增殖、侵袭转移相关基因蛋白表达的影响 ±s
注:与SSBP1siRNA组比较,*P<0.05
3 讨论
研究发现,线粒体基因突变和功能障碍是恶性肿瘤发生发展的重要因素[13]。而单链DNA结合蛋白(SSBP1)是调控线粒体生成和功能的关键因子,主要参与线粒体DNA(mtDNA)的复制、转录、损伤后修复过程[14-16]。SSBP1表达失调与mtDNA复制速度、稳定性及线粒体功能障碍密切相关。有研究证实,乳腺癌、大肠癌、卵巢癌、骨肉瘤、肾癌等恶性肿瘤组织异常高表达SSBP1,SSBP1可能作为潜在的肿瘤标志物,这对于肿瘤诊断、疗效及预后评估具有重要临床价值[17,18]。
研究发现,线粒体途径是诱导细胞凋亡的主要途径。Wong等[19]研究表明SSBP1基因通过影响线粒体凋亡途径参与恶性肿瘤的发生发展。已知多种蛋白参与和调控了线粒体凋亡过程,其中Bcl-2家族蛋白发挥了重要作用。Bax是主要的促凋亡蛋白,活化后从细胞核转位进入线粒体,从而介导线粒体凋亡;而Bcl-2是主要的抗凋亡蛋白,能够抑制caspase-3活化发挥抗凋亡作用[20-22]。PCNA是细胞增殖过程中特异性表达的一种核蛋白,可作为反映细胞增殖活性的敏感指标[23]。MMP-9属于MMPs家族,其水平越高表示肿瘤侵袭转移能力越强[24]。
为探讨SSBP1参与肝癌发生发展的具体机制,本研究观察靶向沉默SSBP1对肝癌HepG2细胞增殖、侵袭转移的影响。结果表明,SSBP1 siRNA转染后HepG2细胞SSBP1 mRNA和蛋白表达显著降低,表明利用siRNA干扰SSBP1基因成功,SSBP1表达被显著抑制。与对照组和空白转染组比较,转染SSBP1 siRNA后HepG2细胞增殖能力、G2期和S期细胞比例、线粒体膜电位明显降低,G1期细胞比例、细胞凋亡率明显升高(P<0.05);同时细胞增殖相关基因PCNA、凋亡抑制基因Bcl-2、转移相关基因MMP-9蛋白表达显著下调,凋亡诱导基因Bax蛋白表达显著上调(P<0.05),提示SSBP1基因参与肝癌的发生发展,下调SSBP1表达能够有效调控肝癌细胞周期、增殖及侵袭转移过程。
综上所述,本实验表明下调SSBP1表达能够明显抑制肝癌细胞增殖、侵袭转移,降低线粒体膜电位,并诱导肝癌细胞凋亡。本实验首次证实肝癌细胞过表达SSBP1基因与细胞增殖、侵袭转移有关。因此,SSBP1有可能成为防治肝癌的潜在靶点,利用特异性拮抗剂或siRNA造成SSBP1基因沉默有可能成为治疗肝癌的有效措施。本研究为肝癌的靶向治疗提供了新思路和新策略。但是SSBP1调控肝癌细胞增殖、侵袭转移的具体分子机制仍需进一步研究。
1 Fitzmaurice C,Dicker D.Global Burden of Disease Cancer Collaboration,The global burden of cancer 2013.JAMA Oncol,2015,1:505-527.
2 Jemal A,Bray F,Center MM,et al.Global cancer statistics.CA Cancer J Clin,2011,61:69-90.
3 Uchiyama H,Minagawa R,Itoh S,et al.Favorable outcomes of hepatectomy for ruptured hepatocellular carcinoma:retrospective analysis of primary r0-hepatectomized patients.Anticancer Res,2016,36:379-385.
4 Yeh JH,Hung CH,Wang JH,et al.Modifiable prognostic factors of hepatocellular carcinoma in patients with non-surgical treatment.PLoS One,2015,147:2851-2853.
5 Tan ZM,Sun BC.Effects of antiviral therapy on preventing liver tumorigenesis and hepatocellular carcinoma recurrence.World Journal of Gastroenterology,2013,19:8895-8901.
6 葛益谋.金属蛋白酶-9、血管内皮生长因子在原发性肝癌外周血液中变化的临床意义.广西医科大学学报,2012,21:846-848.
7 Ba Q,Hao M,Huang H,Hou J,et al.Iron deprivation suppresses hepatocellular carcinoma growth in experimental studies.Clin Cancer Res,2011,17:7625-7633.
8 McKinney EA,Oliveira MT.Replicating animal mitochondrial DNA.Genet Mol Biol,2013,36:308-315.
9 Ye Y,Huang A,Huang C,et al.Comparative mitochondrial proteomic analysis of hepatocellular carcinoma from patients.Proteom Clin Appl,2013,7:403-415.
10 Yu J,Liang QY,Wang J,et al.Zinc-finger protein 331,a novel putative tumor suppressor,suppresses growth and invasiveness of gastric cancer.Oncogene,2013,32:307-317.
11 Jiang HL,Sun HF,Gao SP,et al.SSBP1 Suppresses TGF-β-Driven Epithelial-to-Mesenchymal Transition and Metastasis in Triple- Negative Breast Cancer by Regulating Mitochondrial Retrograde Signaling.Cancer Res,2016,76:952-964.
12 Batra R,Harder N,Gogolin S,et al.Time-lapse imaging of neuroblastoma cells to determine cell fate upon gene knockdown.PloS one,2012,7:50988.
13 van Loon B,Samson LD.Alkyladenine DNA glycosylase (AAG) localizes to mitochondria and interacts with mitochondrial singlestranded binding protein ( mtSSB).DNA Repair,2013,12:177-187.
14 Douglas C.Wallace.Mitochondria and cancer.Nat Rev Cancer,2012,12:685-698.
15 Ruhanen H,Borrie S,Szabadkai G,et al.Mitochondrial singlestranded DNA binding protein is required for maintenance of mitochondrial DNA and 7S DNA but is not required for mitochondrial nucleoid organisation.Biochim Biophys Acta,2010,1803:931-939.
16 龙盘,常淞.mtSSBP1 与相关性疾病的研究进展.医学综述,2015,21:3867-3870.
17 Malik AN,Czajka A.Is mitochondrial DNA content a potential biomarker of mitochondrial dysfunction?Mitochondrion,2013,13:481-492.
18 Marvin Edeas,Volkmar Weissig.Targeting mitochondria:Strategies,innovations and challenges The future of medicine will come through mitochondria.Mitochondrion,2013,13:389-390.
19 Wong TS,Rajagopalan S,Townsley FM,et al.Physical and functional interactions between human mitochondrial single-stranded DNA-binding protein and tumour suppressor p53.Nucleic Acids Res,2009,37:568-581.
20 Hüttemann M,Pecina P,Rainbolt M,et al.The multiple functions of cytochrome c and their regulation in life and death decisions of the mammalian cell:from respiration to apoptosis.Mitochondrion,2011,11:369-381.
21 Westphal D,Dewson G,Czabotar PE,et al.Molecular biology of Bax and Bak activation and action.Biochim Biophys Acta,2011,1813:521-31.
22 Czabotar PE,Lessene G,Strasser A,et al.Control of apoptosis by the BCL-2 protein family:implications for physiology and therapy.Nat Rev Mol Cell Biol,2014,15:49-63.
23 姚合梅,王政,崔瑾,等.苗药验方皮部熏洗对骨关节炎大鼠软骨细胞凋亡与增殖的影响.浙江中医药大学学报,2013,27:89-896.
24 黄竹英,李松,汪平帮,等.VEGF与MMP-9在原发性肝癌中的表达及意义.国际检验医学杂志,2013,31:1353-1355.
Effects of RNAi-mediated SSBP1 gene silencing on proliferation,invasion and metastasis of human hepatocellular carcinoma HepG2 cells in vitro
MARongfen,CHENQiuying,LIUYanfen.
DepartmentofClinicalLaboratory,People’sHospitalofTanshanCity,Hebei,Tangshan063001,China
Objective To investigate the effects of RNAi-mediated SSBP1 gene silencing on proliferation,invasion and metastasis of human hepatocellular carcinoma HepG2 cells in vitro.Methods The high effective targeting SSBP1 siRNA was obtained by construction and screening,which was transfected into HepG2 cells by liposomes mediated transient transfection.The cells were divided into 3 groups: negative control group,blank transfection group (blank control group) and transfection group.The expression levels of SSBP1 mRNA and protein were detected by Real time-PCR and Western-Blot after the transfection.CCK-8 method was used to detect cell proliferation.Flow cytometry was used to detect the cell cycle,cell apoptosis and mitochondrial trans-membrane potential. The scuffing test and Transwell invasion assay were used to detect invasion and metastasis ability of cells. Moreover the expressions of gene and protein related with cell proliferation,invasion and metastasis were detected by Western Blot.Results The SSBP1 siRNA could obviously inhibit the expressions of SSBP1 mRNA and protein.As compared with negative control group and blank control group,after HepG2 cells were transfected by SSBP1 siRNA,the cell proliferation ability, cell proportion at phase G2and phase S, mitochondrial trans-membrane potential were obviously decreased,however, the cell proportion at phase G1and cell apoptosis rate were significantly increased (P<0.05). Meanwhile the expression levels of PCNA,Bcl-2 and MMP-9 proteins were obviously down-regulated,however, the expression levels of Bax proteins were significantly up-regulated (P<0.05).Conclusion The targeting silencing of SSBP1 gene can inhibit proliferation,invasion, metastasis of HepG2 cells,and can induce cell apoptosis via mitochondria pathway.
hepatocellular carcinoma; mitochondria single chain -DNA-binding protein;cell proliferation; invasion and metastasis
10.3969/j.issn.1002-7386.2017.17.004
063001 河北省唐山市人民医院检验科
R 735.7
A
1002-7386(2017)17-2578-04
2017-03-19)

