肠道内、外感染气单胞菌的菌种分布特征及毒力基因分析
李淑妃,陈晓,2,陈瑜,2(.浙江大学医学院附属第一医院检验科,杭州 30003;2.浙江大学传染病诊治国家重点实验室协同创新中心,杭州 30003)
·临床实验研究·
肠道内、外感染气单胞菌的菌种分布特征及毒力基因分析
李淑妃1,陈晓1,2,陈瑜1,2(1.浙江大学医学院附属第一医院检验科,杭州 310003;2.浙江大学传染病诊治国家重点实验室协同创新中心,杭州 310003)
目的 了解肠道内、外分离气单胞菌菌种分布及毒力基因谱的差异,探究该菌致病性与感染部位的关系。方法 收集2013年5月至2015年9月急性腹泻患者及肠道外标本来源的气单胞菌156株,采用PCR方法检测其18种毒力基因hlyA、aerA、act、alt、ast、aexT、ascV、aopP、ascF-G、gcat、tapA、fla、Ser、exu、ahyB、eprCAI、lip、laf,并统计分析肠道内、外来源气单胞菌毒力基因谱的差异。结果 156株气单胞菌中肠道内气单胞菌79株,以豚鼠气单胞菌为主(51.9%);肠道外气单胞菌77株,主要为嗜水气单胞菌(48.1%)和豚鼠气单胞菌(39.0%)。gcat、act、fla、ahyB、exu、lip基因在肠道内、外气单胞菌中的检出率较高(>45.57%),而aexT、aopP、ascF-G、ascV则较低(<20.78%);gcat、ahyB、laf、ast、exu、lip、hlyA、aerA在肠道内气单胞菌中的检出率显著低于肠道外(P<0.05);嗜水气单胞菌gcat、ahyB、exu、lip、eprCAI、hlyA在肠道外的检出率显著高于肠道内(P<0.05);豚鼠气单胞菌lip、hlyA在肠道外的检出率显著高于肠道内(P<0.05),而aopP反之;维氏气单胞菌毒力基因在肠道内、外的检出率差异无统计学意义。结论 肠道内、外气单胞菌感染的菌种分布及携带的毒力基因谱存在差异,临床需区别对待。
气单胞菌;毒力基因;肠道内;肠道外
气单胞菌(Aeromonas)作为一种人畜共患病原菌,其感染类型在临床上可分为肠道内和肠道外感染,前者主要表现为腹泻等胃肠炎症状,后者主要发生于肝硬化及恶性肿瘤等免疫功能低下患者,以血流感染和皮肤及软组织感染为主,预后差,病死率高[1]。气单胞菌致病涉及由不同基因编码的多种毒力因子,包括Ⅲ型分泌系统(aopP、aexT、ascF-G和ascV等基因)、外毒素(act、ast、alt、hlyA和aerA等基因)和与侵袭力相关的黏附因子(tapA基因)、胞外酶(gcat、ahyB、exu、lip、eprCAI、Ser等基因)、鞭毛(fla和laf基因)等[1]。然而,目前国内有关肠道内、外来源气单胞菌的菌种分布及毒力特征的研究少有报道。为更好地了解该菌的致病能力,现就临床分离肠道内、外来源气单胞菌的菌种分布及毒力基因谱特征进行分析,结果如下。
1 材料与方法
1.1 菌株来源 收集2013年5月至2015年9月浙江大学医学院附属第一医院门诊及急诊急性腹泻(急性腹泻:每日排便≥3次,且大便性状有改变:呈稀便、水样便、黏脓便或脓血便等,持续时间≤14 d[2-3])患者粪便来源的气单胞菌;收集同期住院患者血液、胆汁、尿液和痰液等(剔除同一患者重复菌株)来源的气单胞菌,共156株。采用Vitek 2 Compact微生物鉴定仪进行细菌鉴定,对于仪器不能区分的嗜水气单胞菌和豚鼠气单胞菌辅以VP生化试验鉴定(阴性判读为嗜水气单胞菌)。
1.2 主要试剂及仪器 各种微生物培养基(杭州天和公司);Vitek 2 Compact微生物鉴定仪及配套鉴定板条(法国生物梅里埃公司);DNA Engine PCR扩增仪、Mini-Protean 3水平电泳仪、Gel Doc XR+凝胶成像系统(美国Bio-Rad公司);引物由上海生工公司合成;Taq DNA聚合酶、100 bp DNA marker(TaKaRa公司)。
1.3 毒力基因测定 用热裂解法提取菌株基因组DNA。用PCR方法检测菌株18种毒力基因,参照文献[4-9]合成引物,见表1,引物由上海生工公司合成。PCR反应体系50 μL,包括10×PCR buffer(含Mg2+)5.0 μL,dNTPs(2.5 mmol/L)5 μL,Taq DNA聚合酶(5 U/μL)0.3 μL,上、下游引物(10 μmol/L)各1.0 μL,模板DNA 3.0 μL,ddH2O 34.7 μL。PCR反应参数:94 ℃ 5 min;94 ℃ 30 s,退火温度(见表1)30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。扩增产物经15 g/L琼脂糖凝胶电泳,紫外灯下观察结果。
1.4 测序验证 各毒力基因随机选取1株PCR阳性产物送上海生工公司进行基因测序,测序结果经美国国家生物技术信息中心/局部序列比对基本检索工具(National Center for Biotechnology Information/Basic Local Alignment Search Tool,NCBI/BLAST)比对确认。

表1 气单胞菌毒力基因PCR检测相关引物
1.5 统计学分析 用SPSS 20.0软件进行。率的组间比较采用χ2检验、连续性校正的χ2检验或Fisher检验。以P<0.05为差异有统计学意义。
2 结果
2.1 菌种分布 共检出气单胞菌156株,包括豚鼠气单胞菌71株、嗜水气单胞菌57株、维氏气单胞菌28株。其中肠道气单胞菌79株,以豚鼠气单胞菌(41株,51.9%)为主,其次为嗜水气单胞菌(20株,25.3%)和维氏气单胞菌(18株,22.8%);肠道外气单胞菌77株,包括嗜水气单胞菌(37株,48.1%)、豚鼠气单胞菌(30株,39.0%)和维氏气单胞菌(10株,13.0%)。
2.2 肠道内、外气单胞菌的毒力基因检出结果 经PCR及测序验证,除tapA仅在肠道外气单胞菌中检出,其他基因在肠道内、外气单胞菌中均有不同程度的检出率,见表2。gcat、act、fla、ahyB、exu、lip在肠道内、肠道外气单胞菌中的检出率较高(>45.57%);aexT、aopP、ascF-G、ascV在肠道内、肠道外气单胞菌中检出率较低(<20.78%)。gcat、ahyB、laf、ast、exu、lip、hlyA、aerA在肠道内气单胞菌中的检出率显著低于肠道外气单胞菌(P<0.05)。
2.3 肠道内、外不同气单胞菌菌种的毒力基因检出结果tapA基因仅在肠道外分离的3种气单胞菌中检出,见表3。嗜水气单胞菌gcat、ahyB、exu、lip、eprCAI、hlyA在肠道外的检出率显著高于肠道内(P<0.05),而ascV在肠道内嗜水气单胞菌中未检出;豚鼠气单胞菌lip、hlyA在肠道外的检出率显著高于肠道内(P<0.05),而aopP反之;维氏气单胞菌毒力基因在肠道内、外的检出率差异无统计学意义,而ahyB未在肠道外检测到。

表2 肠道内、外气单胞菌的毒力基因检出结果比较
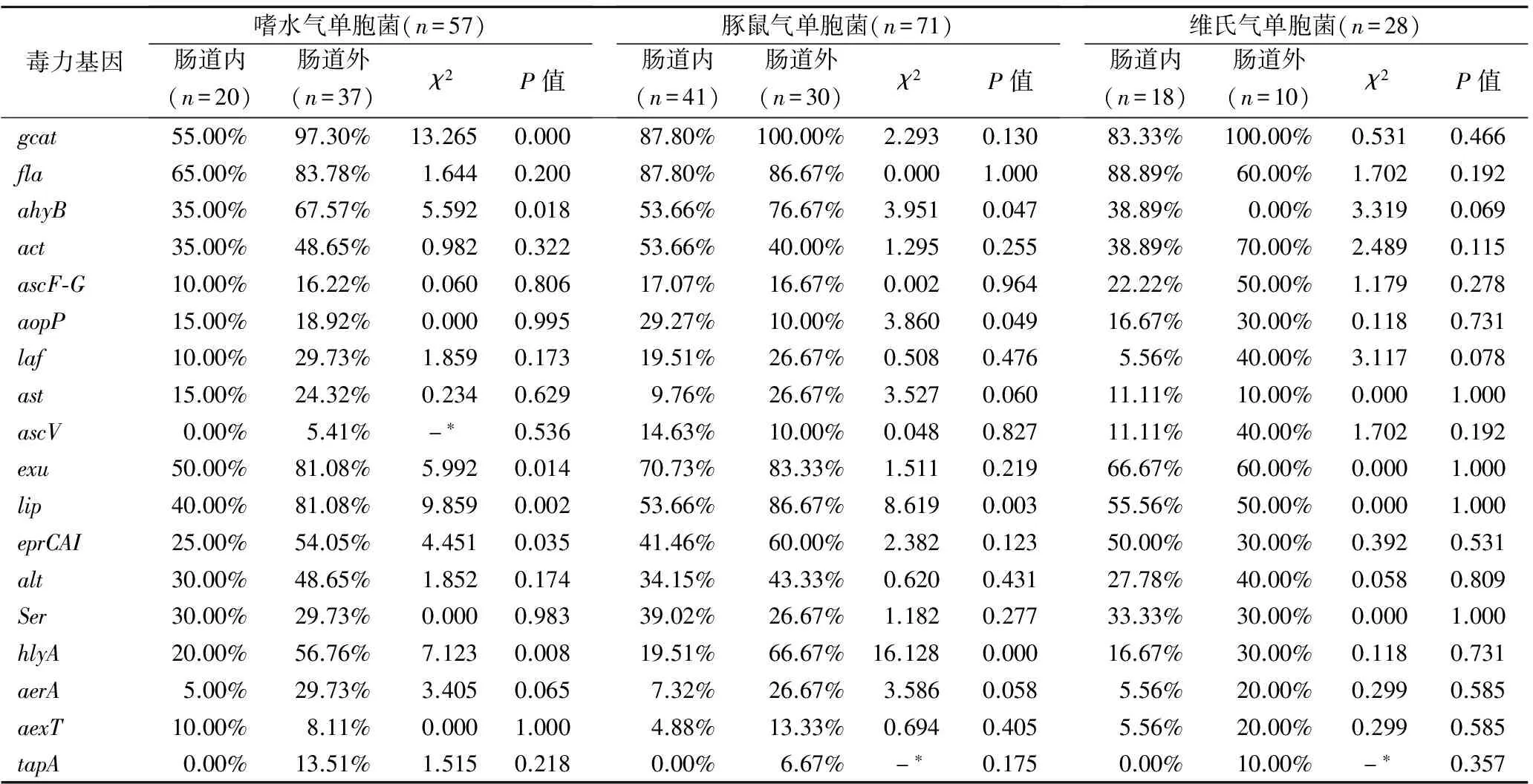
表3 嗜水、豚鼠、维氏气单胞菌毒力基因在肠道内、外的检出率比较
注:*表示采用Fisher精确检验。
3 讨论
本研究发现,本地区肠道气单胞菌属感染以豚鼠气单胞菌为主,然而在巴西、中国香港和巴基斯坦腹泻患者中则分别以嗜水气单胞菌、温和气单胞菌和脆弱气单胞菌及中间气单胞菌最为常见[10-12];而肠道外气单胞菌感染以嗜水气单胞菌为主,与北京地区气单胞菌属的流行状况相近[13]。这些差异的存在可能是与地域环境尤其是水源的差异和病原鉴定方法的不同有关。
毒力基因检测发现,绝大多数(96.79%)的气单胞菌至少携带一种毒力基因。Santos等[6]发现产溶血毒素的气单胞菌具有致病潜能。本研究发现>90%的肠道内及肠道外气单胞菌均具有溶血活性,而毒力基因检测结果显示与溶细胞作用相关的act、hlyA、aerA、gcat、lip基因在肠道内、外气单胞菌中均有不同程度的检出。act是一种细胞毒性肠毒素基因,介导溶血性及细胞毒性,诱导上皮细胞和巨噬细胞凋亡[1]。Wong等[7]研究表明hlyA和aerA可通过在细胞表面形成跨膜孔的方式介导溶血活性。携带gcat的气单胞菌具有脂肪酶或磷脂酶活性,可与lip通过消化质膜的方式溶解红细胞[14]。
gcat、ahyB、exu、lip、hlyA、tapA在肠道内、外的分布差异具有统计学意义。具体表现为嗜水气单胞菌的gcat、ahyB、exu、lip、hlyA基因和豚鼠气单胞菌的lip和hlyA基因在肠道内、外的分布差异具有统计学意义;而tapA均只在肠道外嗜水、豚鼠和维氏气单胞菌中检出,提示气单胞菌部分毒力基因的分布与其来源部位、菌种类型相关。鞭毛是气单胞菌的重要毒力因子。本研究中fla和laf2个鞭毛相关基因在肠道内、外气单胞菌中均有检出,而laf在肠道气单胞菌的检出率明显较低,其原因可能与两者致病机制的不同有关。fla编码的极性鞭毛主要介导细菌生物膜的形成和定植,而laf编码的侧鞭毛则介导细菌对上皮细胞的黏附,引起严重的痢疾样感染[1]。痢疾样腹泻可伴有出血,气单胞菌则可通过破损的肠道黏膜进入机体引起肠道外感染。Ⅲ型分泌系统作为近年来细菌致病机制的研究热点,其相关基因ascF-G、aopP、ascV、aexT在本研究中的检出率却并不高,与Aravena-Román等的研究结果相一致[15]。而Burr和Frey在40株杀鲑气单胞菌中发现aopP、ascV、aexT的检出率高达80%~93%[9],Aravena-Román等也发现ascV、aexT在环境来源气单胞菌中检出率高于临床来源气单胞菌,从而提出T3SS毒力基因分布具有菌种及来源的相关性[15],与本研究的结果相一致。此外,ahyB、exu、eprCAI和Ser等胞外酶基因和肠毒素基因ast和alt在肠道内、外气单胞菌中的普遍检出表明气单胞菌的致病机理十分复杂,需要更深入的研究。
鉴于气单胞菌的毒力基因分布与其菌种和来源有关,故临床上在治疗气单胞菌引起的感染时,要根据其感染部位和菌种类型加以区别对待。
[1]Janda JM, Abbott SL. The genusAeromonas: taxonomy, pathogenicity, and infection[J]. Clin Microbiol Rev, 2010, 23(1): 35-73.
[2]World Health Organization. The treatment of diarrhea: a manual for physicians and other senior health workers[R].Geneba:WHO, 2005.
[3]World Health Organization. WHO recommended surveillance standards[R]. Geneva: WHO, 1999.
[4]Nawaz M, Khan SA, Khan AA,etal. Detection and characterization of virulence genes and integrons inAeromonasveroniiisolated from catfish[J]. Food Microbiol, 2010, 27(3): 327-331.
[5]Sun J, Zhang X, Gao X,etal. Characterization of virulence properties ofAeromonasveroniiisolated from diseased Gibel Carp(Carassius gibelio)[J]. Int J Mol Sci, 2016, 17(4): 496.
[6]Santos JA, Gonzalez CJ, Otero A,etal. Hemolytic activity and siderophore production in differentAeromonasspecies isolated from fish[J]. Appl Environ Microbiol, 1999, 65(12): 5612-5614.
[7]Wong CY, Heuzenroeder MW, Flower RL. Inactivation of two haemolytic toxin genes inAeromonashydrophilaattenuates virulence in a suckling mouse model[J]. Microbiology, 1998, 144(Pt 2): 291-298.
[8]Sreedharan K, Philip R, Singh IS. Characterization and virulence potential of phenotypically diverseAeromonasveroniiisolates recovered from moribund freshwater ornamental fishes of Kerala, India[J]. Antonie Van Leeuwenhoek, 2013, 103(1): 53-67.
[9]Burr SE, Frey J. Analysis of type Ⅲ effector genes in typical and atypicalAeromonassalmonicida[J]. J Fish Dis, 2007, 30(11): 711-714.
[10]Chu YW, Wong CH, Tsang GK,etal. Lack of association between presentation of diarrhoeal symptoms and faecal isolation ofAeromonas spp. amongst outpatients in Hong Kong[J]. J Med Microbiol, 2006, 55(Pt 3): 349-351.
[11]Bauab TM, Levy CE, Rodrigues J,etal. Niche-specific association ofAeromonasribotypes from human and environmental origin[J]. Microbiol Immunol, 2003, 47(1): 7-16.
[12]Albert MJ, Ansaruzzaman M, Talukder KA,etal. Prevalence of enterotoxin genes inAeromonasspp. isolated from children with diarrhea, healthy controls, and the environment[J]. J Clin Microbiol, 2000, 38(10): 3785-3790.
[13]陈素明,鲍春梅,崔恩博,等. 北京地区气单胞菌的流行状况及耐药性分析[J]. 中国抗生素杂志, 2011, 36(5): 374-376.
[14]Pemberton JM, Kidd SP, Schmidt R. Secreted enzymes ofAeromonas[J]. FEMS Microbiol Lett, 1997, 152(1): 1-10.
[15]Aravena-Román M, Inglis TJ, Riley TV,etal. Distribution of 13 virulence genes among clinical and environmentalAeromonasspp. in Western Australia[J]. Eur J Clin Microbiol Infect Dis, 2014, 33(11): 1889-1895.
(本文编辑:周万青,刘群)
Distribution characteristics and virulence gene analysis of intestinal and extraintestinalAeromonas
LIShu-fei1,CHENXiao1,2,CHENYu1,2
(1.DepartmentofClinicalLaboratory,theFirstAffiliatedHospital,CollegeofMedicine,ZhejiangUniversity,Hangzhou310003,Zhejiang; 2.StateKeyLaboratoryforDiagnosisandTreatmentofInfectiousDiseases,CollaborativeInnovationCenterforDiagnosisandTreatmentofInfectiousdiseases,ZhejiangUniversity,Hangzhou310003,Zhejiang,China)
Objective To investigate the species distribution and the difference of virulence gene spectra ofAeromonasisolated from intestinal tract and extraintestinal body fluid, and the correlation of their pathogenicity with infection sites. Methods A total of 156Aeromonasstrains isolated from the fecal specimens of patients with acute diarrhea and extraintestinal specimens were collected during May 2013 and September 2015. Eighteen virulence genes in these strains, includinghlyA,aerA,act,alt,ast,aexT,ascV,aopP,ascF-G,gcat,tapA,fla,Ser,exu,ahyB,eprCAI,lipandlaf, were detected by polymerase chain reaction(PCR). Last, the differences of virulence gene spectra between intestinal and extraintestinalAeromonaswere analyzed. Results Among 156Aeromonasstrains, 79 were from fecal specimens, and 77 from extraintestinal specimens.Aeromonascaviae(A.caviae, 51.9%) was the most common species in the intestinal strains, whileAeromonashydrophila(A.hydrophila, 48.1%) andA.caviae(39.0%) were the main pathogens in extraintestinal infections. The most prevalent virulence genes in intestinal and extraintestinalAeromonasweregcat,act,fla,ahyB,exuandlip(>45.57%), whileaexT,aopP,ascF-GandascVwere less frequently detected(<20.78%). The detection rates ofgcat,ahyB,laf,ast,exu,lip,hlyAandaerAgenes in intestinalAeromonaswere significantly lower than those in extraintestinal isolates(P<0.05). The detection rates ofgcat,ahyB,exu,lip,eprCAIandhlyAgenes in extraintestinalA.hydrophilawere significantly higher than those in intestinalA.hydrophila(P<0.05). The detection rates oflipandhlyAgenes in extraintestinalA.caviaewere significantly higher than those in intestinalA.caviae(P<0.05), while that ofaopPgene was just the reverse. There was no significant difference in the detection rates of virulence genes between intestinal and extraintestinalAeromonasveronii. Conclusion There are significant differences in the species distribution and virulence genes ofAeromonasisolated from intestinal and extraintestinal specimens, indicating that clinicians should treat them differentially.
Aeromonas; virulence gene; intestinal; extraintestinal
10.13602/j.cnki.jcls.2017.07.06
“十二五”国家科技重大专项(2012ZX10004-210)。
李淑妃,1992年生,女,硕士研究生,从事肠道微生物研究。
陈瑜,主任技师,博士研究生导师,E-mail:chenyyu@sina.com。
R446.5
A
2017-03-27)

