基于转录组序列的夏蜡梅SSR位点特征与引物开发
黄耀辉,张 超,周莉花,赵宏波
(浙江农林大学 风 景园林与建筑学院,浙江 临 安311300)
基于转录组序列的夏蜡梅SSR位点特征与引物开发
黄耀辉,张 超,周莉花,赵宏波
(浙江农林大学 风 景园林与建筑学院,浙江 临 安311300)
夏蜡梅Sinocalycanthus chinensis是中国二级濒危植物。为了开发基于夏蜡梅转录组序列的表达序列标签SSR(EST-SSRs)引物,从夏蜡梅转录组数据库组装的125 014条unigene序列中检测到26 564个简单重复序列(SSRs),平均密度为5.18 kb,最丰富的类型是2碱基重复、单碱基重复和3碱基重复,分别占总SSRs的42.43%,29.50%和23.25%,其中主导类型是(AG/CT)n。利用L16(45)正交试验获得夏蜡梅的最优简单重复序列-聚合酶链式反应(SSR-PCR)体系:20.0μL含Taq酶0.6 U(1 U=16.67 nkat),镁离子(Mg2+)1.50mmol·L-1,三磷酸碱基脱氧核苷酸(dNTP)0.25mmol·L-1,引物0.20μmol·L-1,DNA 75 ng。针对所有EST-SSRs共设计出15 585对符合要求的引物,随机选择了220对进行聚合酶链式反应(PCR)扩增,其中120对(54.55%)成功扩增出产物,14对在不同种群个体中扩增出多态,平均扩增等位基因数为2.64,7对在同一种群个体中扩增出多态。这些多态引物的开发可为夏蜡梅的遗传分析提供更丰富的标记。图3表4参35
植物学;夏蜡梅;ESR-SSR;引物筛选;扩增体系;正交实验
简单重复序列(simple sequence repeat,SSR)是一种由1~6个核苷酸为单位多次串联重复组成的核苷酸序列,广泛分布于生物基因组中[1-2]。SSR分子标记技术具有共显性、多态性高、遗传信息量大、稳定性高、重复性好等优点[3-5],已被广泛应用于遗传多样性分析、种质鉴定、基因定位、功能基因挖掘等领域[1,6-7]。传统的基因组SSR标记技术(genomic-SSR)需要构建DNA文库,耗时耗力[6,8]。表达序列标签SSR(expressed sequence tag SSR,EST-SSR),来源于基因的转录组区,相对genomic-SSR更简便,经济且信息量大、通用性好[6,9]。宋跃朋等[10]利用杨树Populus对genomic-SSR和EST-SSR这2种标记的遗传差异做了比较研究,发现在鉴定基因型方面EST-SSR相对更精确。目前,基于转录组序列的SSR标记在很多植物上得到应用[11-16]。夏蜡梅Sinocalycanthus chinensis隶属于蜡梅科Calycanthaceae夏蜡梅属Sinocalycanthus,为第三纪孑遗植物[17],其系统地位特殊,且具有较高的观赏价值。由于自然种群规模小,生境片段化使夏蜡梅不同种群间存在不同距离的隔离[18],种群间基因交流少,内部近交率增加,导致种群内遗传多样性低,种群间遗传分化增加[19-24]。尽管已有多种标记[同工酶[19],随机扩增多态DNA(RAPD)[20,22,24],ISSR[21,24]和扩增片段长度多态性分子标记(AFLP)[25]]用于夏蜡梅的遗传多样性和交配系统分析,但之前标记均是显性标记,且所揭示的多态性信息普遍偏低。因此,开发共显性的SSR标记用于夏蜡梅的遗传多样性相关研究势在必行。本研究以之前获得的夏蜡梅转录组数据,分析SSR位点特征,设计相应引物,建立最优的SSR-PCR体系,从不同水平(种群间和种群内)筛选高效、多态的引物。
1 材料和方法
1.1 试验材料
夏蜡梅不同野生种群材料:选择浙江天台的经过坪(JGP)和捣臼孔(DJK),浙江临安的龙塘山(LTS),西坑(XK),前坑(QK)和安徽绩溪的龙须山(LXS)等6个野生种群,采集新鲜叶片,硅胶干燥低温保存,随机选取个体3个·种群-1。
同一野生种群材料:选择经过坪(JGP)种群,按比例估算采样植株的间距和大小,采集24个个体叶片样本(间隔距离视种群大小适当调整),各年龄级采集数量大致相等,硅胶干燥低温保存。
1.2 基因组DNA的提取
实验采用十六烷基三甲基溴化铵(CTAB)法提取基因组DNA[26]。
1.3 SSR位点开发与引物设计
基于夏蜡梅转录组数据库组装的125 014条unigene序列,运用MISA进行SSR位点搜索[27]:重复单元长度为1~6个核苷酸,最少重复次数为12,6,5,5,4,4次;SSR位点侧翼序列的长度≥150 bp。据检测到的位点,用Primer 3.0引物批量设计程序设计引物[4]:引物序列中没有SSR;序列在DNA保守序列区内;长度为15~30 bp;上下游引物不存在互补序列;引物自身不存在互补序列;引物退火温度(Tm)为53~63℃;上下游引物的Tm值相差≤5℃;鸟嘌呤和胞嘧啶所占的比率(GC含量)为40%~ 60%。随机挑选2~6核苷酸重复基序的引物220对(编号:XLM001~XLM220),由上海生工生物工程股份有限公司合成,用于后续SSR引物筛选。
1.4 聚合酶链式反应(PCR)扩增体系的优化
以夏蜡梅叶片DNA为模板,SSR-PCR扩增程序参考WU等[28]:94℃预变性3 min;30个循环(94℃变性30 s,最佳Tm退火30 s,72℃1min),72℃延伸10min。
SSR-PCR扩增采用20.0μL体系,利用五因素四水平正交试验设计L16(45)(表1)筛选Taq酶,镁离子(Mg2+),DNA,dNTP和引物5种因素的浓度,建立夏蜡梅SSR-PCR扩增体系。选择编号XLM78和XLM91的2对引物用于优化PCR扩增体系。
PCR扩增产物用体积分数为8%非变性聚丙烯酰胺凝胶电泳检测,160 V电压,90min,银染显色。
1.5 SSR引物筛选和验证
利用优化的PCR扩增体系和6个种群(个体3个·种群-1)对合成的220对引物进行多态性筛选[29]:根据条带扩增情况统计能够稳定扩增出清晰目的条带的引物;根据条带迁移情况统计目标范围内出现相异条带的引物;根据条带大小的不同分类标记带型 “A,B,C,D,…”,纯合子条带可记为“AA,BB,CC,DD,…”,杂合子条带根据带型表示为 “AB,BD,CD,BD,…”,带型数据可用于后续遗传多样性等遗传分析。
利用同一种群(JGP)的24个个体对筛选的种群间多态性引物进行PCR扩增,统计目标范围内出现相异条带的引物。读带方法同上。
表1 夏蜡梅SSR-PCR扩增体系(20.0μL)正交设计表L16(45)Table 1 Orthogonal experimental design of SSR-PCR technique system for Sinocalycanthus chinensis
2 结果与分析
2.1 夏蜡梅转录组SSR位点的分布频率与基序特征
从夏蜡梅125 014条unigene序列 (总覆盖长度为137.52 Mb)中检测到22 113条序列上包含了共26 564个SSR位点(21.25%),平均密度为5.18 kb。其中含有2个或2个以上SSR位点的序列有3 664条(16.57%),复合SSRs有1 066条(4.01%)。
夏蜡梅不同重复类型的EST-SSRs分布频率不同(表2)。最丰富的类型是二碱基重复型,占42.43%;其次是单碱基重复型和三碱基重复型,分别占了29.50%和23.25%; 四碱基重复型、五碱基重复型、六碱基重复型较少,占4.82%。从重复次数看(表2),二碱基及二碱基以上重复型中,以5~7次重复为主,其次是8~10次重复:二碱基重复型集中在6~10次;三碱基重复型、四碱基重复型集中在5~6次;五碱基重复型、六碱基重复型集中在4次。从SSR重复基元出现的频率看(表3):单碱基重复型以(A/T)n形式为主,占99.07%;(C/G)n极少,仅占0.93%。二碱基重复型以(AG/CT)n形式为主,占85.33%,占SSR总数的36.21%;(CG/CG)n最少仅占本类基序比例的0.21%,占SSR总数的0.09%。碱基重复型以(AAG/CTT)n形式最多,占 40.8%,其次是(ATC/ATG)n和(AGC/CTG)n,分别占 18.26%和11.69%,(ACT/AGT)n最少,占1.12%。四碱基重复型、五碱基重复型、六碱基重复型的重复基元种类较多,占SSR总数比例较少(4.82%)。

表2 夏蜡梅EST-SSR的分布特征Table 2 Distribution of the SSRmotifs in Sinocalycanthus chinensis
表3 夏蜡梅EST-SSR主要基序类型分布Table 3 Distribution of types for EST-SSR in Sinocalycanthus chinensis
2.2 夏蜡梅SSR-PCR体系优化结果
正交实验结果(图1)显示:5,9,10,13号组合均可扩增出比较清晰、稳定的带且杂带较少(XLM078扩增结果),重复试验(XLM091扩增结果),结果一致。
选用6个种群的18个样本(随机选择样本3个·种群-1)验证以上4种体系。结果发现:仅9号组合可重复稳定地扩增出清晰的条带(图2)。为此选择9号体系作为夏蜡梅SSR-PCR最优体系:20.0μL体系含Taq酶0.6 U(1 U=16.67 nkat),镁离子 1.50 mmol·L-1,dNTP 0.25 mmol·L-1,引物 0.20μmol·L-1,DNA 75.0 ng。
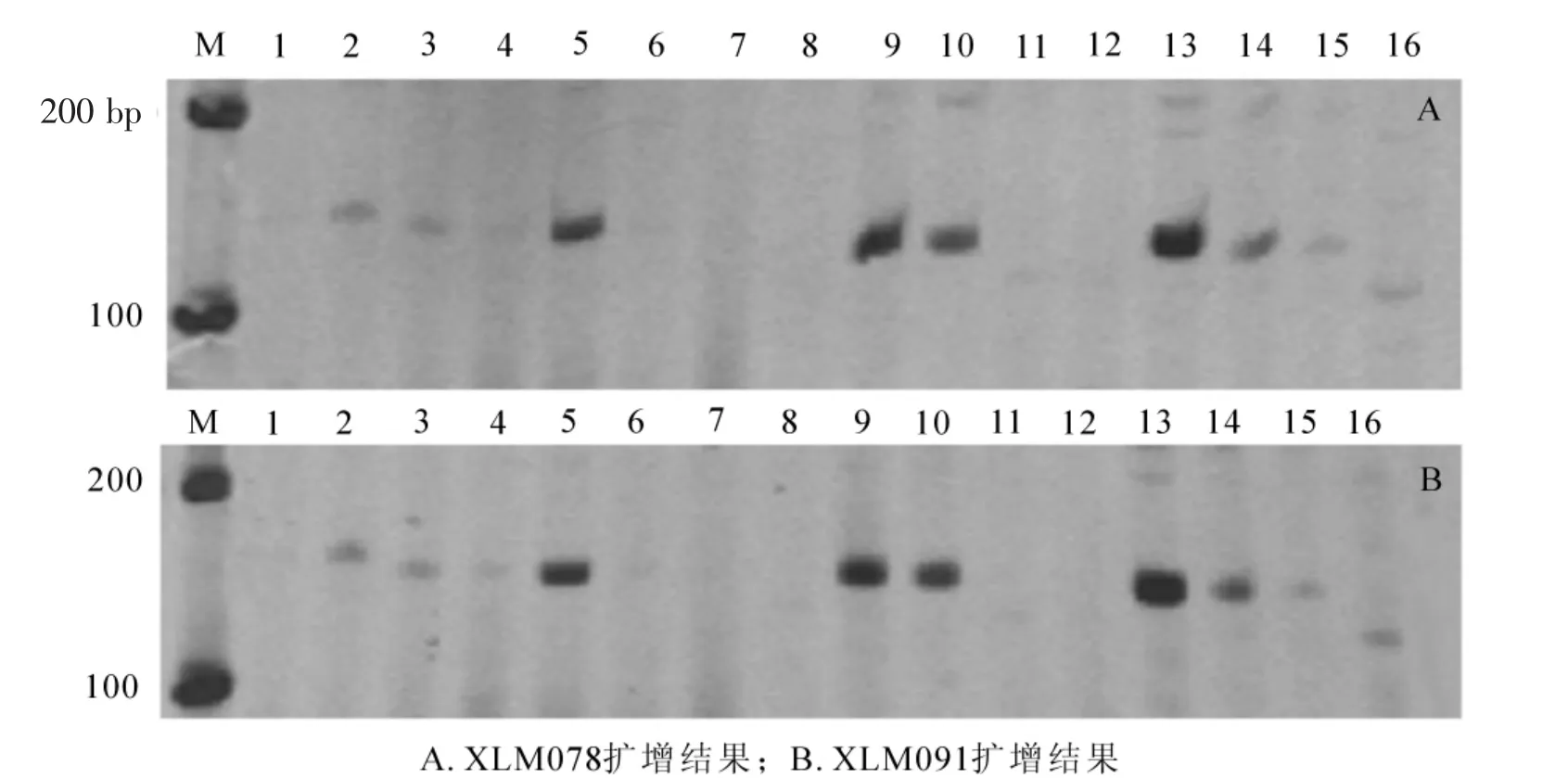
图1 正交实验SSR-PCR产物电泳图谱Figure 1 Electrophoresis pattern of orthogonal test for SSR-PCR products
2.3 SSR引物筛选和验证
试验选取6个种群的18个样本(随机选择样本3个·种群-1)对随机选择合成的220对引物(编号:XLM001~XLM220)进行PCR扩增,结果表明(表4):总体而言,共120对引物扩增出特异性产物,有效扩增率为54.54%,其中14对引物扩增出多态性条带,占引物总数的6.36%。220随机引物对应的SSRs中,最多的为二碱基重复型、三碱基重复型,最少的为四碱基重复型。各自类型的引物中,可成功扩增出产物的引物比例与总体(54.54%)相差不大,其中最高的是四碱基重复型(64.29%),最低的为三碱基重复型(48.57%);就多态性引物所占各自类型引物总量的比例而言,二碱基重复型SSRs对应的引物最高(7.79%),其次是四碱基重复型(7.14%),最低的为五碱基重复型(4.35%)。14对多态性引物共扩增出37个等位基因,平均扩增为2.64,其中五碱基重复型SSRs对应的引物扩增的等位基因数最高(平均为4),其次是四碱基重复型(平均为3),最低的为六碱基重复型(平均为2)。
选取经过坪(JGP)种群24个样本,对上述具有种群间多态性的14对引物进行SSR-PCR扩增。结果发现:7对引物(3.18%)扩增出多态性条带:即XLM003,XLM007,XLM021,XLM035,XLM078,XLM139和XLM146,其中XLM007和XLM139扩增产物条带比较丰富,等位变异较多,XLM007在经过坪(JGP)种群的个体中扩增出4个(图3A),XLM139扩增出5个(图3B)。
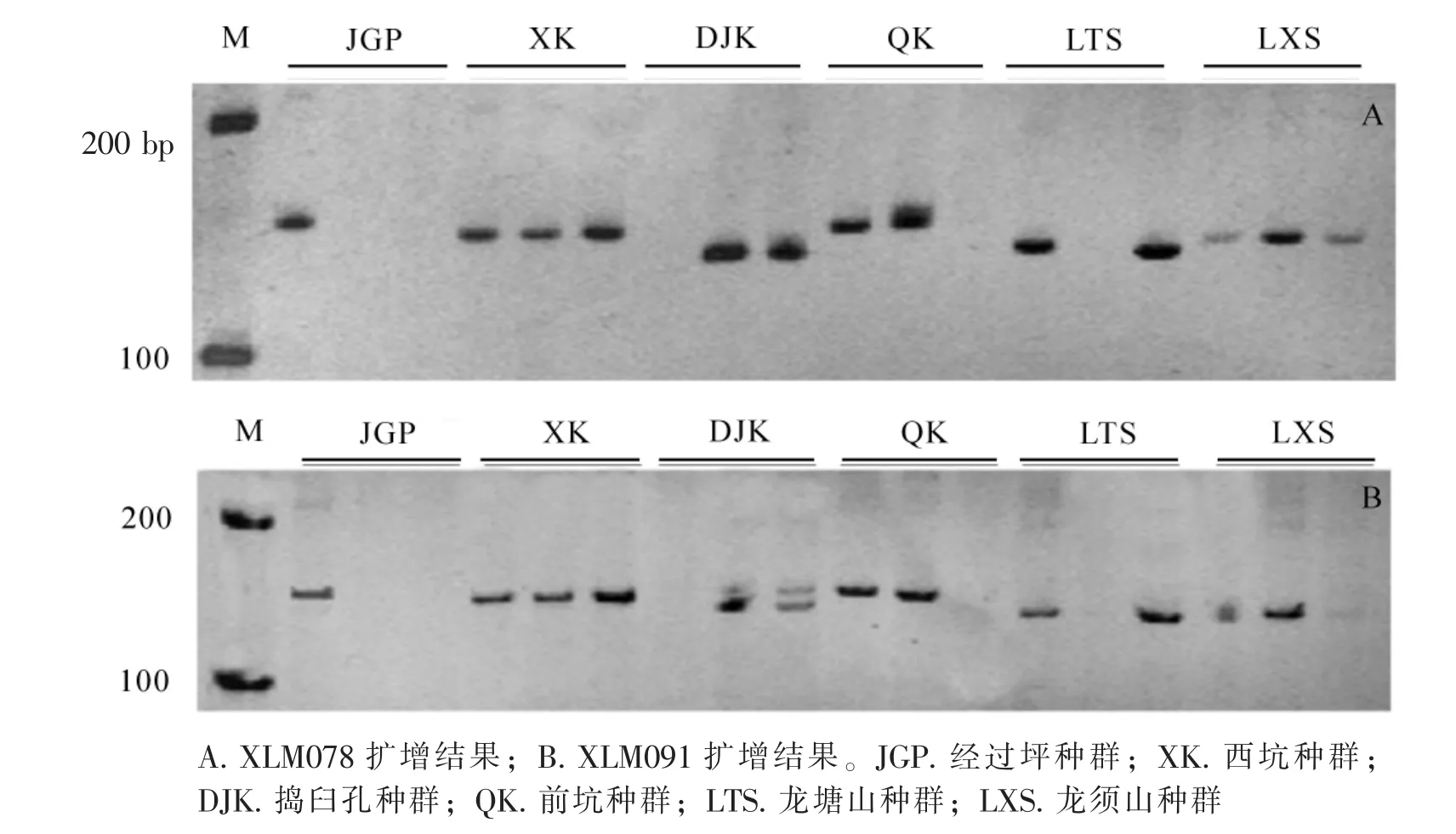
图2 不同种群夏蜡梅DNA的PCR产物电泳图谱Figure 2 Electrophoresis pattern of DNA PCR products in different population of Sinocalycanthus chinensis

表4 220对引物的对应的SSR类型与扩增情况Table 4 SSR types and amplification results of 220 primers
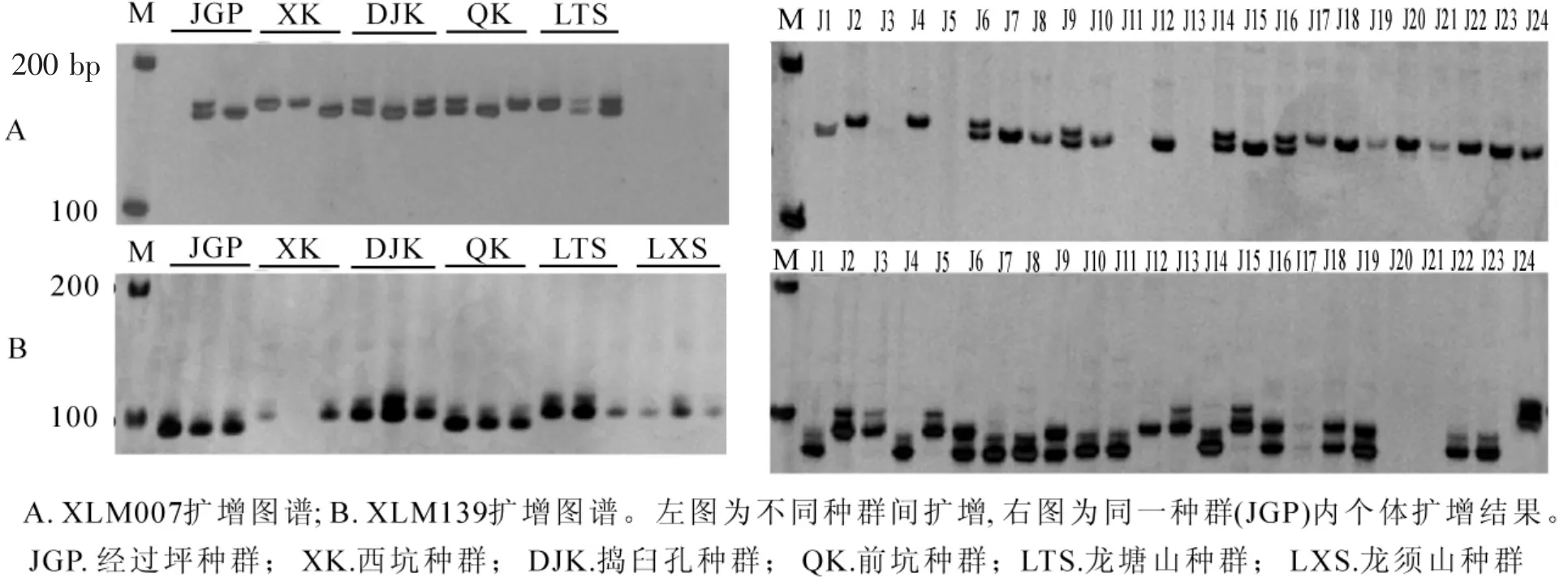
图3 引物XLM007和XLM139扩增产物的电泳图谱Figure 3 Electrophoresis pattern of amplification by primer XLM007 and XLM 139
3 讨论
3.1 EST-SSRs分布频率与特征
夏蜡梅转录组序列中的SSRs较为丰富,从125 014条unigene序列中共检测到26 564个SSR位点,发生频率为21.25%,平均5.18 kb就有1个SSR位点,其中含有2个或2个以上SSR位点的序列有3 664条(16.57%),复合SSRs有1 066条(4.01%)。就SSR位点发生频率而言,夏蜡梅比蜡梅Chimonanthuspraecox(12.35%)[3]高,相较其他观赏植物,如百合Lilium(5.98%)[31],蝴蝶兰Phalaenopsis(3.19%)[32],辣椒Capsicum annuum(7.83%)[4],南方红豆杉Taxus chinensis var.mairei(2.24%)[5]和红掌Anthurium andraeanum(12.70%)[16]等也是如此;就SSR位点平均密度而言,夏蜡梅(1/5.18 kb)与蜡梅(1/5.00 kb)[30]相差无几,显著高于上述几种观赏植物。SSRs发生频率和平均密度除了因物种的不同而差异较大外,还可能与搜索标准,数据库的大小有关[4]。
研究表明:多数物种的EST-SSRs中以三碱基重复型出现的频率较高[33]。夏蜡梅不同重复类型的EST-SSRs最丰富的类型是二碱基重复型,占42.43%,其中以(AG/CT)n形式的二碱基重复型最多,占总SSRs的36.21%,这与蜡梅[44.81%,(AG/CT)n占总SSRs的39.85%][30]一致;三碱基重复型中都以(AAG/ CTT)n为主要的重复基元[30],两者四、五、六碱基重复型所占比例都相对较少;就重复次数而言,除单核苷重复外,两者都以4~10次重复为主[30]。有所不同的是,夏蜡梅转录组中单碱基重复型[基本为(A/ T)n]所占数量仅次于二碱基重复型,蜡梅转录组中三碱基重复型[比例最大的为(AAG/CTT)n]要多于单碱基重复型[30]。形成这种差异的原因除了基于植物本身的EST-SSR特点不同外,与转录组数据来源和数量也有关系[4]。
3.2 引物有效性评价
根据22 113条EST序列中检测到的26 564个SSR-ESTs,共设计出15 585对引物。在随机选择220对引物中,有120对引物(54.55%)成功扩增出产物,14对(6.36%)在不同种群个体中扩增出多态性条带,平均位点数为2.64;7对(3.18%)在经过坪种群内不同个体间扩增出多态性条带,说明夏蜡梅EST-SSR位点多,但多态性较低,原因是基因转录区的具有较高的保守性,使得EST-SSR引物具有较强的种属间通用性,可以用于亲缘关系较远的物种[30,34]。胥猛等[35]基于鹅掌楸Liriodendron的6 520条EST设计了176对SSR引物,66对扩增出多态性条带,这66对中85%在鹅掌楸中有扩增,54%在白玉兰Michelia alba中有扩增。宋跃朋等[10]在比较杨树Populus基因组SSR和EST-SSR这2种标记的遗传差异性中发现其EST-SSR多态性明显低于基因组SSR,保守性很强,通用性高。夏蜡梅EST-SSR引物的种属间通用性还有待验证。
夏蜡梅EST-SSR引物中有14对(6.36%)在6个种群间扩增出多态性条带,而仅7对(3.18%)在同一种群(JGP)中有多态性条带,可能源于其种群自然分布的特点:种群规模小,片段化分布,不同种群间相互隔离,基因流阻断,种群内部近交率增加,种群间遗传分化增加,导致种群内遗传多样性低,大部分遗传多样性来自种群间[19-24]。
本研究开发了基于夏蜡梅转录组的SSR标记,为夏蜡梅种群的遗传多样性分析、交配系统估算、遗传图谱构建等提供了新的途径,同时夏蜡梅EST序列的开发为挖掘新的SSR提供了丰富的资源。
[1] KALIA R K,RAIM K,KALIA S,et al.Microsatellitemarkers:an overview of the recent progress in plants[J].Euphytica,2011,177(3):309-334.
[2] 李满堂,张仕林,邓鹏,等.洋葱转录组SSR信息分析及其多态性研究[J].园艺学报,2015,42(6):1103-1111.
LIMantang,ZHANG Shilin,DENG Peng,et al.Analysis on SSR information in transcriptome of onion and the polymorphism[J].Acta Hortic Sin,2015,42(6):1103-1111.
[3] 吕婧,时秋香,任毅,等.黄瓜23对高多态性SSR标记的筛选与评价[J].园艺学报,2011,38(11):2140-2148.
LÜJing,SHIQiuxiang,REN Yi,et al.Screening and evaluation of 23 high polymorphism SSR markers in cucumber[J].Acta Hortic Sin,2011,38(11):2140-2148.
[4] 刘峰,王运生,田雪亮,等.辣椒转录组SSR挖掘及其多态性分析[J].园艺学报,2012,39(1):168-174.
LIU Feng,WANG Yunsheng,TIAN Xueliang,et al.SSR mining in pepper(Capsicum annuum L.)transcriptome and the polymorphism analysis[J].Acta Hortic Sin,2012,39(1):168-174.
[5] 李炎林,杨星星,张家银,等.南方红豆杉转录组SSR挖掘及分子标记的研究[J].园艺学报,2014,41(4):735-745.
LIYanlin,YANG Xingxing,ZHANG Jiayin,et al.Studies on SSRmolecularmarkers based on transcriptome of Taxus chinensis var.mairei[J].Acta Hortic Sin,2014,41(4):735-745.
[6] 陈怀琼,隋春,魏建和.植物SSR引物开发策略简述[J].分子植物育种,2009,7(4):845-851.
CHEN Huaiqiong,SUIChun,WEI Jianhe.Summary of strategies for developing SSR primer[J].Mol Plant Breed, 2009,7(4):845-851.
[7] 程小毛,黄晓霞.SSR标记开发及其在植物中的应用[J].中国农学通报,2011,27(5):304-307.
CHENG Xiaomao,HUANG Xiaoxia.Development and application of SSR markers in plants[J].Chin Agric Sci Bull, 2011,27(5):304-307.
[8] 李齐发,赵兴波,罗晓林,等.牦牛基因组微卫星富集文库的构建与分析[J].遗传学报,2004,31(5):489-494.
LIQifa,ZHAO Xingbo,LUO Xiaolin,et al.Construction and identification on enriched microsatellite library from yak genome[J].Acta Genet Sin,2004,31(5):489-494.
[9] 李小白,向林,罗洁,等.转录组测序(RNA-seq)策略及其数据在分子标记开发上的应用[J].中国细胞生物学报, 2013,35(5):720-726.
LIXiaobai,XIANG Lin,LUO Jie,et al.The strategy of RNA-seq,application and development ofmolecularmarker derived from RNA-seq[J].Chin JCell Biol,2013,35(5):720-726.
[10] 宋跃朋,江锡兵,张曼,等.杨树Genomic-SSR与EST-SSR分子标记遗传差异性分析[J].北京林业大学学报, 2010,32(5):1-7.
SONG Yuepeng,JIANG Xibing,ZHANG Man,et al.Genetic differences revealed by genomic-SSR and EST-SSR in poplar[J].JBeijing For Univ,2010,32(5):1-7.
[11] 关媛,李效尊,潘俊松,等.黄瓜果实EST-SSR标记的开发[J].分子植物育种,2007,5(5):725-728.
GUAN Yuan,LIXiaozun,PAN Junsong,et al.Development of EST-SSRs in cucumber fruit[J].Mol Plant Breed, 2007,5(5):725-728.
[12] LURO F L,COSTANTINO G,TEROL J,et al.Transferability of the EST-SSRs developed on Nules Clementine(Citrus clementina Hort ex Tan)to other citrus species and their effectiveness for genetic mapping[J].BMC Genom, 2008,9(1):287.doi:10.1186/1471-2164-9-287.
[13] 王丽鸳,姜燕华,段云裳,等.茶树EST-SSRs分布特征及引物开发[J].植物遗传资源学报,2009,10(4):511-516.
WANG Liyuan,JIANG Yanhua,DUAN Yunshang,et al.Characterization of EST-derived microsatellites and development of SSR-markers in tea(Camellia sinensis)[J].JPlant Genet Resour,2009,10(4):511-516.
[14] ZENG Shaohua,XIAO Gong,GUO Juan,et al.Development of a EST dataset and characterization of EST-SSRs in a traditional Chinesemedicinal plant,Epimedium sagittatum(Sieb.et Zucc.)Maxim [J].BMCGenom,2010,11(1): 94.doi:10.1186/1471-2164-11-94.
[15] ZHANG Haiyang,WEILibin,MIAO Hongmei,et al.Development and validation of genic-SSR markers in Sesame by RNA-seq[J].BMCGenom,2012,13(1):316.doi:10.1186/1471-2164-13-316.
[16] 郁永明,田丹青,潘晓韵,等.基于红掌转录组序列的SSR标记分析与开发[J].分子植物育种,2015,13(6): 1349-1354.
YU Yongming,TIAN Danqing,PAN Xiaoyun,et al.Mining and developing SSR molecularmarkers based on transcriptome sequences of Anthurium[J].Mol Plant Breed,2015,13(6):1349-1354.
[17] 张若蕙,刘洪谔.世界蜡梅[M].北京:中国科学技术出版社,1998.
[18] 谈探,金则新,李均敏,等.濒危植物夏蜡梅遗传分化研究[J].西北林学院学报,2008,23(2):77-82.
TAN Tan,JIN Zexin,LI Junmin,et al.Genetic differentiation of Calycanthus chinensis an endangered plant[J].J Northwest For Univ,2008,23(2):77-82.
[19] 周世良,叶文国.夏蜡梅的遗传多样性及其保护[J].生物多样性,2002,10(1):1-6.
ZHOU Shiliang,YEWenguo.The genetic diversity and conservation of Sinocalycanthus chinensis[J].Biodivers Sci,2002,10(1):1-6.
[20] LI Junmin,JIN Zexin.High genetic differentiation revealed by RAPD analysis of narrowly endemic Sinocalycanthus chinensis Cheng et S.Y.Chang,an endangered species of China[J].Biochem Syst Ecol,2006,34(10):725-735.
[21] 金则新,李钧敏.珍稀濒危植物夏蜡梅遗传多样性的ISSR分析[J].应用生态学报,2007,18(2):247-253.
JIN Zexin,LI Junmin.ISSR analysis on genetic diversity of endangered relic shrub Sinocalycanthus chinensis[J]. Chin JAppl Ecol,2007,18(2):247-253.
[22] 张文标,金则新,李均敏.不同生境夏蜡梅群体遗传多样性的RAPD分析[J].植物研究,2007,27(3):313-318.
ZHANGWenbiao,JIN Zexin,LIJunmin.Genetic diversity of Sinocalycanthus chinensis in four different habitats revealed by RAPD[J].Bull Bot Res,2007,27(3):313-318.
[23] LIJunmin,JIN Zexin,GU Jingjing.Genetic isolation by distance in the endangered plant Sinocalycanthus chinensis endemic to China[J].Pak JBot,2012,44(4):1275-1280.
[24] 汪琼,姚青菊,徐增莱,等.基于ISSR和RAPD标记的4个夏蜡梅种群的遗传多样性研究[J].广西植物,2013, 33(1):30-34.
WANG Qiong,YAO Qingju,XU Zenglai,et al.Genetic diversity of four populations of Calycanthus chinensis based on ISSR and RAPDmarkers[J].Guihaia,2013,33(1):30-34.
[25] ZHAO Hongbo,ZHOU Lihua,LIU Huahong,et al.Genetic effects of differentmatingmodes in Sinocalycanthus chinensis (Cheng et S.Y.Chang)Cheng et S.Y.Chang,an endangered species endemic to Zhejiang Province,China[J].Biochem Syst Ecol,2014,54:8-14.
[26] 王关林,方宏筠.植物基因工程[M].2版.北京:科学出版社,2002.
[27] GUO Shaogui,ZHENG Yi,JOUNG JG,et al.Transcriptome sequencing and comparative analysis of cucumber flowerswith different sex types[J].BMCGenom,2010,11(1):384.doi:10.1186/1471-2164-11-384.
[28] WU Jing,CAIChangfu,CHEN Fangyun,et al.Characterisation and development of EST-SSR markers in tree peony using transcriptome sequences[J].Mol Breed,2014,34(4):1853-1866.
[29] 袁阳阳,王青锋,陈进明.基于转录组测序信息的水生植物莕菜SSR标记开发[J].植物科学学报,2013,31(5):485-492.
YUAN Yangyang,WANG Qingfeng,CHEN Jinming.Development of SSR markers in aquatic plant Nymphoides peltata(Menyanthaceae)based on information from transcriptome sequencing[J].Plant Sci J,2013,31(5):485-492.
[30] 李响,杨楠,赵凯歌,等.蜡梅转录组EST-SSR标记开发与引物筛选[J].北京林业大学学报,2013,35(增刊1): 25-32.
LIXiang,YANG Nan,ZHAO Kaige,et al.Development and primer selection of EST-SSR molecularmarkers based on transcriptome sequencing of Chimonanthus praecox[J].JBeijing For Univ,2013,35(supp 1):25-32.
[31] 杨素丽,明军,刘春,等.基于EST信息的百合SSR标记的建立[J].园艺学报,2008,35(7):1069-1074.
YANG Suli,MING Jun,LIU Chun,et al.Datamining for simple sequence repeatsmarker development in expressed sequence tags from Lilium L.[J].Acta Hortic Sin,2008,35(7):1069-1074.
[32] 张水明,陈程,龚凌燕,等.蝴蝶兰EST资源SSR标记分析与开发[J].园艺学报,2012,39(6):1191-1198.
ZHANG Shuiming,CHEN Cheng,GONG Lingyan,et al.Analysis of SSRs information and development of SSR markers from Phalaenopsis ESTs[J].Acta Hortic Sin,2012,39(6):1191-1198.
[33] LIANG Xuanqiang,CHEN Xiaoping,HONG Yanbin,et al.Utility of EST-derived SSR in cultivated peanut(Arachis hypogaea L.)and Arachis wild species[J].BMC Plant Biol,2009,9(2):35-48.
[34] TING N C,ZAKIN M,ROSLIR,et al.SSRmining in oil palm EST database:application in oil palm germplasm diversity studies[J].JGenet,2010,89(2):135-145.
[35] 胥猛,李火根.鹅掌楸EST-SSR引物开发及通用性分析[J].分子植物育种,2008,6(3):615-618.
XU Meng,LIHuogen.Development and characterization ofmicrosatellitemarkers from expressed sequence tags for Liriodendron[J].Mol Plant Breed,2008,6(3):615-618.
Development and primer screening of SSRmarkers based on transcriptome sequences in Sinocalycanthus chinensis
HUANG Yaohui,ZHANG Chao,ZHOU Lihua,ZHAO Hongbo
(School of Landscape Architecture,Zhejiang A&FUniversity,Lin’an 311300,Zhejiang,China)
Sinocalycanthus chinensis,a national second-class endangered species in China with high value for research and appreciation,was used to develop expressed sequence tag (EST)-simple sequence repeat(SSR)primers,which were assembled from transcriptome sequencing data.Then,the best SSR-Polymerase chain reaction PCR technique was determined and primer pairswere designed and amplified to investigate genetic diversity.Results showed 26 564 SSRs were detected from 125 014 unigene sequences.Among these unigene sequences,the average density was 5.18 kb,and themost abundant types were di-nucleotide repeats (42.43%)with (AG/CT)nrepeats occupying the largest number,mono-nucleotide repeats (29.50%),and tri-nucleotide repeats (23.25%).Total 15 585 primer pairs can be designed according to distribution of the EST-SSR locus. The best SSR-PCR reaction system in S.chinensis was gained by an L16(45)orthogonal experimental design, namely a 20.0μL SSR-PCR system containing 0.6 U(1 U=16.67 nkat)Taq enzyme,Mg2+1.50mmol·L-1,dNTP 0.25 mmol·L-1,primer 0.20μmol·L-1,and DNA 75 ng.Randomly 220 primer pairs were selected to amplify and 120 primer pairs (54.55%)was successful with bands.Among these primer pairs,14 pairs can amplify polymorphism among different populations (average allele number was 2.64)and seven primer pairs can amplify polymorphism within the population.These polymorphic primers could provide more abundantmarks for the genetic analysis of S.chinensis.[Ch,3 fig.4 tab.35 ref.]
botany;Sinocalycanthus chinensis;EST-SSR;primer screening;amplification system;orthogonaldesign
S718.4
A
2095-0756(2017)04-0589-08
10.11833/j.issn.2095-0756.2017.04.004
2016-03-31;
2016-07-26
国家自然科学基金资助项目(31401902,31101571)
黄耀辉,从事园林植物种质资源研究。E-mail:huangyaohui_ok@126.com。通信作者:赵宏波,教授,博士,从事观赏植物遗传育种和植物繁殖生态研究。E-mail:zhaohb@zafu.edu.cn

