基于改进的DNA连接酶链式反应发现NOD2基因多态性与冠心病的相关性
李扬,杨熹,田小利
基于改进的DNA连接酶链式反应发现NOD2基因多态性与冠心病的相关性
李扬,杨熹,田小利
目的:本研究改进基于脱氧核糖核酸(DNA)连接酶的中低通量基因分型方法,并用此方法进行核苷酸结合寡聚结构域(NOD)样受体基因NOD1和NOD2与冠心病的关联分析。
方法:通过多重聚合酶链式反应(PCR)预扩增增加连接模板分子数量,提高特异性连接效率,优化DNA探针设计,摸索连接反应温度、时间、循环圈数及连接酶种类实现等位基因特异性连接,通过荧光掺入PCR和毛细管电泳实现多种等位基因特异性产物一次性检测。Sanger测序验证此方法准确性后,利用此方法对NOD1和NOD2基因上的单核苷酸多态性(SNP)位点在1 555例冠心病患者和1887例对照受试者中进行分型和关联分析。
结果:通过优化反应条件和等位基因特异性探针设计原则,实现10 ng DNA样本一次性分型30个等位基因多态性位点。基于改进的DNA连接酶中低通量基因分型方法,NOD1、NOD2基因与冠心病的关联分析发现,NOD2基因上rs1861759和rs751271位点在非高血压条件下与冠心病相关(P均<0.05),经Bonferroni多重检验校正后,相关性仍然显著(P均<0.05)。
结论:本研究优化了DNA连接酶链式反应技术在设计上的关键点,联合使用多重PCR和毛细管电泳,建立了一种高准确性、低成本、满足基于中低通量位点分型的临床分子诊断和科研需求的基因分型新方法,并用该方法发现NOD2基因上的位点rs1861759和rs751271在非高血压条件下与冠心病相关。
冠状动脉疾病;基因分型;DNA连接酶类;多态性,单核苷酸
(Chinese Circulation Journal, 2017,32:569.)
随着精准医疗理念的推广,基因分型在临床诊断、个体化用药指导等方面的应用需求与日剧增[1]。基因分型方法需兼顾人力、物力及时间成本,同时需要一个合适的通量符合对应的要求。常见的单一位点基因检测手段有Sanger测序、限制性片段长度多态性[2]、单链构象多态性分析[3]、等位基因特异性聚合酶链式反应(PCR)[4]、TaqMan[5]、连接酶检测反应(LDR)[6]等。虽然这些方法精确度高,但如果需要检测比较多的单核苷酸多态性(SNP)位点,速度慢,成本高。高通量基因分型的手段有二代测序、杂交芯片等,可以一次分型百万或更多的位点,但涉及复杂的生物信息学分析,得到较多冗余信息,成本昂贵[7,8]。如果需要分型的位点达不到如此通量,高通量基因分型手段也不是很好的选择。本研究拟建立一种基于脱氧核糖核酸(DNA)连接酶的基因分型新方法,可高准确性、低成本、快速分型十几个甚至几十个位点,满足基于中低通量位点分型进行的临床分子诊断和科研需求。
模式识别受体是一类能特异识别病原微生物的某些结构、进化上保守的分子,其募集接头蛋白并激活下游的相关信号通路,从而启动机体的免疫应答,在宿主的先天性免疫中发挥着重要作用。目前已报道的模式识别受体主要包括Toll 样受体、核苷酸结合寡聚结构域(NOD)样受体、维甲酸诱导基因(RIG)样受体等[9]。炎症和先天性免疫在动脉粥样硬化病理过程中起非常重要的作用,Toll样模式识别受体与动脉粥样硬化性疾病如冠心病的密切关系已经逐渐被大家所认识[10,11]。与Toll样受体不同,NOD样受体主要识别细胞内的细菌产物。目前,NOD样受体与冠心病的关系还不清楚。NOD样受体家族中,NOD1和NOD2是最早被发现的两类分子。NOD1在多种细胞中表达,而NOD2主要表达在巨噬细胞和树突状细胞[12]。本研究将利用建立的基因分型新方法进行NOD1和NOD2与冠心病的关联分析。

表1 基因位点rs1861759和rs751271预扩增引物和探针序列
1 材料与方法
1.1 通用试剂
EasyTaq DNA聚合酶及缓冲液购自北京全式金生物公司;10 mmol dNTP购自赛百盛生物公司;DNA marker购自博迈德生物公司;T4多聚核苷酸激酶及缓冲液购自NEB公司;Taq DNA连接酶及缓冲液购自NEB公司;pIRES-neo质粒购自Clontech公司;Hi-Di甲酰胺等片段分析试剂购自Life Technologies公司。
1.2 DNA探针及引物
DNA 探针和引物(包括荧光修饰引物)由赛百盛生物公司合成,用于扩增的通用引物序列是:T7m: GGATACGACTCACTATAGGT;M13m: GGTAAGGACGACGGTCCAGT;M13Rm: GCGGATAACAAGTTCACACT。位点rs1861759和rs751271预扩增引物和探针序列见表1。
1.3 冠心病患者和对照受试者
本研究中,1 555例冠心病患者资料来自解放军总医院心内科,冠心病、高血压及糖尿病的诊断标准如之前已发表文章所述[13],1 887例对照受试者资料来自北京大学体检中心。
1.4 多重PCR预扩增
预扩增引物设计使待检测位点位于产物中部,每对引物单独使用时,10 ng基因组模板DNA进行35圈扩增有明显目的条带,允许有少量非特异扩增。20 μl多重PCR体系使用1 μl引物混合物(5 μmol/引物)进行第一轮扩增。
1.5 用于连接酶链式反应(LCR)的DNA探针设计
如图1所示,每个SNP位点需要6条DNA探针进行LCR反应。其中,rsX-A、rsX-G、rsX-NC和rsX-NT为等位基因特异性探针,其5’或3’末端为特定等位基因。rsX-N、rsX-NC、rsX-NT序列与基因组匹配。rsX-A和rsX-G包括与基因组序列匹配的部分(黑色线)和与通用引物T7m[6-羧基荧光素(FAM)蓝色荧光标记]或M13Rm[5-六氯荧光素(HEX)绿色荧光标记]结合序列(蓝色线),其中一个等位基因的特异探针还有3个与基因组不互补的碱基(黄色线)以控制两个等位基因的终产物有3个碱基的长度差异。rsX包括与基因组序列匹配的部分(黑色线)和与质粒模板结合的序列(粉红色)。质粒序列将用于扩增产生含有stuffer序列和M13m(通用引物)结合序列的长探针,同时该序列决定stuffer序列的长度,从而控制该位点扩增终产物的大小。
1.6 DNA探针预处理和多重LCR
设计合成的rsX-N、rsX-A、rsX-G不需要处理,rs-NC和rsNT需要磷酸化处理,rsX需要磷酸化之后通过PCR产生长度90~250 bp的DNA单链探针。磷酸化使用T4多聚核苷酸激酶,DNA长探针制备为不对称PCR(引物M13m和磷酸化的rsX比例1:20)。
对探针进行预混合:(1)LA,未磷酸化组:rsX-A、rsX-G和rsX-N,每个终浓度为1 μmol;(2)LB,磷酸化组:Pi-rsX-NC和Pi-rsX-NT,终浓度1 μmol;(3)LC:长度特异性序列等体积混合。Taq DNA连接酶催化的多重LCR体系包括:LA,0.2 μl;LB,0.2 μl;LC,1 μl。反应条件为94℃,20 s;48℃,150 s;循环35圈;4℃结束反应。
1.7 荧光掺入PCR
荧光引物(M13Rm-HEX/T7m-FAM)
和通用引物M13m的PCR扩增体系10 μl,
包含10倍稀释的连接产物1 μl。反应条件:
55℃退火,30 s;72℃延伸,30 s;循环25圈。
1.8 片段分析
HiDi甲酰胺与Liz 500以60:1比例混合,
9 μl混合液加1 μl荧光掺入PCR产物,94℃变
性2 min后立刻冰置,冷却后进行毛细管电泳。
片段分析结果使用GeneMarker V1.6分析。
1.9 电泳结果判读
对于不确定峰型归类的样本均进行1
代测序验证,若不确定样本超过5%则该位点视为分型失败;成功判定的样本,也按一定的比例抽样进行1代测序验证,若准确率低则该位点也被视为分型失败。
1.10 统计学方法
利用SPSS软件中二分类Logistic回归分析每个SNP位点在危险因素校正和不校正情况下与冠心病的相关性。多重检验校正采用Bonferroni校正方法。P<0.05视为有统计学意义。

图1 基因分型流程图
2 结果
2.1 总体基因分型流程(图1)
该方法分为四步:(1)基因组DNA预扩增,使包含待分型位点的基因组区域得以富集;(2)等位基因特异性连接反应,只有与模板完全匹配的探针可以高效完成连接反应;(3)荧光掺入PCR,通过荧光标记的通用引物往连接产物中掺入荧光,同时扩增富集连接产物;(4)产物通过毛细管电泳进行片段分析,同一位点的不同等位基因有3个碱基(黄色标记)的差异可以被毛细管电泳分辨,不同位点设计不同的填充序列长度(建议差异大于10 bp),也可被分辨。考虑到毛细管电泳的分辨率和电泳效果,终产物最好100 ~250 bp。因此,单色荧光可一次分型约15个位点,设置两种荧光不相互干扰的通用引物可使通量加倍。
2.2 预扩增PCR
比较理想的多重引物预扩增结果在100~200 bp之间有一条较粗的明亮条带。
2.3 连接酶反应系统优化
DNA连接酶的选择:常见的耐热DNA连接酶有Taq DNA连接酶以及9N DNA连接酶,通过比较我们选择了特异性更好的Taq DNA连接酶。值得注意的是,Taq DNA连接酶缓冲液含有氧化型辅酶I,长期保存必须放在-80℃,而短期保存必须放在-20℃。LCR探针设计优化:rsX和rsX-N探针无特异性问题,和模板配对的部分一般为19 bp。其他四条探针涉及到特异性配对的问题,需要符合以下条件:(1)SNP位点本身在序列一端;(2)GC含量适当,长度在11~15个碱基,3GC+AT=24-27(不包括SNP位点本身);(3)避免简单重复序列;(4)选择SNP两翼的序列中更接近上述条件的做等位基因特异性探针。LCR温度方面,经过对比,我们发现48℃特异性最强,而且SNP之间的信号更加均一。
2.4 荧光染料的选取
荧光引物PCR中,我们尝试了FAM、TET、HEX、TAMRA、ROX多种荧光染料修饰引物。综合考虑光谱兼容性以及引物修饰难度,最终选择FAM和HEX两种荧光修饰公共引物。
2.5 片段分析
荧光掺入PCR的产物因有荧光标记需要避光保存并尽快上机,上机前终产物电泳结果在150~250 bp长度范围内可见连续条带。上机毛细管电泳结果如图2所示,每一个峰代表一种终产物。
2.6 NOD1和NOD2基因关联分析
我们用该方法对NOD1和NOD2基因上的8个SNP位点(NOD1基因位点包括rs41524946、rs2284358、rs17159048、rs1558068;NOD2基因位点包括rs2111235、rs8057341、rs1861759、rs751271)在两个人群中进行了分型,人群1包括784例冠心病患者和895例对照受试者;人群2包括474例冠心病患者和486例对照受试者(表2)。

图2 毛细管电泳结果

表2 进行NOD1和NOD2基因分型的两个人群的基本资料比较
8个位点中,rs1558068位点因峰形图不确定归类的样本超过5%被视为分型失败,其余7个位点可以成功分型,每个位点抽检32个样本进行1代测序验证结果吻合率达到100%,认为分型结果准确可靠(图3)。
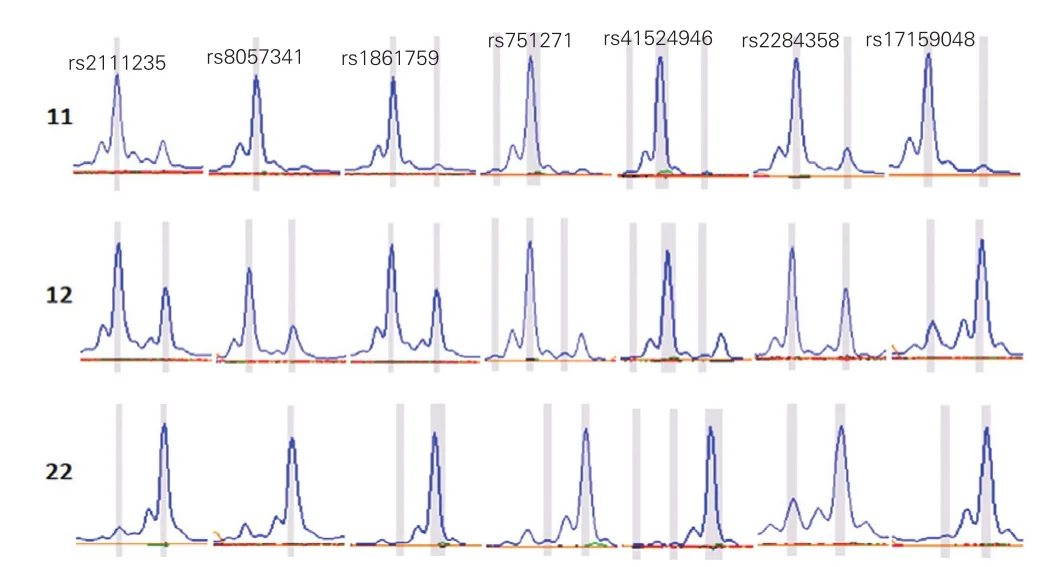
图3 NOD的7个SNP位点三种基因型的分型电泳图举例
与冠心病关联分析的结果显示,7个位点等位基因分布频率在冠心病患者和对照受试者中无明显区别,说明NOD1和NOD2基因与冠心病发病不相关。当以性别进行分层分析时,NOD2基因的两个位点rs8057341和rs751271在女性中显示出与冠心病一定的相关性,合并两个人群后,非校正P值达到0.002和0.003,但经过危险因素校正后并不相关。当以是否合并高血压进行分层分析时,结果显示,在合并高血压的人群,7个位点与冠心病无明显相关性。但在非高血压人群,NOD2基因的4个位点都显示出一致的相关性(表3)。
合并两个人群并进行危险因素校正后,其中的两个位点rs1861759和rs751271的P值达到0.001和0.002,经过多重检验Bonferroni校正后P值分别为0.007和0.014,仍然有统计学意义。另外两个位点在合并人群中P值达到0.015和0.011,但经多重检验Bonferroni校正后没有统计学意义。我们计算了rs1861759和rs751271位点的统计检验效能,如果设置检验阈值(α)为0.05,两位点的检验效能分别为0.944和0.766。
为了进一步提高检验效能,得到更加可靠的结论,我们在另一个非高血压人群(人群3)中用直接测序的方法检测了目标位点rs1861759和rs751271,并进行与冠心病的关联分析(表4)。结果发现,两位点在非高血压人群中仍然显示与冠心病相关(表5)。三个人群合并后,设置α为0.05,计算两个位点的统计效能分别为0.998和0.917;如果考虑多重检验,以最严格的Bonferroni方法计算,设置α= 0.05/7,计算两个位点的统计效能分别为0.981和0.760。
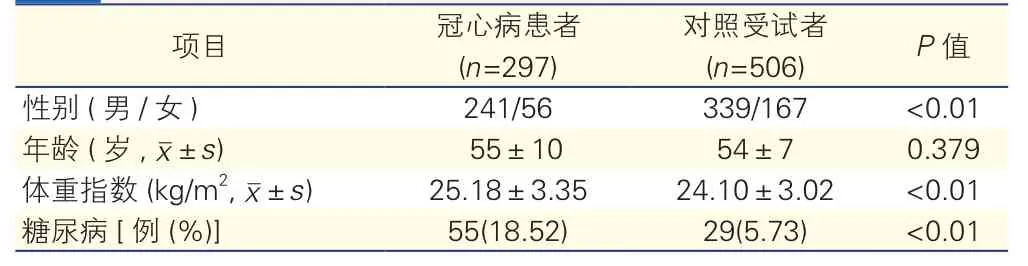
表4 非高血压人群(人群3)的基本资料(n=803)

表3 候选基因位点与不合并高血压的冠心病的关联分析

表5 基因位点rs1861759和rs751271与不合并高血压的冠心病关联分析
3 讨论
本研究优化了DNA连接酶链式反应技术在设计上的关键点,联合使用多重PCR和毛细管电泳,建立了一种基于DNA连接酶链式反应的中低通量基因分型新方法,填补了中低通量位点分型缺乏准确、快速而又低成本方法的空缺。方法创新点在于:(1)在探针上加入人工特异片段控制产物长度和扩增的特异性,实现多个位点的分型在同一个反应中进行;(2)确定等位基因特异性探针设计原则,增加连接反应特异性,进而提高分型准确性;(3) 掺入荧光引物,借助高分辨率毛细管凝胶电泳,实现多个位点一次性检测,增加通量。4)与多重PCR结合,降低模板DNA用量。
由于实验中每个位点涉及多种DNA探针,探针制备成本较高,步骤较多。因此,本方法对应于中低通量位点、大样本量的分型需求,这样分型条件一旦确定可以分型大量样本,平均到每个样本的分型成本和工作量较低。除了经济成本低,本方法的样本成本也很低。通常一代测序每个位点需要至少10 ng的模板DNA,而本方法平均每个位点仅用低于1 ng的模板DNA。第三,本方法快速而准确性高,从预扩增到拿到结果1天内完成,且准确性与一代测序相当。
由结果可见,同一批电泳的不同位点的峰型图高低并不完全一致,同一位点的两个等位基因的高度也不一致,这是由于多步反应中不同位点或同一位点的两个等位基因反应效率差异导致的。如果位点间差异过大,甚至有的位点没有出峰,可以调整预扩增反应引物的浓度来平衡不同位点产物量。对于同一位点两个等位基因高度差异问题,只要分型结果中有明确的3种类型的峰型图,可以准确判定3种基因型,即可不必调整,否则需要调整LCR过程中探针的用量。不同位点3种基因型的峰型图是有差异的,所以每做新位点时需要首先通过一代测序明确3种基因型的峰图形式。
越来越多的研究揭示,细菌感染在动脉粥样硬化发病机制中的重要作用[14-16]。因此,细胞内识别细菌产物的NOD样受体有可能与冠心病的发生相关。应用新的分型方法,我们对NOD样受体NOD1和NOD2基因进行了与冠心病的关联分析。在非高血压人群,NOD2基因的两个位点rs1861759和rs751271与冠心病相关。然而,在本研究中,我们并没有发现NOD1基因的多态性位点与冠心病的相关性。NOD1在多种细胞中表达,而NOD2主要表达在巨噬细胞和树突状细胞[12];此前的研究也发现NOD1和 NOD2两基因的表达调控模式不尽相同[17],暗示这两个基因在动脉粥样硬化病理过程中作用可能不同。当然,由于我们的样本数量是有限的,本研究并不能排除NOD1基因与冠心病的相关性。
综上所述,本研究建立了一种基于DNA连接酶的基因分型新方法,该方法准确、快速而又低成本,适用于一般实验条件下中低通量位点、大样本量的分型需求。应用该新方法,我们完成了NOD样受体基因NOD1和NOD2与冠心病的关联分析,发现NOD2基因上位点rs1861759和rs751271在非高血压条件下与冠心病相关。
[1] Kotze MJ, Luckhoff HK, Peeters AV, et al. Genomic medicine and risk prediction across the disease spectrum. Crit Rev Clin Lab Sci, 2015, 52: 120-137.
2] Osadnik T, Strzelczyk JK, Lekston A, et al. The association of functional polymorphisms in genes encoding growth factors for endothelial cells and smooth muscle cells with the severity of coronary artery disease. BMC Cardiovasc Disord, 2016, 16: 218.
[3] Volckmar AL, Song JY, Jarick I, et al. Fine mapping of a GWAS-derived obesity candidate region on chromosome 16p11.2. PLoS One, 2015, 10: e0125660.
[4] Kumar GR, Spurthi KM, Kumar GK, et al. Genetic polymorphisms of eNOS (-786T/C, Intron 4b/4a & 894G/T) and its association with asymptomatic first degree relatives of coronary heart disease patients. Nitric Oxide, 2016, 60: 40-49.
[5] Reis ST, Viana NI, Leite KR, et al. Role of genetic polymorphisms in the development and prognosis of sporadic and familial prostate cancer. PLoS One, 2016, 11: e0166380.
[6] Choi W, Jung GY. Highly multiplex and sensitive SNP genotyping method using a three-color fluorescence-labeled ligase detection reaction coupled with conformation-sensitive CE. Electrophoresis, 2017, 38: 513-520.
[7] Pinto AM, Ariani F, Bianciardi L, et al. Exploiting the potential of next-generation sequencing in genomic medicine. Expert Rev Mol Diagn, 2016, 16: 1037-1047.
[8] Rabbani B, Nakaoka H, Akhondzadeh S, et al. Next generation sequencing: implications in personalized medicine and pharmacogenomics. Mol Biosyst, 2016, 12: 1818-1830.
[9] Onoyama S, Ihara K, Yamaguchi Y, et al. Genetic susceptibility to Kawasaki disease: analysis of pattern recognition receptor genes. Hum Immunol, 2012, 73: 654-660.
[10] Lin J, Kakkar V, Lu X. Essential roles of toll-like receptors in atherosclerosis. Curr Med Chem, 2016, 23: 431-454.
[11] Seneviratne AN, Monaco C. Role of inflammatory cells and toll-like receptors in atherosclerosis. Curr Vasc Pharmacol, 2015, 13: 146-160.
[12] Caruso R, Warner N, Inohara N, et al. NOD1 and NOD2: signaling, host defense, and inflammatory disease. Immunity, 2014, 41: 898-908.
[13] Jiang F, Dong Y, Wu C, et al. Fine mapping of chromosome 3q22.3 identifies two haplotype blocks in ESYT3 associated with coronary artery disease in female Han Chinese. Atherosclerosis, 2011, 218: 397-403.
[14] 赵玫, 白小涓. 冠心病危险因素的研究进展. 中国循环杂志, 2000, 15: 127-128.
[15] Fong IW. Emerging relations between infectious diseases and coronary artery disease and atherosclerosis. CMAJ, 2000, 163: 49-56.
[16] Ott SJ, El Mokhtari NE, Musfeldt M, et al. Detection of diverse bacterial signatures in atherosclerotic lesions of patients with coronary heart disease. Circulation, 2006, 113: 929-937.
[17] King AE, Horne AW, Hombach-Klonisch S, et al. Differential expression and regulation of nuclear oligomerization domain proteins NOD1 and NOD2 in human endometrium: a potential role in innate immune protection and menstruation. Mol Hum Reprod, 2009, 15: 311-319.
Association Study of NOD2 Gene and Coronary Artery Disease Based on Optimized DNA Ligase Chain Reaction
LI Yang, YANG Xi, TIAN Xiao-li.
Vascular Biology Laboratory, Beijing Anzhen Hospital Affiliated to Capital Medical University, Beijing Institute of Heart, Lung and Blood Vessel Diseases, Beijing (100029), China
TIAN Xiao-li, Email: tianxiaoli@ncu.edu.cn
Objective: Based on optimized method of DNA ligase chain reaction in medium/low throughput genotyping, we assessed the relationship between NOD-like receptor genes NOD1, NOD2 and coronary artery disease (CAD) occurrence.
Methods: A multiplex PCR was conducted to enrich DNA template; probe design, annealing temperature, time and number of circulation of PCR were opfimizecl for allele specific ligation; allele specific products were identified by fluorescence PCR and capillary electrophoresis; the accuracy was verified by Sanger sequencing. Single nucleotide polymorphisms (SNPs) on NOD1 gene and NOD2 gene were examined in 1555 CAD patients and 1887 control subjects; the relationship between SNPs and CAD occurrence was studied.
Results: Based on optimized PCR condition and allele specific probe design, 30 allele loci genotyping can be genotyped by 10ng DNA template at one time. Association study presented that rs751271 and rs1861759 on NOD2 gene were related to non-hypertensive CAD, all P<0.05; with Bonferroni correction, such correlation was still significant, all P<0.05.
Conclusion: We optimized DNA ligase chain reaction and established a novel high accuracy, low cost method for thedemand of medium/low throughput genotyping in clinical molecular diagnosis. With this method, we identified that rs1861759 and rs751271 on NOD2 gene were associated with non-hypertensive CAD.
Coronary atery disease; Genotype; DNA ligases; Polymorphism, mononucleotide
2017-01-17)
(编辑:朱柳媛)
973项目(2013CB530700);国家自然科学基金重点项目(81130003);国家自然科学基金面上项目(81070262)
100029 北京市,首都医科大学附属北京安贞医院 北京市心肺血管疾病研究所 血管生物研究室(李扬);北京大学分子医学研究所(杨熹);南昌大学人类衰老研究所 生命科学学院(田小利)
李扬 助理研究员 博士 研究方向:心血管疾病遗传学研究 Email: liyanganzhen@126.com 通讯作者:田小利 Email: tianxiaoli@ncu.edu.cn中图分类号:R54 文献标识码:A 文章编号:1000-3614(2017)06-0569-06 doi:10.3969/j.issn.1000-3614.2017.06.010

