黄瓜耐镉相关基因CsNAC019的克隆及表达分析
李慧园,田春育,郑玉莹,武涛
(东北农业大学园艺园林学院/农业部东北地区园艺作物生物学与种质创制重点实验室,哈尔滨 150030)
黄瓜耐镉相关基因CsNAC019的克隆及表达分析
李慧园,田春育,郑玉莹,武涛
(东北农业大学园艺园林学院/农业部东北地区园艺作物生物学与种质创制重点实验室,哈尔滨 150030)
【目的】研究黄瓜耐镉(Cd)分子机制,结合生物信息学分析获得一个 NAC转录因子家族基因CsNAC019,对其进行克隆和表达分析,为进一步将该基因用于黄瓜耐Cd品种的改良与选育提供试验基础。【方法】通过PCR技术扩增CsNAC019全长;利用NCBI和DNAMAN进行序列比对和保守结构域分析;利用Expasy、TMHMM等在线软件进行氨基酸组成、稳定系数、亲水系数分析;通过 PlantCARE在线工具进行启动子序列分析;采用MEGA 6.0软件根据NJ方法,构建系统进化树;采用qRT-PCR技术检测Cd胁迫后黄瓜CsNAC019在叶中的表达情况,并用DPS 7.05数据处理系统软件进行方差及显著性分析。【结果】CsNAC019含有典型的NAC保守结构域。通过BLAST分析比对,该基因在氨基酸序列上与拟南芥NAC019存在60%的同源性,两者在从N端到C端有A、B、C、D、E 5个氨基酸相对保守区的序列高度一致。该基因CDS序列全长为960 bp,编码319个氨基酸,编码蛋白质分子量为35.66 kD,理论等电点为8.72,不稳定系数为68.18,平均亲水系数为-0.483。启动子分析表明,除含有真核生物启动子固有的TATA和CAAT 元件外,CsNAC019的启动子序列还存在G-box、ABRE、W-box、P-box、TCA-element、TC-richrepeats、HSE等多种响应逆境胁迫的顺式元件。系统进化分析表明,CsNAC019与甜瓜、桃、苹果、柑桔、葡萄、大豆等15种植物NAC转录因子有64%—96%的同源性,其中与甜瓜的NAC蛋白同源性最高,为96%。通过qRT-PCR分析发现,与对照相比,CsNAC019在Cd胁迫条件下表达量明显上调(8.2倍)。【结论】CsNAC019为黄瓜Cd胁迫诱导表达基因,推测其可能通过调节下游基因的表达调控黄瓜对Cd胁迫的应答。
黄瓜;Cd;CsNAC019;基因克隆;表达分析
0 引言
【研究意义】黄瓜(Cucumis sativus L.)是中国设施蔬菜生产中的主要栽培作物之一,由于温室大棚内化肥、农药等的连年使用,黄瓜易受到重金属的毒害,其中,镉(Cd)污染较为严重。近些年,Cd污染事件频发,例如,2009年8月在湖南浏阳以及2012 年1月在广西龙源出现的Cd污染事件等。Cd是生态系统中危害最大的重金属之一[1],具有分解周期长、移动性大、毒性高、难降解等特点,在生产活动中容易被作物吸收富集,不仅严重影响作物的产量和品质,而且可以通过食物链在人体中积累危害人体健康[2]。黄瓜对重金属敏感、运输效率高,是研究重金属污染的可行材料[3]。近几年,随着工业进程的快速发展,Cd进入生态系统危害人类健康的现象日益严重[4],直接危害人类的健康。就中国而言,受污染区域已多达11个省市的 25个地区[5]。因此,有效提高黄瓜的耐Cd能力、培育出耐Cd品种、减少Cd的危害对食品安全问题具有重大意义。此外,国家有关食品中 Cd限量指标包括黄瓜在内的蔬菜标准是 0.05 mg·kg-1(GB 2762—2005),因此,耐Cd黄瓜品种除了要在Cd污染环境下保持较好的生长势和产量之外,还需要保证黄瓜果实内Cd含量低于0.05 mg·kg-1,以保证食品安全。【前人研究进展】目前关于植物耐 Cd机制的研究表明,植物可通过区域化、固定钝化及形成金属硫蛋白、植物螯合肽、应急作用、排外作用等来抵抗或减弱Cd的毒害程度[6]。WEBER等[7]研究发现,AtPME3的过表达能够显著增强拟南芥对Zn、Cd离子的敏感性。BHUIYAN等[8]、SHIGAKI等[9]和ARAZI 等[10]研究发现ABC 转运蛋白家族基因、拟南芥中的CAX、烟草中的NtCBP4等。目前已鉴定的金属阳离子运载蛋白家族主要有CDF家族、ZIP家族、NRAMP家族、重金属ATPases家族、ABC转运蛋白家族等[6]。此外研究表明,NAC转录因子是至今发现的植物基因组中最大的转录因子家族之一,NAC转录因子不仅参与植物的多个生长发育过程如种子萌发、顶端分生组织的形成、细胞次生根的合成、次生壁的合成、开花、衰老、侧根形成等,还参与植物对生物以及非生物胁迫的响应[5,11-17]。NAC转录因子直接参与或通过调控参与干旱、高盐应答基因的表达,在植物抗干旱、高盐等非生物逆境胁迫中起重要作用。前人研究证明,水稻SNAC1、OsNAC6/SNAC2、OsNAC5、OsNAC10、OsNAC045以及拟南芥ATAF1、ANAC019、ANAC055 和ANAC072/ RD26均能够响应干旱、高盐、高温胁迫[18-20]。NAC019在番茄中同源转录因子为LeJA2和SlNAC019,LeJA2作为一个保卫细胞特异定位的转录因子,通过直接调控脱落酸(ABA)合成相关基因的表达参与番茄的抗逆反应[21];而SlNAC019则作用于番茄茉莉酸信号通路中,作为一个次级转录因子调控抗虫和抗病相关基因的表达[22]。【本研究切入点】为了研究黄瓜耐Cd胁迫分子机制,前期对拟南芥NAC转录因子家族耐逆基因进行分析[23-26],根据基因表达模式以及与黄瓜基因组同源比对结果,对潜在的黄瓜耐Cd相关基因进行筛选,结果表明,NAC019可能参与Cd胁迫响应。通过同源比对得知,CsNAC019与NAC019蛋白有较高的同源性,推测CsNAC019在功能上与NAC019具有一定的相似性。虽然NAC019 等NAC转录因子的抗逆性研究已有报道,但是NAC转录因子参与 Cd等重金属胁迫响应的相关研究的报道较少。【拟解决的关键问题】本研究对黄瓜CsNAC019进行分离,获得该基因全长CDS序列,并阐述该基因的蛋白结构、系统进化情况及对启动子序列进行分析,并研究其在 Cd胁迫下基因的表达情况,对CsNAC019参与Cd胁迫下的机理提出假说,为研究黄瓜耐Cd的分子机制和耐Cd的黄瓜新品种选育提供理论依据。
1 材料与方法
1.1 材料与试验设计
供试材料为黄瓜 649。大肠杆菌 DH5α菌株、pEASY-T1克隆载体、DNA凝胶回收试剂盒、质粒提取试剂盒和Taq DNA Polymerase购自北京全式金生物技术(TransGen Biotech)有限公司;Trizol购自Invitrogen公司;SYBR Green PCR Master Mix荧光定量试剂盒、cDNA第一链合成试剂盒购自东洋纺(TOYOBO)公司;测序由北京金唯智生物科技有限公司完成;其他生化试剂均为进口或国产分析纯试剂。
浸种催芽后,黄瓜种子于人工气候室(昼夜温度为26℃/18℃,光周期为16 h/8 h,平均湿度为60%—70%)中进行播种培养,使用全蛭石为基质,浇灌营养液栽培。待其播种后5 d(子叶展平)时进行水培,用含有CdCl2(300 μmol·L-1)的营养液进行处理,基础营养液配方(1.512 mmol·L-1NaH2PO4·2H2O、0.257 mmol·L-1Na2HPO4·12H2O、1.5 mmol·L-1MgSO4·7H2O、4 mmol·L-1Ca(NO3)2·4H2O、6 mmol·L-1KNO3、8.6 μmol·L-1C10H12FeN2NaO8·3H2O、10.3 μmol·L-1MnSO4、1 μmol·L-1CuSO4·5H2O、30 μmol·L-1H3BO3、24 nmol·L-1Na6Mo7O24·4H2O和130 nmol·L-1CoCl2·6H2O)参考文献[27],用KCl和KNO3调整营养液中K+和Ca2+的平衡。在播种后12 d采摘第1片真叶,取5株长势一致的黄瓜作为重复,液氮速冻后,存放于-80℃保存备用。
1.2 总RNA提取及cDNA第一链合成
采用Trizol法提取各部位总RNA,用分光光度计检测吸光度及浓度;cDNA第一链的合成使用TOYOBO公司的ReverTra Ace qPCR RT Kit反转录试剂盒,具体操作按照说明书进行。
1.3 引物设计与合成
以黄瓜基因组数据库中序列号为Csa3M852470.1的基因CDS序列为模板,利用Primer Premier 5.0软件设计克隆引物和qRT-PCR引物(表1)。引物由上海生工生物工程技术服务有限公司合成。
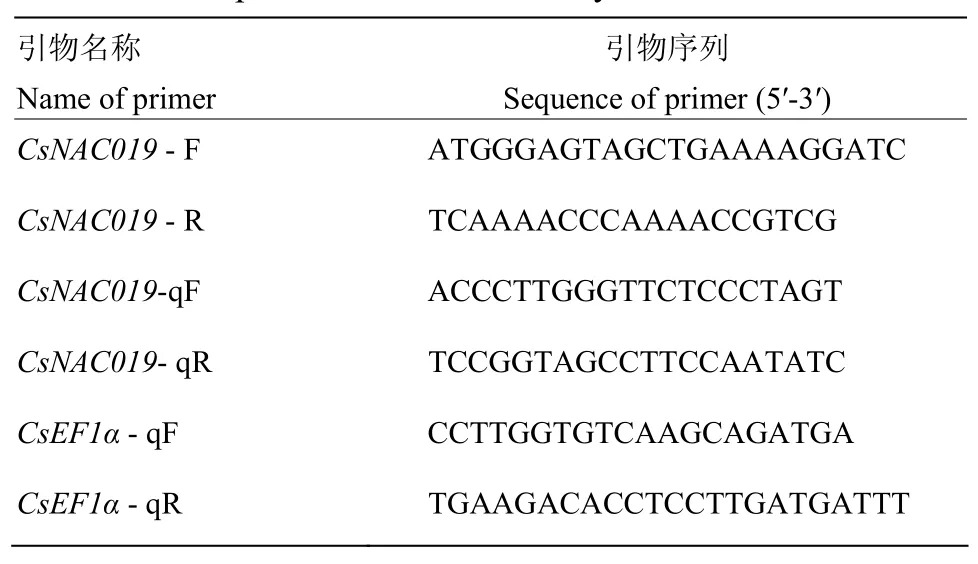
表1 本研究所用引物Table 1 The primers used in the study
1.4 CsNAC019的克隆与序列分析
以649黄瓜叶片cDNA为模板,利用PCR技术进行 CsNAC019的克隆,20 μL反应体系包括10×TaqBuffer 2 μL、dNTP Mix 2 μL、CsNAC019-F(10 μmol·L-1)和CsNAC019-R(10 μmol·L-1)各0.5 μL、TaqDNA聚合酶(5 U·μL-1)0.2 μL、模板cDNA(约200 ng·μL-1)2 μL和ddH2O 12.8 μL。反应条件为96 ℃ 5 min;95℃ 30 s,58℃ 30 s,72℃ 60 s,28个循环;72℃ 10 min。反应结束后,1%琼脂糖凝胶电泳检测并进行目的条带的回收,与pEASY-T1克隆载体连接,转入大肠杆菌DH5α感受态细胞中,菌液铺平板37℃培养过夜,挑斑,摇菌。再进行质粒提取,酶切鉴定后送阳性样本测序。
利用NCBI在线Blast下载不同植物中NAC蛋白序列,进行多重序列比对后导入 MEGA 6.0(http:// www.megasoftware.net/history.php),根据NJ方法(执行参数:Bootstrap method 1000;Poissonmodel;Pairwise deletion)分析,进而构建系统进化树。利用DNAMAN、DNAStar进行序列比对和分析。利用在线软件Expasy、TMHMM等进行氨基酸组成、稳定系数、亲水系数等分析。
1.5 CsNAC019的启动子序列分析
从黄瓜基因数据库中获取CsNAC019的启动子区域,即开放阅读框上游的1 500 bp,使用在线工具 PlantCARE(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)分析启动子区域的顺式作用元件。
1.6 Cd胁迫条件下的表达分析
以300 μmol·L-1Cd处理的植株叶cDNA为模板,进行qRT-PCR,反应体系为20 μL,包括SYBR Green PCR Master Mix 10 μL、CsNAC019-qF(10 μmol·L-1)和CsNAC019-qR(10 μmol·L-1)各0.5 μL,cDNA(约150 ng·μL-1)1 μL和ddH2O 8 μL。反应条件为96℃ 1 min;95℃ 15 s;56℃ 15 s;72℃ 45 s,45个循环,以CsEF1α作为参照基因(表1)。
采用 2-ΔΔCT相对定量分析方法计算出基因的相对表达量,并用DPS 7.05数据处理系统软件进行方差及显著性分析。
2 结果
2.1 黄瓜耐Cd胁迫相关基因CsNAC019的获得
通过对前期拟南芥NAC转录因子家族耐逆基因进行分析,根据基因表达模式以及与黄瓜基因组同源比对结果,对潜在的黄瓜耐Cd相关基因进行筛选,同时确保与重金属相关的该基因功能没有被报道,初步确定了拟南芥 NAC转录因子 NAC019作为研究对象。然后利用 DNAMAN软件,对CsNAC019和NAC019的氨基酸序列进行比对,同源性为60%。NAC转录因子具有显著的结构特点,即蛋白的N端含有高度保守约150个氨基酸组成的NAC结构域,NAC结构域从N端到C端包含5个保守的亚结构域A、B、C、D、E。两者序列比对结果分析显示A、B、C、D、E 5个亚结构域氨基酸相对保守区的序列高度一致(图 1)。由于序列的相似性可推断功能也具有一定的相似性,最终确定NAC019在黄瓜中的同源基因 CsNAC019作为试验研究对象。
2.2 黄瓜耐Cd胁迫相关基因CsNAC019的克隆
以300 μmol Cd处理的黄瓜叶片cDNA为模板,CsNAC019-F和CsNAC019-R为引物进行PCR扩增,扩增出1条约960 bp的亮带(图2),回收目标条带,并与pEASY-T1克隆载体连接,送上海生工生物工程技术服务有限公司测序,结果与黄瓜基因组数据库Csa3M852470.1基因编码区序列完全一致。

图1 CsNAC019与NAC019保守结构域的氨基酸比较Fig. 1 Alignments of NAC domain between CsNAC019 and NAC019
2.3 黄瓜耐Cd胁迫相关基因CsNAC019的序列分析
CsNAC019的cDNA序列及其推导氨基酸组成如图3所示,共编码319个氨基酸。利用NCBI对该基因的氨基酸序列保守结构域分析发现,其蛋白属于NAM superfamily超基因家族之一(图4)。经过在线软件Expasy-ProtParam(http://www.expasy.org/)分析该基因所编码蛋白的理化性质,结果表明,预测的CsNAC019蛋白的分子量为35.66 kD,理论等电点为8.72,不稳定系数为 68.18,平均亲水系数为-0.483,因此其属于不稳定型的疏水蛋白。利用 TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/)跨膜结构区预测表明:CsNAC019蛋白不具有跨膜蛋白结构。
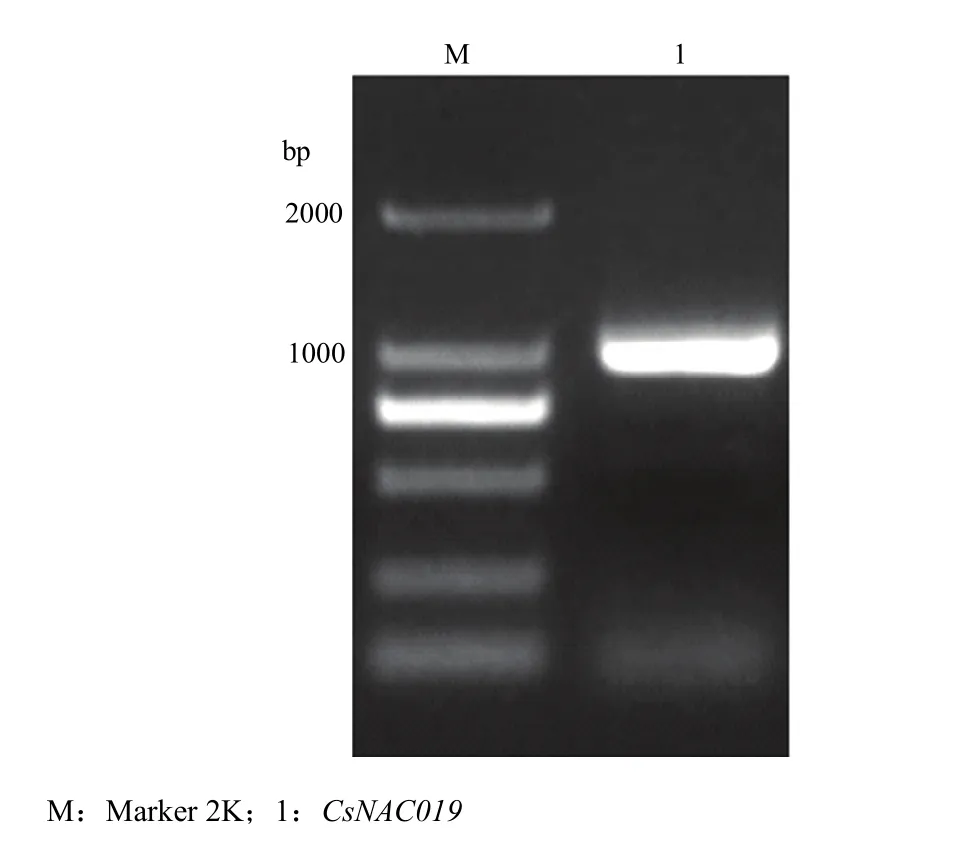
图2 CsNAC019的扩增Fig. 2 Amplification of the CsNAC019 gene

图3 CsNAC019的CDS序列及其氨基酸序列Fig. 3 Nucleotide and amino acid sequence of CsNAC019
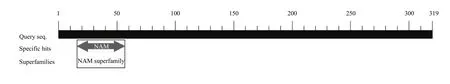
图4 CsNAC019蛋白的保守结构域分析Fig. 4 Conserved domain of CsNAC019 protein
2.4 黄瓜耐Cd胁迫相关基因CsNAC019启动子顺式元件分析
利用Plantcare 对 CsNAC019起始密码子上游1 500 bp的启动子区域进行分析(表2),结果表明,除含有真核生物启动子固有的TATA和CAAT元件外,还包含1个G-box光响应元件、1个ABREABA响应元件、2个W-box真菌诱导响应元件、1个P-box赤霉素响应元件、3个TCA-element水杨酸响应元件、2个TC-richrepeats、2个HSE热胁迫响应元件等多种响应逆境胁迫的顺式元件。
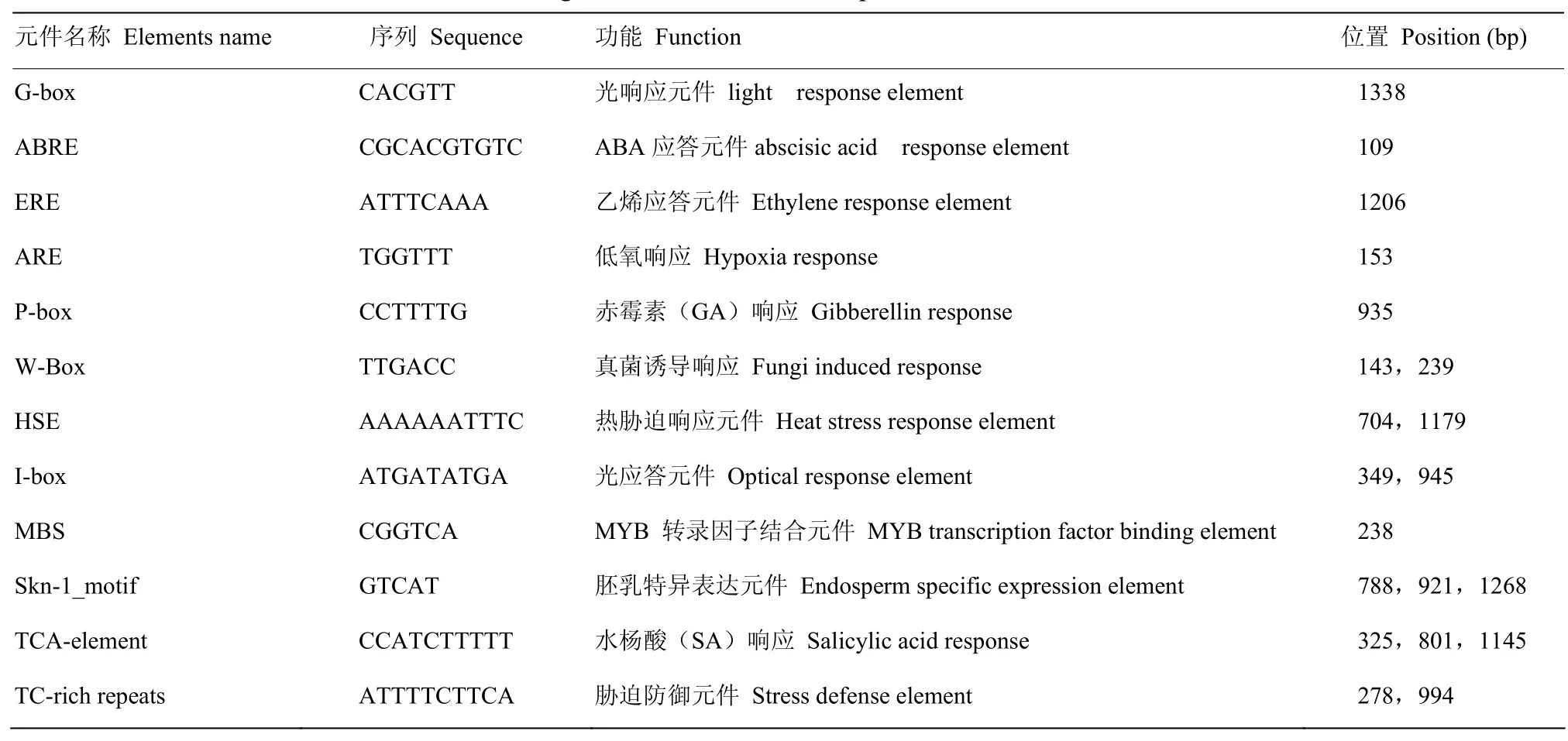
表2 CsNAC019启动子区的顺式作用元件分布及功能Table 2 Distribution and function of cis-acting elements in CsNAC019 promoter
2.5 黄瓜耐Cd胁迫相关基因CsNAC019系统进化分析
利用DNAMAN 8.0软件翻译获得CsNAC019的氨基酸序列,在NCBI(http://www.ncbi.nlm.nih.gov/)蛋白数据库中比对发现,该蛋白与梅花、桃、苹果等15种植物NAC转录因子有64%—96%的同源性,其中与甜瓜的 NAC蛋白同源性最高,为 96%。将CsNAC019与这 15条转录因子序列进行多重比较并利用MEGA 6.0软件构建系统进化树。这16条蛋白序列明显地聚类为2组,CsNAC019与甜瓜、大豆为第一类,其中,黄瓜与甜瓜同属葫芦科甜瓜属作物,亲缘关系最近;而梅花和桃等则为第二类(图5)。
2.6 黄瓜耐Cd胁迫相关基因CsNAC019的表达分析
利用qRT-PCR技术对CsNAC019在Cd胁迫条件下叶片中的表达情况进行分析(图 6)。结果表明,与对照相比,CsNAC019在Cd胁迫条件下的表达量上调明显,提高了8.2倍。
3 讨论

图5 CsNAC019蛋白与其他植物NAC蛋白系统进化树分析Fig. 5 Phylogentic tree of CsNAC019 and some other NAC proteins
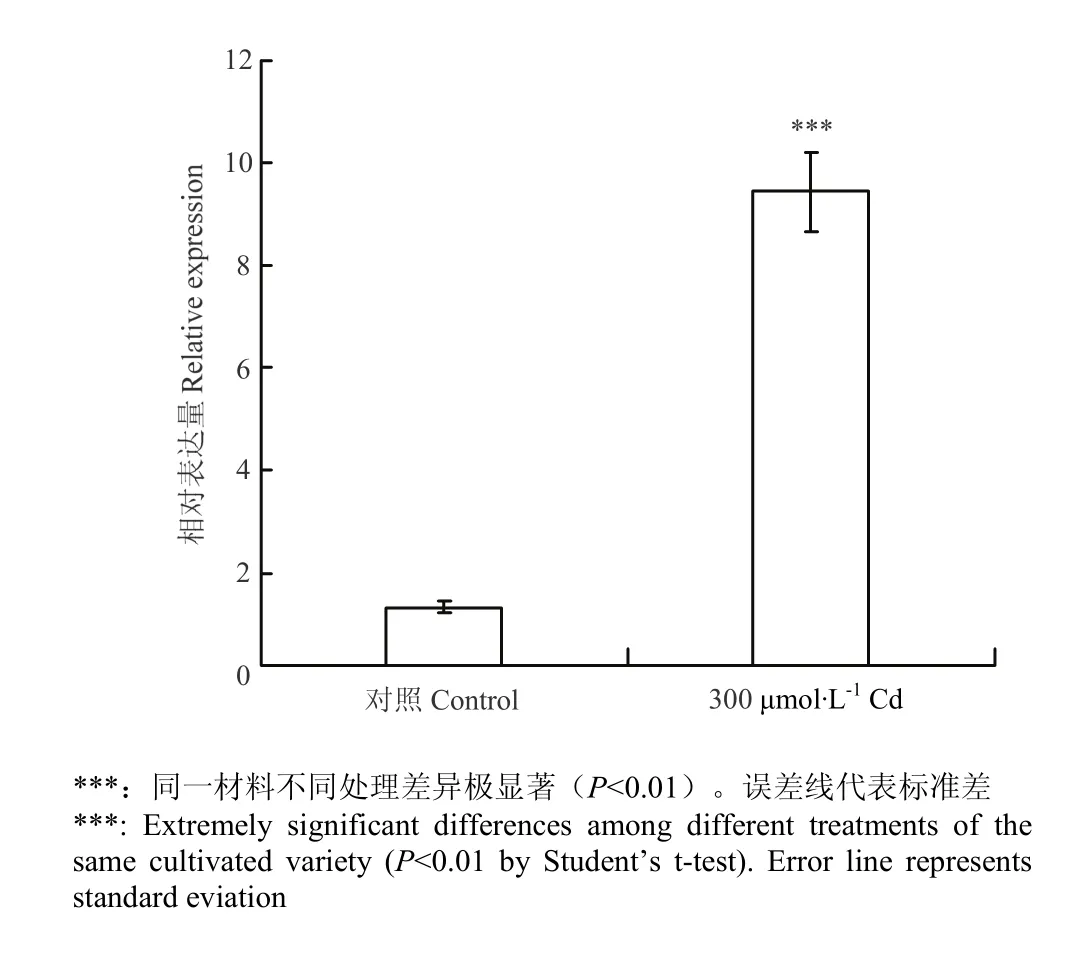
图6 CsNAC019在Cd胁迫诱导后的相对表达量Fig. 6 The relative expression of CsNAC019 under Cd stress
本研究利用生物信息学方法得到NAC019在黄瓜中的同源基因 CsNAC019。通过对 Cd胁迫下黄瓜CsNAC019表达的分析,该基因的表达受Cd胁迫的诱导,这些耐Cd基因是植物在逆境下增强耐性的重要分子基础。利用PCR克隆技术,成功获得Cd相关基因CsNAC019的CDS全长。通过保守结构域分析表明,该基因编码的蛋白是植物NAM superfamily超基因家族蛋白;在启动子序列分析中,发现多个与逆境响应有关的重要元件,如ABRE、ARE、HSE等。ABRE、G-box都是ABA诱导启动子表达的顺式作用元件[28-29]。该基因其氨基酸序列与其他植物 NAC转录因子编码的氨基酸序列同源性较高,其中,与同属葫芦科的甜瓜同源性最高。
NAC转录因子是植物中最大的一类转录因子,目前已在拟南芥、水稻、小麦等20多种植物中发现了NAC转录因子[30],NAC转录因子具有显著的结构特点,即蛋白的N端含有高度保守的约150个氨基酸组成的NAC结构域。同时,NAC结构域包含5个保守的亚结构域A、B、C、D、E,其中亚结构域的功能可能与结合 DNA和参与发育时期调控等有关[31-32]。NAC基因家族中的很多成员在植物对非生物逆境胁迫反应中存在差异表达特征,这种在非生物逆境胁迫反应中的差异表达可能反映了 NAC转录因子在植物抗非生物胁迫中具有一定的生物学功能。当植物处于干旱、高盐的胁迫下,植物细胞会感知外界的胁迫信号,通过一系列复杂的信号传导途径如ABA信号途径,把信号传递到胁迫应答的转录因子,由各类转录因子启动胁迫应答反应基因表达,从而激活植物抗逆反应,降低或消除干旱、高盐逆境所造成的伤害。干旱、高盐等逆境胁迫或ABA能诱导NAC019编码NAC转录因子的表达[6]。此外,NAC蛋白还会与其他蛋白发生互作,在拟南芥中NAC019能在体外或者体内特异地与CATGTG序列结合,并激活报告基因 GUS的转录。同时,NAC019能与ZFHD1蛋白直接结合,这种结合是激活下游基因转录所必需的[33-34]。
现有研究表明,植物通过不同的机制,例如区域化、固定钝化及形成金属硫蛋白、植物螯合肽、应急作用、排外作用等来抵抗或减弱 Cd的毒害程度[6]。目前,尽管NAC转录因子在不同作物中已有了较为广泛的研究,但是关于该基因对重金属响应的研究尚不明确。本研究对CsNAC019在Cd胁迫条件下的表达进行分析,发现该基因在 300 μmol·L-1Cd胁迫下表达量显著上调,这说明该基因是响应Cd胁迫后上调表达的基因。CsNAC019通过激活ABA通路来抵抗干旱、高盐等逆境胁迫的机理已研究的比较清楚,也暗示 Cd胁迫可能激发了CsNAC019的上调表达,从而激活了一系列复杂的信号传导途径如ABA信号途径,把信号传递到胁迫应答的转录因子,由各类转录因子启动胁迫应答反应基因表达,从而激活植物抗逆反应,降低或消除Cd逆境所造成的伤害。
目前,关于黄瓜耐 Cd相关基因的报道较少。本研究通过克隆获得CsNAC019,在Cd胁迫条件下,与对照相比,该基因表达显著上调,这一结论将为后续研究黄瓜耐 Cd生理及分子机制提供思路和线索,也将为下一步利用转基因技术培育黄瓜耐 Cd新品种奠定基础。
4 结论
从黄瓜Cd胁迫条件下的叶片中分离出NAC转录因子家族基因CsNAC019,全长为960 bp,编码319个氨基酸,编码蛋白质分子量为35.66 kD。该基因为一个新的Cd胁迫诱导表达基因,推测其可能通过调节下游基因的表达来调控黄瓜对Cd胁迫的应答。
[1] CHRISTENSEN T H. Cadmium soil sorption at low concentrations: V. Evidence of competition by other heavy metals. Water Air & Soil Pollution, 1987, 34(3): 293-303.
[2] 李婧, 周艳文, 陈森, 高小杰. 我国土壤Cd污染现状、危害及其治理方法综述. 安徽农学通报, 2015, 21(24):104-107. LI J, ZHOU Y W, CHEN S, GAO X J. Actualities, damage and management of soil cadmium pollution in China. Anhui Agricultural Science Bulletin, 2015, 21(24):104-107. (in Chinese)
[3] MORENO-CASELLES J, MORAL R, PÉREZ-ESPINOSA A, PÉREZ-MURCIA M D. Cadmium accumulation and distribution in cucumber plant. Journal of Plant Nutrition, 2000, 23(23): 243-250. [4] 和莉莉, 李冬梅, 吴钢. 我国城市土壤重金属污染研究现状和展望.土壤通报, 2008, 39(5): 1210-1216. HE L L, LI D M, WU G. Current status and prospect of heavy metal pollution in urban soils of China. Chinese Journal of Soil Science, 2008, 39(5): 1210-1216. (in Chinese)
[5] GONG W Q, PAN G X. Issues of grain Cd uptake and the potential health risk of rice production sector of China. Science and Technology Review, 2006, 24(5): 43-48.
[6] 刘明浩, 陈光辉, 王悦. 植物耐镉机制研究进展. 作物研究, 2015(1): 101-105. LIU M H, CHEN G H, WANG Y. Research progress on mechanisms of plant resistance to cadmium. Crop Research, 2015(1): 101-105. (in Chinese)
[7] WEBER M, DEINLEIN U, FISCHER S, ROGOWSKI M, GEIMER S,TENHAKEN R, CLEMENS S. A mutation in the Arabidopsis thaliana cell wall biosynthesis gene pectin methylesterase 3 as well as its ab-errant expression cause hypersensitivity specifically to Zn. The Plant Journal, 2013, 76(1): 151-164.
[8] BHUIYAN M S U, MIN S R, JEONG W J, SULTANA S, CHOI K W, LEE Y, LIU J R. Overexpression of AtATM3 in Brassica juncea confers enhanced heavy metal tolerance and accumulation. Plant Cell, Tissue and Organ Culture, 2011, 107(1): 69-77.
[9] SHIGAKI T, SREEVIDYA C, HIRSCHI K D. Analysis of the Ca2 +domain in the Arabidopsis H+/Ca2+antiporters CAX1 and CAX3. Plant Molecular Biology, 2002, 50(3): 475-483.
[10] ARAZI T, SUNKAR R, KAPLAN B, FROMM H. A tobacco plasma membrane calmodulin-binding transporter confers Ni2+tolerance and Pb2+hypersensitivity in transgenic plants. The Plant Journal, 1999, 20(2): 171-182.
[11] VROEMEN C W, MORDHORST A P, ALBRECHT C, KWAAITAAL M A, VRIES S C.The CUP-SHAPED COTYLEDON3 gene is required for boundary and shoot meristem formation in Arabidopsis. The Plant Cell, 2003, 15: 1563-1577.
[12] KIM Y S, KIM S G, PARK J E, PARK H Y, LIM M H, CHUA N H, PARK C M. A membrane-bound NAC transcription factor regulates cell division in Arabidopsis. The Plant Cell, 2006, 18: 3132-3144.
[13] HAO Y J, WEI W, SONG Q X, CHEN H W, ZHANG Y Q, WANG F, ZOU H F, LEI G, TIAN A G, ZHANG W K, MA B, ZHANG J S, CHEN S Y. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants.The Plant Journal, 2011, 68: 302-313.
[14] JENSEN M K, KJAERSGAARD T, NIELSEN M M, GALBERG P, PETERSEN K, OSHEA C, SKRIVER K. The Arabidopsis thaliana NAC transcription factor family: structure-function relationships and determinants of ANAC019 stress signaling. Biochemical Journal, 2010, 426(2) : 183-196.
[15] ZHAO Q, GALLEGO-GIRALDO L, WANG H, ZENG Y, DING S Y, CHEN F, DIXON R. An NAC transcription factor orchestrates multiple features of cell wall development in Medicago truncatula. The Plant Journal, 2010, 63(1): 100-114.
[16] 申玉华, 徐振军, 杨晓坡, 相吉山, 文静, 黄文婕. 紫花苜蓿NAC转录因子 MsNAC1基因的克隆、生物信息学分析及非生物逆境胁迫下的表达分析. 植物遗传资源学报, 2014, 15(6): 1312-1319. SHEN Y H, XU Z J, YANG X P, XIANG J S, WEN J, HUANG W J. Cloning and bioinformatics analysis of an novel NAC transcription factor MsNAC1 from Medicago sativa L.and detection of its expression under abiotic stresses. Journal of Plant Genetic Resources, 2014, 15(6): 1312-1319. (in Chinese)
[17] 李小兰, 胡玉鑫, 杨星, 于晓东, 李秋莉. 非生物胁迫相关NAC转录因子的结构及功能. 植物生理学报, 2013, 49(10): 1009-1017. LI X L, HU Y X, YANG X, YU X D, LI Q L. Structure and functions of NAC transcription factors involved in abiotic stress. Plant Physiology Journal, 2013, 49(10): 1009-1017. (in Chinese)
[18] 孙利军, 李大勇, 张慧娟, 宋凤鸣. NAC转录因子在植物抗病和抗非生物胁迫反应中的作用. 遗传, 2012, 34(8): 993-1002. SUN L J, LI D Y, ZHANG H J, SONG F M. Functions of NAC transcription factors in biotic and abiotic stress responses in plants. Hereditas, 2012, 34(8): 993-1002. (in Chinese)
[19] TAKASAKI H, MARUYAMA K, KIDOKORO S, ITO Y, FUJITA Y, SHINOZAKI K, YAMAGUCHI S K, NAKASHIMA K. The abiotic stress-responsive NAC-type transcription factor OsNAC5 regulates stress-inducible genes and stress tolerance in rice. Molecular Genetics and Genomics, 2010, 284(3): 173-183.
[20] JEONG J S, KIM Y S, BAEK K H, JUNG H, HA S H, CHOI D Y, KIM M, REUZEAU C, KIM J K. Root-specific expression of OsNAC10 improves drought tolerance and grain yield in rice under field drought conditions. Plant Physiology, 2010, 153(1): 185-197.
[21] 杜敏敏, 邓磊, 李传友. 番茄中两个高度同源的 NAC类转录因子通过不同的机制调控病原菌诱导的气孔子关闭和重新开张. 遗传, 2014, 36(8): 847. DU M M, DENG L, LI C Y. Two highly homologous NAC transcription factors in tomato are regulated by different mechanisms to regulate pathogen-induced stomatal closure and reopening. Hereditas, 2014, 36(8): 847. (in Chinese)
[22] ZHU M, HU Z , ZHOU S, WANG L, DONG T. Molecular characterization of six tissue-specific or stress-inducible genes of NAC transcription factor family in tomato (Solanum lycopersicum). Journal of Plant Growth Regulation, 2014, 33(4): 730-744.
[23] JENSEN M K, KJAERSGAARD T, NIELSEN M M, GALBERG P, PETERSEN K. The Arabidopsis thaliana NAC transcription factor family: Structure-function relationships and determinants of ANAC019 stress signalling. Biochemical Journal, 2010, 426(2): 183-196.
[24] BU Q, JIANG H, LI C B, ZHAI Q, ZHANG J. Role of the Arabidopsis thaliana NAC transcription factors ANAC019 and ANAC055 in regulating jasmonic acid-signaled defense responses. Cell Research, 2008, 18(7): 756-767.
[25] JIANG H, LI H, BU Q, LI C. The RHA2a-interacting proteins ANAC019 and ANAC055 may play a dual role in regulating ABA response and jasmonate response. Plant Signaling & Behavior, 2009, 4(5): 464-466.
[26] WELNER D H, ERNST H A, OLSEN A N, GROSSMANN J G, HELGSTRAND C. Structural characterization of ANAC019, a member of the NAC family of plant transcription factors. Acta Crystallographica, 2001, 64(a1): C306-C307.
[27] FUJIWARA T, HIRAI M Y, CHINO M, KOMEDA Y, NAITO S. Effects of sulfur nutrition on expression of the soybean seed storage protein genes in transgenic petunia. Plant Physiology, 1992, 99(1): 263-268.
[28] 何访, 梅文莉, 郭冬, 李辉亮, 彭世清. 植物激素应答元件研究进展. 热带作物学报, 2015, 36(1): 211-218. HE F, MEI W L, GUO D, LI H L, PENG S Q. Advancement of phytohormone response cis-elements. Chinese Journal of Tropical Crops, 2015, 36(1): 211-218. (in Chinese)
[29] 张毅, 尹辉, 李丹, 朱巍巍, 李秋莉. 植物启动子的化学因素诱导元件. 植物生理学报, 2007, 43(4): 787-794. ZHANG Y, YIN H, LI D, ZHU W W, LI Q L. The Cis-elements of plant chemic inducible promoters. Plant Physiology and Molecular Biology, 2007, 43(4): 787-794. (in Chinese)
[30] RIECHMANN J L, HEARD J, MARTIN G, REUBER L, JIANG C Z, KEDDIE J, ADAM L, PINEDA O, RATCLIFFE O J, SAMAHA R R, CREELMAN R, PILGRIM M, BROUN P, ZHANG J Z, GHANDEHARI D B, SHERMAN K, YU G L. Arabidopsis transcription factors: Genome-wide comparative analysis among eukaryotes. Science, 2000, 290: 2105-2110.
[31] ERNST H A, OLSEN A N, SKRIVER K, LARSEN S, LEGGIO L L. Structure of the conserved domain of ANAC, a member of the NAC family of transcription factors. EMBO Reports, 2004, 5(3): 297-303.
[32] JENSEN M K, RUNG J H, GREGERSEN P L, GJETTING T, FUGLSANG A T, HANSEN M, JOEHNK N, LYNGKJAER M F, COLLINGE D B. The HvNAC6 transcription factor: A positive regulator of penetration resistance in barley and Arabidopsis. Plant Molecular Biology, 2007, 65 (1/2): 137-150.
[33] TRAN L S P, NAKASHIMA K, SAKUMA Y, SIMPSON S D, FUJITA Y, MARUYAMA K, FUJITA M, SEKI M, SHINOZAKI K, YAMAGUCHI-SHINOZAKI K. Isolation and functional analysis of Arabidopsis stress -inducible NAC transcription factors that bind to a drought-responsive cis-element in the early responsive to dehydration stress 1 promoter. Plant Cell Online, 2004, 16(9): 2481-2498.
[34] 李伟, 韩蕾, 钱永强, 孙振元. 植物NAC转录因子的种类、特征及功能. 应用与环境生物学报, 2011(4): 596-606. LI W, HAN L, QIAN Y Q, SUN Z Y. Characteristics and functions of NAC transcription factors in plants. Chinese Journal of Applied and Environmental Biology, 2011(4): 596-606. (in Chinese)
(责任编辑 李莉)
Cloning and Expression Analysis of Cadmium Tolerance Related Gene CsNAC019 in Cucumber
LI HuiYuan, TIAN ChunYu, ZHENG YuYing, WU Tao
(Horticultural and Landscape Architecture College, Northeast Agricultural University/Key Laboratory of Biology and Germplasm Creation for Horticultural Crops, Harbin 150030)
【Objective】 To understand the molecular mechanism of Cd tolerance in cucumber, a NAC transcription factor CsNAC019 was obtained by using bioinformatics analysis. CsNAC019 was cloned and its expression was analyzed under Cd stress. The results of the present study will provide an experimental foundation for breeding of cucumber cultivars with high capacity for Cd tolerance.【Method】The full length sequence of CsNAC019 was cloned by PCR. Sequence comparison and conserved domain were analyzed by NCBI and DNAMAN. Amino acid composition, stability coefficient, and hydrophilic coefficient were analyzed by online software Expasy and TMHMM. The promoter region was analyzed by online software PlantCARE. Phylogenic tree was constructed by MEGA 6.0 according to the NJ method. The real-time PCR (qRT-PCR) was used to analyze the expression pattern of CsNAC019 under Cd treatments. The variance and significance were analyzed by DPS 7.05.【Result】CsNAC019 contained a typical conserved NAC domain. BLAST analysis revealed that CsNAC019 had 60% homology with NAC019 in the amino acid sequence, and the five relatively conserved regions of its amino acid sequences, A, B, C, D and E, were highly consistent between them from the N-terminal to the C-terminal. The full-length CDS of CsNAC019 was 960 bp, which encoded 319 amino acids with a molecular weight of 35.66 kD. The theoretical isoelectric point is 8.72, the instability coefficient is 68.18, and the average hydrophilic coefficient is -0.483. Besides the eukaryotic promoter TATA and CAAT natural elements, promoter analysis showed that the promoter sequence of CsNAC019 also had cis elements in response to stress, such as G-box, ABRE, W-box, P-box, TCA-element, TC-richrepeats, and HSE, etc. Phylogenetic analysis showed that CsNAC019 had 64%-96% homology with NAC transcription factors of other 15 plants (melon, peach, apple, citrus, grape and soybean, etc). CsNAC019 had the highest homology, 96%, with the NAC sequence of melon. qRT-PCR analysis revealed that the expression of CsNAC019 was significantly increased (8.2 folds) compared with the control under Cd tolerance condition.【Conclusion】CsNAC019 is a Cd tolerance induced gene. It was speculated that CsNAC019 may regulate the cucumber to response to Cd tolerance by regulating the expression of downstream genes.
cucumber; Cd; CsNAC019; gene clone; expression analysis
2016-11-25;接受日期:2017-03-06
国家自然科学基金青年基金(31101545)、黑龙江省普通本科高等学校青年创新人才培养计划(UNPYSCT-201500)、东北农业大学“青年才俊”项目(14QC07)、东北农业大学“学术骨干”项目(16XG05)
联系方式:李慧园,E-mail:tarasland@163.com。通信作者武涛Tel:0451-55190214;E-mail:wutao@neau.edu.cn

