茶氨酸合成酶基因的SNP挖掘和遗传定位
李小杰,马建强,姚明哲,陈亮
中国农业科学院茶叶研究所 国家茶树改良中心,浙江 杭州 310008
茶氨酸合成酶基因的SNP挖掘和遗传定位
李小杰,马建强,姚明哲*,陈亮*
中国农业科学院茶叶研究所 国家茶树改良中心,浙江 杭州 310008
茶氨酸合成酶(Theanine synthetase, TS)基因是茶树茶氨酸代谢过程中的关键酶基因。本研究以氨基酸含量差异明显的亲本及其杂交所得F1子代为研究材料,克隆TS基因的cDNA序列,挖掘其SNPs位点,并成功将杂合SNP位点定位在遗传连锁群上。研究结果显示,通过序列比对在亲本间检测到3个SNPs,验证得到1个杂合的位点SNP735。将此位点成功转化为dCAPS标记,该标记在子代中的基因型分离比为1∶1,利用该群体已构建的茶树遗传图谱进行遗传定位,将dCAPS标记定位在连锁群LG03上,相邻标记为TM299和TM517。联合此标记及其相邻标记与游离氨基酸总含量和茶氨酸含量进行统计分析,表明具有显著的相关性。
茶树;TS;SNP;dCAPS标记;定位;茶氨酸
游离氨基酸是影响茶叶品质的主要功能成分之一,其中茶氨酸是茶叶的标志性物质,占游离氨基酸总量的50%~70%[1-3],具有降血压与安神等特殊的生理功能[4],是近年来的研究热点。
茶氨酸合成酶(Theanine synthetase, TS)是茶树茶氨酸合成代谢途径中的关键酶之一。Yukitaka等[5]最先从茶树嫩梢中克隆出两条茶氨酸合成酶基因序列TS1和TS2,并通过原核表达方法证实它们具有茶氨酸合成酶的活性,其登录号分别为DD410895和DD410896。李娟等[6]克隆了TS1的cDNA全长序列(JN226569)。在NCBI已经收录的茶树基因序列中,TS基因与部分谷氨酰胺合成酶(Glutamine synthetase, GS)基因(AB115183、JQ925873)序列相似性较高,推测TS基因可能是GS基因家族成员。陈琪等[7]利用原核表达和生物信息学方法对TS1(DD410895)和GS(AB115184)进行功能验证与结构预测分析,认为两个基因开放阅读框(Open read frame, ORF)序列内3个碱基的差异,造成了二者功能的不同,并初步推断TS基因属于GS基因家族成员。由于目前茶树尚无参考基因组序列,TS基因的功能与其来源问题仍需要深入研究。
本研究以茶树品种迎霜(YS)与北跃单株(BD)及其杂交所得F1代为材料,通过克隆TS基因的cDNA序列,进行SNPs挖掘与分析,将检测到的SNP位点转化为dCAPS标记,对F1群体进行基因型鉴定,实现TS基因在遗传图谱上的定位,并验证了dCAPS标记与游离氨基酸总含量和茶氨酸含量变异的相关性,以期为茶树分子标记辅助育种提供一定的理论基础[8-10]。
1 材料与方法
1.1 材料
以茶树品种迎霜与北跃单株及其杂交所得F1代为试验材料,采摘长势一致的茶树叶片,用液氮冷冻处理,保存在-80℃中备用[11]。
1.2 方法
1.2.1 目标基因的扩增
采用RNA提取试剂盒RNApure Plant Kit (DNase I)(康为),按说明书操作提取亲本及F1代叶片的RNA。用微量紫外检测仪ND-1000UV-Vis测定其浓度和纯度,1.2%琼脂糖凝胶电泳检测RNA质量。采用cDNA合成试剂盒 PrimeScript™II 1st Strand cDNA Synthesis Kit (Takara),将提取的RNA反转录成cDNA,并稀释到200 ng·μL-1待用。
TS基因参考序列登录号为JN226569。采用Primer Premier 5.0软件设计PCR扩增所用的特异引物,其中TS-10引物参考李娟等[6]发表的序列,引物序列详见表1,由上海华津生物技术公司合成。PCR扩增体系为50 µL,其中10×PCR Buffer 5 µL,dNTP(2 mmol·L-1)5 µL,MgSO4(1.5 mmol·L-1)3 µL,模板cDNA 2 µL,上、下游引物(10 mmol·L-1)各1.5 μL,KOD-Plus-Neo酶(1 U·μL-1)1 μL,加水至终体积50 µL。反应程序为:94℃ 2 min;98℃ 10 s,55℃ 30 s,68℃ 30 s,68℃ 7 min,30个循环。PCR扩增产物采用1.2%的琼脂糖凝胶电泳,120 V电压电泳30 min后进行鉴定[12-13]。将具有目标单一条带的PCR产物送交上海华津生物公司测序。
1.2.2 SNPs挖掘与dCAPS标记的转化
利用序列比对软件Seqman比较亲本及子代PCR产物测序峰图,进行SNPs挖掘。当同一位置的测序峰图出现差异,即视为SNP位点;当同一位点呈大小相近的双峰,即视为杂合SNP位点[14-15]。对筛选出的SNP杂合位点(表2),利用dCAPS Finder 2.0(http://helix. wustl.edu/dcaps/dcaps.html)和Primer Premier 5.0软件进行错配引物和序列另一侧引物的设计[16-17]。共设计了5对引物,详见表3。PCR反应体系50 µL,其中10×PCR Buffer (Mg2+plus) 5 µL,dNTP Mixture (2.5 mmol·L-1each) 4 µL,模板cDNA 2 µL,上、下游引物(10 mmol·L-1)各1 μL,Taq酶(5 U·μL-1)0.25 μL,加水至终体积50 µL。PCR反应程序:98℃ 2 min;98℃ 10 s,61℃ 30 s,72℃30 s,72℃ 7 min,30个循环。对PCR产物进行酶切鉴定,反应体系20 µL,其中PCR产物10 µL,Buffer 2 µL,限制性内切酶Eco47 III (Takara) 1 µL,ddH2O 7 µL。酶切反应温度37℃,反应时长5 h。酶切产物用2.2%的琼脂糖凝胶电泳进行鉴定。
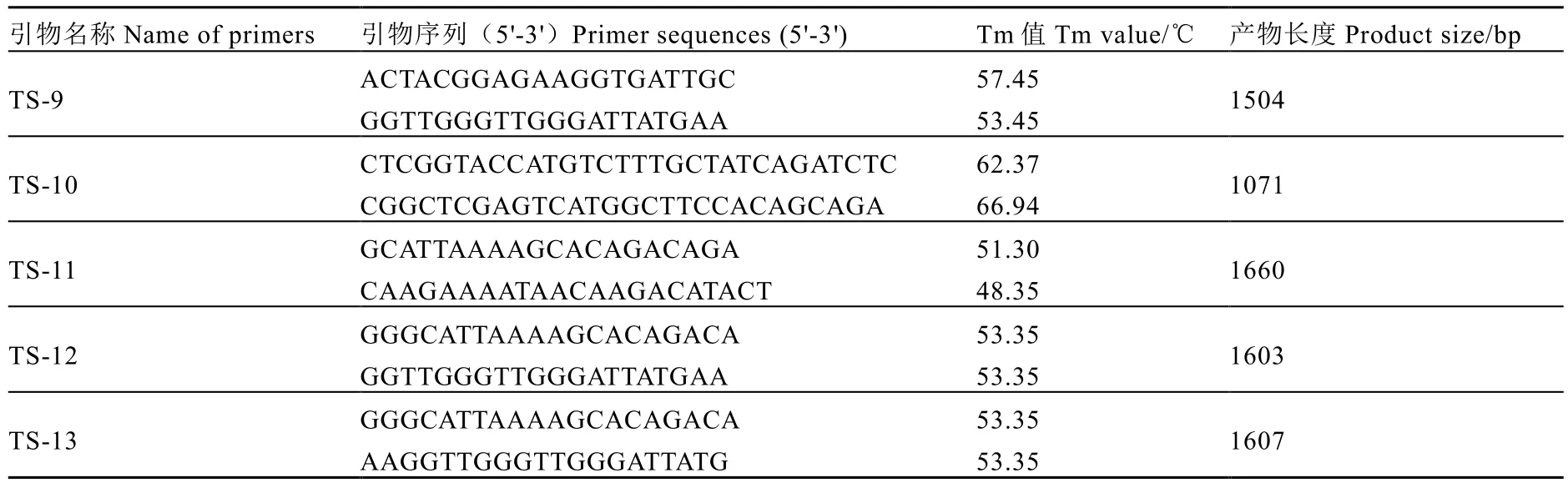
表1 引物信息Table 1 The information of primers
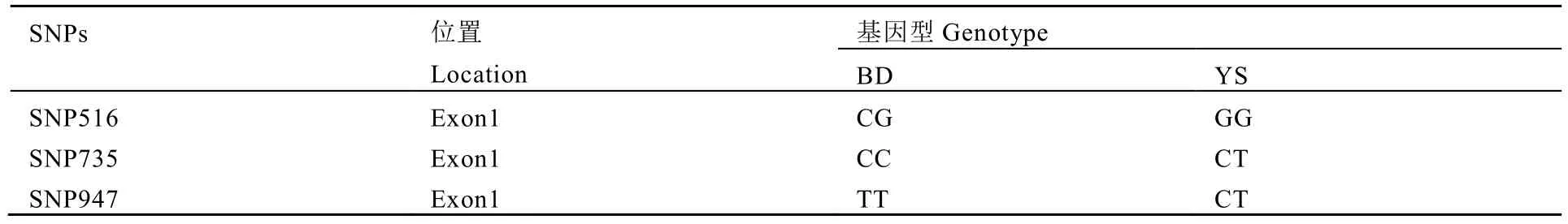
表2 SNPs位点信息Table 2 The information of SNPs locus

表3 dCAPS标记引物信息Table 3 The information of dCAPS marker primers
1.2.3 酶切带型分析与基因定位
为了验证酶切结果的准确性,将F1子代酶切前的PCR产物进行测序验证。通过序列峰图比对,检测每个子代的测序峰图与酶切结果是否一致。统计子代基因型数据,结合该群体构建的遗传图谱[18],利用JoinMap软件将杂合SNP位点转化的dCAPS标记定位到图谱上。
1.2.4 dCAPS标记与氨基酸含量的相关性分析
按照《茶树种质资源描述规范和数据标准》中描述的方法采制茶样[19],采摘春季一芽二叶。用烘干机120℃热风杀青5 min,80℃烘干至恒重,制成生化茶样。样品提取与测定参考GB/T 30987—2014[20],仪器为赛卡姆全自动氨基酸分析仪(s433D);所用试剂购自赛卡姆(北京)科学仪器有限公司。
利用MapQTL6软件,分析dCAPS标记及其在连锁群上的相邻标记与茶氨酸含量及游离氨基酸总含量的相关性。
2 结果与分析
2.1 SNPs挖掘
在11对PCR特异引物中,筛选出5对引物(表1)能扩增出目标序列单一条带。最终选择能扩出单一条带,且测序峰图质量好的TS-10引物对目标基因进行扩增。PCR产物测序长度约980 bp,位于TS基因的开放阅读框中(TS基因的开放阅读框为1 071 bp)。对亲本测序峰图进行比对,共检测到3个SNPs位点,其中在ORF序列第735和947位发生了转换(C/T),第516位发生了颠换(C/G)(表2)。第735位的SNP确认为杂合位点,其中“YS”基因型为杂合(C/T),“BD”基因型为纯合(CC),子代基因型表现为杂合(C/T)或者纯合(CC)(图1)。

图1 SNP735杂合位点的验证Fig. 1 Testing for the heterozygous SNP735
2.2 dCAPS标记的转化与在F1中的分离
经过电泳鉴定,5对dCAPS引物均能获得目标序列单一条带。经过酶切实验筛选,选择条带最清晰、最易鉴定带型的MY1引物进行PCR扩增(图2),相应的Eco47Ⅲ作为反应时的限制性内切酶,成功将TS基因的SNP杂合位点转化为dCAPS标记。
对亲本和148个子代的PCR产物进行酶切分析,发现与测序验证结果一致。对F1子代个体基因型进行统计分析,发现该位点在群体中的分离比接近于1∶1,验证了该SNP杂合位点的可靠性。
2.3 基因定位及dCAPS标记与茶氨酸的相关性
利用JoinMap软件进行连锁分析,结果显示基于杂合位点SNP735转化的dCAPS标记被定位在连锁群LG03[18]上(图3)。由图3可以看出,TS基因位于3号连锁群上约40 cm的位置,相邻的两个标记分别是TM299和TM517。
MapQTL6软件检验结果显示,F1群体茶氨酸、游离氨基酸含量变异与该dCAPS标记及其相邻的TM299和TM517标记存在显著相关性。但由于茶氨酸、游离氨基酸含量易受环境影响,不同年份间性状与标记的关联表现不稳定(表4)。
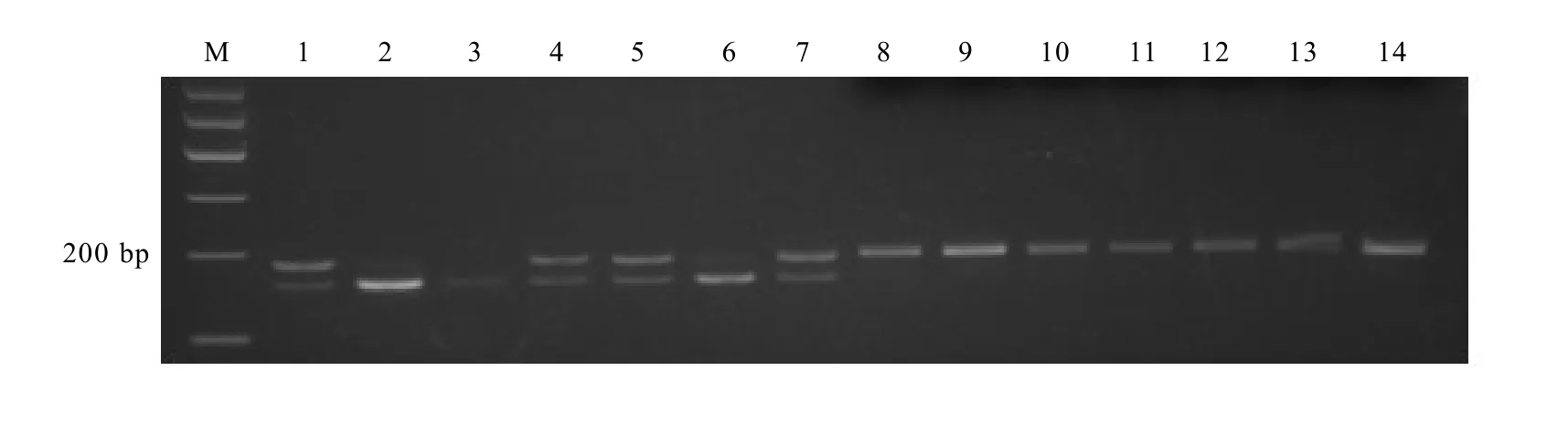
图2 F1子代PCR产物和Eco47Ⅲ酶切结果Fig. 2 The PCR products and the results of restriction endonuclease digestion byEco47Ⅲ in the F1generation

图3 TS基因在遗传图谱第3号连锁群上的定位Fig. 3 Mapping ofTSgene on LG03 linkage group
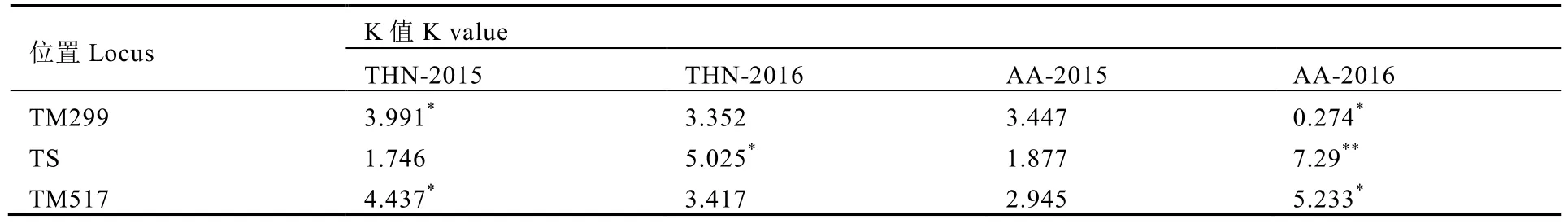
表4 Kruskal-Wallis检验Table 4 Kruskal-Wallis test
3 讨论
本研究通过对一个F1群体进行TS基因的cDNA序列分析,开发SNPs位点,在基因的编码区第735位核苷酸位点发掘到1个SNP杂合位点,成功地转化为dCAPS标记。结合该群体的茶树遗传图谱,将此标记定位到了第3号连锁群上;联合氨基酸表型数据与该标记及其在连锁群上的相邻标记进行统计分析,表明具有显著的相关性。为茶树分子标记辅助育种奠定了一定的理论基础[21-22]。
茶氨酸含量是典型的数量性状,前人研究表明茶氨酸的合成是以谷氨酸和乙胺为底物,在茶氨酸合成酶的催化下生成茶氨酸[23-24]。NCBI收录的茶氨酸合成酶基因序列已有3条,均为cDNA序列。Deng等[25]研究了茶籽苗各部位茶氨酸合成酶基因的表达差异,结果表明TS1在新梢中表达量高于其他部位,根部相对较低;TS2在新梢和根系中表达量相同,但在子叶中的转录水平较低。李春芳等[26]对茶树的13个组织部位进行转录组分析,共在转录组数据库中注释得到9个候选茶氨酸合成酶基因(TS),其中有3个基因在所有检测的组织中均有表达,其余6个基因的表达模式不尽相同。这表明该酶同时受到多个基因调控,而这些基因的作用模式目前尚不清楚,是由多个基因共同作用还是某一个基因起到关键性作用[27-28],这仍需要进行深入的研究。
茶氨酸合成酶基因与谷氨酰胺合成酶基因序列相似度较高,其功能与来源问题一直颇受争议。对茶树TS与GS基因的SNPs挖掘,实现在遗传图谱上的定位,从而确定两个基因的位置关系,可以进一步分析TS基因的来源和与GS基因家族的关系。本研究同时也选取了与TS基因序列差异相对较多的AB115184基因序列,并对茶树GS基因进行了SNPs挖掘,但是在该基因的编码区并未挖掘到SNPs位点。本次对GS基因SNPs的挖掘是在一个F1群体内进行,其遗传多样性有一定的局限性,后续的研究可以在遗传多样性丰富的样本内开展[29],以期挖掘到更多的SNPs。
[1] 陈亮, 虞富莲, 杨亚军. 茶树种质资源与遗传改良[M]. 北京:中国农业科学技术出版社, 2006: 103-104.
[2] 齐桂年. 茶氨酸的研究进展[J]. 贵州茶叶, 2001(2): 15-16.
[3] 朱小兰, 陈波, 罗旭彪, 等. 高效液相色谱法测定茶叶中的茶氨酸[J]. 色谱, 2003, 21(4): 400-402.
[4] 宛晓春. 茶叶生物化学[M]. 3版. 北京: 中国农业出版社, 2003: 32-35.
[5] Okada Y, Koseki M, Chu M. Protein and cDNA sequences of two theanine synthetases fromCamellia sinensis: JP, 2006254780[P]. 2006.
[6] 李娟. 安吉白茶高氨基酸性状相关基因的全长cDNA克隆及功能的初步研究[D]. 长沙: 湖南农业大学, 2011: 40-42.
[7] 陈琪, 江雪梅, 孟祥宇, 等. 茶树茶氨酸合成酶基因的酶活性验证与蛋白三维结构分析[J]. 广西植物, 2015, 35(3): 384-392.
[8] 郑德波, 杨小红, 李建生, 等. 基于 SNP 标记的玉米株高及穗位高QTL定位[J]. 作物学报, 2013, 39(3): 549-556.
[9] 袁金红, 李俊华, 黄小城, 等. 基于全基因组重测序的 SNP 分析在作物基因定位中的研究进展[J]. 植物生理学报, 2015, 51(9): 1400-1404.
[10] 张学铭, 刘博, 胡云艳, 等. 白菜类作物BrFLC5与开花时间相关的dCAPs标记开发[J]. 园艺学报, 2014, 41(10): 2035-2042.
[11] 马春雷, 姚明哲, 王新超, 等. 茶树2个MYB 转录因子基因的克隆及表达分析[J]. 林业科学, 2012, 48(3): 31-37.
[12] 刘凯, 邓志英, 李青芳, 等. 利用高密度SNP遗传图谱定位小麦穗部性状基因[J]. 作物学报, 2016, 42(6): 820-831.
[13] 马建, 马小定, 赵志超, 等. 水稻抗稻瘟病基因Pi35功能性分子标记的开发及其应用[J]. 作物学报, 2015, 41(12): 1779-1790.
[14] 许家磊, 王宇, 后猛, 等. SNP检测方法的研究进展[J]. 分子植物育种, 2015, 13(2): 475-482.
[15] 束永俊, 李勇, 柏锡, 等. 大豆EST-SNP的挖掘, 鉴定及其CAPS标记的开发[J]. 作物学报, 2010, 36(4): 574-579.
[16] 张成才, 王丽鸳, 韦康, 等. 基于茶树SNP的dCAPS标记体系研究[J]. 茶叶科学, 2012, 32(6): 517-522.
[17] 葛乃蓬, 崔龙, 李汉霞, 等. 番茄抗黄化曲叶病毒基因Ty-1的双重SNP标记的开发[J]. 园艺学报, 2014, 41(8): 1583-1590.
[18] Ma J Q, Yao M Z, Ma C L, et al. Construction of a SSR-based genetic map and identification of QTLs for catechins content in tea plant (Camellia sinensis) [J]. PLOS ONE, 2014, 9(3): e93131.
[19] 陈亮, 杨亚军, 虞富莲. 茶树种质资源描述规范和数据标准[M]. 北京: 中国农业出版社, 2005: 70-71.
[20] 谭和平, 徐文平, 赵爱平, 等. GB/T 30987—2014 植物中游离氨基酸的测定[S]. 北京: 中国标准出版社, 2014: 7-24.
[21] Ma J Q, Huang L, Ma C L, et al. Large-scale SNP discovery and genotyping for constructing a high-density genetic map of tea plant using specific-locus amplified fragment sequencing (SLAF-seq) [J]. PLOS ONE, 2015, 10(6): e0128798.
[22] 马建强. 茶树高密度遗传图谱构建及重要性状QTL定位[D].北京: 中国农业科学院, 2013: 61-63.
[23] Sasaoka K, Kito M, Inagaki H. Studies on the biosynthesis of theanine in tea seedlings synthesis of theanine by thehomogenate of tea seedlings [J]. Agricultural and Biological Chemistry, 1963, 27(6): 467-468.
[24] Sasaoka K, Kito M, Onishi Y. Some properties of the theanine synthesizing enzyme in tea seedlings [J]. Agricultural and Biological Chemistry, 1965, 29(11): 984-988.
[25] Deng W W, Ogita S, Ashihara H. Biosynthesis of theanine (γ-ethylamino-l-glutamic acid) in seedlings ofCamellia sinensis[J]. Phytochemistry Letters, 2008, 1(2): 115-119.
[26] Li C F, Zhu Y, Yu Y, et al. Global transcriptome and gene regulation network for secondary metabolite biosynthesis of tea plant (Camellia sinensis) [J]. BMC Genomics, 2015, 16(1): 560.
[27] 张志鹏, 王晓玥, 黄少雄, 等. 植物功能基因单核苷酸多态性的研究进展[J]. 中国农学通报, 2016, 32(20): 25-29.
[28] Palle S R, Seeve C M, Eckert A J, et al. Association of loblolly pine xylem development gene expression with single-nucleotide polymorphisms [J]. Tree Physiology, 2013, 33(7): 763-774.
[29] Jin J Q, Yao M Z, Ma C L, et al. Natural allelic variations of TCS1 play a crucial role in caffeine biosynthesis of tea plant and its related species [J]. Plant Physiology and Biochemistry, 2016(100): 18-26.
SNP Detection and Mapping of Theanine Synthetase Gene in Tea Plant
LI Xiaojie, MA Jianqiang, YAO Mingzhe*, CHEN Liang*
Tea Research Institute of the Chinese Academy of Agricultural Sciences, National Center for Tea Improvement, Hangzhou 310008, China
The theanine synthetase (TS) gene is considered as the key functional gene for the synthesis of theanine in tea plant. In this study, an F1segregating population and its parents were used to detect single nucleotide polymorphism (SNP) in theTSgene for genetic mapping. According to the sequence alignment between the parents, three SNPs were found, and SNP735 was identified to be heterozygous in YS. SNP735 was subsequently transformed into dCAPS marker and used for genotyping in the F1population. The results showed that the segregation ratio of alleles at this SNP locus was close to 1∶1. The dCAPS marker was mapped to LG03 at a position between TM299 and TM517 in the tea genetic map. Meanwhile, significant correlations between the dCAPS marker and the content of theanine and total amino acid were identified in this study.
tea plant,TS, SNP, dCAPS marker, mapping, theanine
Q51;Q52
A
1000-369X(2017)03-251-07
2017-01-17
:2017-03-28
国家茶叶产业技术体系项目(CARS-023)、国家自然科学基金(31170624、31500568)
李小杰,女,硕士研究生,主要从事茶树资源育种研究。*通讯作者

