基于线粒体COI基因序列的内蒙古沙葱萤叶甲种群遗传多样性及遗传分化
张鹏飞,周晓榕,庞保平*,谭 瑶,田 帅
(1.内蒙古农业大学草原昆虫研究中心,呼和浩特 010019;2.克什克腾旗农牧业局,内蒙古赤峰 025350)
基于线粒体COI基因序列的内蒙古沙葱萤叶甲种群遗传多样性及遗传分化
张鹏飞1,周晓榕1,庞保平1*,谭 瑶1,田 帅2
(1.内蒙古农业大学草原昆虫研究中心,呼和浩特 010019;2.克什克腾旗农牧业局,内蒙古赤峰 025350)
沙葱萤叶甲Galerucadaurica(Joannis)是一种近年来在内蒙古草原上猖獗发生为害的新害虫,为明确内蒙古沙葱萤叶甲不同地理种群间的遗传多样性及遗传分化,本研究测定了内蒙古8个地理种群的197头沙葱萤叶甲的线粒体COI基因417 bp序列,利用DnaSP 5.0、Arlequin 3.11和Network 4.6.1.0等软件对沙葱萤叶甲种群间的遗传多样性、遗传分化、分子变异等进行分析。在所分析的197个COI序列中,共有43个变异位点和62个单倍型,其中2个单倍型为各种群共享,每个种群具有3个及以上的独享单倍型。总种群的单倍型多态度指数Hd为0.9466,不同地理种群单倍型多态度Hd在0.8889-0.9600。总群体固定系数Fst为0.0315,基因流Nm为15.37。中性检验结果不显著(Tajima’s D=-1.6300,0.10>P>0.05),说明内蒙古沙葱萤叶甲在较近的历史时期内没有出现种群扩张现象。分子变异分析(AMOVA)结果表明,沙葱萤叶甲的遗传分化主要来自种群内部(96.34%),而种群间未发生明显的遗传分化。各种群间遗传分化程度与地理距离无显著相关性。不同地理种群的单倍型在中介网络图上散布在不同的分布群中,未形成明显的地理分布格局。结果表明沙葱萤叶甲不同地理种群遗传多样性高,基因交流程度高,遗传分化不明显且与地理距离无显著相关性。
沙葱萤叶甲;线粒体COI;遗传多样性;遗传分化;基因流;单倍型
沙葱萤叶甲Galerucadaurica(Joannis)是近年来在内蒙古草原上猖獗发生为害的一种新害虫,该虫从2009年开始在内蒙古草原上大面积暴发成灾(杨星科等,2010),呈现逐年加重的趋势,严重影响了草原生态环境及农牧业的可持续发展。沙葱萤叶甲种群的遗传结构研究,有利于探讨其种群间遗传进化关系,揭示其起源、扩散路线及发生趋势(Roderick and Navajas, 2003)。因此,本文选用线粒体DNA中的COI基因作为分子标记,对内蒙古沙葱萤叶甲8个地理种群的遗传结构进行研究,分析其不同地理种群的遗传多样性及遗传分化,以期揭示其成灾的遗传机制,为指导虫情监测以及综合治理提供理论依据。线粒体DNA是生物体基因组的一部分,作为生物多样性、种群遗传结构及分子系统进化研究中的一种分子标记被广泛应用(Avise, 1987, 1991; Sunnucks, 2000; Behura, 2006)。线粒体DNA作为分子标记的优点如下:第一,线粒体DNA在细胞内大量存在,为母系遗传,且以单拷贝形式存在,扩增较为容易;第二,线粒体DNA在各种生物体内高度保守(Gissietal., 2008);第三,线粒体DNA受环境选择压力较小而具有中性进化性,进化速率较核基因快(Harrison, 1989);第四,由于线粒体具有保守序列区和可变序列区,所以线粒体的扩增引物比较容易设计。特别是线粒体DNA中的COI基因,因较快的进化速率和清晰的进化模式,被广泛应用于多种农林害虫中,如烟粉虱Bemisiatabaci(褚栋等, 2005; Chuetal., 2011)、桔小实蝇Bactroceradorsalis(Liuetal., 2007; Shietal., 2010; Shietal., 2012; 王蒙等, 2014)、银杏大蚕蛾Caligulajaponica(杨宝山等, 2009)、二点委夜蛾Athetislepigone(朱彦彬等, 2012)、亚洲小车蝗Oedaleusasiaticus(高书晶等, 2011; 李云龙等, 2013)、黄胫小车蝗Oedaleusinfernalis(孙嵬等, 2013)、苹果蠹蛾Cydiapomonella(李玉婷等, 2013)、螺旋粉虱Aleurodicusdisperses(于卫卫等, 2013)、西花蓟马Frankliniellaoccidentalis(Brunneretal., 2010; Duanetal., 2013)、小菜蛾Plutellaxylostella(Weietal., 2013)、大豆食心虫Leguminivoraglycinvorella(王红等, 2014)、甜菜夜蛾Spodopteraexigua(王兴亚等, 2014)、假眼小绿叶蝉Empoascavitis(周宁宁等, 2014)、扶桑棉粉蚧Phenacoccussolenopsis(赵静等, 2014)。
沙葱萤叶甲自2009年在内蒙古中部的锡林郭勒草原暴发以来,发生范围不断扩大,2014年已扩大到6个盟市16个旗县,最西端达阿拉善盟的阿拉善右旗,最东端达呼伦贝尔市的新巴尔虎右旗(高靖淳等, 2015)。然而,目前对该虫的发生规律及成灾机制了解得很少,研究主要集中于发生为害(马崇勇等, 2012)、寄主植物选择性(昊翔等, 2014)、抗寒性(高靖淳等, 2015; 李浩等, 2014, 2015)以及药剂筛选(常静等, 2015)等方面。该虫在短短几年之内发生范围如此之广,具有哪些种群遗传学特性?各地虫源究竟是本地虫源还是外地虫源?回答这些问题对揭示该虫的成灾机制以及监测预警和防控具有重要的意义。作者曾应用微卫星标记分析了内蒙古不同地区沙葱萤叶甲种群的遗传多样性(张鹏飞等, 2015)。微卫星是核基因的共显性位点,具有高度的多态性,特别适合于研究近期的种群过程(Nardietal., 2005)。相反,母系遗传的线粒体标记能更深入地了解种群的侵入史和进化过程(Rollinsetal., 2011)。因此,基于本实验室构建的沙葱萤叶甲线粒体全基因组序列中的COI基因序列(Zhouetal., 2015),应用线粒体COI基因作为分子标记,测定了沙葱萤叶甲不同地区种群的线粒体COI基因序列,以期更全面地了解沙葱萤叶甲种群的遗传结构及分化。
1 材料与方法
1.1 供试虫源
2014年4-5月赴内蒙古草原沙葱萤叶甲主要发生区采集幼虫,为尽可能避免采集到同一个体的后代,在每个采集地点选取间隔约1 km的5个点,每个点再选取相距约40-50 m取样点4个,从每个取样点的葱属植物上采集沙葱萤叶甲幼虫3-4头,放于不同的塑料罐中。带回实验室后,冲洗晾干,用无水乙醇浸泡,-80℃保存。样本采集信息见表1。

表1 内蒙古沙葱萤叶甲不同地理种群样品的采集信息
1.2 基因组DNA的提取
取单头沙葱萤叶甲幼虫用液氮研磨后,移入1.5 mL离心管中,使用天根dp304动物基因组DNA提取试剂盒,对单头沙葱萤叶甲样本提取基因组DNA,提取的DNA经1%琼脂糖凝胶电泳检测,-20℃保存备用。
1.3 PCR扩增及序列测定
本研究PCR扩增所用的引物运用软件Primer 5.0(http://www.premierbiosoft.com)依据沙葱萤叶甲线粒体COI基因序列(GeneBank登录号:KR025478)进行设计,引物COI-F:5′-AAAGGCATAACATTAGACCG-3′和COI-R:5′-AGTTAATTCTT GATCCGTGA-3′。
PCR反应体系为50 μL:DNA模板3.0 μL,10×PCR Buffer(Mg2+Free) 5.0 μL,MgCl2(25 mmol/L) 3.0 μL,dNTPs Mixture (各2.5 mmol/L) 4.0 μL,上下游引物(10 μmol/L)各2.0 μL,TaKaRa Taq聚合酶(5 U/μL) 0.5 μL(大连宝生物工程有限公司),超纯水补足至50 μL。反应程序(BIO-RAD PCR仪):94℃预变性5 min; 94℃变性45 s,50℃退火50 s,72℃延伸45 s,共35个循环;72℃延伸10 min,4℃保存。
PCR产物采用1%琼脂糖凝胶检测,然后在凝胶成像分析系统中观察并照相,记录结果。检测结果为阳性后,委托北京六合华大基因科技有限公司进行测序。
1.4 数据分析
采用Codoncode Aligner 3.6.1软件来查看序列的峰图并且进行碱基比对,检测测序的正确性。通过DnaSP 5.0软件(Librado and Rozas, 2009),统计线粒体COI基因序列不同地理种群的单倍型数和出现的频率、单倍型多态度(Hd)、核苷酸多态度(Pi)、核苷酸平均差异数(K)及种群间遗传分化程度(Fst),并计算中性检验(Tajima, 1989)。采用MEGA 5.0软件(Tamuraetal., 2011)对COI基因序列进行序列比对及同源性分析,计算所得序列的保守位点、变异位点、简约信息位点、单一变异位点以及碱基组成,与此同时,以Kimura 2-Paramter模型计算种群间遗传距离并构建UPGMA系统树。应用Arlequin 3.11软件(Excoffieretal., 2005)进行分子变异分析(analysis of molecular variance, AMOVA)。使用GenAlEx 6.41软件(Peakall and Smouse, 2006)检验各种群遗传距离与地理距离的相关性。利用Network 4.6.1.0(Bandeltetal., 1999)基于中点连接法(Median-joining)对所有个体构建单倍型的中介网状关系图。
2 结果与分析
2.1 沙葱萤叶甲COI基因的碱基组成及序列分析
以沙葱萤叶甲的基因组DNA为模板进行PCR扩增,得到8个地理种群197头沙葱萤叶甲的测序结果,经拼接校对,最后获得417 bp基因序列,与GeneBank上已发表的沙葱萤叶甲COI序列(GeneBank登录号:KR025478)一致性高达99%,确认为目的片段。均没有碱基的缺失和插入,其中保守位点(conserved sites)374个,变异位点(variable)43个,变异百分率为10.3%,其中简约信息位点(parsimony informative sites)25个,单一变异位点(singleton variable sites)18个。所测序列中COI基因碱基组成为31.9%A,37.3%T,15.9%G和15.0%C,A+T含量为69.1%,G+C含量为30.9%,具有明显的A/T偏向性。
2.2 沙葱萤叶甲不同地理种群的遗传多样性
沙葱萤叶甲各种群的遗传多样性指标分析结果表明(表2),总种群单倍型多样性(haplotype diversity,Hd)为0.9466,核苷酸多样性(nucleotide diversity,Pi)为0.0078,核苷酸平均差异数(average number of nucleotide differences,K)为3.2707。各种群的单倍型数在10-19,最少为阿巴嘎旗种群(AQ),最多为四子王旗(SQ)和镶黄旗种群(HQ);变异位点数在10-20,最少为鄂托克旗种群(EQ),最多为四子王旗种群(SQ);单倍型多样性Hd在0.8889-0.9600,最低为阿巴嘎旗种群(AQ),最高为四子王旗种群(SQ);核苷酸多样性Pi在0.0068-0.0086,最低为鄂托克旗种群(EQ),最高为阿巴嘎旗种群(AQ);核苷酸平均差异数在2.8167-3.5906,最小为鄂托克旗种群(EQ),最大为阿巴嘎旗种群(AQ)。Tajima’s D检测结果可知(表2),总种群的Tajima’s D值为-1.6300,结果不显著(0.10>P>0.05),表明沙葱萤叶甲种群的COI序列符合中性理论。
2.3 沙葱萤叶甲不同地理种群的遗传分化分析
Fst表示群体间的分化程度,在-1~1之间,Fst值越大表示两个群体的分化程度越高(Hudsonetal., 1992)。根据DnaSP 5.0计算的结果表明(表3),总群体固定系数(Fst)为0.0315,基因流Nm为15.37,种群间Fst在-0.0169~0.1061之间,平均值为0.0305。只有苏尼特右旗(SY)与阿巴嘎旗(AQ)和锡林浩特市(XS)种群间有显著的遗传分化,而其他种群间没有显著的遗传分化。上述结果说明,沙葱萤叶甲不同地理种群间遗传分化程度低,基因交流明显。
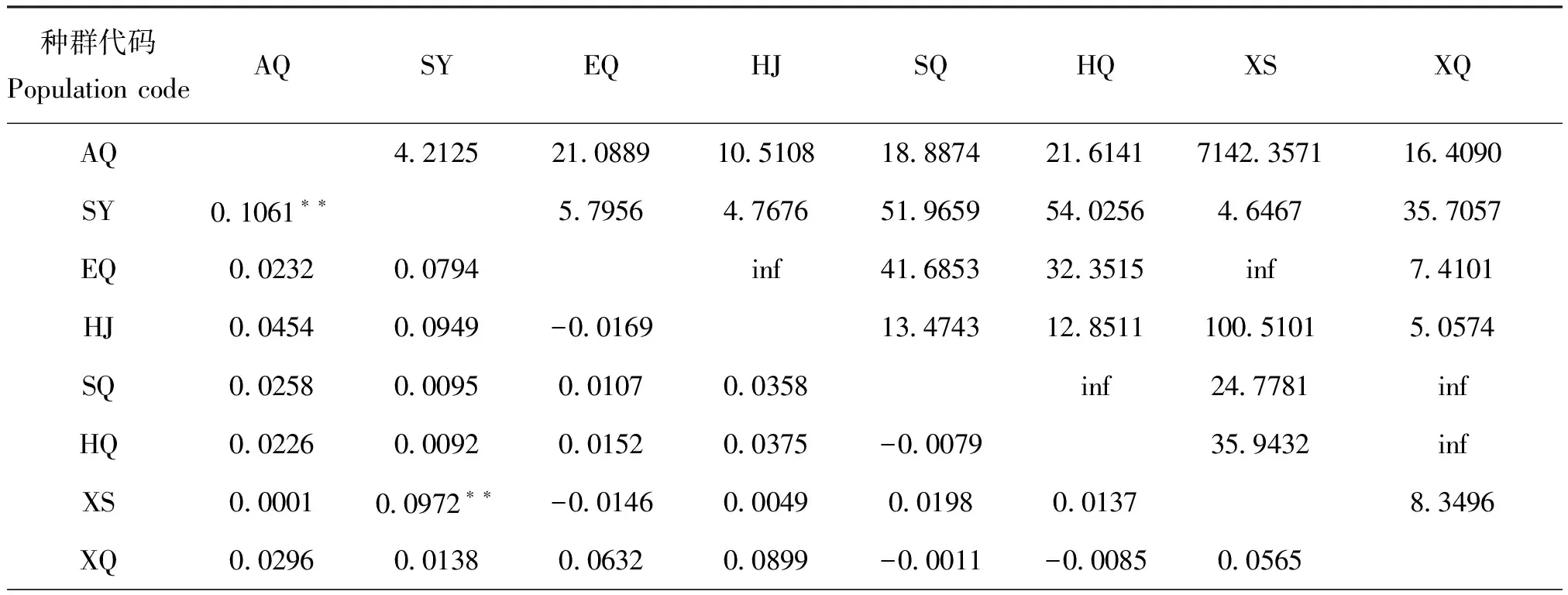
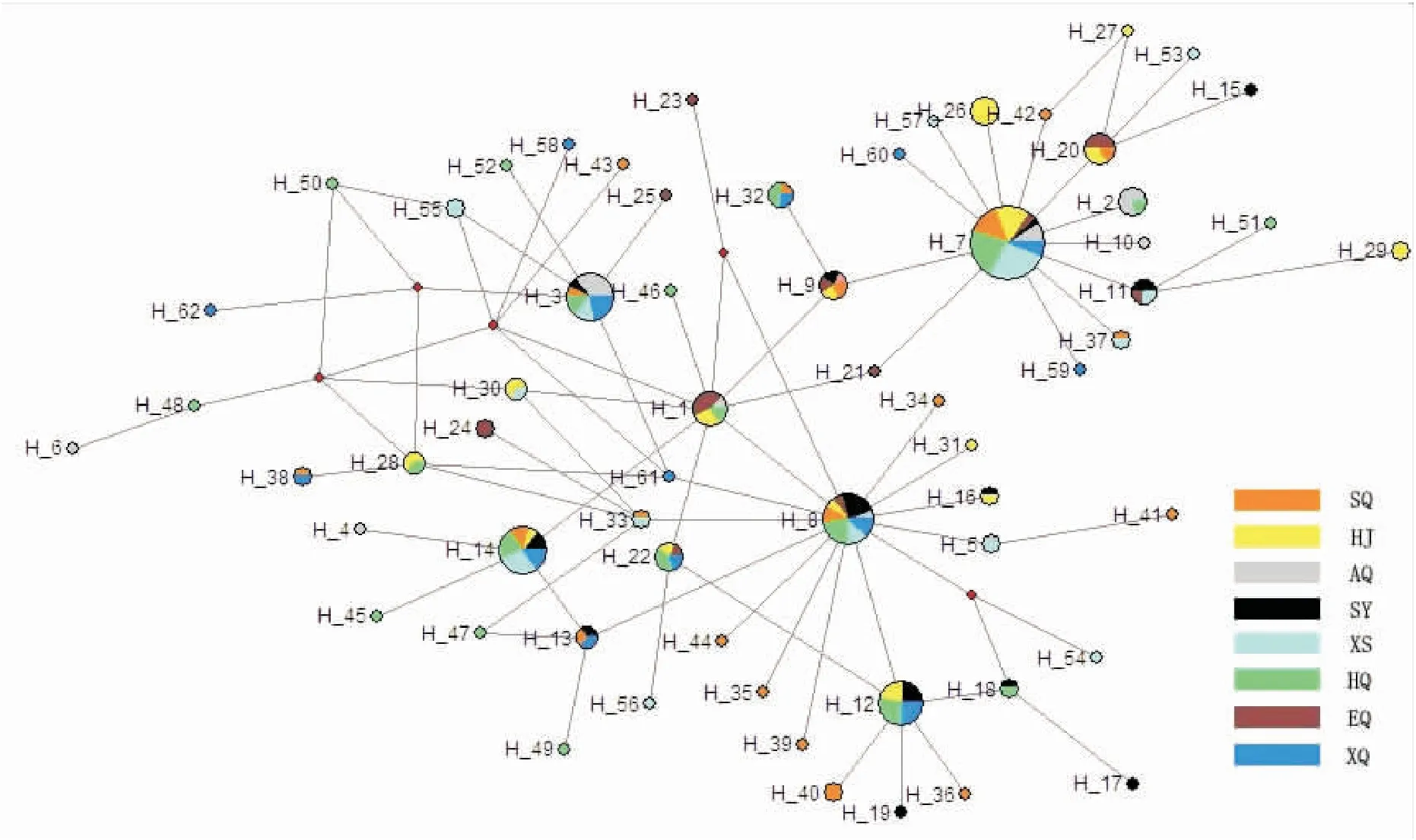
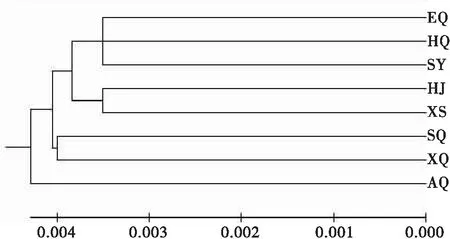
沙葱萤叶甲不同地理种群线粒体COI基因的AMOVA分析结果显示(表4),沙葱萤叶甲变异主要来自个体间,种群间的变异所占的比例非常小(3.66%),而种群内的遗传变异占96.34%,差异显著(0.001 表2 沙葱萤叶甲不同地理种群的遗传多样性指数及中性检测 表3 沙葱萤叶甲不同地理种群间遗传分化系数(Fst)(下三角)和基因流(Nm)(上三角) 注:**0.001 表4 沙葱萤叶甲不同地理种群线粒体COI基因的分子变异分析(AMOVA) 2.4 沙葱萤叶甲不同地理种群的单倍型分析 在197条沙葱萤叶甲COI序列中,共检测出62个单倍型,分别命名为H1-H62 (GenBank登录号:KU057704-KU057765)(表5)。其中共享单倍型21个,独享单倍型41个,单倍型H7和H8是各种群共享的主体单倍型,可认为这两个单倍型是较为原始的、能够适应环境变化并在种群中稳定存在的优势单倍型。单倍型H4、H6和H10独享于阿巴嘎旗种群(AQ),单倍型H15、H17和H19独享于苏尼特右旗种群(SY),单倍型H21和H23-H25独享于鄂托克旗种群(EQ),单倍型H26、H27、H29和H31独享于杭锦旗种群(HJ),单倍型H34-H36和H39-H44独享于四子王旗种群(SQ),单倍型H45-H52独享于镶黄旗种群(HQ),单倍型H53-H57独享于锡林浩特市种群(XS),单倍型H58-H62独享于新巴尔虎右旗种群(XQ)。这些独立存在于各地理种群的独享单倍型则说明各地理种群存在着一定基因交流的同时,也具有一定程度上的遗传分化。 表5 沙葱萤叶甲不同地理种群COI基因单倍型分布 注:加粗单倍型为独享单倍型。Note: Haplotype names in boldface are unique haplotypes. 单倍型网络(图1)总体呈现星状分布图,通过单倍型网络图可清晰地看出各单倍型在各地理种群的分布情况及单倍型之间的演化关系。共享单倍型相互散布在不同的地理种群中,未形成明显的系统地理结构,62个单倍型没有按照地理分布形成明显的族群。 2.5 沙葱萤叶甲不同地理种群系统发育树 Kimura 2-Paramter模型分析沙葱萤叶甲不同地理种群的COI序列计算种群间遗传距离(表6),不同地理种群间的遗传距离在0.007-0.008,遗传距离相差较小。应用Mantel相关性检验表明(图2),种群间遗传距离和地理距离之间未呈现出显著的相关性(r=0.1079,P=0.3560>0.05),说明沙葱萤叶甲不同地理种群间的遗传分化不是由地理距离远近决定的。采用遗传距离基于UPGMA法构建沙葱萤叶甲不同地理种群的系统树(图3),系统发育树并未反映出与地理位置相关的信息,未形成明显的系统地理结构。 表6 沙葱萤叶甲不同地理种群间的遗传距离 图1 沙葱萤叶甲线粒体COI基因各单倍型的中介网络图Fig.1 Median-joining haplotype network of Galeruca daurica based on COI gene of mtDNA注:圆面积代表单倍型出现的频率,彩色扇形面积代表各样品种群在同一单倍型中所占的比例。Note: Circle areas are proportional to haplotype frequencies, while colored portions represent the proportions of the same haplotype that occurs in each sampling region. 图2 沙葱萤叶甲不同地理种群的遗传距离与地理距离的Mantel检验Fig.2 Mantel test between genetic distance and geographic distance among different populations of Galeruca daurica 图3 基于UPGMA法构建的沙葱萤叶甲不同地理种群的聚类图Fig.3 The UPGMA dendrogram of different geographic populations of Galeruca daurica 本研究比较了沙葱萤叶甲8个地理种群线粒体COI基因的部分序列的差异。COI基因是蛋白质编码基因之一,由于蛋白质功能上的需要和三联体密码子结构的限制,缺失和插入很少发生,即使发生也很容易被淘汰,本研究的内蒙古沙葱萤叶甲8个地理种群的COI基因序列没有出现碱基的插入和缺失。A+T含量(69.1%)高于G+C含量(30.9%),表现出明显的碱基A/T偏倚性,与其他昆虫线粒体基因序列碱基组成特点相一致(Jermiinetal., 1994)。 Tajima’s D检验是基于种内多态性的一种中性检验方法,可以反映出物种种群变化动态的历史,被广泛使用。检验值显著大于0时,可反映出种群的动态平衡; 检验值显著小于0时,则可反映出种群的扩张及瓶颈效应(Tajima, 1989; Harpendingetal., 1998)。沙葱萤叶甲总种群和各种群的Tajima’s D检验结果均不显著,表明内蒙古沙葱萤叶甲在较近的历史上未经历群体扩张事件,近几年不同地区沙葱萤叶甲的猖獗发生很可能是当地种群积累的结果。 本研究表明沙葱萤叶甲具有丰富的单倍型多样性(Hd=0.9466),不同地理种群间遗传分化程度低(Fst=0.0315)、基因交流明显(Nm=15.37),遗传分化与地理距离无关。而微卫星分析表明,沙葱萤叶甲种群遗传多样性较低(平均期望杂合度He=0.2680),不同地理种群之间基因流较小(Nm=0.9622),遗传分化程度高(Fst=0.2521),遗传分化与地理距离显著相关(张鹏飞等, 2015)。前人在烟粉虱Bemisiatabaci、中国红光熊蜂Bombusignitus和美洲散白蚁Reticulitermesflavipes等遗传多样性方面也得到了不一致的结果,这说明线粒体DNA的多样性与核基因的遗传多样性并不存在必然的相关性(Shaoetal., 2004; DeHeeretal., 2008; Chuetal., 2011; Duanetal., 2013)。目前,几乎所有应用线粒体DNA序列分析的结果均表明,遗传距离与地理距离无显著相关性(Liuetal., 2007; Brunneretal., 2010; 高书晶等, 2011; Shietal., 2012; 李云龙等, 2013; 孙嵬等, 2013; 李玉婷等, 2013;王红等, 2014; 王兴亚等, 2014; 周宁宁等, 2014)。对于非迁飞性昆虫和非人为传播的昆虫,其遗传距离应与地理距离呈正相关,即地理距离越远,遗传分化越大。本文基于线粒体COI基因序列的分析表明,虽然阿巴嘎旗种群与锡林浩特种群和苏尼特右旗种群遗传分化显著,但种群间的Fst在0.0972-0.1061。根据基因分化系数的值介于0-0.05之间表示种群间遗传分化程度很弱,0.05-0.15为分化中等,0.15-0.25为分化程度较大,大于0.25为分化程度很大(Rousset, 1997), 实际上这3个种群的分化程度并不高。而SSR分析表明上述3个地理种群间遗传分化最小(张鹏飞等, 2015)。实际上,上述3个地区是沙葱萤叶甲最早暴发成灾且距离最近的地区。田间观察表明,沙葱萤叶甲发生环境较为单一,均为分布有葱属植物的荒漠草原和退化草原;幼虫和成虫靠爬行迁移,未发现远距离迁飞的现象;以卵块在牛粪、石块及草丛根部越冬,人为传播的可能性很小。与沙葱萤叶甲同为萤叶甲亚科的双斑长跗萤叶甲Monoleptahieroglyphica和玉米根萤叶甲Diabroticavirgiferavirgifera也未发现具有远距离迁飞的现象(梁日霞等, 2011)。同时,微卫星标记是目前绝大多数情况下检测种群结构最有力的方法(Balloux and Lugon-Moulin, 2002)。因此,我们认为微卫星分析结果可能更好地反映了沙葱萤叶甲种群的遗传多样性和遗传分化。然而,不管是核基因(SSR)还是线粒体基因(COI),我们只是研究了整个基因组的一小部分,而使用更多的遗传标记的方法可以使遗传研究的准确率更高(Queiroz, 1993)。因此,今后需进一步采用多种分子标记方法才能更全面地了解沙葱萤叶甲种群的遗传结构及多样性。 References) Avise JC. Intraspecific phylogeography: The mitochondrial DNA bridge between population genetics and systematics[J].AnnualReviewofEcologyandSystematics, 1987, 18: 489-522. Avise JC. Ten unorthodox perspectives on evolution prompted by comparative population genetic findings on mitochondrial DNA[J].AnnualReviewofGenetics, 1991, 25: 45-69. Balloux F, Lugon-Moulin N. The estimation of population differentiation with microsatellite markers[J].MolecularEcology, 2002, 11: 155-165. Bandelt HJ, Forster P, Rohl A. Median-joining networks for inferring intraspecific phylogenies[J].MolecularBiologyandEvolution, 1999, 16(1): 37-48. Behura SK. Molecular marker systems in insects: Current trends and future avenues[J].MolecularEcology, 2006, 15(11): 3087-3113. Brunner PC, Frey JE. Habitat-specific population structure in native western flower thripsFrankliniellaoccidentalis(Insecta, Thysanoptera)[J].JournalofEvolutionaryBiology, 2010, 23: 797-804. Chang J, Zhou XR, Li HP,etal. Synergistic effects ofMetarhiziumanisopliaemixed with three pesticides againstGalerucadaurica[J].ChineseJournalofPesticideScience, 2015, 17(1): 54-59. [常静,周晓榕,李海平,等. 绿僵菌与3种杀虫剂混用对沙葱萤叶甲的协同作用[J]. 农药学学报, 2015, 17(1): 54-59] Chu D, Gao CS, De Barro P,etal. Investigation of the genetic diversity of an invasive whitefly (Bemisiatabaci) in China using both mitochondrial and nuclear DNA markers[J].BulletinofEntomologicalResearch, 2011, 101: 467-475. Chu D, Zhang YJ, Cong B,etal. Sequence analysis of mtDNA COI gene and molecular phylogeny of different geographical populations ofBemisiatabaci(Gennadius)[J].ScientiaAgriculturaSinica, 2005, 38(1): 76-85. [褚栋, 张友军, 丛斌, 等. 烟粉虱不同地理种群的mtDNA COI基因序列分析及其系统发育[J]. 中国农业科学, 2005, 38(1): 76-85 ] DeHeer CJ, Vaigo EL. Strong mitochondrial DNA similarity but low relatedness at microsatellite loci among families within fused colonies of the termiteReticulitermesflavipes[J].InsectsSociaux, 2008, 55: 190-199. Duan HS, Yu Y, Zhang AS,etal. Genetic diversity and inferences on potential source areas of adventiveFrankliniellaoccidentalis(Thysanoptera: Thripidae) in Shandong, China based on mitochondrial and microsatellite markers[J].FloridaEntomologist, 2013, 96: 964-973. Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): An integrated software package for population genetics data analysis[J].EvolutionBioninformatics, 2005, 1: 47-50. Gao JC, Zhou XR, Pang BP,etal. Effects of low temperature on the survivorship and development of overwintering eggs ofGalerucadaurica(Coleoptera: Chrysomelidae)[J].ActaEntomologicaSinica, 2015, 58(8): 881-886. [高靖淳,周晓榕,庞保平,等. 低温对沙葱萤叶甲越冬卵存活和发育的影响[J]. 昆虫学报, 2015, 58(8): 881-886 ] Gao SJ, Li DW, Liu AP,etal. Sequence analysis of mtDNA COI gene and relationships of geographical populations ofOedaleusasiaticusin Inner Mongolia[J].ActaAgrestiaSinica, 2011,19(5): 846-851. [高书晶,李东伟,刘爱萍,等. 不同地理种群的亚洲小车蝗COI基因序列及其相互关系[J]. 草地学报, 2011, 19(5): 846-851] Gissi C, Iannelli F, Pesole G. Evolution of the mitochondrial genome of Metazoa as exemplified by comparison of congeneric species[J].Heredity, 2008, 101: 301-320. Hao X, Zhou XR, Pang BP,etal. Effects of host plants on feeding amount, growth and development ofGalerucadaurica(Joannis) larvae (Coleoptera: Chrysomelidae)[J].ActaAgrestiaSinica, 2014, 22(4): 854-858. [昊翔, 周晓榕, 庞保平, 等. 寄主植物对沙葱萤叶甲幼虫生长发育及取食的影响[J]. 草地学报, 2014, 22(4): 854-858] Harpending HC, Batzer MA, Gurven M,etal. Genetic traces of ancient demography[J].Proc.Natl.Acad.Sci.USA, 1998, 95(4): 1961-1967. Harrison HR. Animal mtDNA as a genetic marker in population and evolutionary biology[J].TrendsinEcology&Evolution, 1989, 4(1): 6-11. Hudson RR, Slatkin M, Maddison WP. Estimation of levels of gene flow from DNA sequence date[J].Genetics, 1992, 132: 583-589. Jermiin LS, Crozier RH. The cytochrome b region in the mitochondrial DNA of the antTetraponerarufoniger: Sequence divergence in Hymenoptera may be associated with nucleotide content[J].JournalofMolecularEvolution, 1994, 38: 282-294. Liang RX, Wang ZY, He KL,etal. Genetic diversity of geographical populations ofMonoleptahieroglyphica(Motschulsky) (Coleoptera:Chrysomelidae) from north China estimated by mitochondrial COII gene sequences[J].ActaEntomologicaSinica, 2011, 54(7): 828-834. [梁日霞,王振营,何康来,等. 基于线粒体COII基因序列的双斑长跗萤叶甲中国北方地理种群的遗传多样性研究[J]. 昆虫学报,2011,54(7):828-834 ] Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics, 2009, 25 (11): 1451-1452. Li H, Zhou XR, Pang BP,etal. Effects of low temperature stress on the supercooling capacity and development ofGalerucadaurica(Joannis) larvae (Coleoptera: Chrysomelidae)[J].ChineseJournalofAppliedEntomology, 2015, 52(2): 434-439. [李浩,周晓榕,庞保平,等. 低温胁迫对沙葱萤叶甲幼虫过冷却能力及生长发育的影响[J]. 应用昆虫学报, 2015, 52(2): 434-439 ] Li H, Zhou XR, Pang BP,etal. Supercooling capacity and cold hardiness ofGalerucadaurica(Coleoptera: Chrysomelidae)[J].ActaEntomologicaSinica, 2014, 57(2): 212-217. [李浩, 周晓榕, 庞保平, 等. 沙葱萤叶甲的过冷却能力与抗寒性[J]. 昆虫学报, 2014, 57(2): 212-217 ] Liu J, Shi W, Ye H. Population genetics analysis of the origin of the Oriental fruit fly,BactroceradorsalisHendel (Diptera: Tephritidae), in northern Yunnan Province, China[J].EntomologicalScience, 2007, 10: 11-19. Li YL, Li X, Liang TS,etal. Sequence analysis of mtDNA COI gene of 7 geographical populations ofOedaleusasiaticusin Beijing and surrounding regions[J].PlantProtection, 2013, 35(5):117-122. [李云龙,李霞,梁铁双,等. 北京及周边地区7个地理种群的亚洲小车蝗mtDNA COI基因序列分析[J]. 植物保护, 2013, 35(5): 117-122] Li YT, Wang K, Zheng Y,etal. COI gene analysis based genetic differentiation ofCydiapomonella(Lepidoptera: Tortricidae) populations in Hexi Corridor[J].JournalofNorthwestAgriculture&ForestryUniversity(Nat. Sci. Ed.), 2013, 41(9): 85-90. [李玉婷,王康,郑燕,等. 甘肃河西走廊苹果蠹蛾种群遗传分化研究——基于COI基因序列分析[J].西北农林科技大学学报(自然科学版),2013, 41(9): 85-90 ] Ma CY, Wei J, Li HS,etal. Preliminary studies on leaf beetle,Galerucadauricaon grassland[J].ChineseJournalofAppliedEntomology, 2012, 49(3): 766-769. [马崇勇, 伟军, 李海山, 等. 草原新害虫沙葱萤叶甲的初步研究[J]. 应用昆虫学报, 2012, 49(3): 766-769 ] Nardi F, Carapelli A, Dallai R,etal. Population structure and colonization history of the olive flyBactroceraoleae(Diptera,Tephritidae)[J].MolecularEcology, 2005, 14: 2729-2738. Peakall R, Smouse PE. GENALEX6: Genetic analysis in Excel. Population genetic software for teaching and research[J].MolecularEcologyNotes, 2006, 6(1): 288-295. Queiroz A. For consensus (sometimes)[J].SystematicBiology, 1993, 42: 368-372. Roderick GK, Navajas M. Genes in new environments: Genetics and evolution in biological control[J].NatureReviewsGenetics, 2003, 4(11): 889-899. Rollins LA, Woolnough AP, Sinclair R,etal. Mitochondrial DNA offers unique insights into invasion history of the common starling[J].MolecularEcology, 2011, 20: 2307-2317. Rousset F. Genetic differentiation and estimation of gene flow fromF-statistics under isolation by distance[J].Genetics,1997,145(4): 1219-1228. Shao ZY, Mao HX, Fu WJ,etal. Genetic structure of Asian populations ofBombusignitm(Hymenoptera: Apidae)[J].JournalofHeredity, 2004, 95: 46-52. Shi W, Kerdelhué C, Ye H. Genetic structure and inferences on potential source areas forBactroceradorsalis(Hendel) based on mitochondrial and microsatellite markers[J].PLoSONE, 2012, 7(5): e37083. Shi W, Kerdelhué C, Ye H. Population genetic structure of the oriental fruit fly,Bactroceradorsalis(Hendel) (Diptera: Tephritidae) from Yunnan province (China) and nearby sites across the border[J].Genetica, 2010, 138:377-385 Sunnucks P. Efficient genetic markers for population biology[J].TrendsinEcology&Evolution, 2000, 15: 199-203. Sun W, Zhang ZT, Dong H,etal. Analysis of the genetic different geographic populations ofOedaleusinfernalis(Orthoptera: Acrididae) based on mtDNA COI gene sequences[J].ActaEntomologicaSinica, 2013, 56(8): 907-916. [孙嵬, 张柱亭, 董辉, 等. 基于线粒体COI基因序列的黄胫小车蝗不同地理种群的遗传分化及基因流分析[J]. 昆虫学报, 2013, 56(8): 907-916 ] Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J].Genetics, 1989, 123: 585-595. Tamura K, Peterson D, Peterson N,etal. MEGA 5, molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J].MolecularBiologyandEvolution, 2011, 28: 2731-2739. Wang H, Xv ZX, Han LL,etal. Analysis of the genetic diversity in geographic populations ofLeguminivoraglycinivorella(Lepidoptera: Olethreutidae) from northeastern China based on mitochondrial DNA COI gene sequences[J].ActaEntomologicaSinica, 2014, 57(9): 1051-1060. [王红, 徐忠新, 韩岚岚, 等. 基于线粒体COI基因序列的大豆食心虫中国东北地理种群遗传多样性分析[J]. 昆虫学报, 2014, 57(9): 1051-1060] Wang M, Xv L, Zhang RJ,etal. Population genetic differentiation analysis of the oriental fruit fly,Bactroceradorsalis(Diptera: Tephritidae) based on mtDNA COI gene[J].ActaEntomologicaSinica, 2014, 57(12): 1424-1438. [王蒙, 徐浪, 张润杰, 等. 基于线粒体COI基因的桔小实蝇种群遗传分化研究[J]. 昆虫学报, 2014, 57(12): 1424-1438 ] Wang XY, Xv GQ. Genetic differentiation and gene flow among geographic populations ofSpodopteraexigua(Lepidoptera: Noctuidae) in China[J].ActaEntomologicaSinica, 2014, 57(9): 1061-1074. [王兴亚, 许国庆. 中国甜菜夜蛾地理种群的遗传分化与基因流[J]. 昆虫学报, 2014, 57(9): 1061-1074 ] Wei SJ, Shi BC, Gong YJ,etal. Genetic structure and demographic history reveal migration of the diamondback mothPlutellaxylostella(Lepidoptera: Plutellidae) from the southern to northern regions of China[J].PLoSONE, 2013, 8(4): e59654. Yang BS, Hou QJ, Wang H,etal. Sequence variation of COI gene and genetic differentiation among the geographic populations ofCaligulajaponica(Lepidoptera: Saturniidae) in China[J].ActaEntomologicaSinica, 2009, 52(4): 406-412. [杨宝山, 候庆君, 王欢, 等. 不同地理种群银杏大蚕蛾COI基因序列变异与遗传分化[J]. 昆虫学报, 2009, 52(4): 406-412 ] Yang XK, Huang DC, Ge SQ,etal. One million mu of meadow in inner Mongolia suffer from the harm of breaking out ofGalerucadaurica(Joannis)[J].ChineseBulletinofEntomology, 2010, 47(4): 812. [杨星科, 黄顶成, 葛斯琴, 等. 内蒙古百万亩草场遭受沙葱萤叶甲暴发危害[J]. 昆虫知识, 2010, 47(4): 812 ] Yu WW, Yang WY, Du YZ,etal. Sequence and phylogenetic analyses of mtDNA-COI and rDNA-ITS1 genes in populations ofAleurodicusdisperses(Hemiptera: Aleyrodidae) in Hainan, southern China[J].ActaEntomologicaSinica, 2013, 56(11): 1324-1334. [于卫卫,杨文晏,杜予州,等. 海南地区螺旋粉虱种群mtDNA-COI和rDNA-ITS1基因的序列及系统发育分析[J]. 昆虫学报, 2013, 56(11): 1324-1334] Zhang PF, Zhou XR, Pang BP,etal. Microsatellite marker analysis of the genetic diversity ofGalerucadaurica(Coleoptera: Chrysomelidae) populations from Inner Mongolia[J].ActaEntomologicaSinica, 2015, 58(9): 1005-1011. [张鹏飞, 周晓榕, 庞保平,等.内蒙古沙葱萤叶甲种群遗传多样性的微卫星分析[J]. 昆虫学报,2015, 58(9): 1005-1011] Zhao J, Sun Y, Tan YA,etal. Genetic differentiation of different geographical populations ofPhenacoccussolenopsisbased on sequences of COI and 28S rDNA[J].CottonScience, 2014,26(2): 130-137. [赵静, 孙洋, 谭永安, 等. 基于COI及28S rDNA序列分析的扶桑棉粉蚧地理种群的遗传分化研究[J]. 棉花学报,2014, 26 (2): 130-137 ] Zhou NN, Wang MX, Cui L,etal. Genetic variation ofEmpoascavitis(Göthe) (Hemiptera: Cicadellidae) among different geographical populations based on mtDNA COI complete sequence[J].ActaEcologicaSinica, 2014, 34(23): 6879-6889. [周宁宁,王梦馨,崔林,等. 基于COI基因全长序列的假眼小绿叶蝉地理种群遗传分化研究 [J]. 生态学报, 2014, 34(23): 6879-6889] Zhou XR, Han HB, Pang BP,etal. The complete mitochondrial genome ofGalerucadaurica(Joannis) (Coleoptera: Chrysomelidae)[J].MitochondrialDNA, 2015, DOI: 10.3109/19401736.2015. 1060424. Zhu YB, Ma JF, Dong L,etal. Analysis of genetic polymorphism ofAthetislepigone(Lepidoptera: Noctuidae) populations from China based on mtDNA COI gene sequences[J].ActaEntomologicaSinica, 55(4): 457-465. [朱彦彬,马继芳,董立,等. 基于COI基因序列的中国二点委夜蛾遗传多态性分析[J]. 昆虫学报, 2012, 55(4): 457-465] Population genetic diversity and differentiation ofGalerucadaurica(Coleoptera: Chrysomelidae) from Inner Mongolia based on mitochondrial DNACOIgene sequences ZHANG Peng-Fei1, ZHOU Xiao-Rong1, PANG Bao-Ping1*, TAN Yao1, TIAN Shuai2 (1. Research Center for Grassland Entomology, Inner Mongolia Agricultural University, Hohhot 010019, China; 2. Bureau of Husbandry of Hexigten Banner, Chifeng 025350, Inner Mongolia Autonomous Region, China) Galerucadaurica(Joannis) is a new pest causing seriously damages in Inner Mongolia grasslands in recent years. In order to investigate the genetic diversity and differentiation among different geographic populations ofG.dauricain Inner Mongolia, the genetic diversity, genetic differentiation and molecular variance were analyzed using DnaSP 5.0, Arlequin 3.11 and Network 4.6.1.0 based on the 417 bp sequences of mtDNA COI gene of 197 individuals from 8 geographic populations. The 43 variable sites were detected and 62 haplotypes were defined based on the 197 COI sequences, including two haplotypes shared by all the 8 populations and three or more haplotypes unshared by different populations. The total haplotype diversity of all populations was 0.9466, and the range of haplotype diversity of different populations was 0.8889-0.9600. The totalFstandNmwere 0.0315 and 15.37, respectively. A neutral test was not significant (Tajima’s D=-1.6300, 0.10>P>0.05), indicating that there might be no population expansion in recent history. Analysis of molecular variance (AMOVA) showed there was no obvious genetic differentiation among populations with a high proportion of the total genetic variance attributable to variation within populations (96.34%). There was no significant correlation between the genetic differentiation and geographic distance among different populations. The haplotype network showed that the haplotypes were distributed in different clades and no obvious geographic structure had been formed. In conclusion, theG.dauricapopulations are characterized by high genetic diversity, high gene flow, and low genetic differentiation with no significant correlation with geographic distance. Galerucadaurica; mt DNA COI; genetic diversity; genetic differentiation; gene flow; haplotype 国家自然科学基金项目(31360441) 张鹏飞,男,1990年生,硕士研究生,研究方向为昆虫分子生态学,E-mail: 296308147@qq.com *通讯作者Author for correspondence, E-mail: pangbp@imau.edu.cn Received:2016-03-01;接受日期Accepted:2016-05-08 Q963;S433.5 A 1674-0858(2017)02-0332-10 张鹏飞,周晓榕,庞保平,等.基于线粒体COI基因序列的内蒙古沙葱萤叶甲种群遗传多样性及遗传分化[J].环境昆虫学报,2017,39(2):332-341.




3 结论与讨论

